牧草叶绿体基因组研究进展
2015-06-24陶晓丽王彦荣刘志鹏
陶晓丽,王彦荣,刘志鹏
(草地农业生态系统国家重点实验室 兰州大学草地农业科技学院,甘肃 兰州 730020)
牧草叶绿体基因组研究进展
陶晓丽,王彦荣,刘志鹏
(草地农业生态系统国家重点实验室 兰州大学草地农业科技学院,甘肃 兰州 730020)
叶绿体是植物能量转化和光合作用的重要细胞器,也是母本遗传信息的重要载体。叶绿体基因组信息在揭示物种进化、杂交、演变,以及物种鉴定等方面具有重要价值。在其他植物叶绿体基因组研究迅速发展的同时,牧草叶绿体基因组的研究也紧随其后,但针对部分牧草的研究并未深入进行,仅仅是完成了叶绿体基因组全序列的测定。鉴于牧草叶绿体基因组研究的重要性,本文收集整理了近年来的有关资料,从叶绿体DNA的分离、提纯、测序以及牧草叶绿体基因组的研究进展等几个方面归纳了此领域的最新研究成果,旨在使人们进一步了解叶绿体DNA的提纯以及牧草叶绿体基因组的研究现状、今后的发展趋势和应用前景。
牧草;叶绿体;基因组;纯化;测序
叶绿体是光合作用的主要场所,是植物细胞内具有自主遗传信息的半自主性细胞器,它普遍存在于陆生植物、大多数藻类和部分原生生物中[1]。近年来,利用分子生物学数据构建生物类群的系统发生树不断被运用[2],分子系统学研究也在不断发展。随着现代分子生物学研究的不断深入,人们发现对于复杂庞大的核基因组而言,仅仅通过比较个别基因的遗传变异远远不够,而且个别序列在分析物种的地理起源与其系统发生关系方面仍存在些许疑问[3],所以人们开始寻找其他的切入点去解决这些疑问。如Correns[4]1909年通过遗传学分析紫茉莉(Mirabilisjalapa)的叶斑现象,发现了与叶绿体结构和发育有关的遗传因子,而这些现象却不符合孟德尔定律。Ris和Plaut[5]1962年通过对衣藻(Chlamydomonassp.)叶绿体的研究,发现了DNA纤丝。此后,叶绿体DNA在很多植物中先后被发现,并引起了研究人员的高度关注。
由于叶绿体是光能转化成化学能的重要细胞器,而光合作用严格受遗传控制,因而了解叶绿体基因组各部分的功能以及叶绿体在生物进化中的系统发生关系及作用,对今后深入研究作物改良、提高光合效率、探索细胞器的起源和进化,从而充分发挥叶绿体功能具有重要意义[6],而全面分析叶绿体基因组序列是做好这些工作的前提。由于现代分子生物技术的发展,尤其是大规模测序技术的不断发展,人们对叶绿体基因组的研究日益深入。叶绿体基因组研究不仅有助于通过遗传转化体系改良叶绿体功能,选育出有价值的新品种,而且可以在分子水平上揭示光合作用机理、核质互作关系以及物种起源与进化[7]。1986年Shinozaki等[8]获得烟草(Nicotianatabacum)叶绿体基因组全序列,开启了叶绿体基因组测序的先河,同年,Ohyama等[9]又测得地钱(Marchantiapolymorpha)叶绿体基因组的完整序列,发展至今,叶绿体基因组数据库正在迅速充实。2005年,美国国家生物技术中心(The National Center for Biotechnology Information,NCBI)细胞器基因组数据库中收录了超过160种植物的叶绿体基因组全序列,除此之外还有很多植物的叶绿体DNA(cpDNA)全序列的测定正在进行[10]。到2012年3月NCBI中收集了来自不同植物的269条叶绿体基因组全序列[11],截至2015年2月,NCBI数据库中共收集了579条植物的叶绿体基因组的全序列,以及一些植物的部分叶绿体基因组或某些基因的序列,其中包含同一个种的不同亚种和同一种的不同栽培品种的叶绿体基因组全序列,例如水稻(Oryzasativa)就有3个亚种,包括野生稻(O.nivara)[12]、籼稻(O.sativasubsp.indica)[13]和粳稻(O.sativasubsp.japanica)[14],而西尔斯山羊草(Aegilopssearsii)有TA1841、TA1837和TA1926三个栽培品种且都已测序完成。然而,关于牧草叶绿体基因组的研究却不多,据不完全统计,目前NCBI数据库中记载了40种牧草叶绿体基因组全序列,以及一些牧草栽培品种的叶绿体基因组全序列,而有文献报道的牧草叶绿体基因组全序列研究的仅有二十几种,仅是基因库中所记载全序列的一半左右。即有些牧草虽然进行了叶绿体基因组全序列的测序,但未曾对其进行深入研究。因此,本文以前人对牧草叶绿体基因组的研究为前提,搜集整理了此领域的最新成果,并结合叶绿体基因组结构和功能,简要总结了牧草叶绿体基因组的测序方法及研究进展,旨在使人们进一步了解牧草叶绿体基因组目前的研究现状、今后的发展趋势和应用前景。
1 叶绿体基因组全序列的测定
全基因组测序技术的出现对每个生物个体来说都具有里程碑的意义。而叶绿体基因组全序列的测定更是一项细致且复杂的工作,作为一项精细技术,对所提取的叶绿体 DNA(cpDNA)的含量及纯度都有很高的要求[15]。所以要获得较高质量的植物叶绿体DNA全序列,试验设计尤为重要。近年来被子植物包括牧草叶绿体DNA全序列的测定所采用的试验方案主要有以下3种:1)提取并纯化植物叶绿体DNA,再将其随机剪切,鸟枪法克隆,最后测序,这也是最基本的测序方法,但这种试验方案耗时、费力,成本高,而且容易丢失片段,故现在已很少采用;2)提取植物叶绿体DNA,通过Long-PCR扩增,将PCR产物克隆(连接)到载体上,最后测序,这种方案是继第1种方案后使用比较快捷的试验方案;3)Fosmid文库构建,按照fosmid文库构建试剂盒(Fosmid Library Production Kit)说明进行操作。目前普遍采用的是第3种实验方案,同时将其与Long-PCR技术配合使用,使测序工作更加顺利[15]。
1.1 叶绿体DNA的分离和提纯
自1979年Kolondener和Tewari[16]首次从高等植物豌豆(Pisumsativum)获得叶绿体DNA以来,高等植物的叶绿体DNA被抽提并被广泛研究,尤以豆科和禾本科叶绿体DNA研究较多。一般来说,由于叶绿体DNA含量太少,而且有诸多外源因素(如,核DNA、RNA、蛋白质等)的干扰,叶绿体DNA的提取很难,而要获得纯度较高、浓度较大的叶绿体DNA更是难上加难。为了进一步研究叶绿体DNA的功能及与系统发育关系,人们发明了一系列高等植物叶绿体DNA的提取方法,最经典的方法有DNase I法[16]、蔗糖梯度离心法[17]、Percoll梯度法[17]、氯化铯梯度离心法[18],但这几种方法耗时、费力,对仪器的要求很高,而且提取的叶绿体DNA的浓度及纯度也不是很高。除此之外,还有无水法和高低盐pH法[19]等。后来,专家们通过几种方法的结合,发明了几种更为高效地提取植物叶绿体DNA的方法,如赵衍和翁醒华[20]在提取水稻叶绿体DNA时,通过蔗糖梯度离心法与调节pH来改变细胞表面的带电量的方法相结合,快速有效地提取叶绿体DNA;再者,龚小松和阎隆飞[21]在提取高粱叶绿体DNA时,将pH法与高盐缓冲相结合,同样有效地提取了叶绿体DNA。虽然提纯方法不同,但这些方法大都包含了3个最基本的步骤:质体的分离,叶绿体的裂解和叶绿体DNA的纯化,以达到提纯的目的。而在提纯中所面临的共同问题表现在:1)提取足够的叶绿体;2)裂解和释放叶绿体DNA。而由于核DNA依附在叶绿体表面,导致叶绿体分离困难。所以,不同的提纯方法只是在叶绿体颗粒纯化和叶绿体DNA的裂解和释放时所做的处理不同而已。
1.2 叶绿体DNA全序列的测定和组装
DNA测序技术是现代分子生物学研究的重要手段之一。植物遗传信息快速准确的获得对生命科学研究具有重要意义。对于每个物种来说,基因组测序能够真实的反映基因组DNA上的遗传信息,通过与不同个体或群体的比对,进而比较全面的揭示基因组(包括核基因组和质体基因组)的复杂性和多样性,所以基因组测序技术的发展对生命科学研究中具有里程碑的意义。从1977年Sanger等[22]报道的双脱氧链终止序列测定法,到高通量测序为代表的第二代技术,最后到以单分子测序技术为基础的新一代测序技术(第三代测序技术),无一不是测序技术的革命性跨越。而目前,市场上最常用的还是高通量为代表的第二代测序技术。
测序之后的组装也是叶绿体基因组研究的一个重要环节。叶绿体因其庞大的基因容量使得无法一次性完成全序列的测定(现在的单分子测序可能是一个新的亮点)。全序列的组装有两种方法:首先,如果是已知物种叶绿体基因组序列重测序,可以用MAQ进行组装(Map to Reference Genome);另外,如果是对新物种进行从头(De Novo)测序,即用生物信息学的方法对所测得的序列进行拼接、组装[velvet软件进行组装(De Novo Assembly)],从而获得该物种的基因序列图谱,不需要任何序列信息即可对某个物种进行测序。
2 牧草叶绿体基因组
2.1 概述
现代生物技术空前发展,测序技术日益完善, 随之而来的是越来越多的叶绿体基因组序列得到解析。然而关于牧草叶绿体基因组的研究却很少。据不完全统计,目前NCBI数据库中记载了40种牧草叶绿体全基因组的全序列,从NCBI的Genbank中记载的牧草叶绿体基因组的数量和物种数目来看,牧草在叶绿体基因组的研究方面还不甚深入,而且有些牧草也仅限于叶绿体基因组全序列的测定,并未进行深入分析,可见牧草叶绿体基因组研究领域还具有很大的发展空间。
2017年11月,国家13个部门联合印发《关于加强船用低硫燃油供应保障和联合监管的指导意见》,引导企业生产、销售、使用合规的船用低硫燃油,着力解决合规燃油供应不足、市场秩序混乱等突出问题,同步推进制度建设和联合监管。
叶绿体基因组所含有的遗传物质的总和称为叶绿体基因组(Plastome)。叶绿体DNA(cpDNA)一般为双链环状分子,但也有个别物种为线状,如伞藻属(Acetabularia)植物。对于一个植物细胞来说,一般在细胞中包含400~1 600个叶绿体基因组拷贝[23]。通过基因定位和基因测序证实被子植物质体基因组通常是非常保守的,大多数高等植物(包括牧草)叶绿体基因组是高度保守的四分体结构[10]:一个大的单拷贝区域(Large Single Copy,LSC)和一个小的单拷贝区域(Small Single Copy,SSC),中间被一对反向重复序列(Inverted Reapet Sequence,IR)IRa和IRb所分开。对于一对反向重复序列,所含基因序列完全相同,但排列方向相反。也有少数植物如三叶草(Trifoliumsubterraneum)、蒺藜苜蓿(Medicagotruncatula)、鹰嘴豆(Cicerarietinum)等豆科牧草,因一个反向重复序列(IR)完全丢失而具有特殊的叶绿体基因组结构[24]。牧草叶绿体基因组大小在120~170 kb,基因组通常包括120~130个基因,其基因容量和基因顺序非常保守。Jansen等[25]2008年报道了鹰嘴豆的叶绿体基因组大小为125 kb,叶绿体基因组大小变异主要受反向重复区长度变异影响。 叶绿体基因组编码的基因主要分为3类:第1类是与叶绿体基因表达有关的基因(转运RNA、核糖体RNA、编码叶绿体RNA聚合酶的3个亚基等);第2类是与光合作用有关的基因; 第3类包括NADH脱氢酶基因、ORF以及带有内含子的读码结构IRF,但它们的功能目前尚不可知[26]。
2.2 禾本科牧草叶绿体基因组
禾本科牧草又称禾草,是牧草的一个主要类群,也是天然草原的优势植物,在畜牧业生产和维持草原生态系统中发挥着重要作用。由于禾草在畜牧业和生态上的重要性,引起了一些生物学家的极大关注。但由于禾草数量庞大,而经过深入研究的仅仅是其中的一小部分,经过基因库中收录的19种禾草叶绿体基因组全序列分析,发现有文献记载禾草叶绿体基因组全序列研究的仅有9种(表1)。与其他植物相比,禾本科植物叶绿体基因组在进化过程中速度加快[32],并发生了一系列结构上的变化[33],因此,禾草叶绿体基因组的研究为整个叶绿体基因组进化研究提供了很好的研究体系。
在禾草的研究中,通过Southern杂交和PCR分析发现,禾草叶绿体基因组结构上发生了变化。首先是基因数量的变化。禾本科植物叶绿体基因组的基因数目、内含子数目以及基因排列顺序,除了有少数不同外,表现得十分保守。并且,与香蒲(Typhaorientalis)、烟草等其他科植物叶绿体基因组相比,禾本科物种中accD、ycf1、ycf2基因逐渐退化缺失,通过羊茅(Festucaovina)和黑麦草(Loliumperenne)比较发现,羊茅叶绿体基因组中缺失了rps14和ycf4基因,序列比对发现造成突变的原因是配对序列中发生了多次非三倍数碱基的插入、缺失,造成移码突变,进而造成基因的丢失[34];其次是内含子的丢失情况。有些物种还会发生内含子丢失的情况,如禾本科植物最初分化产生的物种发生clpP基因内含子的丢失,随后又发生了rpoC1内含子丢失[31];再次,基因排列顺序也发生变化。研究发现,与烟草、香蒲等其他科物种相比,禾本科植物叶绿体基因组基因的排列顺序发生了3次倒置,而相比较其他物种却不曾发现此种现象[14];最后,IR序列边缘的变化[35]。禾本科植物叶绿体基因组的IR边缘,即IR和SSC/LSC的边界发生变化。禾草叶绿体基因组具有一对反向重复区域IR(IRa和IRb),IR与两个单拷贝区域(LSC和SSC)之间存在4个边界,即IRa/LSC、IRa/SSC、IRb/LSC和IRb/SSC(图1)。在进化过程中,基因组的IR边界不断收缩与扩张,致使有些基因进入单拷贝序列而有些基因则进入IR区域。每个物种在边界处序列复制的程度也不同。而且禾草叶绿体基因上ndhH和ndhF基因所在的位置有所变化[28]。除此之外,禾本科植物叶绿体基因组的研究还涉及了叶绿体基因组的单核苷酸多态性(SNP)研究,这个研究已经在黑麦草上得以验证[29]。
2.3 豆科牧草叶绿体基因组
豆科牧草是牧草的主要来源之一,且蛋白质含量属牧草之最,鉴于其重要性和特殊性,豆科牧草的叶绿体基因组研究受到学者的广泛关注。然而究其研究的数量,发现深入到叶绿体基因组水平的豆科牧草仅仅是冰山一角,据不完全统计,基因库中收录的豆科牧草叶绿体基因组全序列有16种(表2),且大部分为车轴草属(Trifolium)植物。虽然研究种类甚少,但人们仍然发现在进化过程中,豆科牧草叶绿体基因组也发生了一系列结构上的变化,因此,豆科牧草为其他牧草叶绿体基因组进化研究提供了很好的系统。
豆科牧草与禾本科牧草分属不同的科。经过研究发现,其叶绿体基因组结构上的变化也有所不同。第一,叶绿体基因组结构不同。与禾本科牧草不同的是豆科牧草研究中出现因一个反向重复区完全丢失而具有特殊的叶绿体基因组结构,如三叶草、蒺藜苜蓿和鹰嘴豆[25];第二,基因的转移和丢失。在被子植物叶绿体基因组研究中发现,accD,rpl23,rpl22,ycf1,ycf2,rps16,ycf4和infA等基因都部分或全部从豆科植物的叶绿体基因组缺失,有的基因(如infA)甚至还发生了多次缺失事件[40];第三,叶绿体基因组基因的排列顺序发生了倒置。在豆科植物叶绿体基因组中发现大约50 kb[41-42]和78 kb[43]长度片段的倒置;第四,基因内含子的缺失情况。研究发现豆科植物叶绿体基因组中,clpP和rps12基因内含子发生了缺失的情况;第五,豆科植物叶绿体DNA上发生了多次重排[44];第六,豆科牧草叶绿体基因组出现了RNA编辑现象,如百脉根(Lotusjaponicus)[40]。
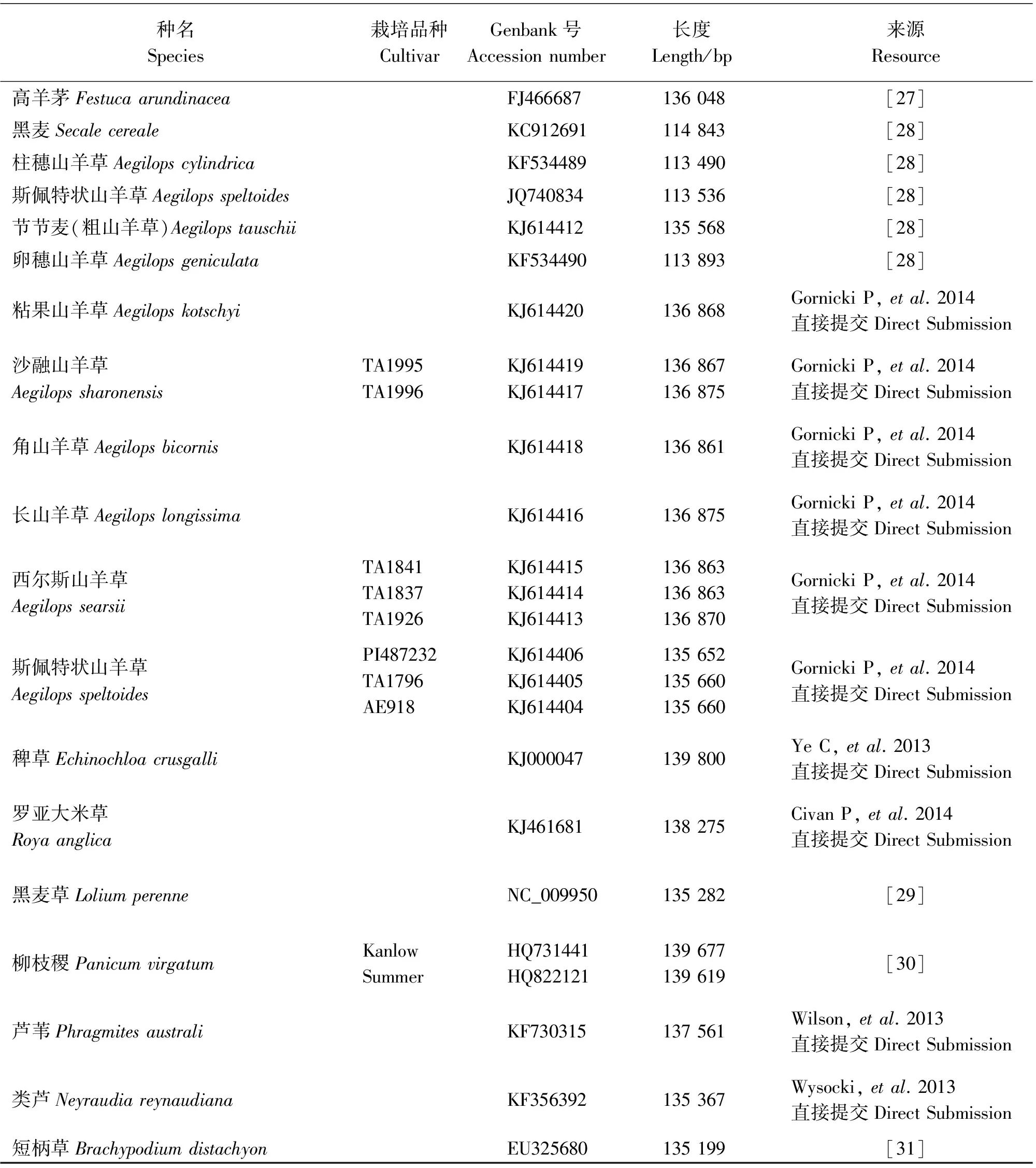
表1 禾本科牧草的叶绿体基因组全序列及GenBank号
注:“来源”一栏中部分序列研究文章已发表,部分序列仅由作者提供,没有发表文章。下同。
Note:“Sources”section of the column sequence of research articles have been published, only partial sequence provided by the author, but the article was not published. The same below.
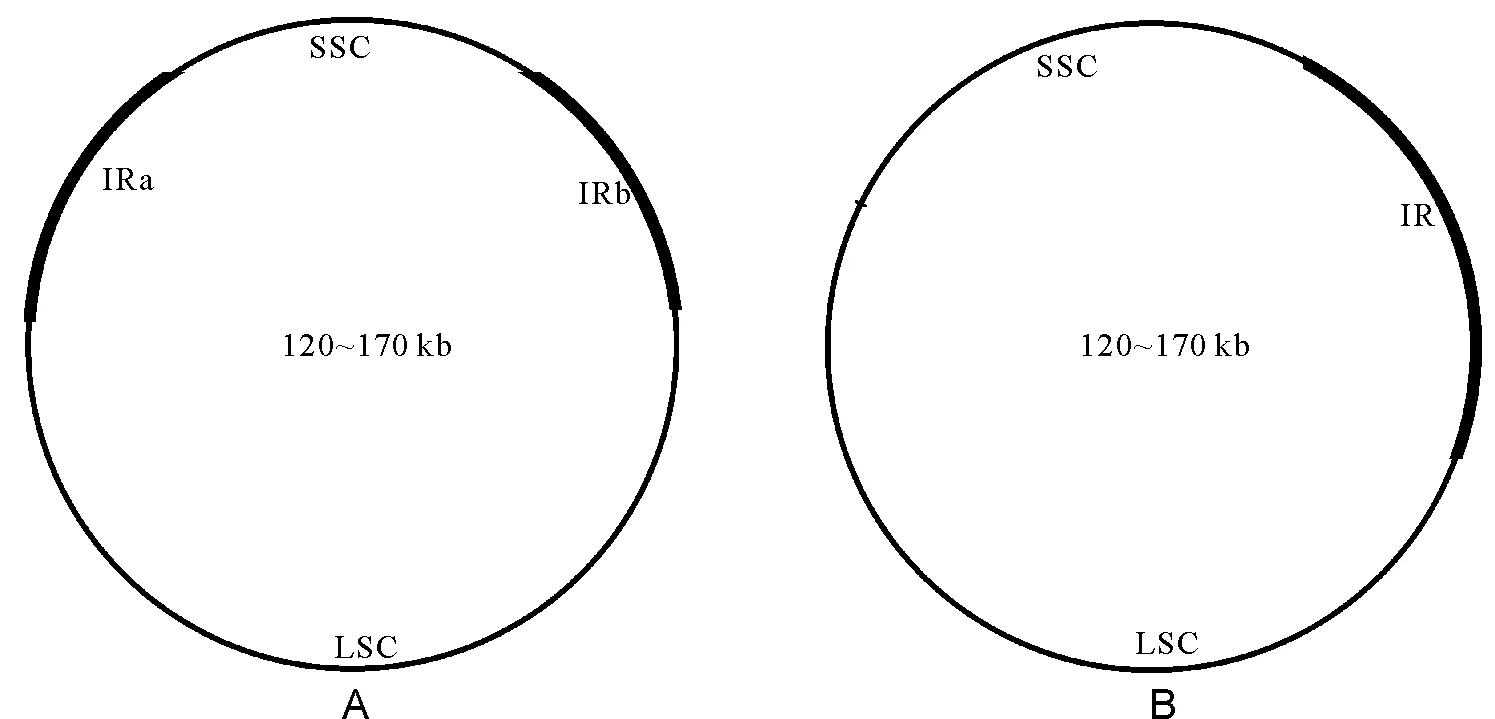
图1 一般叶绿体[1]和特殊叶绿体基因组(豆科)结构示意图Fig.1 Gene schematic diagram of general[1] and special chloroplast genome(Legumes)
注:图中LSC代表叶绿体结构中大的单拷贝区域,SSC代表小的单拷贝区域,A图中IRA和IRB分别表示反向重复序列的两个片段,其序列完全相同,但排列方向相反;B图中IR也代表反向重复序列,但一些植物中一条反向重复序列丢失。
Note: LSC means a large simple copy and SSC means a small simple copy of the chloroplast structure in the
Fig. Both of IRA and IRB are two invert repeat sequences. there are the same sequence, but difference directions in the
Fig.A. IR is the only of invert repeat sequence, because of disappearing of another of some plants in the
Fig.B.

表2 豆科牧草的叶绿体基因组全序列及GenBank号
注:“Trifoliumboissieri”和“Trifoliummeduseum”两个物种,只在NCBI上有记载,没有明确的中文名称。
Note: The two species, “Trifoliumboissieri” and “Trifoliummeduseum”, were merely recorded in NCBI, not named specifically with Chinese.
2.4 其他科牧草叶绿体基因组测序
相对禾草和豆科牧草,其他科牧草则起到了辅助作用,而且这些牧草大部分来源于野生植物。关于野生牧草的研究并不多,只有少数几个物种获得其叶绿体基因组全序列,如菊科的冷蒿(Artemisiafrigida)、蒙大纳蒿(A.montana)、矢车菊(Centaureadiffusa),报春花科的虎尾菜(Penthorumchinense)以及唇形科的牛至(Origanumvulgare)(表3)。
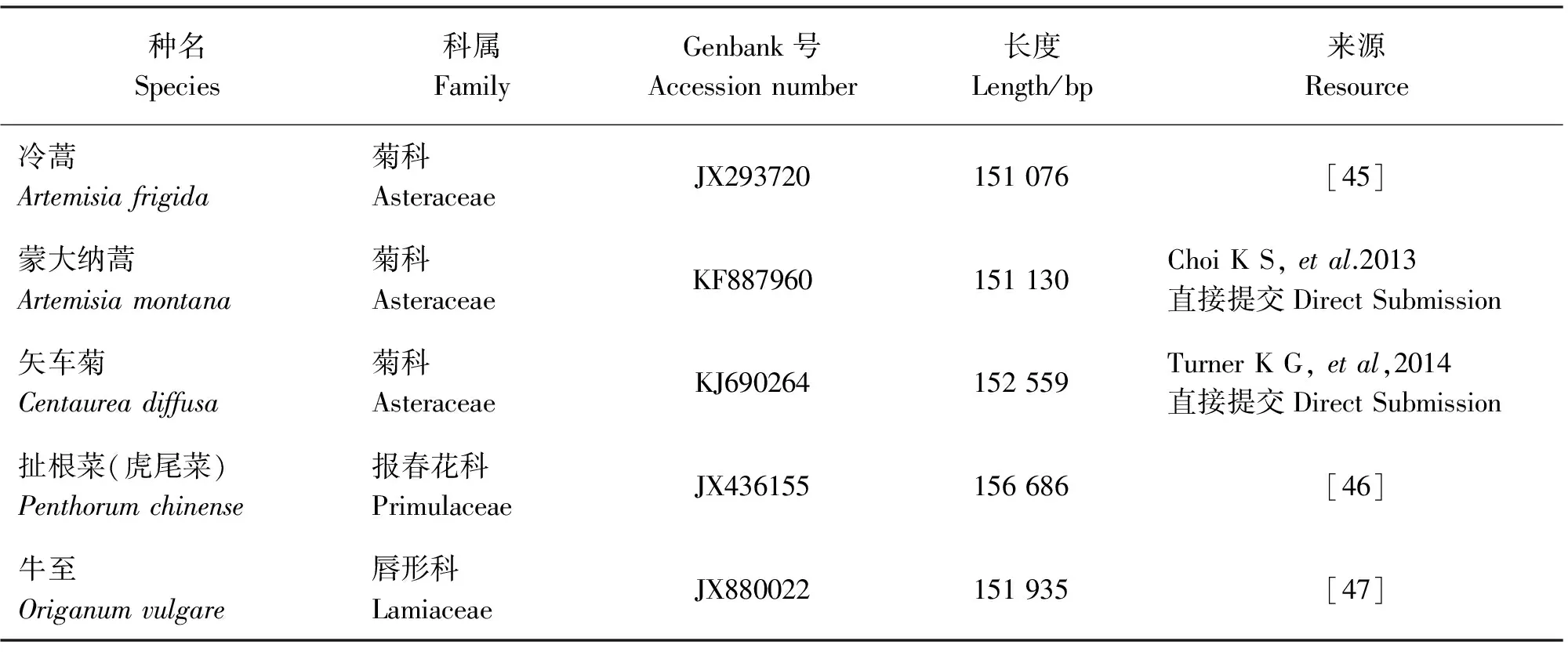
表3 其他科牧草叶绿体基因组全序列及GenBank号
研究表明,叶绿体基因组的结构变化可以解决被子植物的主要分化枝以及很多科植物的系统发育关系[30]。由于来自不同的进化枝,所以,不同科的植物其叶绿体基因组的结构变化也不一样,如菊科[48]、豆科[36]、禾本科[29]等。但是,不论对于哪一科、属的植物,研究方法是一致的,都会涉及叶绿体基因组的结构变化,基因的转移和丢失,叶绿体基因组基因的排列顺序的变化,是否发生倒置,基因内含子的缺失情况以及叶绿体DNA上是否发生重排等等一系列的问题。
对于作为新兴产业的草业,无论是草坪还是牧草,无论是栽培牧草还是天然牧草,其重要性不言而喻,所以牧草叶绿体基因组研究无疑会在这一领域创造出新的辉煌。对其叶绿体基因组的研究,也将不仅仅局限于测序,而是在叶绿体基因组的基因功能、核质互作关系以及系统进化关系等方面做深入研究。每一个经过叶绿体基因组研究的物种都可以作为一个参考样本,为将来的牧草研究导航。
3 展望
虽然叶绿体基因组的研究在迅速发展,而且基因库中叶绿体基因组全序列的数据也在近几年迅速充实,但由于庞大的物种数量,而目前数据库中收录的仅仅是其中很小的一部分,且对于牧草叶绿体基因组研究而言,更是微乎其微,所以牧草叶绿体基因组的全序列有待充实。然而,对于已经测序成功的牧草,其大部分仍然停留在叶绿体基因组全序列的完成和提供,并未进行深入研究。所以学者们以及研究人员需要对其序列进行整合分析,研究叶绿体基因组的基因功能、核质互作关系以及系统进化关系,为社会生产实践做贡献。这是目前亟待需要解决的关键问题。
由于植物叶绿体基因组高拷贝数(拷贝数高的可达数万个)、高保守性的特点,且在IR区域的基因出现加倍现象,因此叶绿体基因组相对于复杂庞大的核基因组更具有特点。Daniell等[49]、Grevich和Daniell[50]的研究表明植物叶绿体中表达的目的蛋白占可溶性蛋白的比例高达46.1%,非常适合作为遗传转化的载体,而且核基因组转化有一些难以克服的缺陷,如核基因组基因数目和功能较叶绿体基因组复杂庞大得多,而且遗传背景复杂,对于外源基因又难以控制等,另外,叶绿体作为半自主细胞器,具有一套自己的遗传系统,可以防止基因污染、基因泄漏等问题,因此,叶绿体遗传转化将成为继核转化之后的一项非常有效的遗传转化新技术。
致谢:该论文是第二届全国草业生物技术大会评选出的优秀论文,并得到中国草业生物技术专业委员会提供的版面费支持。
[1] 邢少辰,CLARKE J L.叶绿体基因组研究进展[J].生物化学与生物物理进展,2008,35(1):21-28.
[2] Liu Z P,Chen Z Y,Pan J,Li X F,Su M,Wang L J,Li H J,Liu G S.Phylogenetic relationships in Leymus (Poaceae:Triticeae) revealed by the nuclear ribosomal internal transcribed spacer and chloroplasttrnL-Fsequences[J].Molecular Phylogenetics and Evolution,2008,46:278-289.
[3] 谢树莲,凌元洁.串珠藻目的现代地理分布和起源[J].山西大学学报·自然科版,2002,25(2):149-155.
[4] Correns C.The foundation of extranuclear inheritance:Plastid and mitochondrial genetics[J].Molecular Genetics and Genomics,2010,283(3):199-209.
[5] Ris H,Plaut W.Ultrastructure of DNA-containing areas in the chloroplast of Chlamydomonas[J].Biology of the Cell,1962,13:383-391 .
[6] 梁明山,陈斌,吴书惠.油菜叶绿体DNA的提取和纯化[J].中国油料,1995,17(2):57-60.
[7] Maliga P.Toward plastid transformation in flowering plants[J].Tibtech,1993(11):101-107.
[8] Shinozaki K,Ohme M,Tanaka M,Wakasugi T,Hayashida N,Matsubayashi T,Zaita N,Chunwongse J,Obokata J,Yamaguchi S K,Ohto C,Torazawa K,Meng B Y,Sugita M,Deno H,Kamogashira T,Yamada K,Kusuda J,Takaiwa F,Kato A,Tohdoh N,Shimada H,Sugiura M.The complete nucleotide sequence of the tobacco chloroplast genome its gene organization and expression[J].The EMBO Journal,1986,5(9):2043-2049.
[9] Ohyama K,Fukuzawa H,Kohchi T,Shirai H,Sano T,Sano S,Umesono K,Shiki Y,Takeuchi M,Chang Z,Aota S,Inokuchi H,Ozeki H.Chloroplast gene organization deduced from complete sequence of liverwortMarchantiapolymorphachloroplast DNA[J].Nature,1986,322:572-574.
[10] Jansen R K,Raubeson L A,Boore J L,Pamphilis C W,Chumley T W,Haberle R C,Wyman S K,Alverson A J,Peery R,Herman S J,Fourcade H M,Kuehl J V,McNeal J R,Leebens-Mack J,Cui L.Methods for obtaining and analyzing whole chloroplast genome sequences[J].Methods in Enzymology,2005,395:348-384.
[11] 张同武.植物细胞器基因组测序,组装及比较基因组学研究[D].杭州:浙江大学博士论文,2012.
[12] Shahid Masood M,Nishikawa T,Fukuoka S,Njenga P K,Tsudzuki T,Kadowaki K.The complete nucleotide sequence of wild rice(Orizynivara) chloroplast genome:First genome wide comparative sequence analysis of wild and cultivated rice[J].Gene,2004,340(1):133-139.
[13] Tang J,Xia H,Cao M,Zhang X,Zeng W,Hu S,Tong W,Wang J,Wang J,Yu J,Yang H,Zhu L.A comparison of rice chloroplast genomes[J].Plant Physiology,2004,135(1):412-420.
[14] Hiratsuka J,Shimada H,Whittier R,Ishibashi T,Sakamoto M,Mori M,Kondo C,Honji Y,Sun C R,Meng B Y,Li Y Q,Kanno A,NishizawaY,Hirai A,Shinozaki K,Sugiura M.The complete sequence of the rice(Oryzasativa) chloroplast genome:Inter molecular recombination between distinct tRNA genes accounts for a major plastid DNA inversion during the evolution of the cereals[J].Molecular and General Genetics,1989,217(2-3):185-194.
[15] 吉莉,谢树莲,冯佳.藻类植物叶绿体基因组研究进展[J].西北植物学报,2010,30(1):208-214.
[16] Kolondener R,Tewari K K.Inverted repeats in chloroplast DNA from high plants[J].Proceedings of the National Academy of Sciences USA,1979,76:41-45.
[17] Plamer J D.Isolation and structural analysis of chloroplast DNA[J].Methods and Enzymology,1986,118:167-186.
[19] Bookjans G,Stummann B M,Henningsen K W.Preparation of chloroplast DNA from pea plastids isolated in a medium of high ionic strength[J].Nalytical Biochemistry,1984,141(1):244-247.
[18] 胡华萃,王进,沈桂芳,唐愫.氯化铯密度梯度离心法纯化叶绿体DNA[J].杭州大学学报,1985,12(3):370-373.
[20] 赵衍,翁醒华.纯化水稻叶绿体DNA的新方法[J].杭州大学学报,1986,13(3):386-389.
[21] 龚小松,阎隆飞.高等植物叶绿体DNA提纯方法的改进[J].科学通报,1991(6):467-469.
[22] Sanger F,Nicklen S,Coulson A R.DNA sequencing with chain terminating inhibitors[J].Proceedings of the National Academy of Sciences USA,1977,74:5463-5467.
[23] Pyke K A. Plastid division and development[J].Plant Cell,1999,11(4):549-560.
[24] Cai Z,Guisinger M,Kim H G,Ruck E,Blazier J C,McMurty V,Kuehl J V,Boore J,Jansen R K.Extensive reorganization of the plastid genome ofTrifoliumsubterraneum(Fabaceae) is as sociated with numerous repeated sequences and novel DNA insertions[J].Journal of Molecular Evolution,2008,67(6):696-704.
[25] Jansen R K,Wojciechowski M F,Sanniyasi E,Lee S B,Daniell H.Complete plastid genome sequence of the chickpea (Cicerarietinum) and the phylogenetic distribution ofrps12 andclpPintron losses among legumes(Leguminosae)[J].Molecular Phylogenetics and Evolution,2008,48:1204-1217.
[26] Raghvendra A S.Photosynhesis:A comprehensive treatise[M].Cambridge:Cambridge University Press,1998:72-86.
[27] Cahoon A B,Sharpe R M,Mysayphonh C,Thompson E J,Ward A D,Lin A.The complete chloroplast genome of tall fescue (Loliumarundinaceum;Poaceae) and comparison of whole plastomes from the family Poaceae[J].American Journal of Botany,2010,97(1):49-58.
[28] Middleton C P,Senerchia N,Stein N,Akhunov E D,Keller B,Wicker T,Kilian B.Sequencing of chloroplast genomes from wheat,barley,rye and their relatives provides a detailed insight into the evolution of the triticeae tribe[J].PLoS ONE,2014,9(3):E85761.
[29] Diekmann K,Hodkinson T R,Fricke E,Barth S.An optimized chloroplast DNA extraction protocol for grasses (Poaceae) proves suitable for whole plastid genome sequencing and SNP detection[J].PLoS ONE,2008,3(7):E2813.
[30] Young H A,Lanzatella C L,Sarath G,Tobias C M.Chloroplast genome variation in upland and lowland switchgrass[J].PLoS ONE,2011,6(8):E23980.
[31] Bortiri E,Coleman-Derr D,Lazo G R,Anderson O D,Gu Y Q.The complete chloroplast genome sequence ofBrachypodiumdistachyon:Sequence comparison and phylogenetic analysis of eight grass plastomes[J].BMC Medical Reserarch Methodology,2008,1:61.
[32] Smith S A,Donoghuem J.Rates of molecular evolution are linked to life history in flowering plants[J].Science,2008,322:86-89.
[33] Katayama H,Ogihara Y.Structural alterations of the chloroplast genome found in grasses are not common in monocots[J].Current Genetics,1993,23(2):160-165.
[34] 唐萍,阮秋燕,彭程.禾本科植物叶绿体基因组结构的系统进化研究[J].中国农学通报,2011,27(30):171-176.
[35] Doyle J J,Davis J I,Soreng R J,Garvin D,Anderson M J.Chloroplast DNA inversions and the origin of the grass family (Poaceae)[J].Proceedings of the National Academy of Sciences USA,1992,89:7723-7726.
[36] Kato T,Kaneko T,Sato S,Nakamura Y,Tabata S.Complete structure of the chloroplast genome of a legume,Lotusjaponicus[J].Mutation Reserarch-DNA Repair,2000,7(6):323-330.
[37] Sveinsson S,Cronk Q.Evolutionary origin of highly repetitive plastid genomes within the clover genus(Trifolium)[J].BMC Evolutionary Biology,2014,14(1):228.
[38] Magee A M,Aspinall S,Rice D W,Cusack B P,Semon M,Perry A S,Stefanovic S,Milbourne D,Barth S,Palmer J D,Gray J C,Kavanagh T A,Wolfe K H.Localized hypermutation and associated gene losses in legume chloroplast genomes[J].Genome Reserach,2010,20(12):1700-1710.
[39] Martin G E,Rousseau-Gueutin M,Cordonnier S,Lima O,Michon-Coudouel S,Naquin D,de Carvalho J F,Ainouche M,Salmon A,Ainouche A.The first complete chloroplast genome of the Genistoid legumeLupinusluteus:Evidence for a novel major lineage-specific rearrangement and new insights regarding plastome evolution in the legume family[J].Phyton-Annales Rei Botanicae,2014,113(7):1197-1210.
[40] Millen R S,Olmstead R G,Adams K L,Palmer J D,Lao N T,Heggie L,Kavanagh T A,Hibberd J M,Gray J C,Morden C W,Calie P J,Jermiin L S,Wolfe K H.Many parallel losses of infA from chloroplast DNA during angiosperm evolution with multiple independent transfers to the nucleus[J].Plant Cell,2001,13(3):645-658.
[41] Palmer J D,Thompson W F.Rearrangements in the chloroplast genomes of mung bean and pea[J].Proceedings of the National Academy of Sciences USA,1981,78:5533-5537.
[42] Doyle J J,Doyle J L,Palmer J D.The distribution and phylogenetic significance of a 50 kb chloroplast DNA inversion in the flowering plant family Leguminosae[J].Molecular Phylogenetics and Evolution,1996,5:429-438.
[43] Bruneau A,Doyle J J,Palmer J D.A chloroplast DNA inversion as a subtribal character in the Phaseoleae (Leguminosae)[J].Australian Systematic Botany,1990,15:378-386.
[44] Palmer J D,Osorio B,Thompson W F.Evolutionary significance of inversions in legume chloroplast DNAs[J].Current Genetics,1988,14(1):65-74.
[45] Liu Y,Huo N X,Dong L L,Wang Y,Zhang S X,Young H A,Feng X X,Gu Y Q.Complete chloroplast genome sequences of mongolia medicine artemisia frigida and phylogenetic relationships with other plants[J].PLoS ONE,2013,8(2):E57533.
[46] Dong W,Xu C,Cheng T,Lin K,Zhou S.Sequencing angiosperm plastid genomes made easy:A complete set of universal primers and a case study on the phylogeny of saxifragales[J].Genome Biology Evolution,2013,5(5):989-997.
[47] Lukas B,Novak J.The complete chloroplast genome ofOriganumvulgare(Lamiaceae)[J].Gene,2013,528(2):163-169.
[48] Jansen R K,Palmer J D.A chloroplast DNA inversion marks an ancient evolutionary split in the sunflower family(Asteraceae)[J].Proceedings of the National Academy of Sciences USA,1987,84:5818-5822.
[49] Daniell H,Khan M S,Allison L.Milestones in chloroplast genetic engineering:An environmentally friendly era in biotechnology[J].Trends in Plant Science,2002,7:84-91.
[50] Grevich J J,Daniell H.Chloroplast genetic engineering:Recent advances and future perspectives[J].Critical Reviews in Plant Sciences,2005,24:83-107.
(责任编辑 王芳)
本刊如有印装质量问题,请将原杂志寄回编辑部,由本部负责调换。
Progress in chloroplast genome analysis of herbage
TAO Xiao-li, WANG Yan-rong, LIU Zhi-peng
(Key Laboratory of Grassland Farming Systems, College of Pastoral Agriculture Science and Technology, Lanzhou University, Lanzhou 730020, China)
Chloroplasts of plant is an important organelle of photosynthesis and energy conversion and it’s also an important carrier of female genetic information. Chloroplast genome information is valuable in revealing the evolution of species, hybrids, evolution, species identification and other aspects. With the rapid development of plant chloroplast genome research, researches in herbage chloroplast genome are also executed by leaps and bounds whereas some herbage have not been studied in-depth and only limited in the chloroplast genome sequences. As the importance of chloroplast genome research, the recent relevant information was collected and the latest research progress was summarized in this field from several aspects including isolation of chloroplast DNA, purification, sequencing to understand more about chloroplast DNA purification and the research of status, future trends and prospects of forage chloroplast genome.
herbage; chloroplast; genome; purification; sequencing
LIU Zhi-peng E-mail:lzp@lzu.edu.cn
10.11829j.issn.1001-0629.2015-0105
2015-02-14 接受日期:2015-04-21
国家重点基础研究发展计划(973计划)——重要牧草、乡土草抗逆优质高产的生物学基础(2014CB138704);国家自然科学基金面上项目——紫花苜蓿种子发育分子机理的研究(31272492)
陶晓丽(1990-),女,甘肃武威人,在读硕士生,主要从事草坪生物学与生物技术研究。E-mail:taoxl14@lzu.edu.cn
刘志鹏(1979-),男,陕西咸阳人,副教授,博士,主要从事草类作物分子育种研究。E-mail:lzp@lzu.edu.cn
S540.1;Q943.2
A
1001-0629(2015)06-0978-10
陶晓丽,王彦荣,刘志鹏.牧草叶绿体基因组研究进展[J].草业科学,2015,32(6):978-987.
TAO Xiao-li,WANG Yan-rong,LIU Zhi-peng.Progress in chloroplast genome analysis of herbage[J].Pratacultural Science,2015,32(6):978-987.
