水稻S5-ORF3蛋白质的进化分析
2014-08-29张达瀚
张达瀚,张 屹
(1. 华中农业大学生命科学技术学院,湖北武汉 430070;2.河北科技大学理学院,河北石家庄 050018)
水稻S5-ORF3蛋白质的进化分析
张达瀚1,张 屹2
(1. 华中农业大学生命科学技术学院,湖北武汉 430070;2.河北科技大学理学院,河北石家庄 050018)
生殖隔离在形成新物种和物种同一性的保持中有重要作用,因而在水稻优化育种中有重要的意义。S5位点是一个已克隆的控制水稻生殖隔离的基因,它产生的S5-ORF3,ORF4和ORF5蛋白质共同调控着indica-japonica杂交后代的育性,特别是S5-ORF3蛋白质在打破水稻亚种indica-japonica之间的生殖隔离与促进物种间的基因交流中发挥重要作用。针对S5-ORF3的进化分析对于研究它的功能和起源很重要,但目前尚无报道。本文基于序列和进化分析,提出S5-ORF3为一种定位在内质网中的新的HSP70家族蛋白,但缺乏HSP70的C端的多肽结合结构域,推测S5-ORF3可能通过影响alpha-淀粉酶的合成来作用于内质网压力;找到了ORF3的19条同源蛋白,并用极大似然算法构建了可靠的进化树,发现水稻的S5-ORF3与节节麦的luminal-binding蛋白进化关系最为密切;作了置信度100%的S5-ORF3蛋白质的三维结构预测,预测了居中的配体绑定位点;发现了水稻的S5-ORF3与其他HSP70蛋白相比独特的基序,为进一步研究S5-ORF3的作用机理和演化历史提供了线索和数据支持。
水稻;S5-ORF3 蛋白;进化树;正向选择;配体结合
生殖隔离限制了物种间的基因交流,一般来说,水稻的2个亚种indica和japonica间产生的后代是不育的[1-2]。研究发现,在水稻基因组的S5位点有一个与生殖隔离相关的“破坏与保护蛋白系统”,该系统由3个紧密连锁的基因ORF3,ORF4,ORF5组成,这三者共同调控着indica-japonica杂交后代的育性,ORF4+与ORF5+导致了内质网的压力,这种压力导致杂交水稻雌配子败育,而ORF3+作为保护者又会阻止内质网压力的产生,从而抑制雌配子败育并打破生殖隔离,使indica和japonica之间的基因可以交流[3]。研究发现S5-ORF3与HSP70有一定的同源性。本研究希望知道哪些蛋白与ORF3同源,它们的进化关系如何;水稻的S5-ORF3有哪些正向选择位点一直伴随和影响着这组蛋白质从古至今的功能变化;水稻的S5-ORF3蛋白质的结构和与配体的结合位点是怎样的等等,这些对于进一步理解这个关键的蛋白质有重要的意义。本研究重点进行S5-ORF3的进化分析,为进一步理解水稻生殖隔离和基因交流提供线索和数据支持。
植物的HSP70是由多个基因家族编码的。HSP70家族按表达情况可分为HSC70 (heat shock cognate protein 70)和 HSP70两大类。HSC70在所有细胞内都能表达,同时也受一定程度的热诱导,是结构型HSP;HSP70在正常情况下表达很少或不表达,而在热刺激或其他胁迫下表达迅速增强,属于诱导型的HSP。根据它在植物中的亚细胞定位的位置,HSP70可分为4个亚族[4]:1)C端含有EEVD基序的HSP70,定位于细胞质中;2)含HDEL基序的HSP70,定位在内质网中,也称BiP蛋白;3)含PEAEYEEAKK的HSP70,定位于线粒体中;4)含PECDVLDADFTDSK的HSP70,定位于质体中。总体来说,HSP70易于存在于细胞器中。但是水稻的S5-ORF3蛋白质中没有这几种基序(motif),只含有一个与内质网中的HSP70相似的基序HDLL。从严格意义上说,水稻的S5-ORF3是一种新的HSP70。
与S5-ORF3最相近的这种定位在内质网中的HSP70,即BiP蛋白,全名叫 the luminal binding protein, 是一种分子伴侣。在内质网压力情况下,BiP的过表达有助于alpha-淀粉酶[5]的合成。但目前S5-ORF3的热激诱导实验还没有做过,不能断定它是否为一种HSC70,也不能断定S5-ORF3缓解内质网压力的机制是否也与alpha-淀粉酶的合成有关。
1 材料与方法
1.1数据
水稻的 S5-ORF3 蛋白质有4 条:JX138498—JX138501,可从NCBI上下载得到。选择indicagroup cultivar Nanjing 11 为 Query Sequence,通过blastp (protein-protein blast) 方法,从NCBI上执行同源搜索而得到100条蛋白质序列(数据库更新至2014-01-25)。由于hypothetical protein与predicted protein,partial protein以及putative protein不能保证蛋白质的准确性和完整性,所以舍去在说明行中含有partial,hypothetical, predicted,candidate division,uncluture,putative单词的序列,同时也舍去没有明确说明所属物种的序列。然后,在余下的序列中为每个物种保留一条ident值最高的序列。最终,得到S5-ORF3的19条同源序列。加上水稻的S5-ORF3蛋白质4条,此次蛋白质进化分析所用的蛋白质序列共23条,它们所对应的物种名字及Gene Bank ID如下所示:
japonicagroup cultivar balilla 水稻培育品种balilla: gi|406066535
indicagroup cultivar Nanjing 11 水稻培育品种南京11号: gi|406066539
cultivar02428 水稻培育品种02428: gi|406066554
cultivardular 水稻培育品种dular: gi|406066557
aegilopstauschii节节麦: gi|475588157
populustrichocarpa毛果杨: gi|566196410
glycinemax大豆: gi|2642238
ricinuscommunis蓖麻: gi|255555659
solanumlycopersicum番茄: gi|350539203
hordeumvulgaresubsp.vulgare大麦: gi|476003
triticumurartu乌拉尔图小麦: gi|474200923
theobromacacao可可: gi|508777841
cyanophoraparadoxa蓝载藻: gi|255977229
capsicumsp. JYM-2013 辣椒: gi|528746217
nicotianatabacum烟草: gi|729623
gossypiumhirsutum陆地棉: gi|211906506
cucumissativus黄瓜: gi|6911549
arabidopsisthaliana拟南芥: gi|15241844
vitisvinifera葡萄: gi|297742397
zeamays玉米: gi|162457723
maluspumila苹果: gi|57639078
pseudotsugamenziesii花旗松: gi|7635897
spinaciaoleracea菠菜: gi|3913786
1.2方法
S5-ORF3的同源蛋白搜索用blastp (protein-protein blast)方法,进化树的绘制用 Mega5[6]的Max-likelihood方法,保守motif的发现用MEME[7],正向选择位点用Mrbayes 3.1.2[8]计算,蛋白质结构与配体搜索用网上软件Phyre2[9]和3DligandSite[10]执行。图1示出了水稻4条S5-ORF3蛋白质与其19条同源蛋白序列的联配情况。
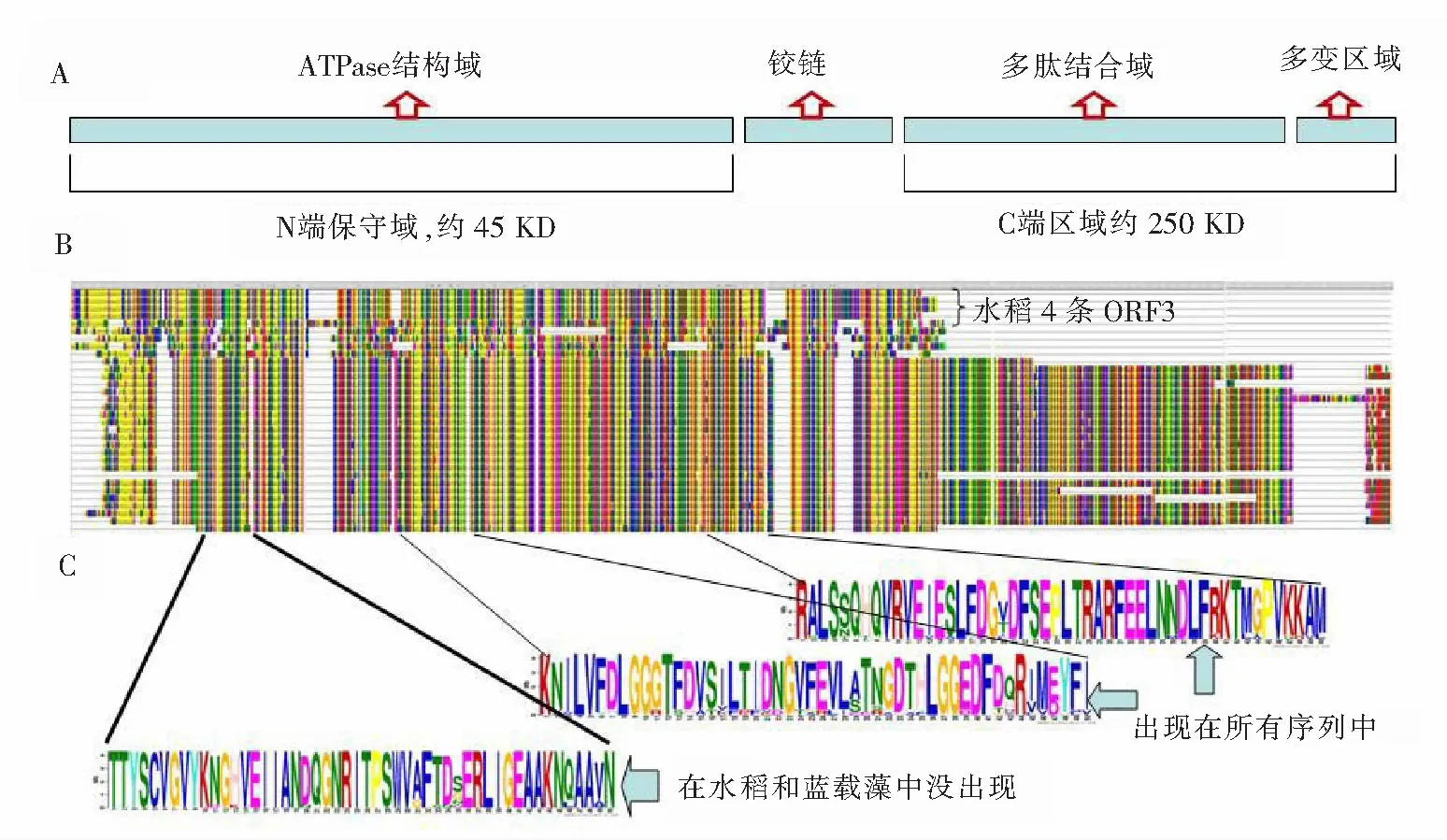
图1 4条S5-ORF3蛋白质与其19条同源蛋白序列的联配Fig.1 Alignment of 4 S5-ORF3 proteins and its 19 homologous sequences
A—HSP 70的结构示意图,KD为蛋白质质量单位,千道尔顿;B—23条序列的联配图,其中,水稻的4条ORF3序列(JX138498—JX138501)在最上方,其下面紧挨着的是节节麦的同源序列,它们与其他序列(包括HSP 70)相比,明显缺乏后面的C-端区域;C—S5-ORF3蛋白质及其同源序列中置信度最高的3个基序(motif),其中第3个motif“TTYS…”motif在水稻的S5-ORF3与蓝载藻Bip蛋白中没有,另外2种motif则是所有序列所共有的
2 结果与讨论
用Nanjing 11 S5-3(indicagroup cultivar Nanjing 11 gene的产物)在NCBI中用blastp进行搜索,找到19条有代表性的同源序列,发现ORF3的产物蛋白质主要与Luminal-binding protein和HSP70在进化上相关。
2.1与HSP70蛋白质的相关性分析
植物的HSP 70也与植物的胁迫应答机制、分子伴侣的作用及抗逆性的提高有密切关联[11],整体来说(见图1 A),包括N端保守域和C端区域2个部分。ORF3在blastp搜索中,局部结构域展现了与HSP 70的高度相关性,如图1 B 所示,ORF3与HSP 70蛋白的N端出现了大段的连配区域,说明ORF3有ATPase相似的结构域,但缺乏HSP 70后面的C端区域。图1 B 最上面的4条序列是水稻的S5-ORF3序列:JX138498—JX138501,发现在联配图中,“AIAIPRAAPA”为S5-ORF3所特有,而它的同源蛋白质没有氨基酸区域;其他序列所拥有的从“VIPAKKSQTFTTYQDQQTTVTI…”开始的蛋白质区域也是S5-ORF3蛋白质所没有的。笔者推测,这2个片段在决定S5-ORF3与luminal-binding蛋白和HSP 70蛋白在结构和功能差异上发挥重要作用。
2.2与luminal-binding蛋白质的相关性分析
这23条序列的blast联配结果(见图1 B)显示,水稻的S5-ORF3与gi|475590630|gb|EMT21528.1| luminal-binding protein 2 [aegilopstauschii] (节节麦)、gi|255977229|dbj|BAH97106.1| luminal binding protein BipCyanophoraparadoxa(早期散发藻类蓝载藻)最为相似,也就是说它与luminal-binding蛋白质的相似程度最大。luminal-binding 蛋白缓解内质网压力,增强烟草和大豆对于旱灾的抗逆性[5,12-13],而S5-ORF3所产生的蛋白质也有缓解内质网压力的功能[3],所以,推断它与luminal-binding 的共同结构域对于缓解内质网压力是至关重要的。
2.3在水稻S5-ORF3蛋白质及其同源序列中寻找保守基序和正向选择位点
为了在S5-ORF3蛋白质及其同源序列中寻找保守的基序(motif),笔者应用了MEME suite[7],并且把发现的前3个得分最高的motif展示在图1 C中。这些motif的生物学功能有待进一步研究。在图1 C中从上到下,第1个motif的正则表达式为RALSSQHQVRVEIESLFDG[VT]DFSEPLTRARFEELNNDLFRKTMGPVKKAM,E值为7.8×10-734,在所有序列中都存在;第2个motif的正则表达式为KNILVFDLGGGTFDVSILTIDNGVFEVL[AS]TNGDTHLGGEDFD[QH]R[IV]M[ED]YFI,E值为4.0×10-724,是在所有序列中都有的;第3个motif的正则表达式为TTYSCVGVYKNGHVEIIANDQGNRITPSWVAFTD[SG]ERLIGEAAKNQAAVN,E值为3.0×10-720,在水稻的S5-ORF3与蓝载藻中没有第3个motif。
在S5-ORF3及其同源体的进化历史中,每个氨基酸或密码子都是一个位点。有许多位点是保守的,因为它们担负着重要的结构和功能,若变化就会导致个体死亡;有的则只进行着中性的漂变,密码子只作同义变化;还有一些位置则担负起功能进化的角色,随着岁月的流逝、物种的进化,它们通过适当的同义和异义的突变使蛋白质和个体更能适应环境与条件,这些密码子为“正向选择位点”。但是,通过MrBayes3.1.2[8]软件的计算,并没有发现S5-ORF3的正向选择位点,因为位点的最大正向选择概率仅为0.132 5,这是不显著的。缺乏正向选择位点应该与HSP70家族蛋白质的保守性有关。
2.4水稻的4条S5-ORF3蛋白质与其19条同源序列所做的进化树
用Mega5中的ML算法构建进化树并且用bootstrap 500次以检查分枝的置信度。如图2所示,S5-ORF3在包括藻类在内的很多植物中都有同源体,水稻的S5-ORF3蛋白质序列与节节麦和蓝载藻的luminal-binding 蛋白有非常紧密的亲缘关系。该进化树的根部分支的bootstrap置信度很高,从而是可靠的。从进化树上看,与ORF3同源的蛋白质序列整体上可分为2组,一组是由节节麦的luminal-binding 蛋白与水稻的S5-ORF3组成的,而HSP70与其他物种的luminal-binding蛋白组成另外一组。从连配图(见图1 B)中可见,这2组序列的最大差异在于它们的C端结构域的有无。
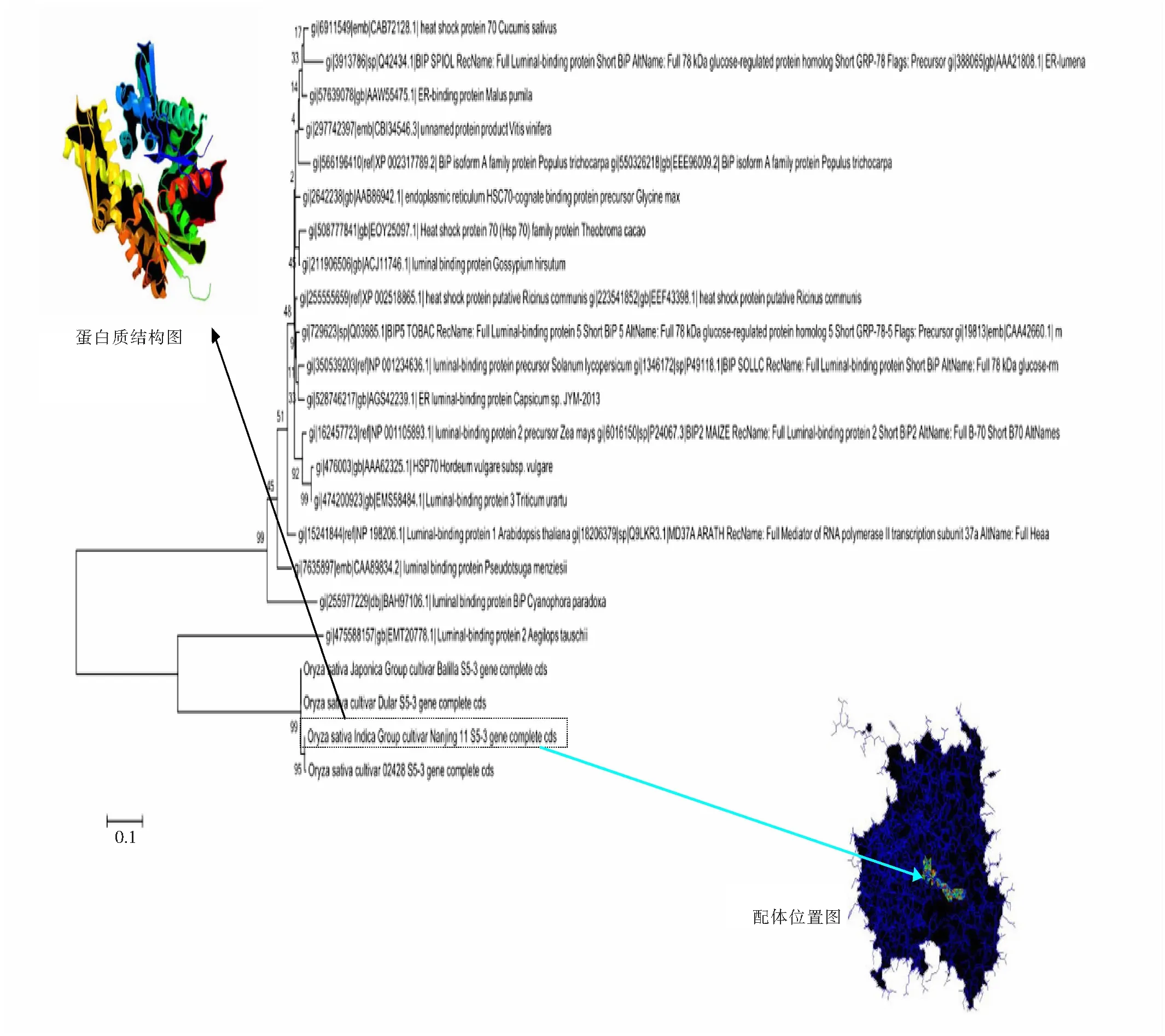
图2 S5-ORF3与19条同源序列的进化树及Nanjing 11 S5-ORF3蛋白质的结构和配体位置图 Fig.2 Evolutionary tree of S5-ORF3 as well as 19 homologous sequences, and the protein structure and the ligand site of Nanjing 11 S5-ORF3
很明显,水稻的4条S5-ORF3蛋白质序列与节节麦和蓝载藻的luminal-binding 蛋白有非常紧密的亲缘关系。其中,结构图由网络服务器Phyre2 预测产生。PDB库中的模板为c3d2fc,这个结构预测可信度100%。N端为红色,C端为紫色。3DligandSite作了配体绑定位点预测,Nanjing 11 S5-ORF3的蛋白质中部有18个可能的配体结合位点。
2.5水稻S5-ORF3蛋白质的结构及可能配体结合位点的预测
用网络服务器Phyre2[9]来预测S5-ORF3 Nanjing 11的蛋白质结构。因为Nanjing 11是典型的ORF3+序列,这个序列对于ORF4和ORF5有显著的上位效应。该蛋白质的结构(如图2的左上方“蛋白质结构图”所示)由15个alpha螺旋、18个beta片和一些coil组成。整体来说,该蛋白质结构来源于sse1p和HSP 70的组合,对于PDB库中的模板c3d2fc的覆盖度为83%,模型预测可信度100%,这应该是得益于HSP70家族的蛋白质保守性很强。此外,发现在S5-ORF3的中部有一个空洞,有可能是配体的结合位置。另外,预测了S5-ORF4的三维结构,是2个巨大的alpha螺旋被一条细小的coil所连接。为了进一步研究S5-ORF3的配体结合的性质,把用网络服务器Phyre2预测得到的S5-ORF3结构的PDB原子坐标文件输入配体结合位点预测软件,3D ligandSite[10]来预测ORF3可能的配体结合位点。这个软件是依据结构的相似性来预测结合位点的,适用于这种保守性很强的蛋白质的配体结合点的预测工作。预测结果如图2右下方的“配体位置图”所示,该配体位于S5-ORF3蛋白质的中部。预测得到的可能的18个配体绑定位点为68HIS, 70GLY,71ASN, 72THR, 73ASN, 260HIS, 262GLY, 263GLY, 264ARG, 291GLY, 292ASP, 329GLU, 332LYS, 333LYS, 336SER, 400GLY, 401SER, 403ARG。
3 结 语
S5-ORF3的热激表达效应、缓解内质网压力的机制是否也与alpha-淀粉酶的合成有关,它中部的配体是什么,S5位点中其他的调控杂交不育的因素,如非编码RNA 及其可能所引发的的上位效应等[14-16],仍需要做进一步的实验研究。
致 谢:感谢华中农业大学张启发院士和欧阳亦聃副研究员的热情帮助。
/
[1] OUYANG Y D, CHEN J J, DING J H, et al. Advances in the understanding of inter-subspecific hybrid sterility and wide-compatibility in rice[J]. Chinese Sci Bull, 2009, 54: 2332-2341.
[2] OUYANG Y D, LIU Y G,ZHANG Q F. Hybrid sterility in plant: Stories from rice[J].Current Opinion in Plant Biology,2010,13:1-7.
[3] YANG J Y, ZHAO X B, CHENG K, et al. A killer-protector system regulates both hybrid sterility and segregation distortion in rice[J]. Science, 2012, 337:1336-1340.
[4] GUY C L, LI Q B. The organization and evolution of the spinach stress 70 molecular haperone gene family[J]. Plant Cell, 1998, 10: 539-556.
[5] NATHALIE L C, EDITH V D, JURGEN D. Overexpression of the luminal binding protein BIP alleviates ER stress[J]. Biology of the Cell, 1998, 90: 109-130.
[6] TAMURA K, PETERSON D, PETERSON N,et al.MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28: 2731-2739.
[7] BAILEY T L, BODEN M, BUSKE F A, et al.MEME SUITE: Tools for motif discovery and searching[J].Nucleic Acids Research, 2009, 37:202-208.
[8] HUELSENBECK J P, RONQUIST F. MrBayes: Bayesian inference of phylogenetic trees[J]. Bioinformatics,2001, 17(8):754-755.
[9] KELLEY L A, STERNBERG M J E. Protein structure prediction on the web: A case study using the phyre server[J].Nature Protocols, 2009, 4:363-371.
[10] WASS M N, KELLEY L A , STERNBERG M J. 3D ligandsite: Predicting ligand-binding sites using similar structures[J]. Nucleic Acid research,2010, 38:469-473.
[11] 齐 妍,徐兆师,李盼松,等.植物热激蛋白70的分子作用机理及其利用研究进展[J]. 植物遗传资源学报,2013, 14(4):507-511. QI Yan, XU Zhaoshi, LI Pansong, et al.Research progress on molecular mechanism and application of HSP70 in plants[J]. Journal of Plant Genetic Resources, 2013, 14(4):507-511.
[12] VALENTE M A S, FARIA J A Q A, SOARES-RAMOS J R L, et al.The ER luminal binding protein (BiP) mediates an increase in drought tolerance in soybean and delays drought-induced leaf senescence in soybean and tobacco[J]. Journal of Experimental Botany, 2009, 60:533-546.
[13] DENECKE J, GOLDMAN M H S, DEMOLDER J, et al. The tobacco luminal binding protein is encoded by a multigene family[J]. The Plant Cell, 1991, 3:1025-1035.
[14] LISA M S, DETLEF W.On epigenetics and epistasis: Hybrids and their non-additive interactions[J].The EMBO Journal, 2012, 31: 249-250.
[15] DING J H, LU Q, OUYANG Y D, et al, A long noncoding RNA regulates photoperiod-sensitive male sterility, an essential component of hybrid rice[J]. PNAS, 2011, 109:2654-2659.
[16] 黄海云,张 屹.基因组装配中存在重复序列叠加时重叠群计数的推广的Lander-Waterman定理[J].河北科技大学报报,2012,33(5):384-385. HUANG Haiyun, ZHANG Yi.Generalized Lander-Waterman theorem under repeat collapse in genome assembly[J].Journal of Hebei University of Science and Technology, 2012,33(5):384-385.
Evolution analysis of rice protein S5-ORF3
ZHANG Dahan1, ZHANG Yi2
(1. College of Life Science and Technology, Huazhong Agricultural University, Wuhan Hubei 430070, China; 2.School of Science, Hebei University of Science and Technology, Shijiazhuang Hebei 050018, China)
Reproductive isolation is important in speciation and maintaining species identity, so it is very significant in optimization of breeding rice. S5 locus is a cloned gene controlling the reproductive isolation, which produces proteins S5-ORF3, ORF4 and ORF5 that work together to regulate fertility in indica-japonica hybrids, and especially the S5-ORF3 protein plays a very important role in distorting reproductive isolation among indica-japonica and enhancing gene flow among populations. The evolutionary analysis of S5-ORF3 is significant for studying its function and origin, which is still no reported. In this paper, based on sequence and evolution analysis, it is proposed that the S5-ORF3 is a new HSP70 family protein which is located in endoplasmic reticulum (ER) and lacks of multi-peptide binding domain. Evolution analysis suggests that S5-ORF3 might act on ER stress via affecting alpha-amylase synthesis; moreover, 19 homologous proteins of S5-ORF3 are found, ML phylogenetic tree is constructed, and it is found that there is close evolution relationship between rice S5-ORF3 and a luminal-binding protein of Aegilops tauschii; the 3D structure of S5-ORF3 with confidence of 100% is predicted, as well as a ligand-binding site located in the middle of S5-ORF3. The specific motif of rice S5-ORF3 among HSP70 family is also found. This work may provide some clues and data for studying function and evolution history of rice S5-ORF3.
rice; protein S5-ORF3; phylogenetic tree; positive selection; ligand-binding
2014-04-04;
2014-05-06;责任编辑:张士莹
国家自然科学基金(J1103510,11171088);教育部重点项目(212011);中央高校基本科研业务费专项资金(2014BC020);河北省自然科学基金(A2011208002)
张达瀚(1996-),男,河北石家庄人,主要从事植物生物学方面的研究。
张 屹教授,博士。E-mail:zhaqi1972@163.com
1008-1542(2014)04-0397-06
10.7535/hbkd.2014yx04015
Q816
A
张达瀚,张 屹.水稻S5-ORF3蛋白质的进化分析[J].河北科技大学学报,2014,35(4):397-403.
ZHANG Dahan, ZHANG Yi.Evolution analysis of rice protein S5-ORF3[J].Journal of Hebei University of Science and Technology,2014,35(4):397-403.
