海绵和海鞘中可培养放线菌的分离与多样性比较
2013-10-13信艳娟ChristopherFRANCO柯才焕
杨 琪, 信艳娟, Christopher FRANCO, 柯才焕, 张 卫,,
(1. 厦门大学 海洋与地球学院, 福建 厦门 361005; 2. 中国科学院 大连化学物理研究所海洋生物产品工程实验室, 大连 116023; 3. Flinders Centre for Marine Bioproducts Development; 4. Department of Medical Biotechnology, School of Medicine, Flinders University, Adelaide, South Australia, 5042, Australia)
随着陆栖微生物在抗生素、酶、酶抑制剂和多糖等生物活性物质方面的大规模开发和应用, 通过寻找新种属或特殊性状的微生物来开发新型天然活性物质的难度越来越大。近年来研发重点转向了前景更为广阔而研发较少的海洋微生物资源, 并成为海洋生物技术开发的主要内容。作为海洋微生物资源开发的前提, 海洋微生物多样性的研究得到了迅速发展[1]。
海绵动物是迄今已知海洋天然产物的最大来源。由于海绵的底栖过滤性摄食和选择性消化等生理特性, 其体内蕴藏了丰富的微生物, 同时海绵中分离得到的丰富天然产物有许多是由其共生微生物产生的[2]。目前海绵相关微生物多样性和活性产物的研究已经获得了很多成果。与活跃的海绵相关微生物研究相比较, 同属于滤食性海洋无脊椎动物的海鞘动物, 虽然已从其体内发现了多种生理活性物质[3], 但是其相关微生物多样性的研究仍处于起步阶段, 研究报道极少。放线菌是一大类有生理活性的次级代谢产物的生产者, 目前所使用的抗生素中, 70%是由放线菌产生, 并且来源于海洋微生物的活性物质中有一半以上来自放线菌[4]。
因此, 真正全面认识海鞘相关放线菌的多样性,将海绵相关放线菌多样性研究方法应用于海鞘, 比较两种动物相关放线菌多样性水平之间的差异, 获得更多的放线菌资源并加以开发应用,是一个很有价值的研究课题。作者选取地域差别较大的中国福建东山湾海域的海绵(山海绵和网架海绵)和中国海南陵水海域的海鞘(皱瘤海鞘和乳突皮海鞘)为研究对象, 比较不同种属之间及不同物种之间相关放线菌多样性差异和特异性关系, 验证海鞘具有高的相关放线菌多样性及存在种属间差异的理论假说, 证明海鞘可作为除海绵之外另一个极具潜力的海洋放线菌和天然活性产物的来源。
1 材料和方法
1.1 样品采集
海绵活体样本采自福建古雷东山湾附近海域潮间带, 经鉴定为山海绵科(Mycalidae)的山海绵属(Mycalesp.)(图 1-A)和网架海绵科(Dictyonellidae)的网架海绵属(Stylissasp.)(图1-B)。海鞘活体样本采自海南陵水县附近水域, 经鉴定为皱瘤海鞘(Styela plicata)(图1-C)和乳突皮海鞘(Molgula manhattensis)(图 1-D)。
1.2 培养基和试剂
根据文献[5]报道, 选择4种有效放线菌分离培养基进行改良, 制备 M1-M4, 另选用放线菌通用培养基高氏一号和细菌通用培养基2216E, 成分见表1。
Taq DNA聚合酶, HhaI(10 U/μL)由BioLabs提供;DNA纯化试剂盒由 Mo BIO提供; DNA Marker由TakaRa提供; 16S rRNA 基因扩增引物 27F(5’-AGAGTTTGATCMTGGCTCAG-3’)、765R (5’- CTGT TTGCTCCCCACGCTTTC-3’)、704F (5’- GTAGCG GTGAAATGCGTA-3’)和 1492R (5’- CGGCTACCTT GTTACGAC-3’)由 GeneWorks提供。

图1 海绵和海鞘样品照片Fig. 1 Photos of sponges and ascidians
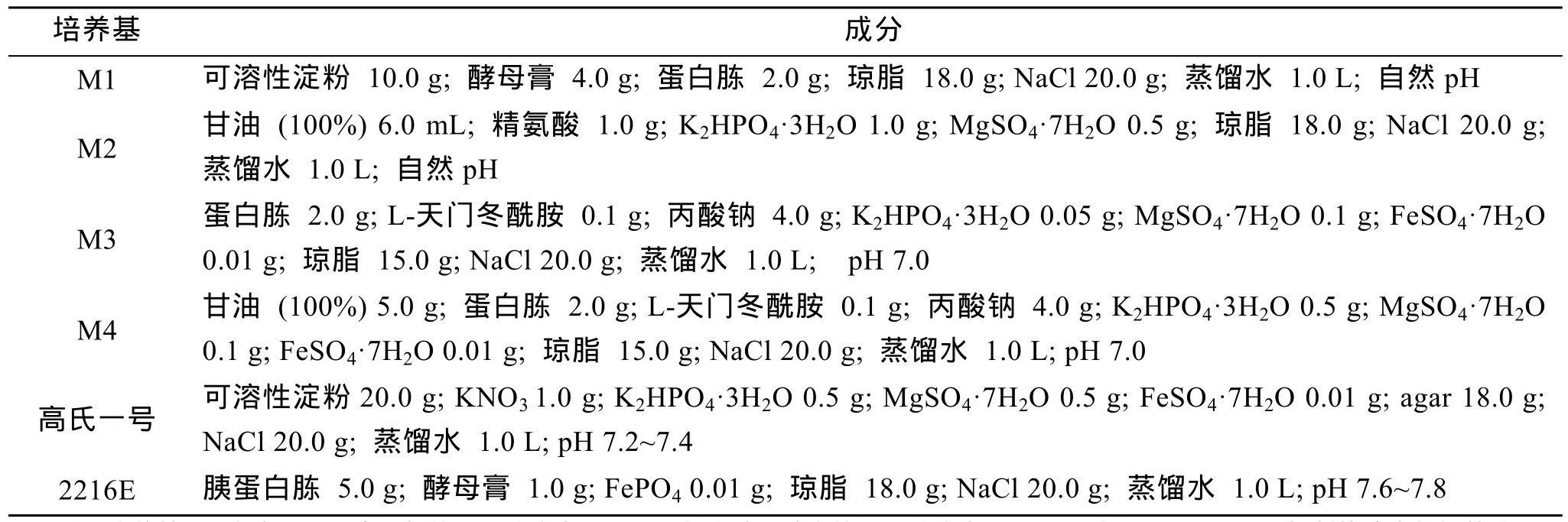
表1 海绵和海鞘放线菌分离培养基及其成分[5]Tab.1 Composition of six different kinds of media used for the isolation of actinomycetes from marine sponges and ascidians
1.3 海绵和海鞘中放线菌的分离、纯化和保藏
将样品在无菌条件下切块, 洗涤, 除去表面附着的海水中微生物和其他杂质; 称量样品5.0 g加入5 mL无菌海水研成匀浆, 为0 #菌液。取1 mL 0#菌液加入9 mL无菌海水制成1 #菌液, 同样方法制备2#菌液。分别取上述菌液 200 μL涂布培养基平板,每种浓度的菌液涂 3个平板; 置于 28℃培养箱中倒置培养7天左右, 观察菌落生长情况。当出现放线菌菌落后, 挑取单菌落划线, 重复分离直至获得纯化菌株。挑取纯化单菌落加入装有1.8 mL 20%甘油水溶液的冻存管中, -80℃保藏。
1.4 基因组DNA提取和16S rRNA基因的PCR扩增
取纯化放线菌菌株2~3环, 采用改良细菌基因组DNA提取方法提取放线菌基因组DNA[6]。用两对引物27F和765R、704F和1492R分别扩增16S rRNA基因。PCR 反应体系(50 μL): 5 μL 10 × ThermoPol Buffer, 1 μL dNTPs(各 10 mmol/L), 1 μL 引物27F(704F)和 1 μL 765R(1492R)(20 μmol/L), 2 μL 模板DNA(≥200 ng), 1 μL TaqDNA 聚合酶(5 U/μL)和 39 μL的纯水。PCR反应条件: 94℃, 2 min; 94℃, 1 min, 52℃,1 min, 72℃, 2 min, 40个循环; 72℃, 延伸10 min; 以20℃终止反应。
1.5 16S rRNA基因限制性片断长度多态性分析(RFLP)和序列分析
16S rRNA基因扩增产物在限制性内切酶 HhaI作用下于37℃反应16 h, 进行琼脂糖凝胶电泳。同时16S rRNA基因扩增产物经纯化后进行测序分析。测序结果与NCBI的GenBank进行Blast比对, 用序列分析软件MEGA5.1构建系统发育树。
2 结果
2.1 菌株分离
根据菌落形态特征以及菌株基丝、气丝的形态特征, 可判断用 6种有效的分离培养基从山海绵和网架海绵中分别分离出放线菌 51株和 36株, 共计87株; 从皱瘤海鞘和乳突皮海鞘中分别分离得到放线菌80株和31株, 共计111株(表2)。
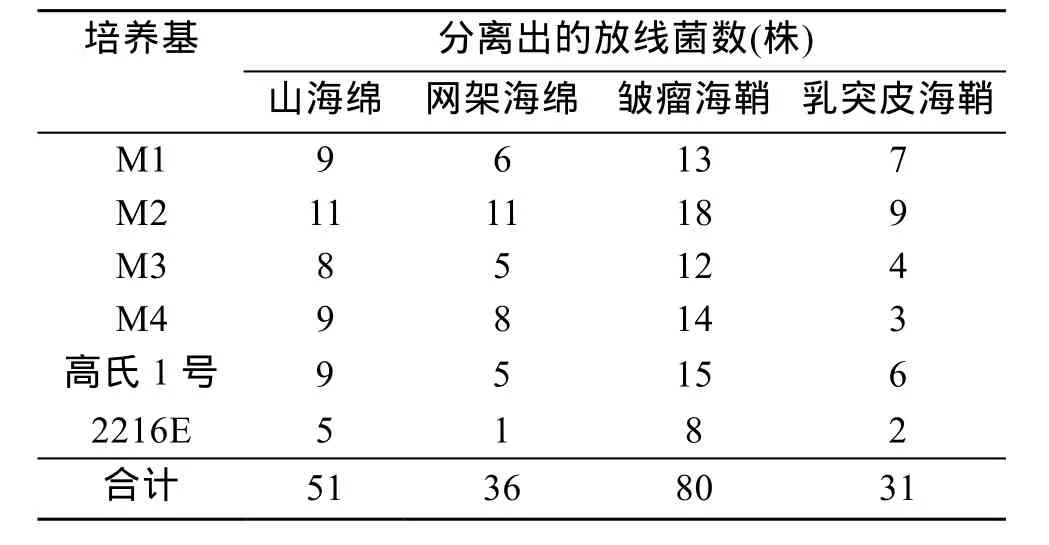
表2 海绵和海鞘在不同培养基中分离得到的放线菌数量Tab. 2 The number of actinobacteria isolated from sponges and ascidians in different isolation media
2.2 RFLP分析
将 198株放线菌的 16S rRNA基因进行 HhaIRFLP分析。结果表明, 198株菌可分成38个RFLP图谱类型(部分代表带型见图2-A和图2-B), 有效的RFLP分组能将菌株初步归类, 为选择代表性菌株进行测序提供依据。结合RFLP带型和测序结果绘制表3和表4。
2.3 16S rRNA基因序列分析
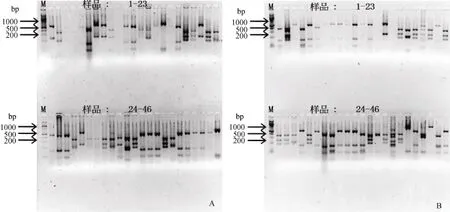
图2 网架海绵和皱瘤海鞘部分相关放线菌16S rRNA基因的RFLP代表性结果Fig. 2 Restriction fragment length polymorphism (RFLP)analysis of some representative actinobacteria isolated from marine sponge Stylissa sp. and marine ascidian Styela plicata
将测得的序列信息分别输入 GenBank中比较,进行属一级的归类, 并构建系统发育树(图3~图6)。

表3 海绵相关放线菌16S rRNA基因的序列分析和RFLP分析Tab. 3 Sequence and RFLP analysis of 16S rRNA gene of actinobacteria from marine sponges Mycale sp. and Stylissa sp.
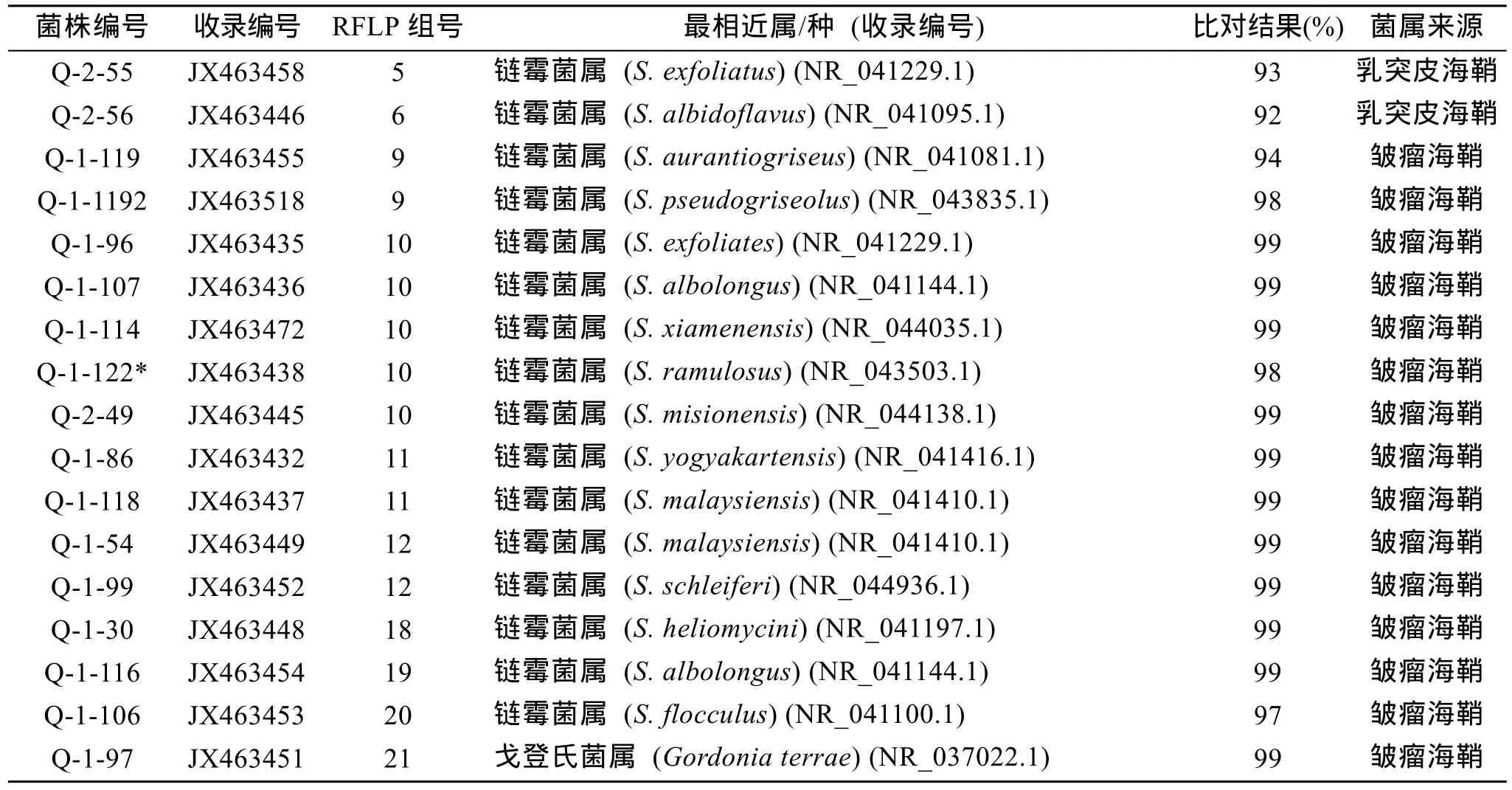
表4 海鞘相关放线菌16S rRNA基因的序列分析和RFLP分析Tab.4 Sequence and RFLP analysis of 16S rRNA gene of actinobacteria from marine ascidians Styela plicata and M. manhattensis
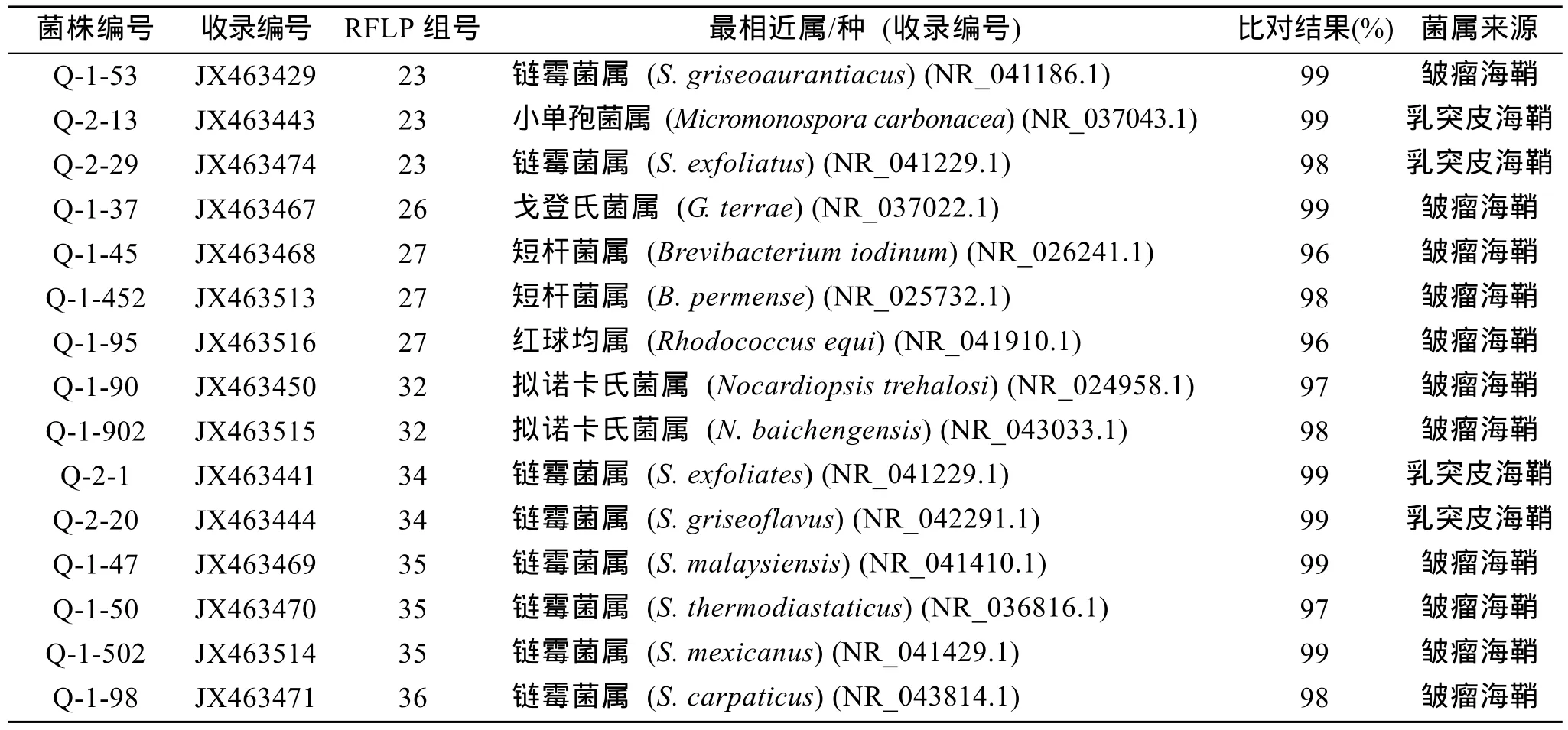
续表
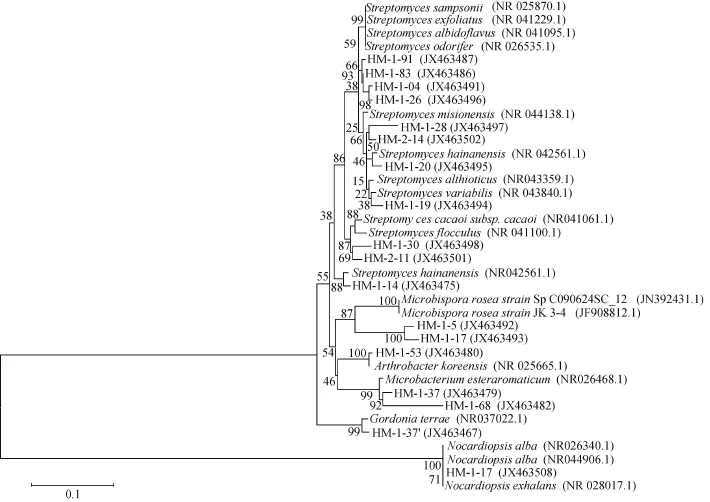
图3 山海绵放线菌16S rRNA基因序列N-J系统发育树分析Fig. 3 Neighbor-joining phylogenetic representation of actinobacteria associated with Mycale sp. and their closest NCBI(BLASTn)relatives based on 16S rRNA gene sequences
测序结果表明, 55株代表性放线菌(从典型RFLP带型和各种属独有带型组中挑选)中有 12株属于稀有放线菌, 其他属于链霉菌, 该菌属在 4个样品中都表现出丰富的种类。12株稀有放线菌分属以下7个属: 拟诺卡氏菌、戈登菌、微杆菌、短杆菌、红球菌、小单孢菌和节细菌。其中山海绵中得到拟诺卡氏菌、节细菌、戈登菌和微杆菌属, 网架海绵中得到短杆菌; 皱瘤海鞘中得到拟诺卡氏菌、戈登菌、微杆菌、短杆菌和红球菌, 乳突皮海鞘中得到小单孢菌。
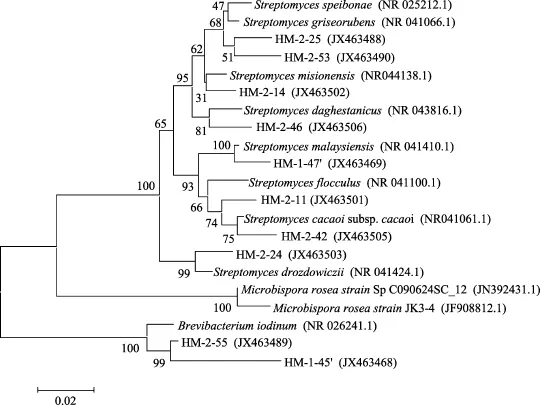
图4 网架海绵放线菌16S rRNA基因序列N-J系统发育树分析Fig. 4 Neighbor-joining phylogenetic representation of actinobacteria associated with Stylissa sp. and their closest NCBI(BLASTn)relatives based on 16S rRNA gene sequences
3 讨论
微生物与海鞘共生的现象能够使海鞘产生具有活性的次级代谢产物, 有很多是具有价值的药用化合物[7-9], 表现出抗肿瘤、抗病毒、抗微生物等生理活性, 以抗肿瘤活性最为引人注目[3]。但至今, 除证实海鞘有共生蓝藻细菌外[10], 其相关微生物的研究并不多。文献中应用培养方法获得了一种海鞘的微生物群落信息以及一些分离菌株[11-13]。但由于培养分离方法的局限性, 只能检测到存在于特定生存环境中的少数微生物[14-15]。利用 DGGE、克隆和 16S rRNA基因测序等分子手段分析微生物群落, 发现似螺原体细菌在乳突皮海鞘的生殖腺中大量存在[16]。但文献中对海鞘相关放线菌的报道极少。
本研究探讨了海鞘中相关放线菌的多样性, 是目前为止极少数海鞘相关微生物多样性报道中专门针对放线菌的研究。Menezes等[17]用7种添加营养丰富的人工海水并减少碳源的分离培养基从海鞘Didemnumsp.中分离出3个放线菌属: 短小杆菌、微球菌和戈登菌。从海鞘Didemnum Ligulum中分离出4个放线菌属: 短小杆菌、节细菌、短杆菌和诺卡氏菌。而本研究从皱瘤海鞘中分离出6个放线菌属, 从乳突皮海鞘中得到 2个放线菌属, 可初步验证海鞘具有高的放线菌多样性, 且存在显著的种属间差异,其中皱瘤海鞘相关放线菌的多样性是目前为止海鞘个体中可培养放线菌多样性最高的研究结果。可见分离培养结合 16S rRNA基因测序的方法能很好的揭示海鞘相关放线菌的多样性, 并且所选用的 6种分离培养基能够有效分离海鞘中相关放线菌, 优于其他研究中的分离培养基。

图5 皱瘤海鞘放线菌16S rRNA基因序列N-J系统发育树分析Fig. 5 Neighbor-joining phylogenetic representation of actinobacteria associated with Styela plicata and their closest NCBI(BLASTn)relatives based on 16S rRNA gene sequences
海绵来源的放线菌多样性研究已经广泛开展,目前已分离出纤维菌属(Cellulosimicrobium)、酸微菌属(Acidimicrobium)、考克氏菌属(Kocuria)、疣胞菌属(Verrucosispora)等22个属[18]。张海涛等[19]用16S rRNA基因扩增和RFLP及测序分析相结合的方法从大连海域繁茂膜海绵中分离出 7个放线菌属。信艳娟等从大连海域繁茂膜海绵中筛选放线菌 9个属[2],是目前多样性最高的研究结果。本研究从山海绵中分离出5个放线菌属, 网架海绵中分离出2个放线菌属, 虽然数量并不是最多, 但可表明海绵相关放线菌高的多样性水平。进而与海鞘相关放线菌多样性平行对比, 证明海鞘中具有丰富的放线菌资源, 在本研究中还表现出高于海绵来源的放线菌多样性, 是一种很有潜力的放线菌来源。同时, 海鞘中发现8种可能的新放线菌菌株, 数量上远高于在海绵(2种)中的发现。
比较海绵之间、海鞘之间相关放线菌多样性, 可发现无论数量上还是多样性水平上都存在显著性差异(表3和表4)。这可能与其生长环境和生理结构的差异有密切关联。质地松软, 滤水系统发达, 生活于潮间带的山海绵比质地坚硬、骨针结构多的网架海绵更易富集海水中的微生物, 同时表面粗糙的皱瘤海鞘则比表皮光滑透明的乳突皮海鞘容易积累微生物, 因而表现出更高的可培养放线菌多样性。但是,更重要的原因是研究手段的局限性, 分离培养手段无法获得稀有的难培养的种类。高浓度的营养物质可能比较适合生长速度快且对高浓度营养物质有适应能力的放线菌, 但对生长速度较慢的放线菌则可能有抑制作用[20-21]。特别是放线菌可以“活的非可培养”状态存在[22], 这更增加了培养难度。因此, 基于目前使用的分离培养方法并不能全面准确的反映海绵和海鞘中放线菌的多样性信息, 仍需应用新的分子多样性研究与培养相结合的方法进一步深入研究。

图6 乳突皮海鞘放线菌16S rRNA基因序列N-J系统发育树分析Fig. 6 Neighbor-joining phylogenetic representation of actinobacteria associated with M. manhattensis and their closest NCBI(BLASTn)relatives based on 16S rRNA gene sequences
Li等[23]和 Webster等[24]都利用添加海绵组织提取液的放线菌培养基, 成功分离到稀有放线菌以及先前没有分到的菌株。这也为今后的分离培养研究提供了可选择的思路。同时也需结合分子生物学的研究手段从基因角度分析, 与分离培养方法互为补充, 提供分子信息指导, 以期获得更全面更准确的海绵和海鞘相关放线菌多样性信息, 真正验证海鞘相关放线菌的多样性及其特有的新菌种资源。
4 结语
作者用 6种有效的分离培养基从两种海绵和两种海鞘中分别分离出87株和111株放线菌, 其中有10株为潜在的新菌株, 为海洋放线菌的开发利用提供了潜在的新资源。与海绵中分离得到的 6个放线菌属和2个可能的新菌株相比, 从海鞘中分离到了7个放线菌属和 8个可能的新株。表明海鞘具有较高的放线菌多样性水平, 是除海绵之外另一个极具潜力的海洋放线菌来源, 可成为开发海洋未知放线菌菌株以及海洋天然活性产物的新方向和储备资源。
[1]李越中, 陈琦. 海洋微生物资源多样性[J]. 生物工程进展, 1998, 18(4): 34-40.
[2]信艳娟, 吴佩春, 邓麦村, 等. 繁茂膜海绵中可培养稀有放线菌的多样性[J]. 微生物学报, 2009, 49(7):859-866.
[3]耿越, 张薛, 赵相轩. 海鞘类天然物的最新研究进展[J]. 天然产物研究与开发, 2001, 13(6):73-78.
[4]Takahashi Y, Omura S. Isolation of new actinomycete strains for the screening of new bioactive compounds[J].Gen Appl Microbiol,2003, 49: 141-154.
[5]Zhang H T , Lee Y K, Zhang W, et al. Culturable actinobacteria from the marine spongeHymeniacidon perleve: isolation and phylogenetic diversity by 16S rRNA gene-RFLP analysis[J]. Anton Leeuw,2006, 90:159-169.
[6]Lee Y K, Kim H W, Liu C L, et al. A simple method for DNA extraction from marine bacteria that produce extracellular materials[J]. J Microbiol Meth, 2003, 52:245-250.
[7]Pisut D P, Pawlik J R. Anti-predatory chemical defenses of ascidians: secondary metabolites or inorganic acids?[J]. Exp Mar Biol Ecol, 2002, 270:203-214.
[8]Moss C, Green D H, Pérez B, et al. Intracellular bacteria associated with the ascidianEsteinascidia turbinata: phylogenetic and in situ hybridization analysis[J]. Mar Biol, 2003, 143: 99-110.
[9]Ramasamy M S, Murugan A. Chemical defense in ascidiansEudistoma virideandDidemnum psammathodesin Tuticorn, southeast coast of India: bacterial epibiosis and fouling deterrent activity[J]. Indian J Mar Sci, 2003,32: 337-339.
[10]Hirose E, Maruyama T, Cheng L, et al. Intra- and extracellular distribution of photosynthetic prokaryotes,Prochloronsp. in a colonial ascidian: ultrastructural and quantitative studies[J]. Symbiosis,1998, 25,301-310.
[11]Olgin Uribe G, Abou Mansour E, Boulander A, et al.6-bromoindole-3-carbaldehyde form andAcinetobactersp.bacterium associated with the ascidianStomozoa murrayi[J]. Chem Ecol, 1996, 23, 2507-2521.
[12]Wahl M. Bacterial epibiosis on Bahamian and Pacific ascidians[J]. Exp Mar Biol Ecol,1995, 191, 239-255.
[13]Romanenko L A, Shumann P, Rhode M, et al.Halomonsa halocynthiaesp. nov. isolated form the marine ascidianHalocynthia auranticum.Int[J]. Syst Evol Microbiol,2002,52: 1767-1772.
[14]Amann R I, Ludwig W, Schleifer K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiol Rev,1995, 59: 143-169.
[15]Head I M, Saunders J R, Pickup R W. Microbial evolution, diversity, and ecology: a decade of ribosomal RNA analysis of uncultivated microorganisms[J].Microb Ecol, 1998, 35: 1-21.
[16]Elia T, Mary C, Stefan M, et al. Phylogenetic diversity of bacteria associated with ascidians in Eel Pond(Woods Hole, Massachusetts, USA)[J]. J Exp Mar Biol Ecol, 2007, 342: 138-146.
[17]Menezes CBA, Bonugli-Santos RC. Microbial diversity associated with algae, ascidians and sponges from the north coast of Sao Paulo state, Brazil[J]. Microbiol Res,2010, 165: 466-482.
[18]Taylor M W, Mohamed N M, Enticknap J J, et al.Sponge-associated microorganisms: evolution, ecology and biotechnological potential[J]. Microbiol Mol Biol Rev, 2007, 71: 295-347.
[19]张海涛, 靳艳, 虞星炬, 等. 16S rDNA-RFLP分析繁茂膜海绵可培养放线菌的多样性[J]. 微生物学报,2005, 45(6): 828-831.
[20]岳秀娟, 余利岩. 自然界中难分离培养微生物的分离和应用[J]. 微生物学通报, 2006, 33(3): 77-8l.
[21]张晓华. 海洋微生物学[C]. 青岛: 中国海洋大学出版社, 2007, 332-333.
[22]薛超波, 王国良, 金珊, 等. 海洋微生物多样性研究进展[J]. 海洋科学进展, 2004, 22(3): 377-384.
[23]Li Z Y, Liu Y. Marine sponge Craniella austrialiensis associated bacterial diversity revelation based on 16S rDNA library and biologically active actinomycetes screening,phylogenetic analysis[J]. Lett Appl Microbiol, 2006, 43:410-416.
[24]Webster N S, Wilson K J, Blackall L L, et al.Phylogenetic diversity of bacteria associated with the marine spongeRhopaloeides odorabile[J]. Appl Environ Microbiol, 2001, 67: 434-444.
