新型羟基化酶基因的克隆及其洛伐他汀转化的应用
2013-06-30霍孝雨诸葛斌方慧英宗红宋健诸葛健
霍孝雨,诸葛斌,方慧英,宗红,宋健,诸葛健
1 江南大学生物工程学院工业生物技术教育部重点实验室,江苏无锡 214122
2 江南大学化学与材料工程学院,江苏无锡 214122
他汀类药物作为羟甲基戊二酰辅酶A 还原酶(HMGR)竞争性抑制剂,可以有效抑制胆固醇合成,降低血清胆固醇,是临床上常用降血脂药物[1-2]。研究人员一直致力于寻找更加高效的他汀类物质,其中利用微生物转化是一个重要的途径,如嗜碳链霉菌Streptomyces carbophilus、马杜拉放线菌Actinomadura sp.可将美伐他汀转化为普伐他汀[3-4]。无锡他汀作为具有我国独立自主知识产权的新型他汀,由 Amycolatopsis sp.CGMCC1149转化洛伐他汀而来,其对HMGR 的抑制效果是洛伐他汀的4倍[5]。国内对他汀的生物转化研究很少,因此研究开发无锡他汀有助于打破国外在他汀方面的垄断。
细胞色素P450(Cytochrome P450,P450)是一类自然界广泛存在的羟基化酶,属于含硫醇-血红素的单加氧酶,因其还原型与一氧化碳(CO)复合物在450 nm 处有特征吸收峰而得名[6-7]。本研究室前期研究表明, Amycolatopsis sp.CGMCC1149细胞蛋白中包含具有P450特性的蛋白,并且洛伐他汀转化与此酶酶活变化存在一定关联,该转化过程也可被CO 所抑制,初步推测洛伐他汀羟基化由P450催化完成[8-9]。为获得该羟基化酶,本文从Amycolatopsis sp.CGMCC1149中克隆得到其基因并在大肠杆菌E.coli 中表达,同时建立了一个体外酶催化功能验证系统,首次实现无锡他汀转化底物洛伐他汀的体外羟基化,为无锡他汀大规模制备奠定了基础。
1 材料与方法
1.1 材料
Amycolatopsis sp.CGMCC1149,E.coli JM109,E.coli DH5α及表达载体pEtac 由本研究室保藏;克隆载体pMD18-T,PCR 相关试剂,限制性内切酶EcoRⅠ、Hind Ⅲ购自大连TaKaRa公司;洛伐他汀购自浙江海正药业;铁氧还蛋白(ferredoxin)和铁氧还蛋白还原酶(ferredoxin reductase)购自Sigma-Aldrich 公司;质粒提取试剂盒,胶回收试剂盒购自北京博大泰克;引物由上海生工生物工程服务有限公司合成;基因测序由北京六和华大基因完成。
1.2 方法
1.2.1 羟基化酶基因的克隆
对NCBI 公布的部分放线菌细胞色素P450基因(GenBank Accession No.AB208694,AB426710,AY549201)进行同源性比对,发现存在保守区,根据保守区序列设计简并引物用于扩增羟基化酶的保守区片段。采用Self-Formed Adaptor PCR (SEFA PCR)[10]技术克隆羟基化酶的侧翼序列。引物设计见表1,其中引物DP-F和DP-R 用于扩增保守区序列,引物3SP1、3SP2、3SP3用于扩增3´侧翼序列,5SP1、5SP2、5SP3用于扩增5´侧翼序列。SEFA PCR 反应条件参照参考文献[10]。
1.2.2 产羟基化酶重组菌 E.coli DH5α(pEtac-p450lov)的构建
根据获得的羟基化酶基因序列,设计一对引物P-F 和P-R,分别在两引物5´端引入EcoRⅠ和Hind Ⅲ酶切位点,“_”表示加入的酶切位点(表1)。PCR 程序为:94℃预变性5 min;94℃变性1 min,65℃退火1 min,72℃延伸2 min,30个循环;72℃再延伸10 min。PCR 产物回收后,与质粒pEtac 同时用EcoRⅠ和Hind Ⅲ双酶切,回收目的片段经T4 DNA 连接酶连接得到 pEtac-p450lov。将该重组质粒导入 E.coli DH5α,挑取阳性转化子,提取质粒验证,即得重组菌E.coli DH5α(pEtac-p450lov)。
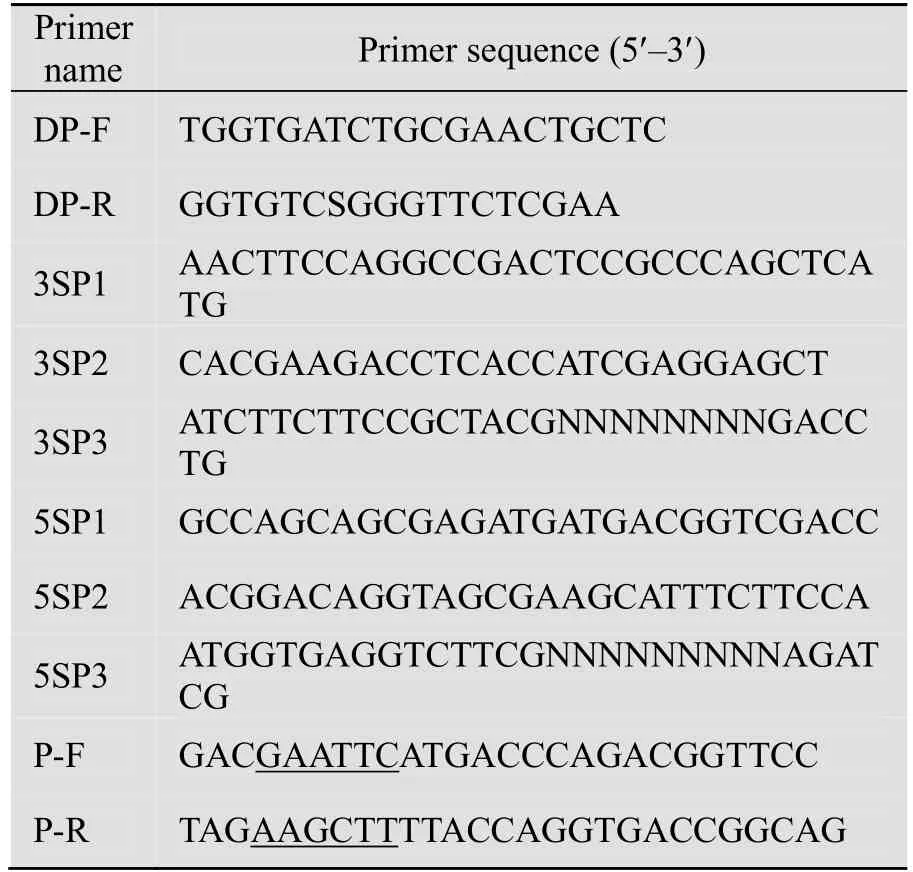
表1 本文所用的引物Table 1 Primers used in this study
1.2.3 重组羟基化酶的诱导表达

1.2.4 体外酶催化系统
体外酶催化系统:0.3 mmol/L 洛伐他汀,0.5 mmol/L NADH,320µg 铁氧还蛋白,0.04 U铁氧还蛋白还原酶,羟基化酶样品,总体积200µL。30℃、150 r/min 水浴反应30 min,105℃、5 min终止反应,HPLC 检测反应产物。检测条件为:Kromasil C18柱(250 mm×4.6 mm,5µm),流动相甲醇-0.5%乙酸(80∶20),流速1 mL/min,进样量20µL,检测波长237 nm,柱温25℃。
2 结果
2.1 羟基化酶基因的克隆
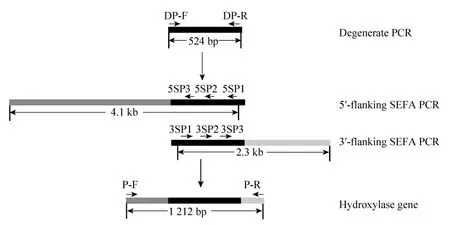
图1 羟基化酶基因克隆路线示意图Fig.1 Schematic diagram of hydroxylase gene cloning.
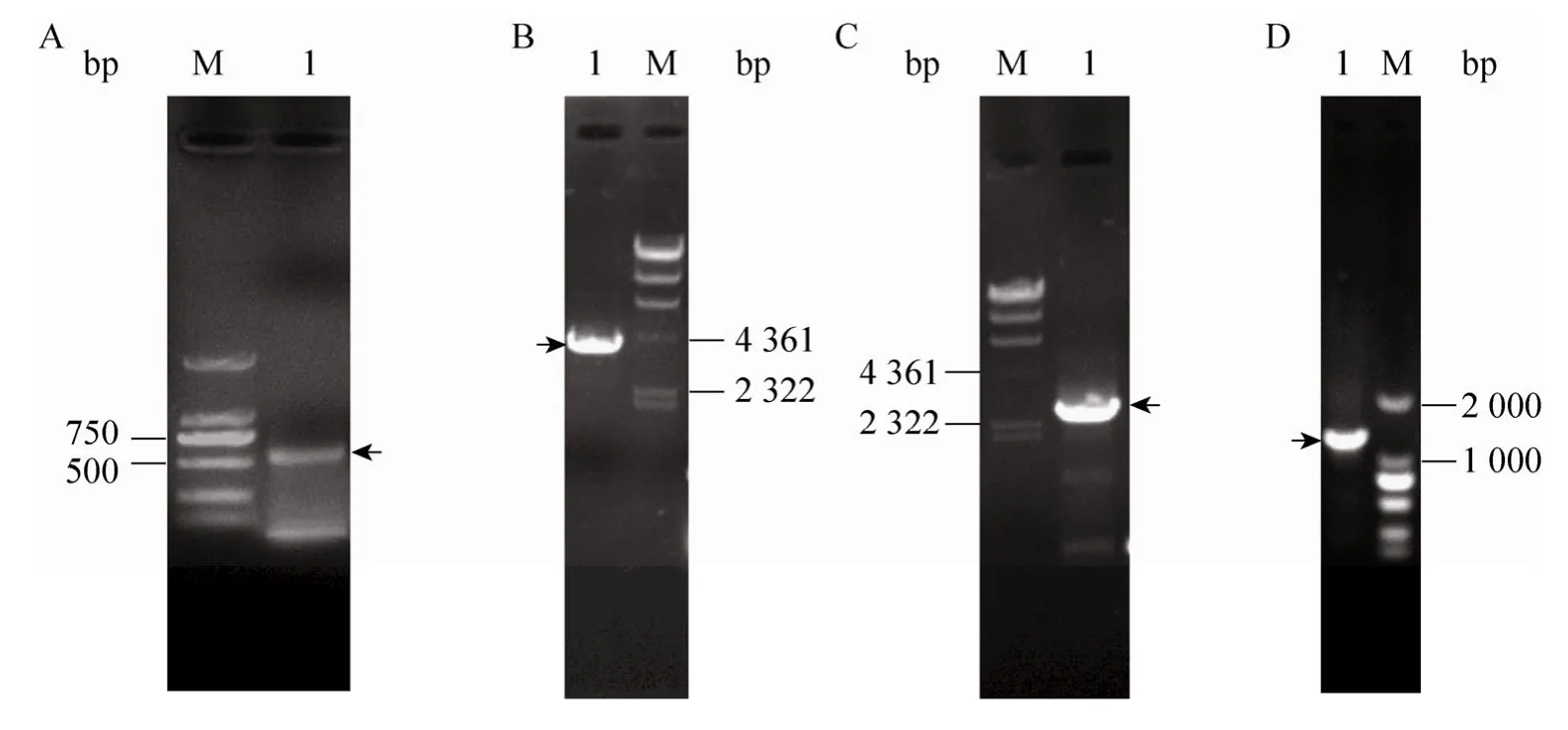
图2 PCR 产物电泳图Fig.2 Results of PCR.(A) Degenerate PCR.(B)5´-flanking SEFA PCR.(C)3´-flanking SEFA PCR.(D)Amplification of the whole hydroxylase gene.M:λ-Hind III digest DNA marker;1:PCR product.
运用简并PCR 和SEFA PCR 技术克隆羟基化酶基因,克隆过程如图1所示。根据P450的保守区设计一对简并引物并进行PCR,扩增得到约0.5 kb 的片段并测序(图2A)。测序得到524 bp的基因片段,将其提交NCBI 进行比对,比对结果表明该片段与 P450基因的相似性高。根据此片段,设计两组SEFA PCR 引物,分别用于扩增其5´和3´侧翼序列,经过两轮SEFA PCR,分别得到5´侧翼约4.1 kb(图2B)和3´侧翼约2.3 kb基因片段(图2C)。将上述得到的片段进行测序并拼接,根据拼接结果设计一对引物P-F 和P-R,PCR 扩增得到约1.2 kb 的片段(图2D),测序结果表明该片段为一个完整开放阅读框,包含1212 bp,编码一个含403个氨基酸的蛋白,蛋白理论等电点为4.7,理论分子量为44.8 kDa。
将该基因序列在NCBI 中进行Blast 比对,发现与P450基因相似性高,其中与远青色拟无枝酸菌 Amycolatopsis azurea 的 P450Um-1(GenBank Accession No.AB208694)[12]的最高(91%)。将该基因编码的氨基酸序列提交细胞色素 P450工程数据库(http://www.cyped.unistuttgart.de/)中CYP 模块(CYP modules)进行二级结构预测[13],结果如图3所示。该蛋白共含有11个α-螺旋,4个β-折叠,一个曲折环(Meander loop)和一个Cys 袋区(Pocket),其中β1含5股(Strand),β2含2股,β3含2股,β4含2股。另外,此蛋白包含几个P450典型功能区,其中[243SHET]为氧结合区(Oxygen binding site),位于螺旋I,与分子氧的结合有关[14];[281EMLR]为离子对结合区(Ion-pair region),位于螺旋K,主要与电子传递链相互作用及血红素结合有关[15];[345FGHGIHQCLG]为血红素结合区(Heme binding region),位于Cys 袋区中,其中Cys 完全保守,与血红素第5配位键结合[16]。因此,可以确定所克隆的基因属于P450基因超家族,并将其命名为 p450lov,编码的蛋白命名为P450Lov,所得的核酸序列及氨基酸序列提交GenBank,基因登录号为KC352683。
2.2 产羟基化酶重组菌 E.coli DH5α(pEtac-p450lov)的构建及诱导表达
根据已得羟基化酶基因序列,设计一对引物进行PCR 得到p450lov 基因片段,并将其插入到pEtac,得到重组质粒pEtac-p450lov(图4A)。对构建好的质粒pEtac-p450lov 进行酶切及PCR 验证,结果表明p450lov 成功连接到pEtac 上(图4B)。将重组质粒pEtac-p450lov 导入E.coli DH5α,得到重组菌 E.coli DH5α(pEtacp450lov),对其进行诱导表达,以E.coli DH5α(pEtac)为对照。不同培养温度(20℃、25℃、30℃、37℃)下重组P450Lov 表达结果如图5所示,20℃时P450Lov 主要集中在破壁上清液中,以可溶性形式存在,随着温度升高上清液中逐渐减少而破壁沉淀中逐渐增多,当温度达到37℃时,P450Lov 主要集中在破壁沉淀,以包涵体形式存在,上清液明显减少。破壁上清液的CO 示差光谱表明(图6),在450 nm 处有明显特征吸收峰,说明重组P450Lov 正确折叠表达,以活性形式存在,其表达量为2.17 nmol/g。

图3 羟基化酶的二级结构Fig.3 Secondary structure of hydroxylase.
图4 重组质粒pEtac-p450lov 的构建及验证Fig.4 Construction and analysis of recombinant plasmid pEtac-p450lov.(A) Recombinant plasmid pEtac-p450lov.(B) Analysis of pEtac-p450lov by restriction enzyme digestion and PCR.M1:λ-Hind III digest DNA marker;M2:DL2000 DNA marker;1:plasmid pEtac-p450lov was digested with EcoR I;2:plasmid pEtac-p450lov was digested with EcoR I and Hind III;3:p450lov gene was PCR amplified using pEtac-p450lov as the template.

图5 羟基化酶的SDS-PAGE 检测Fig.5 SDS-PAGE analysis of hydroxylase.1:lysate supernatant of E.coli DH5α(pEtac);2–5:lysate supernatant of E.coli DH5α(pEtac-p450lov) cultured at 20℃,25℃,30℃,37℃;6–9:lysate precipitant of E.coli DH5α(pEtac-p450lov) cultured at 20℃,25℃,30℃,37℃;M:protein marker.
2.3 羟基化酶的体外催化
利用1.2.4中所述的系统,对羟基化酶进行催化功能验证,结果表明重组P450Lov 可以借助该系统将洛伐他汀羟基化为3-羟甲基洛伐他汀(图7B),而对照组E.coli DH5α(pEtac)细胞破壁上清液则无3-羟甲基洛伐他汀形成(图7A)。由上述结果证实,在Amycolatopsis sp.CGMCC1149中催化洛伐他汀羟基化的反应由P450Lov 催化完成。同时,利用该体系可以实现洛伐他汀的体外羟基化,这为进一步实现无锡他汀的体外转化打下了基础。

图6 羟基化酶的CO 示差光谱Fig.6 CO difference spectrum of hydroxylase.
3 讨论
Amycolatopsis sp.CGMCC1149转化洛伐他汀过程中,高浓度洛伐他汀对菌体有一定毒害作用,从而限制无锡他汀产量的提高,其中利用相关酶进行无锡他汀体外转化是解决此不利影响的手段之一。
P450在自然界分布广泛,催化的典型反应是通过电子传递系统,将分子氧还原,并伴随底物的羟基化[17]。虽然在E.coli 中没有发现P450存在[18],但有些P450可以借助E.coli 的内生电子传递系统来行使完整的催化功能,如灰色链霉菌Streptomyces griseus 的CYP105D1和一些来自人体的P450[19-20]。为了使P450Lov具备催化洛伐他汀的能力,本文首先尝试利用E.coli 的内生电子传递系统来重组P450Lov 的催化活性,结果表明P450Lov 并不能借助该系统来催化洛伐他汀的羟基化。对于细菌来源的P450,一般可用来源菠菜的铁氧化蛋白和铁氧还蛋白还原酶代替自身电子传递系统来行使催化功能[21]。据此本文再次尝试利用铁氧化蛋白和铁氧还蛋白还原酶来建立洛伐他汀的体外羟基化系统。检测结果表明,利用该系统成功实现洛伐他汀的体外羟基化,这也直接证实了Amycolatopsis sp.CGMCC1149转化洛伐他汀过程中的羟基化酶即为细胞色素P450。
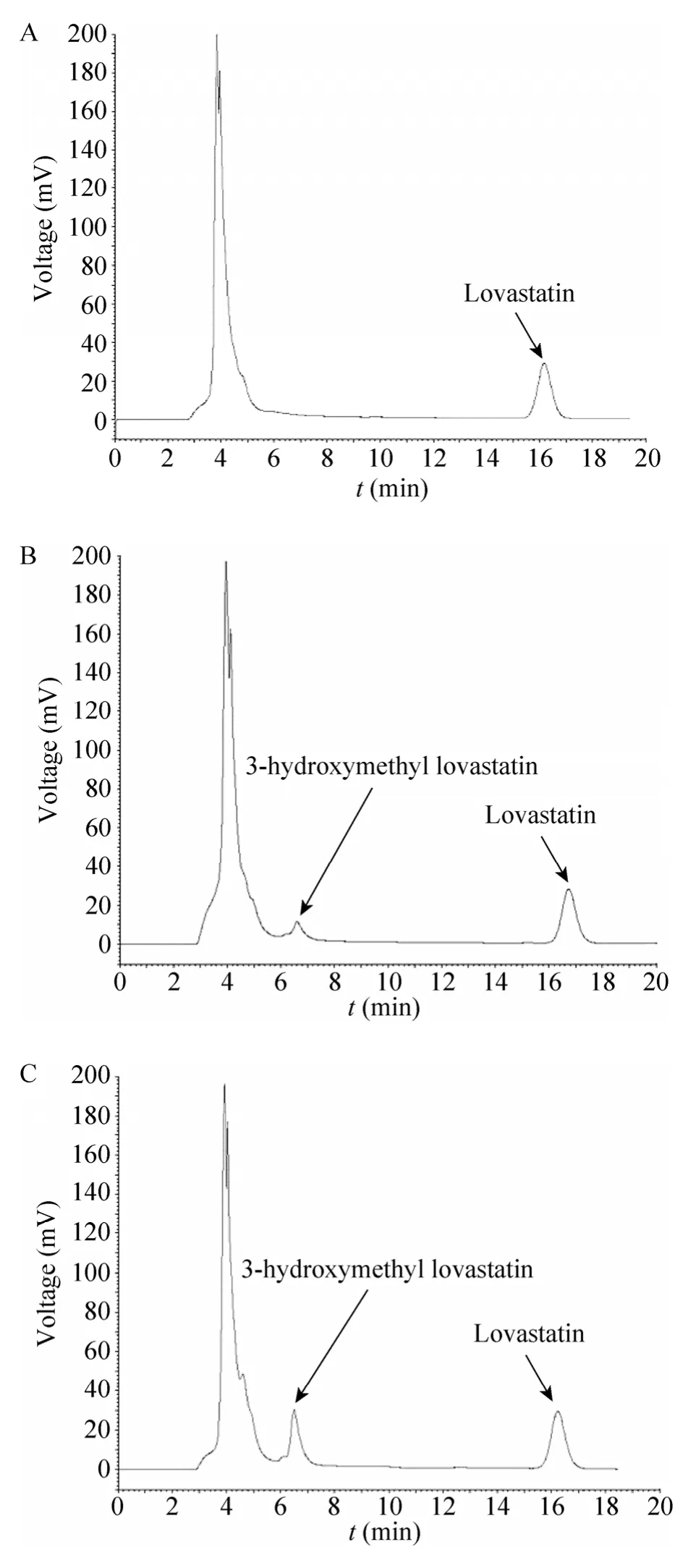
图7 洛伐他汀羟基化产物的HPLC 检测Fig.7 HPLC analysis of lovastatin hydroxylated product.(A) Control group.(B) Experimental group.(C) Adding of 3-hydroxymethyl lovastatin standard.
为进一步提高该系统的催化效率,需要对Amycolatopsis sp.CGMCC1149自身电子传递系统展开更多的研究。此外,本文所建立的体外酶催化系统只是实现了洛伐他汀的体外羟基化,为进一步实现无锡他汀体外转化,需要对转化过程中另一个关键酶——异构酶进行研究。
[1]Tsujita Y,Watanabe Y.Pravastatin sodium:a novel cholesterol-lowering agent which inhibits HMG-CoA reductase.Cardiovasc Drug Rev,1988,7(2):110–126.
[2]Endo A.The discovery and development of HMG-CoA reductase inhibitors.J Lipid Res,1992,33(11):1569–1582.
[3]Matsuoka T,Miyakoshi S,Tanzawa K,et al.Purification and characterization of cytochrome P-450sca from Streptomyces carbophilus.Eur J Biochem,1989,184(3):707–713.
[4]Peng Y,Demain AL.Bioconversion of compactin to pravastatin by Actinomadura sp.ATCC 55678.J Mol Catal B-Enzym,2000,10(1):151–156.
[5]Zhuge B,Fang HY,Yu H,et al.Bioconversion of lovastatin to a novel statin by Amycolatopsis sp..Appl Microbiol Biotechnol,2008,79(2):209–216.
[6]Hannemann F,Bichet A,Ewen MK,et al.Cytochrome P450 systems-biological variations of electron transport chains.BBA-Gen Subjects,2007,1770(3):330–344.
[7]Omura T,Sato R.A new cytochrome in liver microsomes.J Biol Chem,1962,237(4):1375–1376.
[8]Zong H,Zhuge B,Fang HY,et al.Advances in the bioconversion mechanism of lovastatin to wuxistatin by Amycolatopsis sp.CGMCC 1149.Appl Microbiol Biotechnol,2013,97(2):599–609.
[9]Wang X,Zhuge B,Fang HY,et al.Effects of CYP450 on bioconversion of lovastatin by Amycolatopsis sp.ST2710.Ind Microbiol,2010,40(6):28–32(in Chinese).王轩,诸葛斌,方慧英,等.细胞色素P450酶对拟无枝酸菌转化洛伐他汀的影响.工业微生物,2010,40(6):28–32.
[10]Wang S,He J,Cui Z,et al.Self-Formed Adaptor PCR:a Simple and Efficient Method for Chromosome Walking.Appl Environ Microbiol,2007,73(15):5048–5051.
[11]Omura T,Sato R.The carbon monoxide-binding pigment of liver microsomes,evidence for its hemoprotein.J Biol Chem,1964,239(7):2370–2378.
[12]Ueno M,Sasamura S,Yamashita M,et al.Cloning and heterologous expression of P450Um-1,a novel bacterial P450 gene,for hydroxylation of immunosuppressive agent AS1387392.J Antibiot,2010,63(11):649–656.
[13]Sirim D,Widmann M,Wagner F,et al.Prediction and analysis of the modular structure of cytochrome P450 monooxygenases.BMC Struct Biol,2010,10(1):34.
[14]Cupp-Vickery JR,Li H,Poulos TL.Preliminary crystallographic analysis of an enzyme involved in erythromycin biosynthesis:cytochrome P450eryF.Proteins,1994,20(2):197–201.
[15]Graham-Lorence S,Peterson JA.P450s:structural similarities and functional differences.FASEB J,1996,10(2):206–214.
[16]Falquet L,Pagni M,Bucher P,et al.The PROSITE database,its status in 2002.Nucleic Acids Res,2002,30(1):235–238.
[17]Montellano de PRO.Hydrocarbon hydroxylation by cytochrome P450 enzymes.Chem Rev,2010,110(2):932.
[18]Jenkins CM,Waterman MR.Flavodoxin and NADPH-flavodoxin reductase from Escherichia coli support bovine cytochrome P450c17 hydroxylase activities.J Biol Chem,1994,269(44):27401–27408.
[19]Taylor M,Lamb DD,Cannell R,et al.Cytochrome P450105D1(CYP105D1) from Streptommyces griseus:heterologous expression,activity,and activation effects of multiple xenobiotics.Biochem Biophys Res Commun,1999,263(3):838–842.
[20]Iwata H,Fujita K,Kushida H,et al.High catalytic activity of human cytochrome P450 co-expressed with human NADPH-cytochrome P450 reductase inEscherichia coli.Biochem Pharmacol,1998,55(8):1315–1325.
[21]Prior JE,Shokati T,Christians U,et al.Identification and characterization of a bacterial cytochrome P450 for the metabolism of diclofenac.Appl Microbiol Biotechnol,2010,85(3):625–633.
