乙烯信号转导通路研究
2012-08-21张存立郭红卫
张存立 郭红卫
①博士研究生,②教授,北京大学生命科学学院蛋白质与植物基因研究国家重点实验室,北京100871
乙烯信号转导通路研究
张存立①郭红卫②
①博士研究生,②教授,北京大学生命科学学院蛋白质与植物基因研究国家重点实验室,北京100871
植物激素 乙烯 信号转导
作为5大类植物激素之一的乙烯一直是科学家关注和研究的焦点。虽然结构简单,但是气态激素乙烯在植物的生长发育以及胁迫反应中具有重要的作用。通过近20年的研究,科学家已经描绘出一条近似线性的乙烯信号转导通路。在模式植物拟南芥中,这条通路的最上游是由一个多基因家族编码的乙烯的5个受体ETR1,ETR2,ERS1,ERS2和EIN4。与之相结合并共同定位于内质网上的是一个类似Raf的蛋白激酶CTR1。在没有乙烯存在的条件下,受体和CTR1的结合能够协同抑制下游乙烯信号。在这两类负调控因子的下游是乙烯信号的正调控因子EIN2。如果EIN2基因突变,即使有高浓度乙烯存在,植物黄化苗也将表现出完全的乙烯不敏感表型,显示出EIN2在乙烯信号通路中的核心地位。在EIN2的下游是乙烯信号的转录因子家族EIN3以及EILs,它们在响应乙烯信号之后会起始乙烯相关基因的表达。研究还发现,乙烯的转录因子受泛素化降解途径调控,负责识别及结合EIN3等转录因子的F-box蛋白是EBF1和EBF2。EIN5是一种5’→3’外切核酸酶,它能够通过促进EBF1和EBF2的mRNA的降解来拮抗这两个F-box蛋白对EIN3的负反馈调控。最近,有研究表明EIN2同样是一个半衰期很短并经由泛素化降解途径调控的蛋白,而执行调控EIN2任务的是另外两个F-box蛋白ETP1和ETP2。虽然人们对于乙烯信号转导通路的认识取得了巨大进步,但是该信号通路的精细调节机制以及乙烯信号与其他植物激素信号之间的交叉反应还需进行更为深入的研究。
虽然乙烯是一种结构非常简单的气体植物激素,但是它对植物发育以及适应性反应起着非常重要的作用。种子萌发、开花、叶片衰老、果实成熟、叶片剪切、根瘤、细胞程序性死亡以及对非生物胁迫和病原体入侵的反应等生理过程都与乙烯密切相关[1-3]。鉴定乙烯反应的特征性实验是观察生长在暗处并经乙烯处理之后植物黄化苗的“三重反应”。所谓“三重反应”是指黄化苗受乙烯处理后下胚轴和根的伸长被抑制、下胚轴变粗以及顶端弯钩加剧(图1)。科学家通过基于“三重反应”进行遗传筛选的方法,在过去的近20年中找到一系列拟南芥的乙烯突变体(图2)。这些突变体可以被分为3种类型:①乙烯过表达突变体,如eto1,eto2,eto3;②组成型乙烯反应突变体,如ctr1;③乙烯不敏感突变体,如etr1,etr2,ein2,ein3,ein4,ein5以及ein6[1-2,4]。通过对这些拟南芥突变体的深入研究,科学家逐渐描绘出一条近似线性的乙烯信号转导通路(图3)。首先,在上游对乙烯分子的感知是通过一个与内质网膜相结合的受体家族来完成的,其中包括ETR1,ETR2,ERS1,ERS2,EIN4。这些乙烯受体在序列上具有相似性,并且在结构上都与细菌双组分组蛋白激酶相类似[3,5-6]。与乙烯受体相结合并协同抑制下游乙烯信号的是一个属于Raf蛋白激酶家族的负调节因子CTR1蛋白[7-8]。遗传上位于CTR1下游的正调控因子EIN2是一个定位于内质网膜上的大的跨膜蛋白。EIN2的N端与哺乳动物金属离子通道蛋白NRAMP家族有一定的相似性;而其C末端是一个亲水的功能未知的组分。研究发现,EIN2的C末端与已知蛋白没有任何同源性,但是如果在拟南芥中单独转基因表达该区段却足以组成型地激活乙烯和胁迫反应[9]。表型、遗传以及生化的分析结果都显示EIN2蛋白位于乙烯信号通路的一个中心位置。EIN2作为一个正调控因子,其抑制被解除后即可通过正调乙烯信号途径的主要转录因子EIN3和EIL1而将信号通过转录级联方式传递下去,使得下游乙烯应答基因的转录被活化而产生乙烯反应[10-11]。近年来,一些新的科研成果进一步丰富和扩展了乙烯的线性信号转导通路。研究表明,铜离子作为一个辅助因子促进乙烯与受体的结合,而铜离子的转运和浓度梯度的维持对于乙烯与受体的结合是一个必要的过程。一种具有铜离子转运功能的P-type ATPase RAN1在乙烯受体的生物发生以及铜离子的稳态平衡过程中起到重要的调节作用[12]。RTE1是另一类对受体功能起调节作用的乙烯信号的负调控蛋白,它与乙烯受体共定位于内质网膜上并在遗传上位于ETR1的上游,主要通过调节ETR1的活性来调节乙烯反应[13-14]。研究表明,EIN3蛋白可被两个 F-box蛋白EBF1和EBF2识别结合并进入泛素化降解过程[15-17]。有趣的是,研究表明EIN2同样是一个半衰期很短的会被泛素化降解的蛋白,识别和降解EIN2蛋白的F-box蛋白是ETP1和ETP2[18]。5’→3’外切核酸酶EIN5/XRN4蛋白能够通过降解EBF1和EBF2的mRNA来拮抗EBF1和EBF2对EIN3的负反馈调控[19-20]。近期,关于EIN3蛋白磷酸化修饰调节的研究揭示出更为复杂的激素调控网络的存在[21-22]。

图1 拟南芥黄化苗生长在乙烯中(3天)的“三重反应”表型
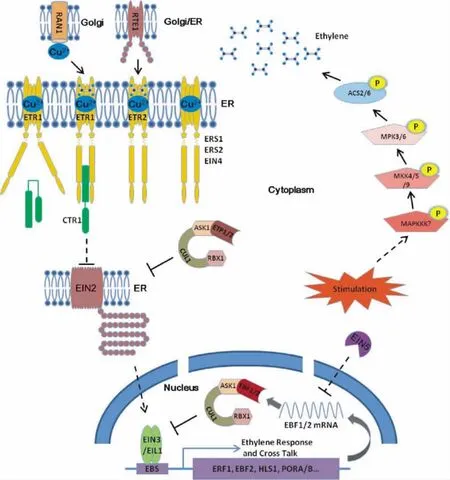
图3 乙烯信号转导通路示意图
1 乙烯受体及其对乙烯信号的感知
植物细胞通过定位于内质网上的受体感知乙烯信号。在拟南芥中共有由一个多基因家族编码的5个乙烯受体,分别是ETR1,ETR2,ERS1,ERS2和EIN4。它们在结构上类似于细菌和真菌中存在的双组分组蛋白激酶[5-6,23]。通过比较其结构特征,发现乙烯受体存在至少3类保守的结构域:N端是一个在铜离子协助下与乙烯结合的跨膜结构域;中间是一个负责不同受体间相互作用的GAF结构域;C端是一个能与下游组分CTR1相互作用的激酶结构域。乙烯5个受体可根据其结构相似性被分为两大类,一类受体包括ETR1和ERS1,另一类受体包括 ETR2,ERS2和 EIN4[6,24]。研究表明,乙烯受体的N端涉及受体的膜定位、乙烯结合以及受体的二聚化等基本功能,因此5个乙烯受体在N端具有较高的相似性[25-26]。乙烯受体的C端则是类似于组蛋白激酶的结构域。但是,体外激酶活性实验表明仅有ETR1,ERS1具有组蛋白激酶活性;其他受体ETR2,ERS2和EIN4则由于缺少组蛋白激酶活性所必需的氨基酸残基而可能作为丝氨酸/苏氨酸蛋白激酶来起作用[27-28]。
由于乙烯结合能力发生突变而产生的受体功能获得型突变体将会导致组成型乙烯不敏感表型的出现,表明乙烯受体作为乙烯信号的负调控因子而存在[6,23,29-30]。同时,乙烯的5 个 受 体 存 在 功 能 上 的 冗 余 ,因为单个受体的功能缺失型突变体表型与野生型类似,但多重功能缺失突变体则具有组成型的乙烯反应表型[31]。进一步研究发现,转基因表达类型Ⅰ受体ETR1或ERS1能够回复功能缺失型双突变体etr1/ers1的组成型乙烯反应表型;转基因表达类型Ⅱ受体ETR2,ERS2或EIN4却不能回复etr1/ers1双突变体的组成型乙烯反应表型[32]。科学家猜测是否因为类型Ⅰ受体独特的组氨酸激酶活性导致了其对功能缺失型双突变体表型的回复作用。但是,转基因表达组氨酸激酶失活的ETR1仍然可以回复功能缺失型双突变体etr1/ers1的表型,说明组氨酸激酶活性并不是类型Ⅰ受体抑制乙烯信号所必需的,而可能只是参与了乙烯信号的某些调节过程[26,33]。还有研究表明,即便受体缺失整个羧基端包括组氨酸激酶区和信号接受区,功能获得型的受体ETR1仍然具有抑制下游乙烯信号的功能,说明受体ETR1的C端对于其抑制功能并非必需[27]。科学家因此推测乙烯受体之间可以通过相互协同作用而维持受体的抑制功能[25,27,34]。乙烯与受体的结合需要铜离子的参与,而在此过程中负责铜离子转运和浓度梯度维持的蛋白是一个具有P-type ATPase活性的 RAN1蛋白[12]。RTE1是另一类进化上非常保守的膜结合蛋白,其转录活性受乙烯调控,并且RTE1通过与乙烯受体相互作用而负调控乙烯反应,暗示乙烯信号的感知可能存在更为精细的调节过程[13,35]。
2 CTR1激酶及其可能介导的MAPK途径
与乙烯受体结合并协同抑制下游乙烯反应的是另一个负调节因子CTR1蛋白。ctr1突变体会表现出组成型的乙烯反应表型[7]。序列分析显示CTR1蛋白是类似于哺乳动物Raf家族的丝氨酸/苏氨酸蛋白激酶。CTR1通过其氨基端与内质网上的乙烯受体的羧基端相结合而被间接锚定在内质网膜上,从而形成受体-CTR1复合体,而且CTR1对下游乙烯信号的负调控功能是依赖于这一蛋白相互 作用的[8,36-37]。研究表明,CTR1的羧基端具有丝氨酸/苏氨酸蛋白激酶活性,而激酶失活的ctr1突变体将会表现出组成型乙烯反应表型[37]。
CTR1的功能依赖于其与乙烯受体的结合。研究发现,由于氨基端发生错义突变而破坏了与受体结合能力的CTR1即便其羧基端激酶活性不受影响,仍然不能再对下游乙烯信号产生抑制作用[37]。基于CTR1在序列上与Raf蛋白激酶的相似性,人们很容易联想到CTR1在乙烯信号通路中是否作为一种MAPKKK通过介导一条MAPK级联反应而激活下游乙烯信号。虽然生化证据显示乙烯反应能够影响植物细胞内的磷酸化水平,但是之前的研究没有直接证据证明乙烯信号通路存在一条由CTR1介导的激活下游乙烯信号通路的MAPK途径[38-40]。有趣的是,多种证据却表明细胞内存在一条MKK4/5/9-MPK3/6途径通过修饰乙烯生物合成限速酶ACS2/6的稳定性而在乙烯生物合成水平而非乙烯信号转导水平对乙烯反应进行调节[41-44]。近期,有研究小组认为在拟南芥原生质体中CTR1可以绕过下游正调控因子EIN2蛋白而直接通过 MKK9-MPK3/6控制乙烯信号的转录因子EIN3蛋白的磷酸化而使其失活,从而抑制下游乙烯信号[21]。但是,另一组科学家在用对应的MKK9基因突变体以及转基因过表达植株进行实验时却表明,在拟南芥体内MKK9并非绕过EIN2蛋白而直接激活乙烯信号,而是通过作用于乙烯生物合成途径而影响乙烯反应[22]。
3 EIN2跨膜蛋白及其谜一样的功能
EIN2蛋白位于CTR1的下游,是乙烯信号通路中的第一个正调控组分。科学家在20多年前筛选乙烯突变体时筛到了它[45]。EIN2基因发生功能缺失型突变可以产生完全的乙烯不敏感表型,暗示EIN2在乙烯信号转导通路中的核心地位[9]。序列分析表明,EIN2基因编码一个12跨膜的大的膜结合蛋白,其N端定位于内质网膜上,C端则游离在胞质中[9,46]。进一步研究发现,EIN2蛋白的N端类似于具有转运二价阳离子功能的NRAMP离子通道家族蛋白;与N端不同是,EIN2的C端为一亲水区,而且序列分析表明EIN2的C端与其他已知蛋白相比没有明显的序列相似性,暗示出EIN2蛋白的C端可能具有非常独特的功能[9]。
虽然EIN2蛋白的N端类似于NRAMP离子通道,但是没有实验证据表明EIN2的N端确实具有离子转运能力,EIN2的N端在乙烯信号转导通路中的功能还有待研究[9]。科学家在研究EIN2的功能时还发现,如果转基因过量表达EIN2蛋白的全长或者其N端,拟南芥暗中或者光下的乙烯反应表型都不能被恢复;但是,转基因单独过量表达EIN2蛋白的C端却能够恢复拟南芥光下生长的幼苗以及成年植株的乙烯反应表型[9]。因此,EIN2蛋白可能是一个双功能信号组分:N端负责接受上游乙烯信号,而且参与乙烯的暗形态反应;C端则负责激活下游乙烯信号[9]。虽然研究发现拟南芥中EIN2蛋白与乙烯受体、COP9光信号复合体组分EER5等蛋白具有一定相互作用,对EIN2功能的研究提供了一定线索,但是EIN2所介导的乙烯信号传递机制仍是一个谜团[47-48]。近期,科学家发现EIN2蛋白也是一种短周期蛋白,可被两个F-box蛋白ETP1和ETP2识别并经由蛋白酶体依赖的泛素化降解途径而降解[18]。此外,在筛选其他激素如生长素、细胞分裂素和脱落酸等信号途径的突变体时也能够筛到对应于EIN2基因的突变体,暗示EIN2可能涉及多个激素信号之间的交叉反应[49-50]。由此可见,EIN2蛋白不仅对于乙烯信号至关重要,而且还参与不同激素信号途径之间的交叉反应。
4 EIN3/EILs在转录水平对乙烯信号的调控
乙烯信号经过传递最终被汇聚到位于EIN2蛋白下游的转录因子家族EIN3以及5个EILs(EIN3-like proteins)等正调控因子。在5个EILs中,EIL1与EIN3的同源性最高,功能上也最为相似[10,22]。研究发现,EIN3与EIL1双基因突变形成的突变体ein3/eil1与ein2一样在黄化苗时期和成年植株中能够表现出完全的乙烯不敏感表型,说明这两个转录因子负责大部分的乙烯信号传递[10,30]。同时,单独转基因过量表达 EIN3或者EIL1,乙烯信号途径都能被组成型地活化;但是,单基因功能缺失突变体ein3-1或eil1-1都只表现出部分乙烯不敏感的表型,说明EIN3及其家族成员不仅正调乙烯反应而且在功能上存在冗余[10]。除了EIL1,拟南芥中还有另外4个EIN3类似蛋白EIL2-EIL5,可能涉及早期乙烯反应的调节过程[51]。
转录因子EIN3和EIL1在细胞核内通过启动转录级联反 应而激活乙烯 应 答 基 因 的 表 达[10-11,52-53]。 研 究 发现,EIN3能够通过二聚化的方式特异性地与一个被称为EIN3结合位点(EIN3 binding site,EBS)的短的回文结构启动子区域相结合并启动ERF1,EDF1-4等初级转录因子的表达;而ERF1可以进一步与启动子区含GCC-box的次级乙烯应答基因结合并起始其转录[11]。有趣的是,研究发现对转录因子EIN3本身的调节发生在蛋白水平而非转录水平上[10]。在乙烯信号通路中,由26S蛋白酶体所介导的泛素化降解途径调节了EIN3蛋白的稳定性,而负责识别和结合转录因子EIN3的有两个F-box蛋白 EBF1和 EBF2[16-17]。EBF1和 EBF2通过介导EIN3等转录因子降解过程而对乙烯信号进行负调[15-17]。另外,EIN3等乙烯转录因子还受到负反馈调节机制的调控,因为乙烯处理能够同时上调EBF2的转录量从而使乙烯反应不至于过强[15,53]。近期研究还发现一个具有5’→3’外切核酸酶活性的蛋白EIN5/XRN4可以通过促进EBF1/EBF2的mRNA的降解来拮抗EBF对EIN3的负反馈调控,这一发现大大扩展了人们对于乙烯信号调控水平多样性和复杂性的认识[19]。
5 乙烯信号与其他信号通路的交叉反应
尽管人们对于乙烯信号转导通路的认识正在逐步清晰,但是对于植物多样化乙烯效应的下游分子网络还知之甚少,而乙烯与其他信号途径的交叉反应为多样化的乙烯反应提供了最为重要的解释[54]。研究表明,乙烯信号与生长素(auxin)、细胞分裂素(cytokinin,CK)、赤霉素(gibberellin,GA)、脱落酸(abscisic acid,ABA)、油菜素内脂(brassinosteroid,BR)、茉莉酸(jasmonic acid,JA)、水杨酸(salicylic acid,SA)等植物激素及植物生长因子,以及光、葡萄糖等环境及营养因子存在着广泛的联系。不同信号途径之间互相协同,在植物的生长发育以及植物应对生物、非生物胁迫等复杂过程中起到非常重要的作用。
5.1 乙烯与生长素
人们早已知道乙烯和生长素在根的伸长、下胚轴的差异化生长以及根毛形成等多种生物学过程中存在广泛的交叉反应[55]。研究发现,许多在根上特异的乙烯不敏感突变体,其生长素的合成或者转运也存在缺陷,暗示乙烯可能通过调节生长素信号来抑制植物根的伸长[56-57]。两个在根上特异的乙烯不敏感突变体wei2和wei7的发现把乙烯信号和生长素信号联系起来[58-59]。WEI2和WEI7分别编码色氨酸生物合成的关键酶邻氨基苯甲酸合成酶的α和β亚基,而色氨酸是多个生长素生物合成途径的共同前体[59]。乙烯通过激活WEI2和WEI7基因的表达促进了根中生长素的合成和积累,从而抑制了植物根的伸长。更为直接的证据来自于另一个根特异的乙烯不敏感突变体wei8的发现[60]。WEI8基因负责编码生长素生物合成途径中的色氨酸氨基转移酶TAA1。研究发现,乙烯处理能够通过特异地在根部分生区促进TAA1及其同源基因TAR2的表达促进生长素IAA(indole-3-acetic acid)的生物合成,从而抑制了根的伸长。另外,乙烯三重反应的短根表型不仅与乙烯调控的生长素在局部的生物合成相关,还与乙烯调控的生长素从分生区到伸长区的分布相关[61-63]。如前所述,乙烯可以通过促进生长素的合成来抑制植物根的伸长。反过来,生长素也可以通过诱导乙烯合成基因ACS4的表达而促进乙烯的生物合成[64]。研究发现,在黄化苗中ACS4基因可以特异地被IAA诱导;同时,在拟南芥生长素抗性突变体axr1-12,axr2-1以及aux1-7中ACS4基因的表达存在缺陷[64]。
在顶端弯钩的发育上,生长素和乙烯同样存在交叉反应。在筛选拟南芥乙烯信号遗传突变体时,科学家发现一个特异的仅在顶端弯钩对乙烯不敏感的突变体hls1[45]。过表达HLS1基因能够产生组成型的下胚轴弯钩表型。乙烯处理能够激活HLS1基因的转录,而在ein2突变体中HLS1基因表达下调[65]。研究表明,hls1突变体对应的基因HLS1编码的蛋白属于N-乙酰转移酶家族成员[65]。此外,HLS1蛋白的缺失可以导致子叶和下胚轴弯钩区域生长素调节基因出现异常的表达[65]。以上结果表明,HLS1可能通过抑制子叶和下胚轴弯钩区域生长素诱导的细胞伸长而介导顶端弯钩的形成。为进一步研究HLS1基因介导的顶端弯钩形成的分子机制,科学家在hls1突变体的基础上筛选到一个可以抑制该突变表型的突变体hss1(HLS1suppressor1)[66]。HSS1基因编码一个生长素应答转录因子ARF2(Auxin Response Factor 2),而 ARF2对于在 HLS1下游调节差异化细胞伸长以及顶端弯钩形成是必需的[66]。乙烯处理能够下调ARF2的蛋白水平,而且这一过程是HLS1依赖的。综上所述,生长素应答转录因子ARF2通过抑制差异化的生长素效应而抑制顶端弯钩的形成;而乙烯通过激活其上游的HLS1蛋白负调ARF2的功能,从而促进顶端弯钩的形成。
5.2 乙烯与细胞分裂素
除了生长素,其他激素也可以诱导乙烯生物合成量的提高,例如细胞分裂素[67-68]。生长素处理可以导致几个ACS蛋白转录量的增加,而细胞分裂素可以增加ACS5蛋白的稳定性[67,69-70]。另一组科学家的研究显示细胞分裂素可以增加一些ACS基因的稳定性[71]。此外,细胞分裂素对乙烯的诱导依赖于典型的细胞分裂素双组分应答通路的存在,包括组氨酸激酶、组氨酸磷酸转移蛋白以及应答调节因子等的存在。这些结果说明,乙烯生物合成途径的限速酶ACS可以被多种激素包括乙烯、生长素、细胞分裂素等通过不同的信号通路介入进行调节,这可能由植物自身组织的特异性及其对不同环境条件的响应来调节乙烯的生物合成量。
5.3 乙烯与赤霉素
赤霉素也是植物生长和发育过程中所必需的一种植物激素。有一个研究组报道,乙烯和赤霉素的交叉反应会影响欧洲山毛榉(FagussylvaticaL.)种子由休眠到萌发的转换[72]。他们发现用GA3或者乙烯利处理山毛榉的种子之后其乙烯生物合成酶FsACO1基因的表达大幅上调,但是乙烯利的调节效应可以被GA的生物合成抑制剂多效唑逆转,说明GA正调控FsACO1基因的表达[72]。GA信号通路的一个关键事件即能够与GA受体GID1相互作用的负调控因子DELLA蛋白在响应GA之后的降解。DELLA蛋白在维持植物体内GA的动态平衡以及在调节GA与其他植物激素信号的交叉反应过程中也发挥着重要作用[73]。在GA存在的情况下,DELLA蛋白被降解,从而下游GA反应的抑制被解除。有科学家观察到乙烯处理可以延迟GA介导的依赖于CTR1的GFP-RGA从根细胞核内的消失,说明乙烯处理可以稳定DELLA蛋白[74]。相似地,还有报道称乙烯可以通过调节DELLA依赖的花分生组织特异性基因来控制花期转换[75]。乙烯信号的激活可以减少具有生物活性的GA水平,因此可以促进DELLA蛋白的积累;而DELLA蛋白积累进而可以延缓植物生命周期,并且可以通过抑制花分生组织特异性基因LEAFY和SOC1的表达延迟开花[75]。最近有研究表明,GA也能够通过诱导HLS1的表达而使黄化苗顶端弯钩加剧[76]。没有GA时,DELLA蛋白能够与EIN3的DNA结合结构域互作从而抑制了EIN3对HLS1基因的转录;施加GA之后,DELLA对EIN3的转录抑制效应被解除,HLS1基因的表达被启动,从而促进了顶端弯钩的形成[76]。
5.4 乙烯与脱落酸
科学家在筛选ABA突变体时发现一个能够使种子在萌发过程中对ABA敏感性增强的突变体era3是由于乙烯信号核心组分EIN2基因突变所致,揭示出ABA信号在此过程中与乙烯信号相互作用并且受到乙烯信号的负调[77]。实际上,当时有两个独立的课题组分别筛选可能影响ABA信号通路的突变并且都鉴定到了乙烯信号的突变体[77-78]。此外,实验显示ctr1和ein2突变体分别对abi1突变体起增强和抑制作用;其他乙烯不敏感突变体也表现出增强的ABA反应,进一步说明在拟南芥种子中乙烯作为ABA信号的一个负调节因子存在[77-78]。与种子中的效应相反,同样这些在种子中表现出增强的ABA反应的乙烯不敏感突变体在根上却表现出对ABA反应的降低[77-78]。更为复杂的是,研究发现虽然外源ABA没有影响到乙烯的生物合成,但是乙烯合成增加突变体可以产生一个与乙烯不敏感突变体相似的在根上对ABA不敏感的表型。很难想像乙烯反应降低和乙烯合成增加都能够在根对ABA的反应上产生相同的效应,但是这也进一步暗示在拟南芥中ETR1应答途径可能调控了不依赖于乙烯的信号。除了种子和根,有科学家在气孔开闭研究过程中同样发现乙烯和脱落酸存在相互作用[79]。他们用乙烯过表达突变体eto1-1以及乙烯不敏感突变体etr1-1,ein3-1研究发现,乙烯能够抑制ABA诱导的气孔关闭[79]。这一结果进一步说明乙烯和脱落酸存在着广泛而复杂的交叉反应,而且其相互作用在不同组织及发育阶段具有不同的效应。
5.5 乙烯与油菜素内脂
油菜素内脂近年也得到了广泛的研究,被称为第六大植物激素。与其他五大类植物激素相比,油菜素内脂具有独特的生理活性,而且含量极低即可发挥生理效应。近期的研究发现,油菜素内脂与乙烯信号在下胚轴伸长以及顶端弯钩形成等方面存在着相互作用及功能冗余[80]。乙烯能够通过控制油菜素内脂的生物合成并且在顶端弯钩建立油菜素内脂的浓度梯度来促进弯钩的形成[80-81]。在控制下胚轴伸长方面,最近的研究发现油菜素内脂可能通过其信号途径中的一个受体样蛋白激酶FERONIA来调节乙烯反应的强度,以此调节拟南芥下胚轴的长度[82]。另外,油菜素内脂处理可以使乙烯生物合成增加[67]。近期研究发现,和细胞分裂素类似,油菜素内脂能够通过促进ACS基因转录以及稳定ACS5蛋白来增加乙烯的生物合成[71]。
5.6 乙烯与茉莉酸
乙烯和茉莉酸之间同样存在着复杂的交叉反应,既相互拮抗,又相互协同。一方面,乙烯可以强烈抑制茉莉酸介导的损伤应答基因的转录,茉莉酸反过来也可以抑制乙烯介导的顶端弯钩形成,说明两种激素之间在一定条件下可能相互拮抗[83-84]。另一方面,在抵抗真菌感染时二者又可以协同作用,植物受到真菌感染即可快速产生乙烯和茉莉酸并使下游防御基因的表达升高,例如ERF1,ORA59以及PDF1.2等防御基因[85-87]。科学家发现,如果单独用乙烯或者茉莉酸处理植物即可分别诱导防御基因的较高水平的表达;有趣的是,如果两种激素同时施加则会使下游防御基因的表达量达到最高值,说明在诱导防御基因表达上两者具有协同效应[85,88]。此外,无论是用乙烯、茉莉酸单独处理或者两者同时处理乙烯不敏感突变体ein2以及茉莉酸不敏感突变体coi1都无法再诱导下游防御基因的表达,暗示植物防御反应的激活依赖于乙烯和茉莉酸两种信号通路的同时存在[88-89]。最近,科学家发现茉莉酸信号通路的负调控因子JAZ蛋白能够通过募集一个RPD3类型的组蛋白去乙酰化酶HDA6调节组蛋白的乙酰化进而抑制乙烯信号转录因子EIN3/EIL1依赖的基因转录,从而抑制了茉莉酸信号通路[90]。研究发现,与施加乙烯可以使EIN3/EIL1蛋白稳定一样,施加茉莉酸也可以通过促进JAZ蛋白的降解而稳定转录因子EIN3/EIL1[90]。另一方面,如果乙烯信号被阻断,转录因子EIN3/EIL1将会被迅速降解而导致植物对乙烯和茉莉酸均不敏感;而当茉莉酸信号被阻断时,将导致JAZ蛋白的大量积累进而强烈抑制转录因子EIN3/EIL1的功能,同样会致使植物对乙烯和茉莉酸不敏感表型的出现[90]。以上结果表明,乙烯信号转录因子EIN3/EIL1可能是乙烯和茉莉酸信号通路的交叉结点。
5.7 乙烯与水杨酸
植物抗病主要涉及3个植物激素,即水杨酸、茉莉酸和乙烯。一般情况下,茉莉酸和乙烯相互协同,茉莉酸/乙烯依赖的防御反应会受到寄生性病原菌以及植食性昆虫的侵害而被诱导激活;水杨酸则往往与茉莉酸/乙烯相互拮抗,水杨酸依赖的防御反应主要受到活体营养的病原菌侵染而激活[91-93]。另一方面,乙烯和水杨酸之间也存在协同关系。之前的研究发现,在乙烯不敏感的烟草中乙烯对于由烟草花叶病毒感染诱发的水杨酸依赖的系统获得性抗性(systemic acquired resistance,SAR)发病是必需的[94]。乙烯可以增强拟南芥对水杨酸的反应,导致水杨酸应答标志基因PR-1表达增强[95-96]。由此可见,乙烯与水杨酸不仅相互拮抗,乙烯还能对水杨酸诱导的PR-1基因表达产生协同效应,而且这一协同效应在乙烯不敏感突变体ein2中被阻断了,说明乙烯对水杨酸的调节依赖于EIN2且需经由乙烯信号途径[96]。
5.8 乙烯与光
植物的生长、发育离不开光,人们对于光信号在植物体内的传导机制研究由来已久。如前所述,乙烯处理能够导致依赖于HLS1的ARF2蛋白水平的降低,进而造成顶端弯钩两侧细胞生长速率产生差异从而加剧弯钩[65,97-98]。研究发现,光和乙烯对顶端弯钩的作用恰好相反,光照能够引起HLS1蛋白水平降低,进而导致ARF2蛋白积累使弯钩消除[66]。由此看出,光、乙烯和生长素在控制植物顶端弯钩形成方面是协同作用的。最近的研究发现,乙烯能够促进植物由暗形态建成到光形态建成的转换[99]。在植物幼苗中,原叶绿酸(protochlorophyllide,Pchlide)需经过光依赖的氧化还原酶POR(protochlorophyllide oxidoreductase)催化形成叶绿酸(chlorophyllide,Chlide),以促进叶绿体的形成而实现光形态建成。黑暗导致的原叶绿酸的过度积累对植物具有光毒性。研究发现,在黑暗中乙烯信号可以通过其转录因子EIN3/EIL1抑制叶绿素前体Pchlide的过度积累;另外,EIN3/EIL1可以直接诱导PORA/B基因的转录从而促进叶绿素合成,从而促进了植物由暗形态建成到光形态建成的转换[99]。
5.9 乙烯与葡萄糖
葡萄糖不仅作为能量供应物质存在,它也是植物生长发育过程中的重要信号分子。科学家在研究葡萄糖不敏感突变体gin1时发现了葡萄糖和乙烯信号之间相互拮抗的关系[100]。乙烯过表达突变体eto1和乙烯组成型信号突变体ctr1表现出葡萄糖不敏感的表型强烈支持了这一拮抗关系。与此相一致的是,几个乙烯不敏感突变体etr1-1,ein2,ein3以及ein6则表现出对葡萄糖超敏感的表型[100-101]。sis1和gin4两个葡萄糖不敏感突变体的发现进一步确定了糖信号和乙烯信号的拮抗关系,因为这两个突变都是ctr1的等位突变形式[101-102]。虽然sis1和gin4在萌发后的发育过程中表现出相似的葡萄糖不敏感表型,但是只有gin4/ctr1在暗中表现出组成型的三重反应表型。如前所述,过表达EIN2的C末端虽然可以部分恢复拟南芥光下生长的幼苗和成株的乙烯反应表型,但是在暗中却仍没有三重反应表型,说明葡萄糖信号可能影响ETR1和EIN2下游的特定乙烯信号途径[9,100-101]。进一步的研究发现,葡萄糖可能通过植物葡萄糖感受蛋白己糖激酶来促进乙烯信号转录因子EIN3蛋白的降解,这与乙烯促进EIN3蛋白的稳定性恰好相反;另外,ein3突变体表现出葡萄糖超敏感表型,而在拟南芥中转基因过表达EIN3蛋白则会降低植物对葡萄糖的敏感性[103]。这一结果说明EIN3可能是葡萄糖和乙烯信号在ETR1和EIN2下游相互拮抗的一个节点。
(2012年2月17日收到)
[1]JOHNSON P R,ECKER J R.The ethylene gas signal transduction pathway:a molecular perspective [J].Annu Rev Genet,1998,32:227-254.
[2]BLEECKERA B,KENDE H.Ethylene:agaseous signal molecule in plants[J].Annu Rev Cell Dev Biol,2000,16:1-18.
[3]KENDRICK M D,CHANG C.Ethylene signaling:new levels of complexity and regulation[J].Curr Opin Plant Biol,2008,11:479-485.
[4]STEPANOVA A N,ECKER J R.Ethylene signaling:from mutants to molecules[J].Curr Opin Plant Biol,2000,3:353-360.
[5]CHANG C,KWOK S F,BLEECKER A B,MEYEROWITZ E M.Arabidopsisethylene-response gene ETR1:similarity of product to two-component regulators [J].Science,1993,262:539-544.
[6]HUA J,SAKAI H,NOURIZADEH S,et al.EIN4 and ERS2 are members of the putative ethylene receptor gene family inArabidopsis[J].Plant Cell,1998,10:1321-1332.
[7]KIEBER J J,ROTHENBERG M,ROMAN G,et al.CTR1,a negative regulator of the ethylene response pathway inArabidopsis,encodes a member of the raf family of protein kinases[J].Cell,1993,72:427-441.
[8]GAO Z,CHEN Y F,RANDLETT M D,et al.Localization of the Raf like kinase CTR1 to the endoplasmic reticulum ofArabidopsisthrough participation in ethylene receptor signaling complexes[J].J Biol Chem,2003,278:34725-34732.
[9]ALONSO J M,HIRAYAMA T,ROMAN G,et al.EIN2,a bifunctional transducer of ethylene and stress responses inArabidopsis[J].Science,1999,284:2148-2152.
[10]CHAO Q,ROTHENBERG M,SOLANO R,et al.Activation of the ethylene gas response pathway inArabidopsisby the nuclear protein ETHYLENEINSENSITIVE3and related proteins[J].Cell,1997,89:1133-1144.
[11]SOLANO R,STEPANOVA A,CHAO Q,et al.Nuclear events in ethylene signaling:A transcriptional cascade mediated by ETHYLENE-INSENSITIVE3 and ETHYLENE-RESPONSE-FACTOR1[J].Genes &Dev,1998,12:3703-3714.
[12]BINDER B M,RODRÍGUEZ F I,BLEECKER A B.The Copper Transporter RAN1 Is Essential for Biogenesis of Ethylene Receptors inArabidopsis[J].J Biol Chem,2010,285:37263-37270.
[13]RESNICK J S,WEN C K,SHOCKEY J A,et al.REVERSION-TO-ETHYLENE SENSITIVITY1,a conserved gene that regulates ethylene receptor function inArabidopsis[J].Proc Natl Acad Sci USA,2006,103:7917-7922.
[14]DONG C H,RIVAROLA M,RESNICK J S,et al.Subcellular co-localization ofArabidopsisRTE1 and ETR1 supports a regulatory role for RTE1 in ETR1 ethylene signaling [J].Plant J,2008,53:275-286.
[15]GAGNE J M,SMALLE J,GINGERICH D J,et al.ArabidopsisEIN3-binding F-box1 and2 form ubiquitin-protein ligases that repress ethylene action and promote growth by directing EIN3 degradation[J].Proc Natl Acad Sci USA,2004,101:6803-6808.
[16]GUO H,ECKER J R.Plant responses to ethylene gas are mediated by SCF(EBF1/EBF2)-dependent proteolysis of EIN3 transcription factor[J].Cell,2003,115:667-677.
[17]POTUSCHAK T,LECHNER E,PARMENTIER Y,et al.EIN3-dependent regulation of plant ethylene hormone signaling by twoArabidopsisF box proteins:EBF1 and EBF2[J].Cell,2003,115:679-689.
[18]QIAO H,CHANG K N,YAZAKI J,et al.Interplay between ethylene,ETP1/ETP2 F-box proteins,and degradation of EIN2 triggers ethylene responses inArabidopsis[J].Genes &Dev,2009,23:512-521.
[19]OLMEDO G,GUO H,GREGORY B D,et al.ETHYLENEINSENSITIVE5 encodes a 5’→3’exoribonuclease required for regulation of the EIN3-targeting F-box proteins EBF1/2 [J].Proc Natl Acad Sci USA,2006,103:13286-13293.
[20]POTUSCHAK T,VANSIRI A,BINDER B M,et al.The exoribonuclease XRN4 is a component of the ethylene response pathway inArabidopsis[J].Plant Cell,2006,18:3047-3057.
[21]YOO S D,CHO Y H,TENA G,et al.Dual control of nuclear EIN3 by bifurcate MAPK cascades in C2H4signaling[J].Nature,2008,451:789-795.
[22]An F Y,Zhao Q,Ji Y S,et al.Ethylene-induced stabilization of ETHYLENE INSENSITIVE3 and EIN3-LIKE1is mediated by proteasomal degradation of EIN3 binding F-Box 1 and 2 that requires EIN2 inArabidopsis[J].Plant Cell,2010,22:2384-2401.
[23]SAKAI H,HUA J,CHEN Q G,et al.ETR2is an ETR1-like gene involved in ethylene signaling inArabidopsis[J].Proc Natl Acad Sci USA,1998,95:5812-5817.
[24]CHANG C,STADLER R.Ethylene hormone receptor action inArabidopsis[J].Bioessays,2001,23:619-627.
[25]SCHALLER G E,BLEECKER A B.Ethylene binding sites generated in yeast expressing theArabidopsisETR1 gene[J].Science,1995,270:1809-1811.
[26]WANG W Y,HALL A E,O’MALLEY R,et al.Canonical histidine kinase activity of the transmitter domain of the ETR1 ethylene receptor fromArabidopsisis not required for signal transmission[J].Proc Natl Acad Sci USA,2003,100:352-357.
[27]GAMBLE R L,QU X,SCHALLER G E.Mutational analysis of the ethylene receptor ETR1.Role of the histidine kinase domain in dominant ethylene insensitivity[J].Plant Physiol,2002,128:1428-1438.
[28]MOUSSATCHE P,KLEE H J.Autophosphorylation activity of theArabidopsisethylene receptor multigene family[J].J Biol Chem,2004,279:48734-48741.
[29]RODRÍGUEZ F I,ESCH J J,HALL A E,et al.A copper cofactor for the ethylene receptor ETR1 fromArabidopsis[J].Science,1999,283(5404):996-998.
[30]ALONSO J M,STEPANOVA A N,SOLANO R,et al.Five components of the ethylene-response pathway identified in a screen for weak ethylene insensitive mutants inArabidopsis[J].Proc Natl Acad Sci USA,2003a,100:2992-2997.
[31]HUA J,MEYEROWITZ E M.Ethylene responses are negatively regulated by a receptor gene family inArabidopsis thaliana[J].Cell,1998,94:261-271.
[32]HALL A E,BLEECKER A B.Analysis of combinatorial lossof-function mutants in theArabidopsisethylene receptors reveals that the ers1etr1 double mutant has severe developmental defects that are EIN2 dependent[J].Plant Cell,2003,15:2032-2041.
[33]BINDER B M,O’MALLEY R C,WANG W Y,et al.Arabidopsisseedling growth response and recovery to ethylene.A kinetic analysis[J].Plant Physiol,2004a,136:2913-2920.
[34]XIE F,LIU Q,WEN C K.Receptor signal output mediated by the ETR1 N-terminus is primarily subfamily Ⅰ receptorsdependent[J].Plant Physiol,2006,142:492-508.
[35]BARRY C S,GIOVANNONI J J.Ripening in the tomato green-ripe mutant is inhibited by ectopic expression of a protein that disrupts ethylene signaling [J].Proc Natl Acad Sci USA,2006,103:7923-7928.
[36]CLARK K L,LARSEN P B,WANG X,et al.Association of theArabidopsisCTR1 Raf-like kinase with the ETR1 and ERS ethylene receptors[J].Proc Natl Acad Sci USA,1998,95:5401-5406.
[37]HUANG Y,LI H,HUTCHISON C E,et al.Biochemical and functionalanalysis of CTR1,aprotein kinase that negatively regulates ethylene signaling inArabidopsis[J].Plant J,2003,33:221-233.
[38]NOVIKOVA G V,MOSHKOV I E,SMITH A R,et al.The effect of ethylene on MAPKinase-like activity inArabidopsis thaliana[J].FEBS Lett,2000,474:29-32.
[39]OUAKED F,ROZHON W,LECOURIEUX D,et al.A MAPK pathway mediates ethylene signaling in plants [J].EMBO J,2003,22:1282-1288.
[40]ECKER J R.Reentry of the ethylene MPK6 module [J].Plant Cell,2004,16:3169-3173.
[41]LIU Y,ZHANG S.Phosphorylation of 1-aminocyclopropane-1-carboxylic acid synthase by MPK6,a stress-responsive mitogen-activated protein kinase,induces ethylene biosynthesis inArabidopsis[J].Plant Cell,2004,16:3386-3399.
[42]JOO S,LIU Y,LUETH A,et al.MAPK phosphorylation-induced stabilization of ACS6 protein is mediated by the noncatalytic C-terminal domain,which also contains the cis-determinant for rapid degradation by the 26S proteasome pathway[J].Plant J,2008,54:129-140.
[43]XU J,LI Y,WANG Y,et al.Activation of MAPK kinase 9 induces ethylene and camalexin biosynthesis and enhances sensitivity to salt stress inArabidopsis[J].J Biol Chem,2008,283:26996-27006.
[44]BETHKE G,UNTHAN T,UHRIG J F,et al.Flg22 regulates the release of an ethylene response factor substrate from MAP kinase 6 inArabidopsisthalianavia ethylene signaling[J].Proc Natl Acad Sci USA,2009,106:8067-8072.
[45]GUZMAN P,ECKER J.Exploiting the triple response ofArabidopsisto identify ethylene-related mutants[J].Plant Cell,1990,2:513-523.
[46]BISSON M M,BLECKMANN A,ALLEKOTTE S,et al.EIN2,the central regulator of ethylene signalling,is localized at the ER membrane where it interacts with the ethylene receptor ETR1[J].Biochem J,2009,424(1):1-6.
[47]CHRISTIANS M J,ROBLES L M,ZELLER S M,et al.Theeer5 mutation,which affects a novel proteasome-related subunit,indicates a prominent role for the COP9 signalosome in resetting the ethylene-signaling pathway inArabidopsis[J].Plant J,2008,55:467-477.
[48]BISSON M M,GROTH G.New insight in ethylene signaling:autokinase activity of ETR1 modulates the interaction of receptors and EIN2[J].Mol Plant,2010,3:882-889.
[49]FUJITA H,SYONO K.Genetic analysis of the effects of polar auxin transport inhibitors on root growth inArabidopsis thaliana[J].Plant Cell Physiol,1996,37:1094-1101.
[50]SU W P,HOWELL S H.A single genetic locus,Ckr1,definesArabidopsismutants in which root growth is resistant to low concentrations of cytokinin[J].Plant Physiol,1992,99:1569-1574.
[51]BINDER B M,MORTIMORE L A,STEPANOVA A N,et al.Short-term growth responses to ethylene inArabidopsisseedlings are EIN3/EIL1 independent [J].Plant Physiol,2004,136:2921-2927.
[52]OHME-TAKAGI M,SHINSHI H.Ethylene-inducible DNA binding proteins that interact with an ethylene responsive element[J].Plant Cell,1995,7:173-182.
[53]ALONSO J M,STEPANOVA A N,LEISSE T J,et al.Genome-wide insertional mutagenesis ofArabidopsisthaliana[J].Science,2003b,301:653-657.
[54]STEPANOVA A N,ALONSO J M.Ethylene signaling and response:where different regulatory modules meet[J].Curr Opin Plant Biol,2009,12:548-555.
[55]STEPANOVA A N,ALONSO J M.Ethylene signaling and response pathway:A unique signaling cascade with a multitude of inputs and outputs[J].Plant Physiol,2005,123:195-206.
[56]ROMAN G,LUBARSKY B,KIEBER J J,et al.Genetic analysis of ethylene signal transduction inArabidopsisthaliana:five novel mutant loci integrated into a stress response pathway[J].Genetics,1995,139:1393-1409.
[57]BENNETT M J,MARCHANT A,GREEN H G,et al.ArabidopsisAUX1 gene:apermease-like regulator of root gravitropism [J].Science,1996,273:948-950.
[58]BARTEL B.Auxin biosenthesis[J].Annu Rev Plant Physiol Plant Mol Biol,1997,48:51-66.
[59]STEPANOVA A N,HOYT J M,HAMILTON A A,et al.A link between ethylene and auxin uncovered by the characterization of two root specific ethylene-insensitive mutants inArabidopsis[J].Plant Cell,2005,17:2230-2242.
[60]STEPANOVA A N,ROBERTSON-HOYT J,YUN J,et al.TAA1-mediated auxin biosynthesis is essential for hormone crosstalk and plant development[J].Cell,2008,133:177-191.
[61]RUZICKA K,LJUNG K,VANNESTE S,et al.Ethylene regulates root growth through effects on auxin biosynthesis and transport-dependent auxin distribution [J].Plant Cell,2007,19:2197-2212.
[62]STEPANOVA A N,YUN J,LIKHACHEVA A V,et al.Multilevel interactions between ethylene and auxin inArabidopsisroots[J].Plant Cell,2007,19:2169-2185.
[63]SWARUP R,PERRY P,HAGENBEEK D,et al.Ethylene upregulates auxin biosynthesis inArabidopsisseedlings to enhance inhibition of root cell elongation[J].Plant Cell,2007,19:2186-2196.
[64]ABEL S,NGUYEN M D,CHOW W,et al.ASC4,aprimary indoleacetic acid-responsive gene encoding 1-aminocyclopropane-1-carboxylate synthase inArabidopsisthaliana:structural characterization,expression inEscherichiacoli,and expression characteristics in response to auxin[J].J Biol Chem,1995,270:19093-19099.
[65]LEHMAN A,BLACK R,ECKER J R.HOOKLESS1,an ethylene response gene,is required for differential cell elongation in theArabidopsishypocotyl[J].Cell,1996,85:183-194.
[66]LI H,JOHNSON P,STEPANOVA A,et al.Convergence of signaling pathways in the control of differential cell growth inArabidopsis[J].Dev Cell,2004,7:193-204.
[67]WOESTE K E,YE C,KIEBER J J.TwoArabidopsismutants that overproduce ethylene are affected in the posttranscriptional regulation of 1-aminocyclopropane-1-carboxylic acid synthase[J].Plant Physiol,1999,119:521-530.
[68]ARTECA R N,ARTECA J M.Effects of brassinosteroid,auxin,and cytokinin on ethylene production inArabidopsis thalianaplants[J].J Exp Bot,2008,59:3019-3026.
[69]VOGEL J P,WOESTE K E,THEOLOGIS A,et al.Recessive and dominant mutations in the ethylene biosynthetic gene ACS5 ofArabidopsisconfer cytokinin insensitivity and ethylene overproduction,respectively [J].Proc Natl Acad Sci USA,1998,95:4766-4771.
[70]CHAE H S,FAURE F,KIEBER J J.Theeto1,eto2,andeto3 mutations and cytokinin treatment increase ethylene biosynthesis inArabidopsisby increasing the stability of ACS protein[J].Plant Cell,2003,15:545-559.
[71]HANSEN M,CHAE H S,KIEBER J J.The regulation of ACS protein stability by cytokinin and brassinosteroid [J].Plant J,2009,57:606-614.
[72]CALVO A P,NICOLÁS C,NICOLÁS G,et al.Evidence of a cross-talk regulation of a GA20-oxidase (FsGA20ox1)by gibberellins and ethylene during the breaking of dormancy inFagussylvaticaseeds[J].Physiol Plantarum,2004,120:623-630.
[73]HIRANO K,UEGUCHI-TANAKA M,MATSUOKA M.GID1-mediated gibberellin signaling in plants [J].Trends Plant Sci,2008,13:192-199.
[74]ACHARD P,VRIEZEN W H,VAN DER STRAETEN D,et al.Ethylene regulatesArabidopsisdevelopment via the modulation of DELLA protein growth repressor function[J].Plant Cell,2003,15:2816-2825.
[75]ACHARD P,BAGHOUR M,CHAPPLE A,et al.The plant stress hormone ethylene controls floral transition via DELLA-dependent regulation of floral meristem-identity genes [J].Proc Natl Acad Sci USA,2007,104:6484-6489.
[76]AN F,ZHANG X,ZHU Z,et al.Coordinated regulation of apical hook development by gibberellins and ethylene in etiolatedArabidopsisseedlings[J].Cell Res,2012,22:915-927.
[77]GHASSEMIAN M,NAMBARA E,CUTLER S,et al.Regulation of abscisic acid signaling by the ethylene response pathway inArabidopsis[J].Plant Cell,2000,12:1117-1126.
[78]BEAUDOIN N,SERIZET C,GOSTI F,et al.Interactions between abscisic acid and ethylene signaling cascades [J].Plant Cell,2000,12:1103-1115.
[79]TANAKA Y,SANO T,TAMAOKI M,et al.Ethylene inhibits abscisic acid-induced stomatal closure inArabidopsis[J].Plant Physiol,2005,138:2337-2343.
[80]DE GRAUWE L,VANDENBUSSCHE F,TIETZ O,et al.Auxin,ethylene and brassinosteroids:tripartite control of growth in theArabidopsishypocotyl[J].Plant Cell Physiol,2005,46(6):827-836.
[81]GENDRONJ M,HAQUEB A,GENDRONB N,et al.Chemical genetic dissection of brassinosteroid-ethylene interaction[J].Mol Plant,2008,1(2):368-379.
[82]DESLAURIERS S D,LARSEN P B.FERONIA is a key modulator of brassinosteroid and ethylene responsiveness inArabidopsishypocotyls[J].Mol Plant,2010,3(3):626-640.
[83]MEMELINK J.Regulation of gene expression by jasmonate hormones[J].Phytochemistry,2009,70:1560-1570.
[84]TURNER J G,ELLIS C E,DEVOTO A.The jasmonate signal pathway[J].Plant Cell,2002,14:S153-S164.
[85]BERROCAL-LOBO M,MOLINA A.Ethylene response factor 1 mediatesArabidopsisresistance to the soilborne fungus Fusarium oxysporum [J].Mol Plant Microbe Interact,2004,17:763-770.
[86]DONG X N.SA,JA,ethylene,and disease resistance in plants[J].Curr Opin Plant Biol,1998,1(4):316-323.
[87]BROWN R L,KAZAN K,MCGRATH K C,et al.A role for the GCC-box in jasmonate mediated activation of the PDF1.2 gene ofArabidopsis[J].Plant Physiol,2003,132:1020-1032.
[88]LORENZO O,PIQUERAS R,SÁNCHEZ-SERRANO J J,SOLANO R.ETHYLENE RESPONSE FACTOR1 integrates signals from ethylene and jasmonate pathways in plant defense[J].Plant Cell,2003,15:165-178.
[89]PENNINCKX I,EGGERMONT K,TERRAS F,et al.Pathogen-induced systemic activation of a plant defensin gene inArabidopsisfollows a salicylic acid-independent pathway[J].Plant Cell,1996,8:2309-2323.
[90]ZHU Z,AN F,FENG Y,et al.Derepression of ethylenestabilized transcription factors (EIN3/EIL1)mediates jasmonate and ethylene signaling synergy inArabidopsis[J].Proc Natl Acad Sci USA,2011,108(30):12539-12544.
[91]KESSLER A,BALDWIN I T.Plant responses to insect herbivory:the emerging molecular analysis[J].Annu Rev Plant Biol,2002,53:299-328.
[92]GLAZEBROOK J.Contrasting mechanisms of defense against biotrophic and necrotrophic pathogens[J].Annu Rev Phytopathol,2005,43:205-227.
[93]HOWE G A,JANDER G.Plant immunity to insect herbivores[J].Annu Rev Plant Biol,2008,59:41-66.
[94]VERBERNE M C,HOEKSTRA J,BOL J F,et al.Signaling of systemic acquired resistance in tobacco depends on ethylene perception[J].Plant J,2003,35:27-32.
[95]LAWTON K A,POTTER S L,UKNES S,et al.Acquired resistance signal transduction inArabidopsisis ethylene independent[J].Plant Cell,1994,6:581-588.
[96]DE VOS M,VAN ZAANEN W,KOORNNEEF A,et al.Herbivore-induced resistance against microbial pathogens inArabidopsis[J].Plant Physiol,2006,142:352-363.
[97]SILK W H,ERICKSON R O.Kinematics of hypocotyl curvature[J].Am J Bot,1978,65:310-319.
[98]HARPHAM N V J,BERRY A W,KNEE E M,et al.The effect of ethylene on the growth and development of widetype and mutantArabidopsisthaliana(L.)Heynh [J].Ann Bot(Lond),1991,68:55-61.
[99]ZHONG S,ZHAO M,SHI T,et al.EIN3/EIL1 cooperate with PIF1to prevent photo-oxidation and to promote greening ofArabidopsisseedlings[J].Proc Natl Acad Sci USA,2009,106:21431-21436.
[100]ZHOU L,JANG J C,JONES T L,et al.Glucose and ethylene signal transduction crosstalk revealed by anArabidopsisglucose-insensitive mutant [J].Proc Natl Acad Sci USA,1998,95:10294-10299.
[101]CHENG W H,ENDO A,ZHOU L,et al.A unique shortchain dehydrogenase/reductase inArabidopsisglucose signaling and abscisic acid biosynthesis and functions[J].Plant Cell,2002,14:2723-2743.
[102]GIBSON S I,LABY R J,KIM D.Thesugar-insensitive1(sis1)mutant ofArabidopsisis allelic toctr1 [J].Biochem Bioph Res Co,2001,280:196-203.
[103]YANAGISAWA S,YOO S D,SHEEN J.Differential regulation of EIN3 stability by glucose and ethylene signalling in plants[J].Nature,2003,425:521-525.
Study of Ethylene Signal Transduction Pathway
ZHANG Cun-li①,GUO Hong-wei②
①Ph.D.Candidate,②Professor,StateKeyLaboratoryofProtein andPlantGeneResearch,SchoolofLifeSciences,PekingUniversity,Beijing100871,China
As one of the five classical phytohormones,ethylene has been focused on by scientists since it was found.Although it has very simple structure,the gaseous phytohormone ethylene has important effects on the developmental processes and stress responses of plant.Through nearly two decades of research,scientists established a largely linear ethylene signal transduction pathway.In the model plantArabidopsis,there are five ethylene receptors ETR1,ETR2,ERS1,ERS2 and EIN4 encoded by a multigene family in the upstream of this signaling pathway.A Raf-like protein kinase CTR1 combines with ethylene receptors and co-localizes in the ER membrane together with these receptors.In the absence of ethylene,receptors and CTR1 can inhibit the downstream ethylene signaling together.A positive regulator EIN2 is the downstream of these two negative regulators.IfEIN2 gene is mutated,the etiolated seedlings of plant will show completely ethylene insensitive phenotype even when high concentration of ethylene exists,which demonstrates that EIN2 plays a key role in ethylene signaling pathway.EIN3 and EILs are transcription factors downstream of EIN2.They will start the transcription of ethylene related genes in response to ethylene signal.It was also found that these transcription factors were regulated by ubiquitin/proteasome degradation pathway.The F-box proteins,which are responsible for recognition and combination of EIN3 protein,are EBF1 and EBF2.EIN5is a 5’→3’exonuclease and antagonizes the negative feedback regulation on EIN3 by promotingEBF1 andEBF2 mRNA decay.Recently,studies have shown that EIN2is also a short half-life protein and will be degraded by the ubiquitin/proteasome pathway.Another two F-box proteins ETP1 and ETP2 are responsible for the regulation of EIN2 protein.Although great progress was made in ethylene signal transduction pathway,further research on the fine-tuning of ethylene signal transduction and the crosstalk between ethylene and other phytohormones still need to be detected.
phytohormone,ethylene,signal transduction
10.3969/j.issn.0253-9608.2012.04.006
(编辑:沈美芳)
