CRISPR系统结构与功能研究进展
2012-05-31黎满香田世成颜运秋
蒋 伟,黎满香,田世成,颜运秋
(湖南农业大学动物医学院,湖南长沙 410128)
在长期的进化过程中,细菌往往通过各种各样的策略来使自己适应各种环境,从而使细菌能在各种复杂的环境中生存。而在基因水平,细菌能获得新的外源基因促使它抵抗环境选择压力,这是细菌进化过程中的一个重要因素。很多细菌和古细菌都是从其他细菌中利用核酸交换来获得外源基因,这种交换被称为基因水平转移(horizontal gene transfer;HGT),例如粪肠球菌中约三分之一的基因组由可移动性片段组成。HGT即是从环境中摄取可移动性片段的DNA(例如质粒,噬菌体等),然后整合到自己的基因组中从而获得相应的表型的过程。然而在这些水平转移中,只有极少一部分能给细菌提供选择优势。为避免冗于的水平转移(即不能给宿主细菌提供选择优势的水平转移),细菌在进化过程中形成了上百种的自我保护机制,例如目前了解最为透切的消除入侵生物的限制性修饰酶系统和表面排斥现象等[1]。
近期研究发现,CRISPR位点 (CRISPR loci)广泛存于细菌及古细菌基因组中。其主要结构以高度保守的重复序列、完全不同的间区相互交替排列而成,在这种重复序列远端还存在与这种重复序相关的蛋白,二者相互辅助以完成其特定的生物学功能。
1 CRISPR位点的发现
Ishino Y等[2]在对大肠埃希菌中的碱性磷酸酶的同工酶Ipa对应的核酸序列进行分析时,发现在该产物编码区的下游存在一个由29个碱基组成的、重复出现的高度保守的核酸序列,两个重复序列之间由长度大致相等的独特的非重复核酸序列间隔开。随后,Mojica F J等[3]在对地中海极嗜盐菌(Haloferaxmediterranei)和沃尔卡尼极嗜盐菌(Haloferax volcanii)的研究中发现了类似的结构。在其他一些细菌的研究中,这种类似的基因结构被广泛报道。随着类似的结构被广泛的发现,直到1991年Hermans P W等才把这种结构正式命名SRSR(short regularly spaced repeats),2002年被Jansen R改名为CRISPR并一直沿用至今[3-7]。随着细菌的全基因组测定的完成,已经在90%的古细菌及40%的细菌基因组中发现CRISPR位点,更令人惊奇的是在大部分已发现该位点的细菌中含有至少1个CRISPR位点,有的甚至含有2个或3个该位点[8]。
2 CRISPR-Cas系统的结构
2.1 CRISPR位点的结构
CRISPR位点由一个前导区(leader)、多个重复序列(repeat)和多个间区(spacer)组成,这些重复序列与间区相互交替出现并串连成一个完整的CRISPR位点(如图1A)。前导区一般是处于CRISPR位点上游,由300bp~500bp碱基组成的一个AT富集的区域。这个区域一般来说在种内是比较保守的,但在种间却有显著的差异[7]。重复序列一般由23bp~50 bp的碱基组成,其平均长度在31bp左右。这段序列在已给定的CRISPR位点的多个重复中相对保守,一般只存在1个~3个碱基差异,这种差异被称为退化重复(degeneration repeats,DRs),但在微生种间甚至于同一个基因组中的两个或多个不同的CRISPR位点间却存在很大的差异。通过对CRISPR DataBase[8]中贮存的所有重复序列进行分析,结果发现这些重复序列间没有明显的联系,但却具有二重对称性,这就意味着它能形成像发夹一样的二极结构。到目前为止,还没有关于这些重复序列具体功能的描述。间区由17bp~84bp碱基组成,平均长度在36bp左右。在同一个CRISPR位点中,基本上没有相同或比较相似的间区。通过长期的研究发现,部分间区序列与已知的或质粒、或噬菌体的序列相匹配[3]。在CRISPR DataBase中的间区序列,只有约2%的序列在Gen-Bank中能找到相应的匹配,这可能是因为目前只有极少的噬菌体和质粒被测序分析,随着测序分析技术的逐渐成熟,这个比例在不断的增加[9]。
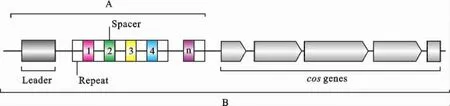
图1 CRISPR位点结构图(A代表CRISPR位点;B代表整个CRISPR系统)Fig.1 The structure of CRISPR-loci(A represents CRISPR loci;B represents the whole element of CRISPR system)
2.2 CRISPR-Cas系统的结构
在生物机体内,最终起到调节生命作用的物质是肽或者蛋白质,而如图1A所示的整个区域都是非编码区,对于实现CRISPR位点的功能显然不现实。因此,在微生长期的进化过程中,为了使CRISPR宿主能适应各种环境,必定有相关蛋白质起着调节作用。Jansen R等[7]对CRISPR侧翼序列进行比对,发现4段基因,并依次命名为cas1、cas2、cas3、cas4,这4段基因通常都与CRISPR位点同时出现于几乎所有CRISPR位点阳性细菌的基因组中,且这4段基因在不同的细菌基因组间具有显著的同源性,说明这4段基因可能与CRISPR位点存在某种关系。这些Cas蛋白或邻接于CRISPR位点下游(图1B),或分散于其他地方,目前发现距CRISPR位点最远的达到了9 kb。通过进一步分析这些蛋白质序列,发现存在高度保守的氨基酸残基或/和功能结构域(amino acid resi-dues or/and functional domains)。这4个蛋白通常以cas4-cas3-cas1-cas2的顺序存在于染色体或质粒上,其中Cas3类似于一种解旋酶,而Cas4类似于RecB家簇的一种半胱氨酸富集的核酸内切酶上。这些酶的功能以及其排列顺序或许预示着这些蛋白质存在一种特定的作用机制。通过大量的序列比对分析发现,Cas1似乎是CRISPR位点中最基础的蛋白质,因为在所有含有CRISPR位点的生物种中都发现了cas1。到目前为止,已有40多种伴随CRISPR位点的Cas蛋白被鉴定[10]。这些蛋白存在多样性,有趣的是每一个CRISPR都含有一个特异性的CRISPR位点关联的被缩写为csx(“x”代表亚型的第一个字母,例如,csy代表Yersina亚型)的基因,因此Haft D H等跟据该位点对45种Cas蛋白的携带情况,将CRISPR位点分成9种亚型(表1)[15]。由表1可以看出cas1-cas6这六种蛋白广泛存在于CRISPR亚型中,可以说是CRISPR位点的核心蛋白,而其中又数cas1最为常见,存在于所有亚型中[11]。但对于普遍存在于所有亚型中的Cas1蛋白的功能却存在较大的争论。Wiedenheft B等从绿脓杆菌中得到的Cas1蛋白有金属依赖性,dsDNA特异性核酸内切酶功能,而Han D H等从Sulpholobus solfataricus中得到的Cas1蛋白却表现为序列非特异性、多位点、高亲合力的核酸结合蛋白的功能[12-14]。
3 CRISPR位点的功能
在自然界中,微生物可以说无处不在。当细菌存在于较为恶劣的环境中时,它该如何抵御外界的干扰呢?从地球上开始有生物至今,在长期的自然选择压力作用下,这些微生物得以进化,使其能在自然选择的压力下存活。在这个漫长的进化过程中,它们形成了许多用于保护自己的,几乎接近完美的功能性系统。这些功能性系统能使其从其他微生物或环境中获得对自己有利的基因,例如基因盒-整合子系统(integron-cassette system)能使细菌从其他供体菌中获得用来抵抗环境中对应药物的耐药基因;但同时也存在另外一些系统限制这种过程的发生,一般来说是限制对自己有害的基因进入自身,从而达到保护自身的目的,例如许多革兰阳性及革兰阴性菌中的表面排拆(surface exclusion)功能,它能从分子水平降低受体菌的接受能力,从而降低自身对有害的基因片段的接受能力[16]。
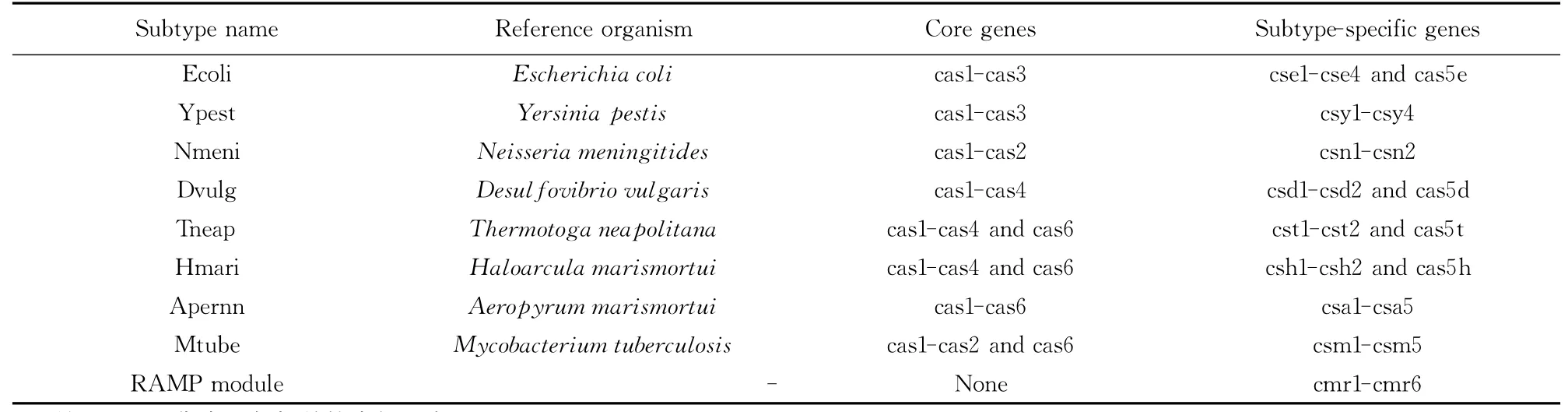
表1 Cas蛋白亚型的分类Table 1 Classification of Cas protein subtypes
在这些自我保护系统中,CRISPR系统亦是其中之一。最早人们对CRISPR系统的认识是通过对噬菌体的研究发现,噬菌体是细菌最为常见的入侵者(invader),为了抵御噬菌体的入侵,即在长期的进化过程中细菌形成了用于抵抗噬菌体入侵的CRISPR系统,从分子水平来说,这个过程可分为三个阶段。(1)合并整合形成新间区;(2)表达加工 CRISPR RNAs(crRNAs);(3)crRNAs干扰入侵的噬菌体[17]。整个过程类似于机体的免疫反应过程,故而此过程被称为遗传记忆(genetic memory)。
3.1 新间区的获得
当细菌与噬菌体共处于同一环境中时,噬菌体为了自身生存,必定定植于细菌表面,然后借助于细菌DNA复制的机制进行繁殖。当噬菌体DNA进入携带有CRISPR系统的细胞膜内时,该宿主体内的CRISPR相关蛋白质复合体中类似于Cas1核酸结合蛋白将会迅速的介导该复合体与噬菌体DNA进行结合,然后通过类似于Cas2核酸内切酶功能等蛋白质的作用,将该DNA切割成17bp~84bp不等的核酸小片段,然后在相关蛋白的作用下,将其中的一个小片段整合至前导区与第一个重复之间形成一个新的间区(图2A)。通过这样一个过程,CRISPR位点就整合进了一个新的间区。这就使宿主快速的适应环境中的入侵者,因此我们也可以将这个阶段称为适应(Adaptation)阶 段。Bolotin A 等[18]研 究 发 现,CRISPR位点中间区的个数与宿主对噬菌体的敏感性呈负相关。进而Barrangou R等利用噬菌体去攻击至少含有一个CRISPR位点的嗜热链球菌(Streptococcusthermophilus),最终在前导序列及第一个重复间区找到一个新的间区,而且变异株对该噬菌体的敏感性降低,证明了该位点具有抵御外源DNA片段的入侵的功能[19-20]。在序列分析中,有些序列来源于细菌质粒,证明CRISPR位点也能从质粒上获得新的间区。
3.2 表达加工处理CRISPR
当新的间区稳定地存在于CRISPR位点后,间区所包含的信息将通过CRISPR途径来保护宿主不受特定的核酸攻击。然而要实现这样一个途径,最基本的就是要将间区加工处理以形成较小的crRNAs。CRISPR的转录首先在闪烁古球菌(Archaeoglobusfulgidus)中被发现[21]。此研究表明位点中的转录首先从前导序列的末端开始,也就是说在前导序列的末端可能含有CRISPR启动子(CRISPR promoter),从启动子开始转录,重复序列和间区先被转录成前体物RNA(precursor RNA;pre-crRNA)。此时核心蛋白Cas1-Cas4将会共同形成一个复合物,这个复合物将pre-crRNA从特异性位点上(此特异性位点具体何在尚不清楚,有报道表明在间区的约第8个碱基处,但也有报道称在重复序列上)剪切成更小的crRNA,随后较小的crRNA与蛋白质形成具有特殊功能的复合物crRNP(crRNA-Cas ribonucleoprotein)[22](图2B)。
3.3 crRNAs干扰入侵核酸
当宿主再次接触到环境中的噬菌体或是质粒时,crRNP将会迅速地通过核酸结合蛋白与进入细胞内的外源核酸特异性结合,这种特异性是由新整合进入间区所存贮的信息决所决定。然后在核酸内切酶的作用下,将目标核酸从3′端开始剪切成适合长度(大约为18bp)的小片段,致使目标核酸无法形使它的功能,噬菌体复制失败,质粒转移失败,从而达到保护宿主的目的(图2C)。
通过以上三个阶段,CRISPR位点行使了它具有的功能。然而它仍然面临所有生理反应所面临的问题,即如何识别自身与非自身核酸,从而避免发生“自身免疫”。在CRISPR位点中,新间区所携带的与crRNA仍然存在着对应的关系。最近一项研究表明,crRNP在体外试验中,能够区别目标核酸与非目标核酸[23],可能是因crRNP中的crRNA与目标核酸存在对应的配对关系,从而能准确的识别目标核酸。
4 展望
到目前为止,CRISPR位点在很多细菌中都有发现。这样普遍的存在于细菌及古细菌中,证明该位点对于细菌的生存进化有着举足轻重的作用。通过进一步深入的研究,探明该位点的作用机制,将使我们对微生物的认识更加深入。一方面,通过该位点的间区序列差异,理论上可以推论该细菌在进化过程的某些细节。另一方面,通过目前对于该位点的研究,如果能将该位点通过分子生物学方法,整合进入工程质粒,然后应用于工业细菌中,例如食物产品及大规模发酵等领域,将会给食品安全带来一个革命性的转折。另外,利用该位点的干扰功能,我们亦可在其中引入某种内源性基因的间区,然后将该质粒转化进入宿主,或许可以达到基因沉默的目的,但这一点尚没有试验证明。此外,CRISPR位点既然能抵御外源基因的入侵,那么由可移动元件介导的耐药基因在细菌的的传播过程是否也能被这种机制所抑制呢?关于这一点尚待试验证明[24]。如果该位点对于某些耐药性质粒或是耐药基因,也能达到沉默的目的,那么,通过对该位点的克隆、转化,将会使全球性的耐药问题得以缓解。
总之,介于该位点中已知的作用机制和有待试验证明的未知的作用机制,该位点对于我们对微生界的认识都有十分显著的作用。因此,对于CRISPR位点的研究,具有无可比拟的重要性。
[1]Asakura Y,Kojima H,Kobayashi I.Evolutionary genome engineering using a restriction-modification system [J].Nucleic Acids Res,2011,39(20):9034-9046.
[2]Ishino Y,Shinagawa H,Makino K,et al.Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion inEscherichiacoli,and identification of the gene product[J].J Bacteriol,1987,169(12):5429-5433.
[3]Mojica F J,Ferrer C,Juez G,et al,Long stretches of short tandem repeats are present in the largest replicons of the ArchaeaHaloferaxmediterraneiandHaloferaxvolcaniiand could be involved in replicon partitioning[J].Mol Microbiol,1995,17(1):85-93.
[4]Hermans P W,van Soolingen D,Bik E M,et al.Insertion element IS987from Mycobacterium bovis BCG is located in a hot-spot integration region for insertion elements inMycobacteriumtuberculosiscomplex strains[J].Infect Immun,1991,59(8):2695-2705.
[5]Klenk H P,Clayton R A,Tomb J F,et al.The complete genome sequence of the hyperthermophilic,sulphate-reducing archaeonArchaeoglobusfulgidus[J].Nature,1997,390(6658):364-370.
[6]Kawarabayasi Y,Hino Y,Horikawa H,et al.Complete genome sequence of an aerobic hyper-thermophilic crenarchaeon,AeropyrumpernixK1[J].DNA Res,1999,6(2):83-101,145-152.
[7]Jansen R,Embden J D,Gaastra W,et al.Identification of genes that are associated with DNA repeats in prokaryotes[J].Mol Microbiol,2002,43(6):1565-1575.
[8]Grissa I,Vergnaud G,Pourcel C.The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J].BMC Bioinformatics,2007,8(172):1-10.
[9]Heidelberg J F,Nelson W C,Schoenfeld T,et al.Germ warfare in a microbial mat community:CRISPRs provide insights into the coevolution of host and viral genomes[J].PLoS one,2009,4(1):e4169.
[10]Hale C R,Zhao P,Olson S,et al.RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex[J].Cell,2009,139(5):945-956.
[11]Haft D H.,Selengut J,Mongodin E F,et al.A guild of 45 CRISPR-associated(Cas)protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes[J].PLoS Comput PLoS Comput Biol,2005,1(6):e60.
[12]Wiedenheft B,Zhou K,Jinek M,et al.Structural basis for DNase activity of a conserved protein implicated in CRISPR-medi-ated genome defense[J].Structure,2009,1(6):904-912.
[13]Han D,Lehmann K,Krauss G.SSO1450—a CAS1protein fromSulfolobussolfataricusP2with high affinity for RNA and DNA [J].FEBS Lett,2009,583(12):1928-1932.
[14]Jore M M,Lundgren M,van Duijn E,et al.Structural basis for CRISPR RNA-guided DNA recognition by Cascade [J].Nat Struct Mol Biol,2011,18(5):529-536.
[15]Marraffini L A,Sontheimer E J.CRISPR interference:RNA-directed adaptive immunity in bacteria and archaea [J].Nat Rev Genet,2010,11(3):181-190.
[16]Possoz C,Gagnat J,Sezonov G,et al.Conjugal immunity ofStreptomycesstrains carrying the integrative element pSAM2is due to the pif gene(pSAM2immunity factor)[J].Mol Microbiol,2003,47(4),1385-1393.
[17]van der Oost J,Jore M M.,Westra E R,et al.CRISPR-based adaptive and heritable immunity in prokaryotes[J].Trends Biochem Sci,2009,34(8):401-407.
[18]Bolotin A,Quinquis B,Sorokin A,et al.Clustered regularly interspaced short palindrome repeats(CRISPRs)have spacers of extrachromosomal origin[J].Microbiology,2005,151(Pt8):2551-2561.
[19]Barrangou R,Fremaux C,Deveau H,et al.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science,2007,315(5819):1709-1712.
[20]Tang T H,Bachellerie J P,Rozhdestvensky T,et al.Identification of 86candidates for small non-messenger RNAs from the archaeonArchaeoglobusfulgidus[J].Proc Natl Acad Sci USA,2002,99(11):7536-7541.
[21]Kunin V,Sorek R,Hugenholtz P.Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J].Genome Biol,2007,8(4):R61.
[22]Marraffini L A,Sontheimer E J.Self versus non-self discrimination during CRISPR RNA-directed immunity[J].Nature,2010,463(7280):568-571.
[23]Horvath P,Barrangou R.CRISPR/Cas,the immune system of bacteria and archaea[J].Science,2010,327(5962):167-170.
[24]Palmer K L,Gilmore M S.Multidrug-resistant enterococci lack CRISPR-cas[J].MBio,2010,1(4):pii:e00227-10.
