基于叶绿体基因组全序列分析真叶植物叶绿体基因的适应性进化*
2012-05-09苏应娟
王 博,高 磊,苏应娟,王 艇
(1.中国科学院武汉植物园//中国科学院植物种质创新与特色农业重点实验室,湖北 武汉 430074;2.中国科学院研究生院,北京 100049;3.中山大学生命科学学院, 广东 广州 510275)
近年来,随着分子生物学,尤其是大规模测序技术的不断革新,叶绿体基因组学研究正被日益推进。自从1986年首次报道烟草Nicotianatabacum[1]和地钱Marchantiapolymorpha[2]的叶绿体基因组全序列以来,来自不同植物的叶绿体基因组全序列数据已有显著增加;至2011年底,被公布的叶绿体基因组全序列已达247个。这些数据资源为深入解析叶绿体基因组的进化提供了条件。
叶绿体基因组一般为双链环形DNA分子,包含4个部分,即大单拷贝区(Large Single copy Region,LSC)、小单拷贝区(Small Single copy Region,SSC)、反向重复区A(Inverted Repeat Region A,IRA)和反向重复区B(IRB);其中两个IR区的序列相同,而方向相反[1]。叶绿体基因按功能可大体分为3类:遗传系统基因、光合系统基因和其它基因[3-5]。遗传系统基因是指与转录和翻译有关的基因,包括编码rRNA、tRNA、核糖体蛋白亚基、RNA聚合酶、RNA成熟酶及叶绿体蛋白酶亚基等产物的基因;光合系统基因是指同光合作用有关的基因,它们负责编码光系统Ⅰ和Ⅱ成员、细胞色素b6f复合体、ATP合酶、NAD(P)H脱氢酶、Rubisco大亚基以及cemA;至于其它基因则功能不一,这涉及一些保守的开放阅读框和出现于部分植物叶绿体基因组的chlB、chlL和chlN。
分析基因的适应性进化对研究基因结构的改变及功能变异具有重要参考价值[6]。对于蛋白质编码基因而言,暗示其发生适应性进化的一个重要特征就是非同义替换率(dN)高于同义替换率(dS);这样,通过估算dN与dS之比ω就可推测蛋白质的进化趋向和所受选择压力。ω的估值等于、小于和大于1即提示进化呈中性、经历负选择与正选择(适应性进化)。同中性进化和负选择相比,正选择极为少见,且往往发生在氨基酸序列的少数位点,或是出现于某个很短的进化历史时期,因而难以检测。近年在最大似然(Maximum Likelihood,ML)估计框架下、基于密码子置换模型建立起来的分析方法,使得检测选择对氨基酸位点的作用有了明显改进。目前,Nielsen和Yang开发的PAML (Phylogenetic Analysis by Maximum Likelihood) 软件已得到广泛运用[7-8]。
陆续有报道指出,clpP[9]、rbcL[10-11]及matK[12]等叶绿体基因曾经历过正选择。然而,对于其他叶绿体基因是否发生适应性进化却所知甚少。另外,虽然现已积累有较为丰富的叶绿体基因组序列数据,但是还未见在叶绿体基因组全序列水平上对真叶植物(蕨类、裸子植物和被子植物)主要类群叶绿体基因的适应性进化进行分析。为此,本文尝试性地开展了这方面的工作,目的是:了解都有哪些叶绿体基因受到正选择作用,明确有哪些正选择基因是属于类群特异的,并初步探讨这些基因发生适应性进化的生物学意义。
1 材料和方法
1.1 序列数据
参考Smith等[13]的蕨类系统,选取蕨类植物已报道的7个叶绿体基因组全序列。根据高磊等[14]利用79个叶绿体基因序列数据构建的系统树,选取7种裸子植物、15种被子植物的叶绿体基因组全序列。从GenBank中获得以上植物的叶绿体基因组全序列数据(表1),鉴定各类群共有的蛋白质编码基因,并依次对其进行适应性进化分析。
1.2 适应性进化分析
基于最大似然模型,利用PAML 软件的codeml程序进行适应性进化分析。本研究采用的是位点间可变ω模型, 该模型假定不同位点具有不同的ω值[8]。使用7种密码子替换模型,分别为M0(单一比率)、Mla(近中性)、M2a(正选择)、M3(离散)、M7(beta)、M8(beta和ω)及M8a。模型M0假定所有位点和分支具相同ω值。模型M1a 假定蛋白质具两类位点:一类为保守位点,0<ω<1;另一类为中性位点,其ω=1。模型M2a增加第三类位点,其ω取值可大于1。模型M3 利用三类位点的离散分布,它们对应的比例p0、p1及p2和ω值ω1、ω2及ω3分别由数据估计得出。模型M7 利用beta分布(p,q);随参数p和q的改变,该分布可取不同形状。模型M8在模型M7基础上增加一类位点,其比例和ω由数据估计得出,这样位点的ω取值就可大于1。模型M8a (beta和ω= 1)同M8类似,只是将ω值固定为1。两两比较模型M1a和M2a、M7和M8以及M8和M8a,即可检验是否存在正选择位点。利用最大似然法估计出的模型参数值,再借助贝叶斯定律就可计算出每个氨基酸所属位点类别的后验概率,进而依据后验概率鉴定正选择位点[8,15]。

表1 植物的种名和叶绿体基因组全序列登录号
2 结果与讨论
2.1 蕨类植物、裸子植物及被子植物叶绿体基因的适应性进化分析
对于蕨类植物,现仅报道了7个叶绿体基因组全序列。它们共有的蛋白质编码基因为77个(附件1),其中有5个基因检测到正选择,分别是cemA、ndhF、ndhK、rps4和ycf2;正选择基因所占比例为6.5%。对于裸子植物,所取的7种代表植物,它们叶绿体基因组共有的蛋白质编码基因数为67个,这当中有5个基因检测到正选择,分别是chlP、petD、rbcL、ycf1和ycf2;正选择基因所占比例为7.5%。在被子植物中,所选15种代表植物的叶绿体基因组共有的蛋白质编码基因为78个,其中有15个基因检测到正选择,它们分别是accD、atpF、ccsA、ndhB、ndhF、ndhK、matK、petD、rpl20、rpl22、rpoC2、rpoC1、rps7、ycf1和ycf2;正选择基因所占比例达19.2%。
本研究在蕨类、裸子及被子植物的叶绿体基因中都检测到有基因发生适应性进化,而且越高等的类群所拥有的受正选择的基因越多。被子植物受正选择的叶绿体基因数远比蕨类和裸子植物为多,这提示叶绿体基因在被子植物对陆地生态系统的适应过程中可能起着重要作用。
2.2 正选择基因的分布和功能类型
检测到的受正选择作用的叶绿体基因在叶绿体基因组的大小单拷贝区和反向重复区均有分布。蕨类的5个正选择基因,有3个位于大单拷贝区、1个位于小单拷贝区、1个位于反向重复区。裸子植物的5个正选择基因,同样是有3个位于大单拷贝区、1个位于小单拷贝区、而1个位于反向重复区。至于被子植物的15个正选择基因,则是有9个位于大单拷贝区,而小单拷贝区和反向重复区各3个(表2)。这些结果似乎表明,发生正选择的叶绿体基因倾向于位于大单拷贝区。
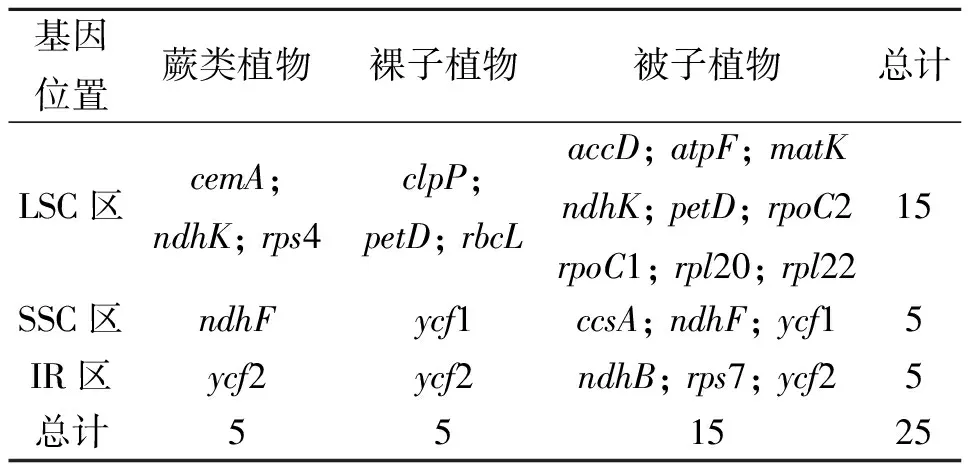
表2 正选择基因所在的叶绿体基因组区域
蕨类植物叶绿体基因组检测到的5个正选择基因,1个属遗传系统基因、3个属光合系统基因、另有1个属其它基因。裸子植物的5个正选择叶绿体基因,1个为遗传系统基因、2个为光合系统基因、另外2个为其它基因。与之相比,被子植物的15个正选择叶绿体基因中,属遗传系统和光合系统的基因各有6个,其余的3个则属其他基因(表3)。可以发现检测出的正选择基因有一半以上是光合系统基因。下面按照不同基因类型分别介绍这些正选择基因的功能,并分析其发生适应性进化的意义。
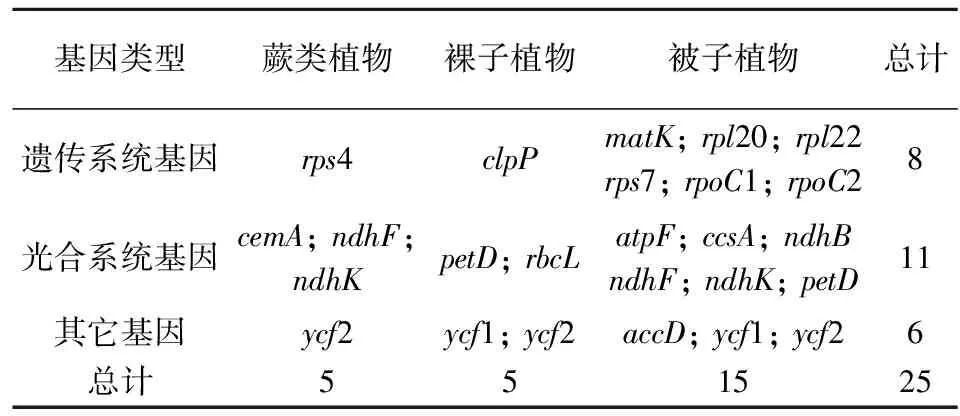
表3 正选择基因类型
2.3 正选择基因的功能及其适应性进化的潜在意义
2.3.1 遗传系统基因 ①clpP基因:clpP基因编码的是Clp蛋白水解酶亚基。该酶位于叶绿体基质,属丝氨酸型蛋白水解酶。在高等植物中,Clp蛋白水解酶的催化亚基ClpP由叶绿体基因clpP编码,而其ATP酶亚基ClpC则由核基因编码[16];它的主要功能是负责降解异常蛋白,以维持叶绿体的正常代谢[17]。我们在裸子植物的clpP基因检测到正选择,显示其适应性进化可能关系着裸子植物叶绿体降解折叠出错蛋白的能力。另有研究发现,clpP的变异可能还会影响细胞对较高水平CO2的适应[18]。②matK基因:matK基因位于叶绿体赖氨酸tRNA基因 (trnKLys)的内含子中,长约1500bp,为单拷贝,负责编码成熟酶。已知该基因编码的成熟酶参与了叶绿体Ⅱ型内含子的剪切[19],涉及对象包括trnK、trnA、trnI、rps12、rpl2和atpF等基因的转录本[20],而且这些基因编码的tRNA和蛋白质产物均为维持叶绿体正常功能所必需。本研究注意到被子植物的matK基因存在适应性进化,这将会对上述基因的转录加工产生影响。Hao等[12]曾专门研究过matK基因的适应性进化,也在被子植物的多个类群检测到正选择。③rps4、rps7、rpl20和rpl22基因:叶绿体拥有60余种核糖体蛋白,其中1/3由叶绿体基因组编码,而其它2/3由核基因组编码[21-24]。rps4、rps7、rpl20及rpl22分别编码核糖体蛋白S4、S7、L20和L22。本文发现蕨类只有rps4基因受到正选择,而被子植物则是rps7、rpl20和rpl22三个基因经历正选择。这些基因的适应性进化会涉及蕨类和被子植物叶绿体的蛋白质合成。张丽君等[25]未在蕨类的rps4基因检测到正选择,其原因可能是由于所选取的代表物种大都属薄囊蕨类。④rpoC1和rpoC2基因:rpoC1和rpoC2基因分别编码叶绿体RNA聚合酶的β’亚基和β’’亚基。不同于细胞核RNA聚合酶,叶绿体RNA聚合酶结构比较简单,能被原核生物RNA聚合酶的抑制剂利福平所抑制。被子植物的这两个基因被检测到受正选择作用,这可能关系着其叶绿体RNA聚合酶的功能行使。
2.3.2 光合系统基因 ①atpF基因;叶绿体ATP合酶(F1F0-ATP酶)由F0和F1两部分组成;疏水的F0镶嵌在膜内、其b亚基保守性较低,而F1则突出于膜外。ATP合酶是能量代谢的关键酶,它既参与光合作用的光合磷酸化反应又催化呼吸作用的氧化磷酸化反应。该酶能利用同光合电子传递或呼吸电子传递耦联的质子运转所形成的跨膜质子动力势,将ADP和无机磷(Pi)合成为ATP[26]。atpF基因编码的就是ATP合酶成员F0的b亚基。被子植物的atpF基因发生适应性进化,会对其叶绿体能量代谢产生一定作用。②cemA基因:cemA基因编码的是叶绿体膜蛋白,但该蛋白的功能尚不清楚[27][28]。Katoh等[29]发现蓝藻集胞藻属的cemA同源物对CO2的传递是必不可少。我们发现蕨类的cemA基因经历正选择,但其进化上的涵义仍待明确。③ccsA基因:ccsA基因负责编码细胞色素c合成蛋白,该蛋白约有250~350个氨基酸,属膜结合蛋白。ccsA的编码产物能与另一基因ccsB的编码蛋白共同构成ccsA复合物[30]。Xie等[31]认为ccsA基因与细胞色素c—血红素的结合有关。这为理解被子植物ccsA基因的适应性进化提供了启示。④ndh基因:ndh基因编码叶绿体NADH脱氢酶。该酶位于类囊体膜 为至少包含11个亚基的复合体[32,33]。高等植物的NADH复合体既参与光合电子传递[34,35],也可作为叶绿体呼吸作用的电子传递载体[36];它能把电子由NADH传递至泛醌,形成跨膜质子梯度,从而推动ATP的合成。此外,该复合体还同胁迫抗性相关[37-40]。在蕨类和被子植物的ndh基因均检测到正选择(表3),它们的适应性进化可能会影响能量转化和抗光氧化胁迫。值得注意的是,很多裸子植物的ndh基因都被丢失或假基因化[41],因此未能进行进化分析。⑤rbcL基因:陆生植物的核酮糖-1,5-二磷酸羧化酶/加氧酶包含大、小亚基各8个,分别为叶绿体rbcL基因和核rbcS基因编码[42]。该酶催化固定CO2的活性位点位于大亚基,特别是其C末端在功能上起重要作用[43]。本研究检测发现裸子植物的rbcL基因受到正选择作用,这有可能改变酶同CO2的结合[44]。刘念等[45]也曾报道裸子植物麻黄属的rbcL基因发生适应性进化。⑥petD基因:叶绿体petD基因负责编码细胞色素b6/f复合体的亚基IV,该亚基对维持复合体的结构和活性十分重要[46]。Chen等[47]发现petD基因的突变可降低衣藻类光合速率。我们注意到裸子植物的petD基因经历了正选择,但其进化方面的意义还有待更多证据。
2.3.3 其它基因 ①accD基因:accD基因编码乙酰辅酶A羧化酶的beta亚基。该酶催化乙酰辅酶A羧化生成丙二酰辅酶A,为脂肪酸和其他次生代谢产物的合成提供底物,是脂肪酸生物合成的关键酶和碳流进入脂肪酸合成的重要调控位点[48]。这为理解accD基因在被子植物发生的适应性进化提供了思路。②ycf1和ycf2基因:ycf1和ycf2基因为叶绿体基因组内两个最大的开放阅读框。尽管有研究认为它们的编码产物对于叶绿体不可缺少[49],但是两者的具体功能究竟如何现在仍不清楚。Wolfe[50]推测ycf2可能编码的是叶绿体ATP酶。我们分析发现,蕨类的ycf1被正选择,而裸子和被子植物的ycf1和ycf2均经历正选择。这一结果的可能进化意义还有待于两基因功能的阐明。
3 结论与展望
我们在蕨类、裸子和被子植物的多个叶绿体基因检测到正选择作用,而且注意到被子植物发生适应性进化的叶绿体基因在数量和比例上都明显比蕨类和裸子植物为多。经受正选择的叶绿体基因主要是遗传系统和光合系统基因。它们的编码产物涉及叶绿体蛋白质合成、基因转录、能量转化与调节及光合作用等诸多过程。推测一些叶绿体功能基因可能在真叶类对陆生生态环境的适应过程中起着重要作用。继续利用下一代测序技术获取数据,并对更多代表植物在叶绿体基因组全序列水平展开分子进化分析,将可为认识真叶植物适应环境改变的微进化机制提供崭新启示。
致谢:感谢中国科学院武汉植物园汪志伟博士提供的宝贵意见。
参考文献:
[1]SHINOZAKI K,OHME M,TANAKA M,et al.The complete nucleotide sequence of the tobacco chloroplast genome: Its gene organization and expression[J].EMBO J,1986,5(9): 2043-2049.
[2]OHYAMA K,FUKUZAWA H,KOHCHI T,et al.Chloroplast gene organization deduced from complete sequence of liverwortMarchantiapolymorphachloroplast DNA[J].Nature,1986,322(6079): 572-574.
[3]WAKASUGI T,TSUDZUKI T,SUGIURA M.The genomics of land plant chloroplasts: Gene content and alteration of genomic information by RNA editing[J].Photosynth Res,2001,70(1): 107-118.
[4]KAHLAU S,ASPINALL S,GRAY J C,et al.Sequence of the tomato chloroplast DNA and evolutionary comparison of solanaceous plastid genomes[J].J Mol Evol,2006,63(2): 194-207.
[5]SHIMADA H,SUGIURA M.Fine structural features of the chloroplast genome: Comparison of the sequenced chloroplast genomes[J].Nucleic Acids Res,1991,19(5): 983-995.
[6]NEI M,KUMAR S.Molecular evolution and phylogenetics[M].USA: Oxford University Press,2000.
[7]YANG Z.PAML 4: Phylogenetic analysis by maximum likelihood[J].Mol Biol Evol,2007,24(8): 1586-1591.
[8]NIELSEN R,YANG Z.Likelihood models for detecting positively selected amino acid sites and applications to the HIV-1 envelope gene[J].Genetics,1998,148(3): 929-936.
[9]ERIXON P,OXELMAN B.Whole-gene positive selection,elevated synonymous substitution rates,duplication,and indel evolution of the chloroplastclpP1 gene[J].PLoS One,2008,3(1): e1386.
[10]KAPRALOV M V,FILATOV D A.Widespread positive selection in the photosynthetic Rubisco enzyme[J].BMC Evol Biol,2007,7: 73.
[11]苏应娟,王艇.水龙骨科附生蕨类Rubisco大亚基的适应性进化:正向选择位点的鉴定[J].中山大学学报:自然科学版,2008,47(5):74-80.
[12]HAO D C,CHEN S L,XIAO P G.Molecular evolution and positive Darwinian selection of the chloroplast maturase matK[J].J Plant Res,2010,123(2): 241-247.
[13]SMITH A R,PRYER K M,SCHUETTPELZ E,et al.A classification for extant ferns[J].Taxon,2006,55(3): 705-731.
[14]GAO L,SU Y J,WANG T.Plastid genome sequencing,comparative genomics,and phylogenomics: Current status and prospects[J].J Syst Evol,2010,48(2): 77-93.
[15]YANG Z H,SWANSON W J,VACQUIER V D.Maximum-likelihood analysis of molecular adaptation in abalone sperm lysin reveals variable selective pressures among lineages and sites[J].Mol Biol Evol,2000,17(10): 1446-1455.
[16]SHANKLIN J,DEWITT N D,FLANAGAN J M.The stroma of higher plant plastids contain ClpP and ClpC,functional homologs ofEscherichiacoliClpP and ClpA: An archetypal two-component ATP-dependent protease[J].Plant Cell,1995,7(10): 1713-1722.
[17]YU A Y,HOURY W A.ClpP: A distinctive family of cylindrical energy-dependent serine proteases[J].FEBS Lett,2007,581(19): 3749-3757.
[18]MAJERAN W,WOLLMAN F A,VALLON O.Evidence for a role of ClpP in the degradation of the chloroplast cytochromeb6fcomplex[J].Plant Cell,2000,12(1): 137-150.
[19]JI Y,FRITSCH P W,LI H,et al.Phylogeny and classification ofParis(Melanthiaceae) inferred from DNA sequence data[J].Ann Bot,2006,98(1): 245-256.
[20]VOGEL J,BORNER T,HESS W R.Comparative analysis of splicing of the complete set of chloroplast group II introns in three higher plant mutants[J].Nucleic Acids Res,1999,27(19): 3866-3874.
[21]刘良式.植物分子遗传学[M].北京:科学出版社,2003.
[22]SUBRAMANIAN A R.Molecular genetics of chloroplast ribosomal proteins[J].Trends Biochem Sci,1993,18(5): 177-181.
[23]DORNE A M,LESCURE A M,MACHE R.Site of synthesis of spinach chloroplast ribosomal proteins and formation of incomplete ribosomal particles in isolated chloroplasts[J].Plant Mol Biol,1984,3(2): 83-90.
[24]GANTT J S.Nucleotide sequences of cDNAs encoding four complete nuclear-encoded plastid ribosomal proteins[J].Curr Genet,1988,14(5): 519-528.
[25]张丽君,陈洁,王艇.蕨类植物叶绿体rps4 基因的适应性进化分析[J].植物研究,2010,30(1): 42-50.
[26]MITCHELL P.Chemiosmotic coupling in oxidative and photosynthetic phosphorylation[J].Biol Rev Camb Philos Soc,1966,41(3): 445-502.
[27]SONODA M,KATOH H,OHKAWA H,et al.Cloning of thecotAgene ofSynechococcusPCC7942 and complementation of acotA-less mutant ofSynechocystisPCC6803 with chimeric genes of the two strains[J].Photosynth Res,1997,54(2): 99-105.
[28]SASAKI Y,SEKIGUCHI K,NAGANO Y,et al.Chloroplast envelope protein encoded by chloroplast genome[J].FEBS Lett,1993,316(1): 93-98.
[29]KATOH A,LEE K S,FUKUZAWA H,et al.cemAhomologue essential to CO2transport in the cyanobacteriumSynechocystisPCC6803[J].Proc Natl Acad Sci USA,1996,93(9): 4006-4010.
[30]HARTSHORNE R S,KERN M,MEYER B,et al.A dedicated haem lyase is required for the maturation of a novel bacterial cytochromecwith unconventional covalent haem binding[J].Mol Microbiol,2007,64(4): 1049-1060.
[31]XIE Z,MERCHANT S.The plastid-encodedccsAgene is required for heme attachment to chloroplastc-type cytochromes[J].J Biol Chem,1996,271(9): 4632-4639.
[32]KUBICKI A,FUNK E,WESTHOFF P,et al.Differential expression of plastome-encodedndhgenes in mesophyll and bundle-sheath chloroplasts of the C4plantSorghumbicolorindicates that the complex I-homologous NAD (P) H-plastoquinone oxidoreductase is involved in cyclic electron transport[J].Planta,1996,199(2): 276-281.
[33]MARTIN M,CASANO L M,SABATER B.Identification of the product ofndhAgene as a thylakoid protein synthesized in response to photooxidative treatment[J].Plant and Cell Physiology,1996,37(3): 293.
[34]JOET T,COURNAC L,HORVATH E M,et al.Increased sensitivity of photosynthesis to antimycin A induced by inactivation of the chloroplastndhBgene.Evidence for a participation of the NADH-dehydrogenase complex to cyclic electron flow around photosystem I[J].Plant Physiol,2001,125(4): 1919-1929.
[35]JOET T,COURNAC L,PELTIER G,et al.Cyclic electron flow around photosystem I in C3plants.In vivo control by the redox state of chloroplasts and involvement of the NADH-dehydrogenase complex[J].Plant Physiol,2002,128(2): 760-769.
[36]CASANO L M,ZAPATA J M,MARTIN M,et al.Chlororespiration and poising of cyclic electron transport.Plastoquinone as electron transporter between thylakoid NADH dehydrogenase and peroxidase[J].J Biol Chem,2000,275(2): 942-948.
[37]WANG P,DUAN W,TAKABAYASHI A,et al.Chloroplastic NAD(P)H dehydrogenase in tobacco leaves functions in alleviation of oxidative damage caused by temperature stress[J].Plant Physiol,2006,141(2): 465-474.
[38]MUNNE-BOSCH S,SHIKANAI T,ASADA K.Enhanced ferredoxin-dependent cyclic electron flow around photosystem I and alpha-tocopherol quinone accumulation in water-stressedndhB-inactivated tobacco mutants[J].Planta,2005,222(3): 502-511.
[39]LI X G,DUAN W,MENG Q W,et al.The function of chloroplastic NAD(P)H dehydrogenase in tobacco during chilling stress under low irradiance[J].Plant Cell Physiol,2004,45(1): 103-108.
[40]QUILES M J.Regulation of the expression of chloroplastndhgenes by light intensity applied during oat plant growth[J].Plant Sci,2005,168(6): 1561-1569.
[41]MARTIN M,SABATER B.Plastidndhgenes in plant evolution[J].Plant Physiol Biochem,2010,48(8): 636-645.
[42]SPREITZER R J,SALVUCCI M E.Rubisco: Structure,regulatory interactions,and possibilities for a better enzyme[J].Annu Rev Plant Biol,2002,53: 449-475.
[43]CURMI P M,CASCIO D,SWEET R M,et al.Crystal structure of the unactivated form of ribulose-1,5-bisphosphate carboxylase/oxygenase from tobacco refined at 2.0-A resolution[J].J Biol Chem,1992,267(24): 16980-16989.
[44]JORDAN D B,OGREN W L.Species variation in the specificity of ribulose biphosphate carboxylase/oxygenase[J].Nature,1981,291: 513-515.
[45]刘念,王庆彪,陈婕,等.麻黄属rbcL基因的适应性进化检测与结构模建[J].科学通报,2010,55(14): 1341-1346.
[46]ESPOSITO D,HIGGS D C,DRAGER R G,et al.A nucleus-encoded suppressor defines a new factor which can promotepetDmRNA stability in the chloroplast ofChlamydomonasreinhardtii[J].Curr Genet,2001,39(1): 40-48.
[47]CHEN X,KINDLE K,STERN D.Initiation codon mutations in theChlamydomonaschloroplastpetDgene result in temperature-sensitive photosynthetic growth[J].EMBO J,1993,12(9): 3627-3635.
[48]SLABAS A R,FAWCETT T.The biochemistry and molecular biology of plant lipid biosynthesis[J].Plant Mol Biol,1992,19(1): 169-191.
[49]DRESCHER A,RUF S,CALSA T J,et al.The two largest chloroplast genome-encoded open reading frames of higher plants are essential genes[J].Plant J,2000,22(2): 97-104.
[50]WOLFE K H.Similarity between putative ATP-binding sites in land plant plastid ORF2280 proteins and the FtsH/CDC48 family of ATPases[J].Curr Genet,1994,25(4): 379-383.
