小麦耐盐性QTL的元分析
2023-10-11程宇坤何万龙耿洪伟
程宇坤,何万龙,任 毅,耿洪伟
(1.新疆农业大学农学院/优质专用麦类作物工程技术研究中心/新疆作物遗传改良与种质创新重点实验室,新疆乌鲁木齐 830052)
小麦耐盐性是由多个基因控制的数量性状,耐盐机理复杂[1]。发掘小麦及其近缘物种中新的耐盐遗传位点并将其运用到育种工作中,已成为培育耐盐小麦品种最经济、有效且安全的方法。
任永哲等[2]以小偃54×京411 RIL群体为材料,检测了盐胁迫下调控小麦苗期性状的QTL,发现多数调控小麦耐盐性的QTL成簇位于3A、4A、4B、5B和5D染色体上。王秀华等[3]在盐胁迫处理下对泰农18×临麦6号RIL群体进行耐盐性QTL定位,在3A、4A、7A和7B染色体上共定位到7个耐盐性QTL。李世姣[4]以CH7034×SY95-71 RIL群体为材料进行耐盐性QTL定位,在小麦5AL染色体上定位到1个稳定的耐盐位点QKna.sx_5A。霍志学[5]以豆麦×矮抗58 RIL群体为材料进行耐盐性QTL定位,共定位到8个QTL,其中Qarl-4A为主效QTL。
数量性状基因座元分析(meta quantitative traits and loci,MQTL)可将不同研究数据进行整合,缩小置信区间[6]。Arcade等[7]开发出用于元分析的BioMercator软件,可将目标性状相关的所有QTL映射整合到一张遗传图谱上,对多个独立试验的QTL数据进行比对分析,并进一步缩小置信区间,获得一致性MQTL。胡雅君等[8]通过对168个控制小麦籽粒可溶性碳水化合物含量的QTL进行元分析,构建一致性图谱,最终获得16个MQTL。王 鹏等[9]对113个粒长QTL和86个粒宽QTL进行元分析,获得18个控制粒长的MQTL和8个控制粒宽的MQTL。程宇坤等[10]通过对342个小麦条锈病抗性QTL,通过元分析获得了194个MQTL。
本研究对已报道的耐盐性QTL[11]进行归纳整理,在Wheat composite 2004图谱的基础上,以Maccaferri等[12]以及DArT网站(https://www.diversityarrays.com/technology-and-resources/geneticmaps/)报道的小麦高密度分子标记图谱作为参考,利用BioMercator v4.2.3软件(http://moulon.inrae.fr/en/logiciels/biomercator/)将这些QTL映射到参考图谱上,构建小麦耐盐性QTL一致性图谱,通过元分析挖掘MQTL,以期为小麦耐盐遗传机制提供理论依据。
1 材料与方法
1.1 材料
以美国农业部公共数据库(http://wheat.pw.usda.gov/)和已发表文献[2,13-44]中控制小麦耐盐性的QTL为研究对象。
1.2 方法
利用BioMercator v4.2.3软件对收集到的小麦耐盐QTL进行整理,包括QTL名称、染色体位置、LOD值、表型变异率、紧密连锁标记、置信区间、群体大小等,其中QTL位置(置信区间和QTL的最大可能位置)和遗传贡献率是进行QTL元分析的两个必要参数。
利用BioMercator v4.2.3软件对位于同一位点不同遗传背景的N个独立存在的目标性状相关的QTL进行运算,计算出同一性状QTL在同一位点或重叠位点上的MQTL。每个MQTL会给出五个模型(1、2、3、4和N),其中赤池信息量准则值(akaike-type criteria values,AIC)最小的模型为最优模型,即真实的QTL模型,按照最大似然函数比方法通过高斯定理计算其置信区间及物理位置。如果某个QTL置信区间未知,可通过Darvasi等[45]应用的的公式来估算95%置信区间。
C.I=530/(N×R2)
(1)
C.I=163/(N×R2)
(2)
其中,C.I为QTL的95%置信区间,N为遗传群体的大小,R2为QTL的表型变异率。公式(1)适用于杂交群体(F2)和回交群体(BC),公式(2)适用于重组自交系群体(RIL)和近等基因系群体(NIL)。若QTL的置信区间已知,根据此公式也可计算出QTL的表型变异率[10]。
2 结果与分析
2.1 小麦耐盐性QTL信息收集和图谱整合结果
从小麦数据库和已发表文献中检索小麦耐盐性QTL,共收集到215个QTL,来自于29不同遗传背景群体,包括Berkut×Worrakatta、SR3×JN177、鲁麦21×山农0431、TK6×DK961等,具体信息见表1。以Maccaferri等[12]以及DArT网站报道的小麦DArT标记图谱作为参考,利用BioMercator v4.2.3软件中的QTL projection功能将收集到的QTL逐一映射到参考图谱上,结果发现共有100个QTL被映射到参考图谱上,这些QTL的LOD值变化范围为1.51~22.35,表型变异率为2.07%~39.21%(表2)。
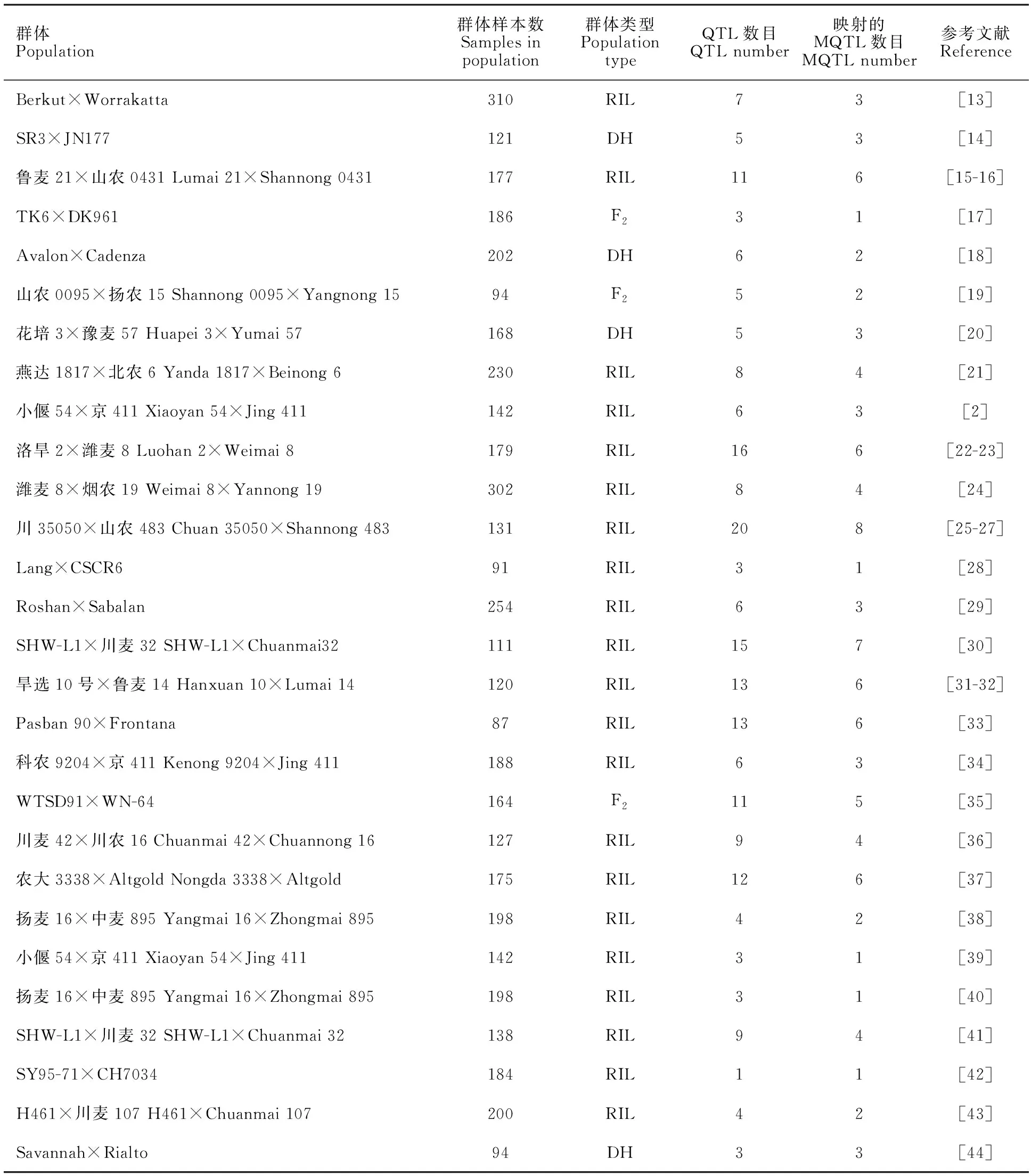
表1 小麦耐盐性QTL数据的整合Table 1 Integration of QTL data for salt tolerance in wheat
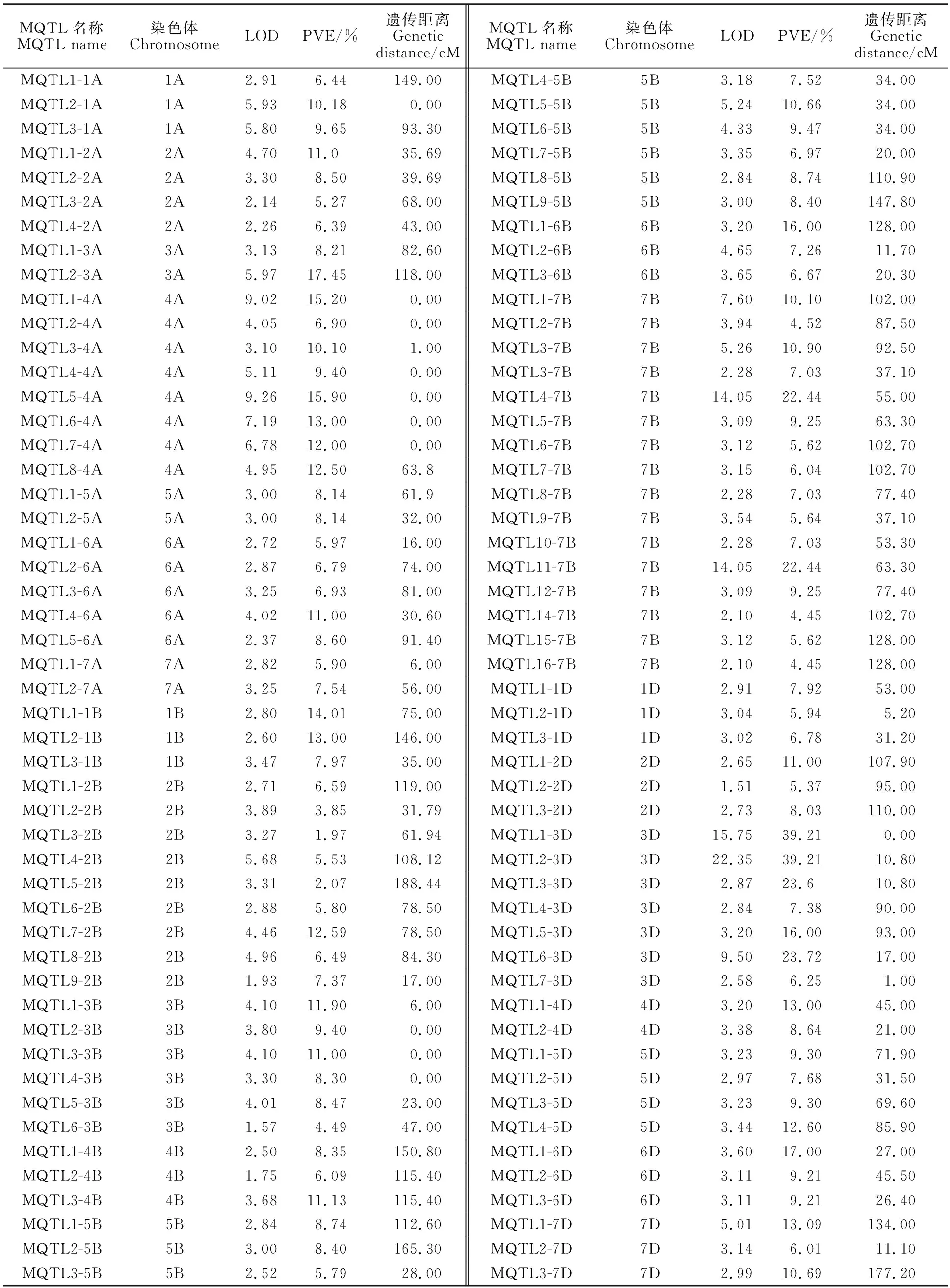
表2 小麦耐盐性QTL的元分析Table 2 Meta-analysis of QTL for salt tolerance in wheat
2.2 小麦耐盐性QTL的元分析结果
利用BioMercator v4.2.3软件对耐盐性QTL进行元分析,以5个模型中AIC值最小为准,确定其MQTL位置,最终将100个耐盐性MQTL映射到小麦21条染色体上(表2)。每条染色体分布的MQTL数目不同,平均每条染色体分布4.7个MQTL。有85个MQTL映射到构建的一致性图谱上,其中在4A染色体上分布的MQTL数目最多,有8个;在6D染色体上分布的MQTL数目最少,只有1个。在4A染色体Xgwm219~Xbarc78标记区间(包含7个MQTL)、3B染色体Xwpt-800213~Xwpt-9432标记区间(包含5个MQTL)和7B染色体Xbarc176~Xwgp45标记区间(包含6个MQTL)分布的MQTL存在重叠区域,即耐盐性QTL热点区域(图1),这些热点区域可能含有控制小麦耐盐性的重要基因。

染色体右侧的竖线表示QTL的置信区间。竖线上方的数字表示MQTL编号。The vertical line on the right side of chromosome means the confidence inderval of QTL. The number above vertical line means the code of MQTL.图1 小麦耐盐性QTL的一致性图谱Fig.1 Consensus map of QTL for salt tolerance in wheat
3 讨论
3.1 参考图谱的选择
元分析中最重要的是参考图谱的选择。在小麦相关性状元分析中最常用的是以Wheat composite 2004为参考图谱,该图谱包含SSR、AFLP、RFLP等标记。随着大量新型标记(如SNP、DArT)被广泛应用到小麦相关性状的QTL定位中,导致新的QTL无法映射到该参考图谱中。本研究在Wheat composite 2004图谱的基础上,以Maccaferri等[12]和DArT 4.0网站报道的小麦分子标记图谱作为参考,该图谱包含SNP、DArT以及SSR等标记,元分析获得的MQTL区间更小。
3.2 小麦耐盐性QTL元分析及一致性图谱的构建
张瑶尧[46]利用163个SSR标记构建F2群体的遗传连锁图谱,并对小麦苗期耐盐性QTL进行定位,结果检测到147个耐盐性QTL,其中9个为一因多效位点,LOD值为2.66~5.12,可解释2.89%~13.98%的表型变异。本研究发现的热点区域共包含18个一因多效位点,LOD值为1.51~22.35,可解释2.07%~39.21%的表型变异。杨彩凤等[21]通过QTL定位,在3A、1B和5B染色体发现了3个与根部耐盐相关的QTL。本研究结果与其结果进行比对后发现,本研究中位于3A染色体Xwmc527~Xwmc264标记区间的QTL,遗传距离从11.12 cM缩短至0.23 cM;位于1B染色体Xbarc119~Xcfe203.2标记区间的QTL,遗传距离从24.34 cM缩短至0.27 cM;位于5B染色体Xbarc36~Xgdm标记区间的QTL,遗传距离从27.84 cM缩短至20.5 cM。说明元分析能更有效的对QTL进行遗传效应解析,进而筛选耐盐相关候选基因。
4 结论
本研究收集到215个小麦耐盐QTL,将整理的QTL信息映射到参考图谱上,最终映射到100个MQTL,发现了3个与耐盐相关的MQTL热点区域。本研究为小麦耐盐新基因甄别及其精细定位提供分子依据。
