甘蔗栽培种单倍体基因组SSR位点的发掘与应用
2020-03-23王恒波祁舒婷陈姝琦郭晋隆阙友雄
王恒波 祁舒婷 陈姝琦 郭晋隆 阙友雄
研究简报
甘蔗栽培种单倍体基因组SSR位点的发掘与应用
王恒波 祁舒婷 陈姝琦 郭晋隆 阙友雄*
福建农林大学农业部福建甘蔗生物学与遗传育种重点实验室 / 国家甘蔗工程技术研究中心, 福建福州 350002
甘蔗是世界上最重要的糖料作物之一, 由于尚未完全破译栽培种基因组, 导致SSR标记匮乏, 难以覆盖全基因组, 限制了甘蔗遗传研究的进展。本研究以栽培种R570的4660个BAC文库片段序列(累计总长为382 Mb, 预测到25,316个编码蛋白基因)组装成的一套甘蔗单倍体基因组的模板, 利用MISA (Microsatellite identification tool)软件, 发掘SSR位点; 并综合分析其与4种禾本科植物(高粱、玉米、水稻和二岁短柄草)SSR位点的分布特征; 选取50对以TG和AG重复基序的SSR引物, 分别利用4个甘蔗属材料(R570、ROC1、LA purple和SES208)和24个重要甘蔗亲本, 对SSR引物进行扩增效率验证和多态性分析。共发掘到27,241个SSR位点, 平均每个BAC片段有6.29个SSR位点, 平均密度为71.33个SSR Mb-1, 远低于高粱的平均密度(350.00个SSR Mb-1)。在重复基序中, 占比前2位的分别为单核苷酸基序(11,079个)和三核苷酸重复基序(6447个), 合计占总SSR位点数的64.33%。与甘蔗不同的是, 4种禾本科植物中的三核苷酸基序类型数量最多、占比最大。此外, 在单核苷酸重复基序中, A/T所占比例最高, 为84.8%, C/G所占比例最低, 为15.2%; 在三核苷酸重复基序中, TGT/ACA所占比例最高, 为16.04%。总之, 禾本科植物基因组富含A/T的基序。在50对SSR引物(TG基序41对和AG基序9对)的多态性验证中, 共有45对(90%)能够扩增出清晰的条带, 其中35对(70%)在4个甘蔗材料上呈现多态性。进一步利用20对多态性较高的SSR引物对24个甘蔗重要亲本材料进行分析, 共扩增到95个等位基因, 平均每对引物扩增4.75个, 验证了这些引物应用于甘蔗遗传多样性研究的可行性。本研究鉴定的甘蔗栽培种单倍体基因组SSR标记, 有效增加了甘蔗遗传研究中可用的分子标记数量, 可直接用于甘蔗群体遗传多样性分析和重要性状遗传机制的解析, 为甘蔗分子育种的深入研究奠定了基础。
甘蔗栽培种; BAC 文库; SSR; 标记开发; 多态性
甘蔗(spp. hybrid)是人类最早利用的C4植物, 是世界上最重要的糖料作物, 其食糖占世界总产的80%, 同时也是一种重要生物能源作物, 其生物乙醇产量占世界总产的40%[1]。甘蔗是一种具有适应性强、生物量高、光合效率高、可连续多年种植及CO2补偿点低的糖料作物。同时, 甘蔗是世界上生物量最大的作物之一, 2017年甘蔗产量已经超过玉米, 位居世界第三[2]。
1887年, 在爪哇和西印度巴巴多斯试验场发现甘蔗种子可以产生幼苗, 开启了甘蔗有性杂交的历史[3]。现代甘蔗栽培种是由甘蔗祖先热带种(L., 2= 80,= 10)和割手密种(L., 2= 40~128,= 8)杂交产生的真正意义上的甘蔗杂种, 为了恢复高含糖量性状, 将杂种后代与热带种回交1次, 母本性状通过2染色体传递给后代, 由于种间杂交和非孟德尔的遗传方式(2+), 造成杂种后代具有高度杂合、多倍体和非整倍体性, 染色体数目在2= 100~130之间[4]。鉴于甘蔗栽培种的多倍及非整倍体的遗传背景, 其复杂性超过了大多数作物, 使相关的遗传研究、育种及基因组测序都面临极大的困难[5-6]。
简单重复序列(simple sequence repeats, SSR)具有高度的多态性、广泛分布于真核生物的基因组[5-6], 且分布随机[7], 但更偏向于低重复、富含基因的区域[8]。SSR位点产生于DNA复制和修复时, DNA聚合酶滑动或不均等重组[9], 因而可以根据在种内或种间产生大量的长度变异[10], 开发和筛选出多态性高、重复性好的SSR分子标记, 进而广泛应用于各种动、植物的品种指纹图谱鉴定[11]、遗传多样性分析[12-14]、遗传图谱构建[15-17]及重要性状(基因)的遗传定位或解析[18]等领域。然而, 相对于其他禾本科植物等模式作物, 甘蔗SSR分子标记开发及遗传连锁图谱的构建都比较落后, 相关的国内外报道较少。Singh等[13]从4085个EST序列中鉴定出351个EST-SSRs, 验证后发现134个有多态性。Shamshad等[19]从NCBI数据库中获得10,000个EST序列, 鉴定出406个SSRs, 验证了63个后发现42个具有多态性。Oliveira等[14]从甘蔗EST数据库中鉴定出2005个SSRs, 验证了342个, 其中224个(65.5%)呈多态性。甘蔗为同源多倍体作物, 染色体数目多(100~130), 遗传背景复杂, 基因组庞大(约10 Gb)[20], 大规模开发甘蔗基因组SSR标记面临很大的困难, 严重制约了甘蔗分子遗传研究相关工作的进展[16]。迄今, 与其他禾本科植物(如高粱、小麦、大麦、水稻、藜麦、玉米、二穗短柄草)相比, 甘蔗已开发的SSR标记数量少、多态性低, 难以满足甘蔗分子标记辅助育种和遗传作图等工作的要求。
甘蔗和高粱同源染色体片段(BAC)的比较分析表明, 二者存在较高的基因共线性和序列保守性, 且甘蔗单倍体基因组大小约为800~900 Mb, 接近高粱基因组大小(750 Mb)[20]。普遍认为, 一个单倍型序列可为其他单倍型同源染色体提供较好的参考。目前, 甘蔗栽培种R570具有一个103,296个克隆的BAC文库, 它代表14×单倍体基因组覆盖率和1.3×R570全基因组覆盖度[21], 已被广泛应用于比较基因组分析[22-24]。Garsmeur等[25]用全基因组分析(WGP)技术将甘蔗栽培种R570的BAC与高粱基因组比对, 确定了一个由4660个甘蔗BAC文库片段组成的覆盖甘蔗单倍体基因组常染色质的BAC的最小标记路径(MTP), 并完成了甘蔗栽培种R570的单倍体基因组测定。目前, 甘蔗栽培种全基因组SSR鉴定尚未报道。本研究旨在分析和验证甘蔗栽培种R570单倍体基因组数据, 利用生物信息学方法发掘SSR位点的特征及其分布规律, 并设计和合成SSR引物, 验证其多态性, 为甘蔗栽培种的分子指纹图谱构建、品种间遗传多样性分析、重要农艺性状的遗传机制研究及分子育种的推进奠定一定的基础。
1 材料与方法
1.1 材料
甘蔗属包括1个割手密种、1个热带种及世界各国培育的甘蔗栽培种, 都来自于广州甘蔗糖业研究所海南甘蔗育种场(表1)。

表1 甘蔗品种资源名称和来源
1.2 基因组序列的来源
通过EMBL-欧洲生物信息学研究所的公共数据库获得甘蔗栽培种R570基因组数据(登录号为ERZ654945), 或者也可以从法国农业研究所甘蔗基因组中心(http:// sugarcane-genome.cirad.fr/)直接获得。其他4种禾本科植物SSR位点序列特征来自郑燕等[26]分析结果。
1.3 SSR位点的查找与SSR引物的开发
应用MISA (Microsatellite identification tool)软件扫描甘蔗栽培种R570的基因组BAC序列[27], 该软件下载自http://pgrc.ipk-gatersleben.de/misa/, 在配置文件中设置核苷酸重复基序(motif)分别为单(mononucleotide repeats MDRs)、二(dinucleotide repeats DNRs)、三(trinucleotide repeats TNRs)、四(tetranucleotide repeats TtNRs)、五(pentanucleotide repeats PNRs)、六(hexanucleotide repeats HNRs), 序列长度分别为10、12、15、16、15、18。对SSR位点两侧各截取200 bp序列设计引物, 借助MISA软件提供的与Primer3的接口工具, 把MISA识别出来的SSR序列转为Primer3需要的格式。用Primer3 (http://frodo.wi.mit.edu/primer3/)在线设计引物, 引物设计参数为primer length: 18~28 bp; annealing temperature: 55~65℃; amplicon size: 100~500 bp; GC content: 45%~65%[13]。
1.4 PCR扩增和电泳分析
PCR 体系为25 μL, 包含DNA (25 ng μL–1) 2.0 μL、正反向引物(10 μmol μL–1)各0.5 μL、2×Plus Master Mix (Dye) 12.5 μL (试剂购自北京康为世纪生物科技有限公司)、9.5 μL ddH2O。鉴于引物较多, 统一使用降落PCR程序, 在T100 Thermal Cycler (Bio-Rad Research, USA)扩增仪上进行。扩增程序为94℃预变性5 min; 94℃变性30 s, 65℃退火30 s, 72℃延伸30 s, 共10个循环, 每个循环退火温度降低0.7℃; 94℃变性30 s, 55℃退火30 s, 72℃延伸30 s, 共25个循环; 最后72℃延伸7 min, 4℃保存。PCR产物经6%非变性聚丙烯酰胺凝胶电泳分离, 140 V恒压下电泳3.0 h, 染色、照相及保存, 其中染色采用电泳后泡染法, 用水和0.1 mol L-1NaCl稀释GelStain 10,000X储备试剂3300倍, 即标准染色液。GelStain染料购自北京全式金生物技术有限公司(货号: GS101-01)。
1.5 数据统计分析
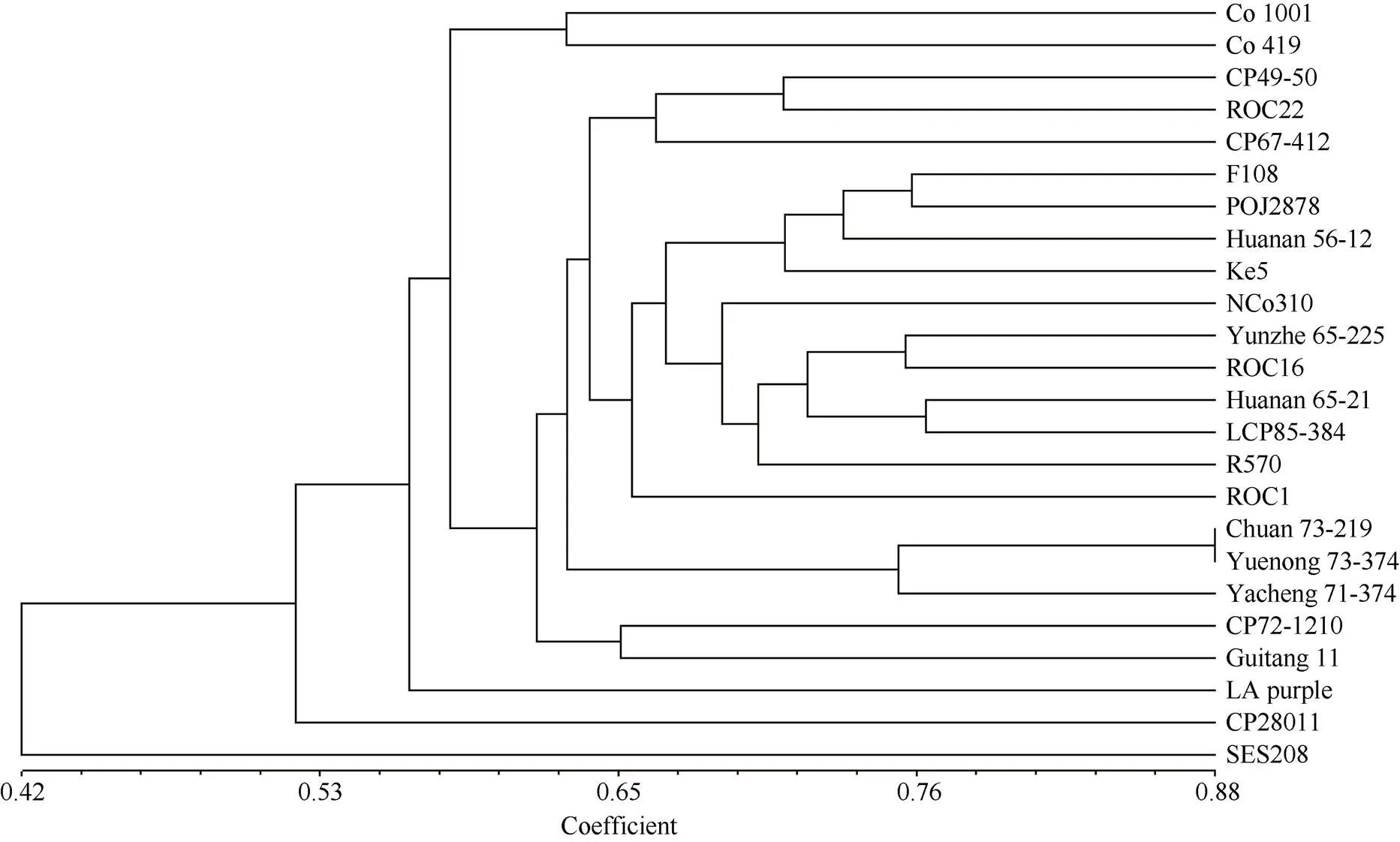
SSR频率是指基因组中出现一个SSR位点的距离, 即每若干kb出现一个SSR位点; SSR丰度是指基因组中所有SSR位点的数量之和; SSR相对丰度是指每百万个碱基中所含的SSR位点数量。按电泳扩增结果, 选择清晰的扩增条带人工读带, 在相同水平迁移位置上, 有条带的记为“1”, 没有条带的记为“0”, 缺失数据记为“-”, 根据统计的结果建立0~1矩阵。利用NTSYS-pc 2.1软件中的子程序SIMQUAL计算样品间的相似性系数(SM), 然后用子程序SAHN中的非加权平均法UPGMA (unweighted pair-group method with arithmetic means)进行聚类分析, 最后利用Tree plot绘制树状聚类图。参考Smith[28]的方法计算引物的多态性信息量(polymorphism information content, PIC)。PIC=1-∑P2, 其中P为某标记扩增的第个等位基因出现的频率, PIC的范围为0~1, 当PIC≥0.5时为高多态性引物; 0.25 利用MISA软件扫描甘蔗栽培种R570的4660个BAC文库序列(总长为382 Mb), 通过分析1~6核苷酸重复基序,设定SSR位点的长度不低于10 bp, 共找到27,241个SSR位点(表1), 平均每1.08个基因或14.01 kb含有1个SSR位点。其中单核苷酸重复基序类型出现的频率最高, 为11,079个位点, 占总数的40.67%; 三核苷酸重复基序类型次之, 为6447个位点, 占总数的23.67%, 两者合计占总数的63.33%。而二、四、五、六核苷酸类型及复合型所占的比例相对较低, 分别为11.97%、4.92%、9.32%和9.45%, 合计占总数的36.67%。 对于不同SSR核苷酸重复基序类型而言, 重复次数越少, 出现的频率越高。在1~6核苷酸重复基序类型中, 优势重复基序类型数量占比最多的重复次数都接近其筛选标准, 平均重复次数分别是11.68、11.23、6.10、4.78、3.38、3.30。在单核苷酸重复基序类型数量中, 优势重复次数最多的是10次重复, 有6297个, 占总位点数23.12%, 与三核苷酸重复基序总数基本相当, 但比二、四、五、六核苷酸重复基序的总数还多。在三核苷酸重复基序类型中, 优势重复次数最多的是5次重复, 有3718个, 占总位点数13.65%, 除了单核苷酸重复类型外, 也都超过了其他4种类型重复基序类型的总数。二、四、五、六核苷酸重复基序类型优势重复次数相对较少, 其中四核苷酸重复基序的总数最少, 其优势重复次数为4次重复, 仅有941个, 占总位点数3.45%。总体而言, 重复次数在3~10之间的总SSR位点数为21,270个, 占总位点数的78.08%。在1~6核苷酸重复基序类型中, 优势重复次数占比分别从56.83%、37.09%、57.67%、70.17%、80.54%和81.74%, 除了二核苷酸重复基序较低外, 其他基序类型呈现出逐步递升趋势。此外, 在设计SSR引物成功率上, 随着核苷酸重复基序类型的增多和优势重复次数的降低, 设计到符合标准的SSR引物的成功率在逐渐下降。 表2 甘蔗栽培种R570基因组上各类核苷酸重复基序分布特征信息 选取了4种具有代表性的禾本科植物(高粱、玉米、水稻、二穗短柄草)和甘蔗比较, 它们的基因组变化范围在272 Mb (二穗短柄草)和2061 Mb (玉米)之间(表3)。SSR数量与基因组大小存在极显著的正相关(= 0.92;< 0.01), 但SSR的相对丰度和频率与基因组大小没有相关性。在1~6核苷酸重复基序中, 水稻SSR位点相对丰度都是最高的, 其次是二穗短柄草(除二和六核苷酸重复基序外), 而甘蔗的SSR相对丰度基本都是最低的(除单核苷酸重复基序外)。总的相对丰度从高到低分别为水稻(566.45)、二穗短柄草(361.15)、高粱(350.00)、玉米(152.54)、甘蔗(71.33), SSR位点分布在禾本科植物中存在丰富的多样性。同时, SSR出现的频率也与SSR数量成正比, 频率从高到低呈现相同的变化趋势。 5种禾本科植物的6种不同核苷酸重复基序的SSR的相对数量(丰度)比较(表3)表明, 除甘蔗的单核苷酸外, 其他物种都表现出三核苷酸的相对数量(丰度)最多, 六核苷酸重复基序次之, 且都是玉米的SSR数量最高, 水稻和甘蔗最少, 而相对数量(丰度)上水稻最高, 玉米和甘蔗最低。 甘蔗和高粱同属于禾本科黍亚科, 两者大约在八至九百万年前由共同祖先分化而来, 且高粱进化相对较慢, 保持了其祖先相对完整的基因组组成[22]。甘蔗和玉米是5个禾本科物种中基因组最大的2个, 因此, 也最有可能出现长SSR序列。从表4可以看出, 所有物种的1~6核苷酸重复基序中, 前3种最长的SSR基序类型基本都是A/T碱基组成的重复基序类型, 而不是C/G重复类型。在单核苷酸重复和二核苷酸重复基序类型中, 最长的核苷酸重复A(88)和AC(910)均出现在玉米的基因组内, 而甘蔗和水稻相对都是最低的。在三核苷酸重复类型中, 最长的核苷酸重复(TGT)369和(ACT)366分别出现在甘蔗和高粱中, 水稻中则最低。对于四核苷酸到六核苷酸重复基序, 长的SSR序列都出现在高粱中, 甘蔗次之。同时, 5个禾本科植物都有一个最长ACAT重复基序, 其余类型除了有1个C/G外, 都由A/T组成。在所有的五核苷酸重复类型中, AATAT重复基序最多, 占到9个, 其余类型只含有1~3个G/C, 说明大多数长的五核苷酸重复类型都是由A/T组成。在六核苷酸重复类型中, 玉米、水稻和二穗短柄草的重复序列长度都远远低于高粱和甘蔗。 表3 5种禾本科植物中1~6核苷酸重复基序类型的SSR数量和相对丰度 表4 5种禾本科植物中前3种最长SSR基序类型 在甘蔗栽培种基因组中, 单、二、三核苷酸重复基序占比达到76.31%以上, 因此分析其优势重复基序和各种类型SSR序列的碱基组成, 对于进一步筛选和验证多态性SSR引物具有重要作用。如图1所示, 从基序结构出现的频率上看, A/T和G/C占各占84.78%和15.22%, 分别是甘蔗栽培种基因组 SSR 中单核苷酸出现频率最高和最低的结构; AT/TA(31.51%)和CG/GC(6.32%)分别是二核苷酸中出现最多和最少的基序结构; 三核苷酸出现频率最多的结构是TGT/ACA(16.04%), 其次是CGC/GCG、CCG/GGC和GCC/CGG基序结构类型, 分别占12.02%、11.31%、10.67%, 合计占34.00%; 最少的则是TCA/AGT, 占0.35%。甘蔗和4种禾本科植物SSR基序都有碱基的偏好性和规律, 在单核苷酸中, 甘蔗、高粱和水稻A/T的比例也都高于G/C的比例, 分别为84.78%、65.60%和64.40%, 但在玉米和二穗短柄草的结果完全相反; 在二核苷酸中, 甘蔗(31.51%)和其他4个物种(22.6%~54.2%)都是AT/TA重复比例最高, 而GC/CG重复的比例最低, 基本都在3.1%~6.6%之间; 在三核苷酸类型中, CGC/GCG、CCG/GGC和GCC/CGG三者合计在所有物种中都最高, 且以水稻(44.75%)最高, 甘蔗(34.0%)次之, 玉米(12.74%)最低[26]。 根据Pan[12]筛选到21对多态性丰富的SSR引物对应的重复基序类型, 它们以TG和AG重复基序为主, 本研究选择以TG和AG基序类型以及重复次数分别在TG(11~69)、AG(23~38)之间的SSR位点, 对其进行引物设计和合成, 共计50对SSR引物, 对4个不同甘蔗属材料(栽培种R570, 栽培种ROC1, 热带种LA purple 和割手密种SES208)进行SSR扩增和多态性筛选。共有45对引物能够扩增出清晰的扩增条带, 其余的5对引物没有扩增条带或者扩增产物量较弱, 其中35对引物在4个材料上呈现多态性(表5), 多态率为70% (35/50), 其中TG重复类型的引物有28对, AG重复类型的引物有7对。图2显示了部分引物筛选扩增结果。为了进一步验证本研究鉴定到SSR引物的多态性, 选用20对多态性较高的SSR引物, 对我国50年来育成163个甘蔗品种的18个骨干亲本(它们的血缘来自热带种、割手密种、大茎野生种和印度种的2~4个种, 且具有较低的共祖系数)、2个甘蔗祖先种(割手密种SES 208和热带种LA purple)和4个重要甘蔗栽培种(LCP85-384、R570、ROC16和ROC22)进行遗传多样性分析。结果20对引物在24个甘蔗实验材料上呈现多态性, 共扩增得到等位基因95个, 每对扩增出1~7个等位基因(表5), 平均每一对引物扩增出4.75个等位基因。图3展示了其中FAFUR-S22引物在24个供试甘蔗材料上的PCR扩增电泳图谱。 图1 1~3核苷酸重复基序类型及数量 图2 7对不同SSR引物在4个甘蔗属材料上扩增的电泳图 1~4: FAFUR-S44; 5~8: FAFUR-S45; 9~12: FAFUR-S46; 13~16: FAFUR-S47; 17~20: FAFUR-S48; 21~24: FAFUR-S49; 25~28: FAFUR-S50。4个扩增产物为SES208 (1, 5, 9, 13, 17, 21, 25)、LA purple (2, 6, 10, 14, 18, 22, 26)、ROC16 (3, 7, 11, 15, 19, 23, 27)和R570 (4, 8, 12, 16, 20, 24, 28); M: 50 bp DNA ladder (3421A)。 1–4: FAFUR-S44; 5–8: FAFUR-S45; 9–12: FAFUR-S46; 13–16: FAFUR-S47; 17–20: FAFUR-S48; 21–24: FAFUR-S49; 25–28: FAFUR-S50. The amplification products of four samples were SES208 (1, 5, 9, 13, 17, 21, 25), LA purple (2, 6, 10, 14, 18, 22, 26), ROC16 (3, 7, 11, 15, 19, 23, 27), and R570 (4, 8, 12, 16, 20, 24, 28). M: 50 bp DNA ladder (3421A). 基于上述20对SSR引物所给出的95种等位基因类型, 对24份甘蔗属材料的UPGMA聚类分析, 供试材料之间的遗传相似系数分布在0.40~0.82之间(图4), 在遗传相似性系数为0.525时, 可将24个甘蔗材料分成5种类型, 第1种类型包含甘蔗栽培种Co 1001和Co 419; 第2种类型有19个甘蔗材料; 第3种类型为1个甘蔗材料热带种类型LA purple; 第4类型为1个甘蔗栽培种材料CP28-11; 第5种类型为1个割手密种SES 208。需要注意的是, 在相似性系数为0.4时, 割手密种SES208与其他甘蔗栽培种和热带种(LA purple)较早分开, 表明割手密种与甘蔗栽培种具有较远的遗传关系。根据张琼等[29]分析结果, CP28-11具有热带种(0.5)、割手密(0.125)和印度种(0.375)的血缘关系, 遗传关系介于割手密和热带种之间。本研究中, 热带种(LA purple)在相似性系数为0.525时与其他栽培种分开, 接着是印度种Co 1001和Co 419在相似性系数为0.551时与其他栽培种分开, 表明印度种亲缘关系介于热带种与甘蔗栽培种之间, 也具有较丰富的遗传多样性。 图3 SSR引物(FAFUR-S22)在24个甘蔗材料上扩增的电泳图 1: Co 1001; 2: Co 419; 3: CP28-11; 4: CP49-50; 5: CP67-412; 6: CP72-1210; 7: F108; 8: NCo310; 9: ROC1; 10: 川73-219; 11: 桂糖11号; 12: 华南56-12; 13: POJ2878; 14: 科5; 15: 崖城71-374; 16: 粤农73-204; 17: 云蔗65-225; 18: 华南56-21; 19: LCP85-384; 20: R570; 21: ROC16; 22: ROC22; 23: LA purple; 24: SES208; M: 50 bp DNA ladder (3421A)。 1: Co 1001; 2: Co 419; 3: CP28-11; 4: CP49-50; 5: CP67-412; 6: CP72-1210; 7: F108; 8: NCo310; 9: ROC1; 10: C73-219; 11: Guitang 11; 12: Huanan 56-12; 13: POJ2878; 14: Ke 5; 15: Yacheng 71-374; 16: Yuenong 73-204; 17: Yunzhe 65-225; 18: Huanan 56-21; 19: LCP85-384; 20: R570; 21: ROC16; 22: ROC22; 23: LA purple; 24: SES208; M: 50 bp DNA ladder (3421A). 表5 具有扩增多态性的甘蔗SSR引物信息表 (续表5) #表示引物对的多态性相对较低。#indicates that the primer pairs have relatively low polymorphism. 图4 基于SSR分子标记的24份甘蔗属材料的UPGMA聚类分析 SSR分子标记具有多态性高、重复性好、操作简便及共显性等优点, 被广泛应用于甘蔗指纹图谱构建[11]、遗传多样性分析[12-14]、遗传连锁图谱构建及QTL定位等方面[15-18]。但目前尚未完全破译甘蔗栽培种基因组, 对于SSR标记开发仅仅停留在BAC文库和表达序列标签(EST)水平。本研究首次利用已经完成的甘蔗栽培种单倍体基因组数据, 分析SSR在基因组分布频率、基序类型及序列特征等相关信息, 对于甘蔗基因组遗传研究和分子标记开发提供重要数据支撑。 本研究从甘蔗栽培种R570的4660个BAC文库序列组装的单倍体基因组(累计总长为382 Mb, 预测到25,316个编码蛋白基因)中, 共发现27,241个SSR位点, 在基因组中1~6核苷酸重复基序中, 单核苷酸重复基序占比最高, 达到40.67%, 其次是三核苷酸(占23.67%)、二核苷酸重复基序(占11.97%)。这与拟南芥[30]、水稻[30]、小麦[31]、玉米[3]、棉花[31]、马铃薯[32]、苹果[15]和葡萄[33]的基因组SSR位点中存在大量的单、二、三核苷酸的结果基本一致; 与高粱[26,34]、根瘤菌和秀丽线虫[35]的研究结果不同, 它们的基因组SSR位点以四、五、六核苷酸重复基序为主。一般情况下, 短重复基序占多数表明物种进化水平相对较高[36], 而长重复基序占多数的物种具有较低的突变频率或较短的进化时间[37]。甘蔗与高粱在八至九百万年前由共同祖先分化而来, 高粱进化相对较慢, 保持了相对完整的祖先基因组构成[22]。本研究结果进一步说明, 与高粱相比, 甘蔗在进化与分类地位中处于相对较高水平, 其基因组也经历了较长的进化时间或具有较高的突变频率。但是, 甘蔗栽培种基因组遗传基础复杂、杂合度高, 尚未完成基因组测序, 本研究仅依据高粱基因组为参考, 利用BAC文库序列拼装一套甘蔗单倍体基因组作为SSR挖掘的模板[25], 基因组数据并不完整。因此, 分析结果尚无法与其他物种上的SSR数据比较, 但是也初步证明了与大多数禾本科植物具有相似的核苷酸基序组成。 在不同物种的SSR位点信息特征研究方面, SSR基序的结构组成, 尤其是1~3核苷酸重复基序具有明显的偏好性和规律性, 但4~6核苷酸重复基序, 由于基序类型呈现指数上升(分别是2,521,020和4092), 碱基偏好性和规律性表现的不明显。本研究结果显示, 甘蔗栽培种基因组序列中, 单核苷酸重复基序类型以A/T出现的次数最多(占84.78%), 这与水稻、高粱[26]、苹果[15]和烟草[38]的研究结果一致, 但是与二穗短柄草、玉米G/C基序结构出现次数不一致[26]; 而二核苷酸重复基序类型则以AT/TA出现的次数最多(占31.51%), CG/GC(占6.32%)最少, 该结果与高粱、烟草、苹果的研究结果一致, 但是在其他禾本科植物小麦[39]、二穗短柄草、水稻和玉米中不一致[26], 它们是以AG重复类型最多; 三核苷酸重复基序类型以TGT/ACA (16.04%)出现的次数最多(占16.04%), 其次是含有CGC/GCG等6类不同组合基序类型(合计占34.0%), 与拟南芥(AAG/TTC)、烟草(AAC/TTG)、苹果(AAC/GTT)基本相似, 与大麦、小麦、玉米、水稻、高粱、黑麦等其他禾本科作物有所不同, 它们以CCG/GCC出现次数最多, 而ATT/TAA最少。综合以上, 甘蔗的1~3核苷酸重复基序均以A和T核苷酸构成的基序为主要类型, 这与真核生物基因组SSR位点分析结果基本一致[36], 产生这样的结果有2种可能原因, 一是甲基化的C残基转变为T[40]; 二是在DNA复制过程中产生SSR位点, 基于A、T结构类型基序比G、C类型需要较少能量, 导致富含A、T碱基结构类型比G、C类型容易产生[38], 但是随着核苷酸重复类型从3增至6时, 这种氢键的能量优势就相对不明显了。 近年来, 随着基因组测序技术进步和测序成本的降低, 已经完成许多生物基因组测序, 这些测序结果对于相应物种基因组SSR标记开发和应用提供了数据支撑, 还可以利用所开发的SSR标记对未测序的近缘物种进行遗传分析, 加快其群体结构分析、遗传多样性研究、亲缘关系鉴定、重要性状的功能基因QTL定位及关联标记开发与应用。本研究通过分析Pan等[12]筛选和鉴定的21对多态性丰富的SSR引物的重复基序类型, 选择了50对SSR引物, 它们由TG(41对)和AG(9对)基序重复类型组成。此外, 利用4个甘蔗属材料进行PCR扩增和多态性分析, 共有45对引物扩增到预期的扩增片段, 其中TG和AG基序类型的分别有37对(扩增效率90.24%)和8对, 扩增效率为88.89%, 远高于Cordeiro等[41]开发甘蔗EST-SSR引物的60%, 也高于Oliveira等[14]开发的甘蔗基因组SSR的70%、Bushman等[42]针对多年生黑麦草开发的基因组和EST-SSR的75 %、Fernandez等[43]获得的甜瓜基因组和EST的79 %和Hwang等[44]发掘的西瓜EST-SSR的79%。此外, 在我们设计的50对SSR引物中, 有35对呈现出明显的多态性, TG和AG基序类型分别有28对和7对, 且AG基序的多态性(77.78%)高于TG类型(68.29%)。 本研究还在对SSR标记初步扩增分析的基础上, 进一步利用24个甘蔗重要亲本材料验证上述20对SSR引物的多态性。结果20对引物扩增出95种不同等位基因, 每对引物扩增1~8个等位基因, 平均每对引物扩增4.75个。与Pinto等[45]的6.04个、Oliveira等[14]的7.55个、Marconi等[46]的6.0个相比, 本研究开发的甘蔗SSR引物扩增出的等位基因偏少, 主要原因应该是不同研究所采用的电泳分离方法存在差异, 当将本研究鉴定的SSR引物应用于甘蔗遗传研究时, 建议进一步优化PCR扩增条件, 同时采用分辨率更高的电泳分离方法, 比如变性聚丙烯酰胺凝胶电泳、毛细管电泳等, 以便更准确和高效地对目标材料进行扩增和多态性分析。同时, 20对SSR引物扩增的多态性信息量(PIC)的变异幅度在0.61~0.92之间, 平均值为0.78, 说明本研究所筛选和鉴定的SSR引物所扩增出来的条带多态性丰富。特别需要指出的是, 川73-219和粤农73-204在相似性系数为0.9时没有分开, 说明两者亲缘关系非常近, 需要用更多SSR引物鉴定和分析。 综上所述, 本研究利用大小约为382 Mb的甘蔗栽培种R570的单倍体基因组, 开发了27,241个SSR位点, 平均每BAC片段有6.29个。通过分析和比较其他物种基因组序列上SSR位点、类型及结构等特征, 设计了50对SSR引物, 分别利用4个甘蔗属近缘种(R570、ROC1、LA purple和SES 208)和24个甘蔗重要杂交亲本材料, 进行扩增效率和多态性分析, 获得了20对引物扩增条带清晰、多态性条带比率高, 具有较好应用前景的SSR标记。这20对引物在24个甘蔗杂交亲本中扩增出95种不同等位基因, 每对引物扩增1~8个等位基因之间, 平均为4.75个。本研究建立的SSR引物筛选和鉴定方法, 对于开发高质量多态性SSR标记具有积极的借鉴作用; 所开发的20对具有良好多态性的SSR标记, 能够为甘蔗及其近缘种的品系鉴定、遗传多样性分析、遗传图谱构建及重要性状的遗传机制解析等提供了分子标记支撑。 [1] 刘燕群, 李玉萍, 梁伟红, 宋启道, 秦小立, 叶露. 国外甘蔗产业发展现状.世界农业, 2015, (8): 147–152. Liu Y Q, Li Y P, Liang W H, Song Q D, Qin X L, Ye L. Current situation of sugarcane industr in the world,, 2015, (8): 147–152 (in Chinese with English abstract). [2] FAOSTAT: http://www.fao.org/faostat/zh/#data?tdsourcetag=s_ pctim_aiomsg. [3] 彭绍光. 甘蔗育种学. 北京. 农业出版社, 1990. pp 4–5. Peng S G. Sugarcane Genetic Breeding. Beijing: Agricultural Press, 1990. pp 4–5 (in Chinese). [4] Hermann S R, Aitken K S, Jackson P A, George A W, Piperidis N, Wei X, Kilian A, Detering F. Evidence for second division restitution as the basis for 2+maternal chromosome transmission in a sugarcane cross.,2012, 187: 359–368. [5] Piperidis G, Piperidis N, D’Hont A. Molecular cytogenetic investigation of chromosome composition and transmission in sugarcane., 2010, 284: 65–73. [6] Wang H B, Chen P H, Yang Y Q, D'Hont A, Lu Y H. Molecular insights into the origin of the brown rust resistance geneamongspecies.,2017, 130: 2431–2443. [7] Smith D, Devey M E. Occurrence and inheritance of microsatellites in., 1994, 37: 977–983. [8] Morgante M, Hanafey M, Powell W. Microsatellites are preferentially associated with nonrepetitive DNA in plant genomes.,2002, 30: 194–200. [9] Levinson G, Gutman G A. Slipped-strand mispairing: a major mechanism for DNA sequence evolution., 1987, 4: 203–221. [10] Michael K, Eva-Maria D, Ugo R, Nicoletta C, Uta-Dorothee I, Manfred K, Wolfgang R M. Haplotype studies support slippage as the mechanism of germline mutations in short tandem repeats., 2004, 25: 3344–3348. [11] Liu X L, Ma L, Chen X K, Ying X M, Cai Q, Liu J Y, Wu C W. Establishment of DNA fingerprint identity for sugarcane cultivars in Yunnan, China.2010, 36: 202–210. [12] Pan Y B. Highly polymorphic microsatellite DNA markers for sugarcane germplasm evaluation and variety identity testing.,2006,8: 246–256. [13] Ram K S, Satya N J, Suhail K, Sonia Y, Nandita B, Saurabh R, Vasudha B, Sanjay K D, Raman K, Sushil SDevelopment, cross-species/genera transferability of novel EST-SSR markers and their utility in revealing population structure and genetic diversity in sugarcane.,2013,524: 309–329. [14] Oliveira K M, Pinto L R, Marconi T G, Mollinari M, Ulian E C, Chabregas S M, Falco M C, Burnquist W, Garcia A A, Souza A P. Characterization of new polymorphic functional markers for sugarcane.,2009, 52: 191–209. [15] 关玲, 章镇, 王新卫, 薛华柏, 刘艳红, 王三红, 乔玉山. 苹果基因组SSR位点分析与应用. 中国农业科学,2011, 44: 4415–4428. Guan L, Zhang Z, Wang X W, Xue H B, Liu Y H, Wang S H, Qiao Y S. Evaluation and application of the SSR loci in apple genome., 2011, 44: 4415–4428 (in Chinese with English abstract). [16] 刘新龙, 毛钧, 陆鑫, 马丽, Karen S A, Jackson P A, 蔡青, 范源洪. 甘蔗SSR和AFLP分子遗传连锁图谱构建. 作物学报, 2010, 36: 177–183. Liu X L, Mao J, Lu X, Ma L, Karen S A, Jackson P A, Cai Q, Fan Y H. Construction of molecular genetic linkage map of sugarcane based on SSR and AFLP markers., 2010, 36: 177–183 (in Chinese with English abstract). [17] Andru S, Pan Y B, Thongthawee S, Burner D M, Kimbeng C A. Genetic analysis of the sugarcane (spp.) cultivar ‘LCP 85-384’: I. linkage mapping using AFLP, SSR, and TRAP markers., 2011, 123: 77–93. [18] Yang X, Islam M S, Sood S, Maya S, Hanson E A, Comstock J, Wang J. Identifying quantitative trait loci (QTLs) and developing diagnostic markers linked to orange rust resistance in sugarcane (spp.)., 2018, 9: 350. [19] Shamshad U H, Kumar P, Singh R K, Verma K S, Bhatt R, Sharma M, Kachhwaha S, Kothari S L. Assessment of functional EST-SSR markers (Sugarcane) in cross-species transferability, genetic diversity among Poaceae plants, and bulk segregation analysis., 2016, 2016: 7052323. [20] Jianping W, Bruce R, Simone M, Qingyi Y, Jan E M, Haibao T, Cuixia C, Fares N, Graham W, John BMicrocollinearity between autopolyploid sugarcane and diploid sorghum genomes., 2010, 11: 261. [21] Laurent G, Paulo A. Sugarcane genomics: depicting the complex genome of an important tropical crop., 2002, 5: 122–127. [22] Jannoo N, Grivet L, Chantret N, Garsmeur O, Glaszmann J C, Arruda P, D’Hont A. Orthologous comparison in a gene-rich region among grasses reveals stability in the sugarcane polyploid genome., 2007, 50: 574–585. [23] Paterson A H, Bowers J E, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov AThe Sorghum bicolor genome and the diversification of grasses.,2009, 457: 551–556. [24] Garsmeur O, Charron C, Bocs S, Jouffe V, Samain S, Couloux A, Droc G, Zini C, Glaszmann J, Van Sluys MHigh homologous gene conservation despite extreme autopolyploid redundancy in sugarcane., 2011, 189: 629–642. [25] Garsmeur O, Droc G, Antonise R, Grimwood J, Potier B, Aitken K, Jenkins J, Martin G, Charron C, Hervouet CA mosaic monoploid reference sequence for the highly complex genome of sugarcane.,2018, 9: 2638–2638. [26] 郑燕, 张耿, 吴为人. 禾本科植物微卫星序列的特征分析和比较. 基因组学与应用生物学, 2011, 30: 513–520. Zheng Y, Zhang Z, Wu W R. Characterization and comparison of microsatellites in Gramineae., 2011, 30: 513–520 (in Chinese with English abstract). [27] Martins W, de Sousa D, Proite K, Guimarães P, Moretzsohn M, Bertioli D. New softwares for automated microsatellite marker development.,2006, 34: e31. [28] Smith J S, Chin E C, Shu H, Smith O S, Wall S J, Senior M L, Mitchell S E, Kresovich S, Ziegle J. An evaluation of the utility of SSR loci as molecular markers in maize (L.): comparisons with data from RFLPS and pedigree.,1997, 95: 163–173. [29] 张琼, 齐永文, 张垂明, 陈勇生, 邓海华. 我国大陆甘蔗骨干亲本亲缘关系分析.广东农业科学, 2009, (10): 44–48. Zhang Q, Qi Y W, Zhang T M, Chen Y S, Deng H H. Pedigree analysis of genetic relationship among core parents of sugarcane in mainland China., 2009, (10): 44–48 (in Chinese with English abstract). [30] Lawson M J, Zhang L. Distinct patterns of SSR distribution in theand rice genomes.,2006, 7: R14. [31] Kantety R V, La Rota M, Matthews D E, Sorrells M E. Data mining for simple sequence repeats in expressed sequence tags from barley, maize, rice, sorghum and wheat., 2002, 48: 501–510. [32] Tang J, Baldwin S J, Jacobs J M, Linden C G, Voorrips R E, Leunissen J A, van Eck H, Vosman B. Large-scale identification of polymorphic microsatellites using an in silico approach., 2008, 9: 374. [33] 蔡斌, 李成慧, 姚泉洪, 周军, 陶建敏, 章镇. 葡萄全基因组SSR分析和数据库构建. 南京农业大学学报, 2009, 32(4): 28–32. Cai B, Li C H, Yao Q H, Zhou J, Tao J M, Zhang Z. Analysis of SSRs in grape genome and development of SSR database., 2009, 32(4): 28–32 (in Chinese with English abstract). [34] 陆景标, 李杰勤, 卢杰, 詹秋文. 高梁非编码区SSR引物设计以及e-PCR的验证. 种子, 2010, 29(9): 1–6. Lu J B, Li J Q, Lu J, Zhan Q W. Design of SSR primers and verification of e-PCR in non-coding regions of sorghum genome., 2010, 29(9): 1–6 (in Chinese with English abstract). [35] 高亚梅, 韩毅强, 汤辉, 孙东梅, 王彦杰, 王伟东. 根瘤菌基因组内简单重复序列的分析. 中国农业科学2008, 41: 2992–2998. Gao Y M, Han Y Q, Tang H, Sun D M, Wang Y J, Wang W D. Analysis of simple sequence repeats in Rhizobium genomes., 2008, 41: 2992–2998 (in Chinese with English abstract). [36] Toth G, Gaspari Z, Jurka J. Microsatellites in different eukaryotic genomes: survey and analysis., 2000, 10: 967–981. [37] Harr B, Schlotterer C. Long microsatellite alleles in Drosophila melanogaster have a downward mutation bias and short persistence times, which cause their genome-wide under representation., 2000, 155: 1213–1220. [38] 童治军, 焦芳婵, 肖炳光. 普通烟草及其祖先种基因组SSR位点分析. 中国农业科学,2015, 48: 2108–2117.Tong Z J, Jiao F C, Xiao B G. Analysis of SSR loci ingenome and its two ancestral species genome.,2015, 48: 2108–2117 (in Chinese with English abstract). [39] Yu J K, La Rota M, Kantety R V, Sorrells M E. EST derived SSR markers for comparative mapping in wheat and rice., 2004, 271: 742–751. [40] Schorderet D F, Gartler S M. Analysis of CpG suppression in methylated and nonmethylated species.,1992, 89: 957–961. [41] Cordeiro G M, Casu R, McIntyre C L, Manners J M, Henry R J. Microsatellite markers from sugarcane (spp.) ESTs cross transferable to erianthus and sorghum.,2001, 160: 1115–1123. [42] Bushman B S, Larson S R, Mott I W, Cliften P F, Wang R R, Chatterton N J, Hernandez A G, Ali S, Kim R W, Thimmapuram JDevelopment and annotation of perennial Triticeae ESTs and SSR markers.,2008, 51: 779–788. [43] Fernandez I, Eduardo I, Blanca J, Esteras C, Pico B, Nuez F, Arus P, Garcia J, Monforte A J. Bin mapping of genomic and EST-derived SSRs in melon (L.).,2008, 118: 139–150. [44] Hwang J Y, Ahn S G, Youl Oh J, Choi Y W, Kang J S, Park Y H. Functional characterization of watermelon (L.) EST-SSR by gel electrophoresis and high resolution melting analysis.,2011, 130: 715–724. [45] Pinto L R, Oliveira K M, Ulian E C, Garcia A A, de Souza A P. Survey in the sugarcane expressed sequence tag database (SUCEST) for simple sequence repeats.,2004, 47: 795–804. [46] Marconi T G, Costa E A, Miranda H R, Mancini M C, Cardoso C B, Oliveira K M, Pinto L R, Mollinari M, Garcia A A, Souza A P. Functional markers for gene mapping and genetic diversity studies in sugarcane.,2011, 4: 264. Development and application of SSR loci in monoploid reference genome of sugarcane cultivar WANG Heng-Bo, QI Shu-Ting, CHEN Shu-Qi, GUO Jin-Long, and QUE You-Xiong* Key Laboratory of Sugarcane Biology and Genetic Breeding (Fujian), Ministry of Agriculture, Fujian Agriculture and Forestry University / Sugarcane Research & Development Center, China Agricultural Technology System, Fuzhou 350002, Fujian, China Sugarcane is one of the most important sugar crops in the world. However, it is difficult to develop SSR on a large scale since the genome of cultivar has not been sequenced, which limits the genetic improvement of sugarcane. In this study, a template of monoploid sugarcane genome was assembled using a set of 4660 BAC library sequences (with a cumulative length of 382 Mb, predicting 25,316 genes) from cultivar ‘R570’. SSR loci were identified by using MISA (Microsatellite identification tool) software. The distribution characteristics of the monoploid genome ‘R570’ was comprehensively analyzed by comparing with the SSR loci of four Gramineae plants (,,, and). Fifty pairs of primers with TG and AG repeat motifs were designed to verify the amplification efficiency and polymorphism by PCR amplification in fourclones (R570, ROC1, LA purple, and SES208) and twenty four core parents of sugarcane. A total of 27,241 SSR loci were identified, with an average of 6.29 SSR loci per BAC clone and an average density of 71.33 SSR Mb–1which was much lower than that of sorghum (350.00 SSR Mb–1). The mono-nucleotide (11,079) and tri-nucleotide repeat motifs (6447) accounted for 64.33% of the total SSR loci. The number and proportion of tri-nucleotide repeat motifs were the largest in the four Gramineae plants. In addition, A/T (accounting for 84.8%) motif had the highest proportion and C/G (accounting for 15.2%) motif the lowest proportion in the mono-nucleotide repeat motifs and TGT/ACA (accounting for 16.04%) motif had the highest proportion in the trinucleotide repeat motifs. In general, the genomes in Gramineae plants are rich in A/T repeat motifs. In the polymorphism validation of 50 pairs of primers (41 pairs of TG motif and 9 pairs of AG motif), 45 pairs of primers (90%) were found to be able to amplify successfully, of which 35 (70%) were polymorphic in 4 sugarcane clones. Furthermore, 20 pairs of polymorphic SSR primers were used to detect 24 core parents of sugarcane, a total of 95 alleles were amplified with an average of 4.75 alleles per primer, verifying the application feasibility of these primers for the genetic diversity analysis in sugarcane. The development of SSR markers from the monoploid genome of cultivars ‘R570’ not only enriches the number of SSR markers available in sugarcane genetic analysis, but also facilitates the genetic diversity analysis of sugarcane population and the genetic mechanism dissection of important agronomic traits, which provides a foundation for the in-depth research of molecular breeding in sugarcane. sugarcane cultivars; bacterial artificial chromosome library; SSR; development; polymorphism 2019-09-11; 2019-12-26; 2020-01-15. 10.3724/SP.J.1006.2020.94135 阙友雄, E-mail: queyouxiong@126.com, Tel: 0591-83852547 E-mail: wanghengbo_0354@126.com, Tel: 0591-83789177 本研究由引进国际先进农业科学技术计划(948计划)项目(2014-S18), 国家现代农业产业技术体系建设专项(CARS-17)和福建农林大学校科技发展专项(KFA18025A)资助。 This study was supported by the Program of Introducing International Super Agricultural Science and Technology (948 Program) (2014-S18), the Agricultural Research System (CARS-17), and Fujian Agriculture and Forestry University Science and Technology Development Special Fund (KFA18025A). URL: http://kns.cnki.net/kcms/detail/11.1809.S.20200115.1136.020.html2 结果与分析
2.1 甘蔗栽培种SSR位点的数量、类型及分布频率

2.2 甘蔗与其他4种禾本科植物SSR位点的数量和频率特征比较
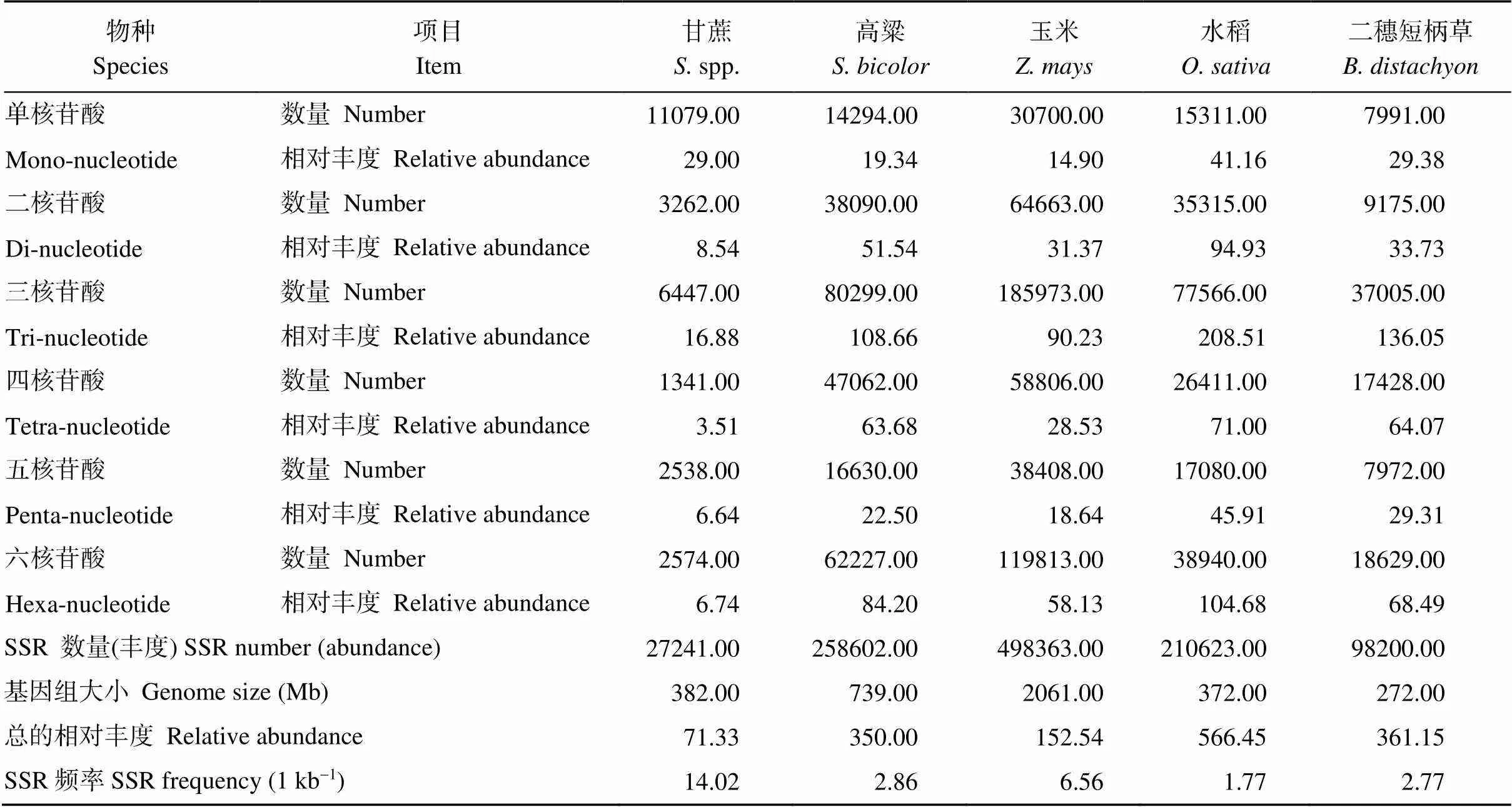

2.3 甘蔗SSR重复基序种类及频率特征分析
2.4 SSR标记的开发及其扩增效率和多态性验证
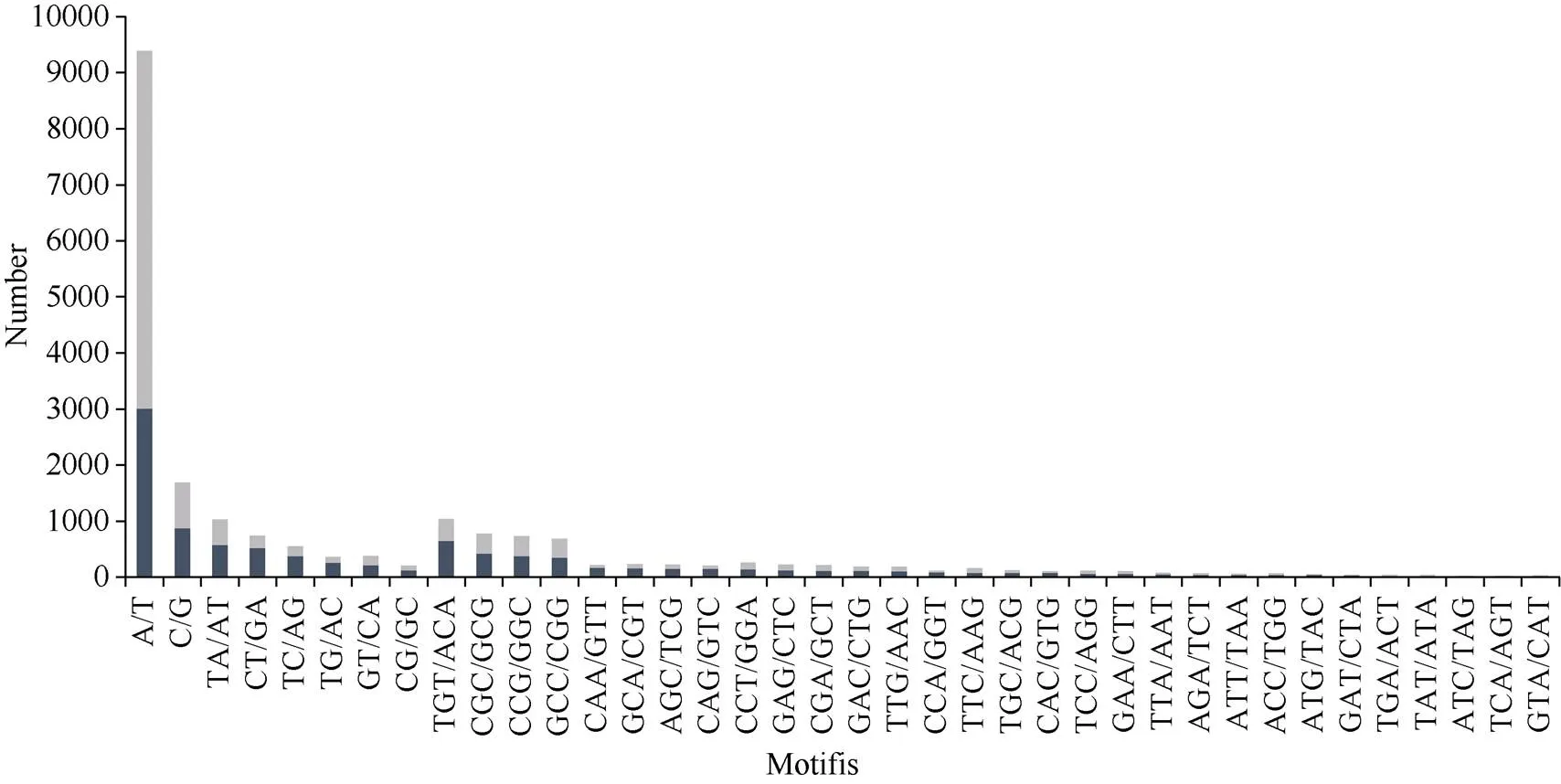
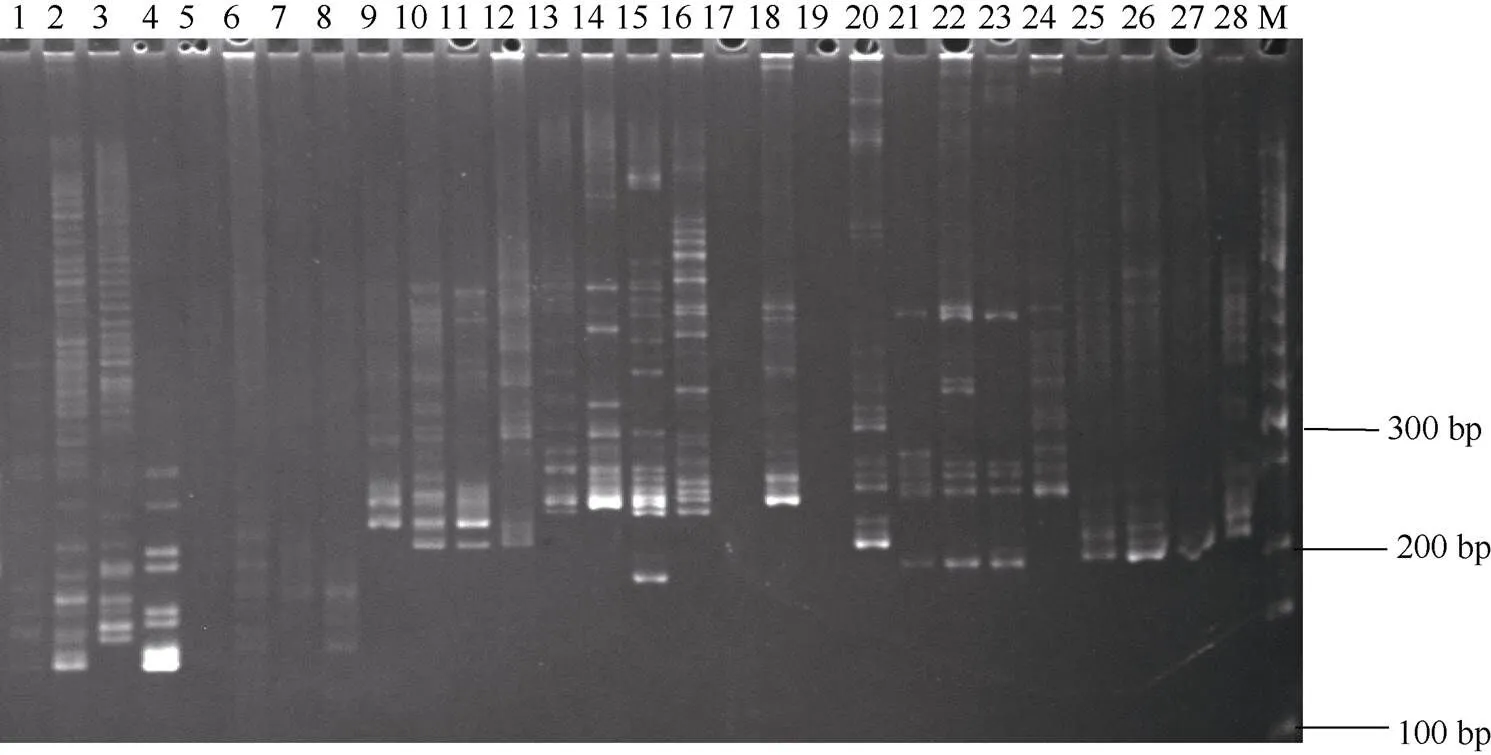
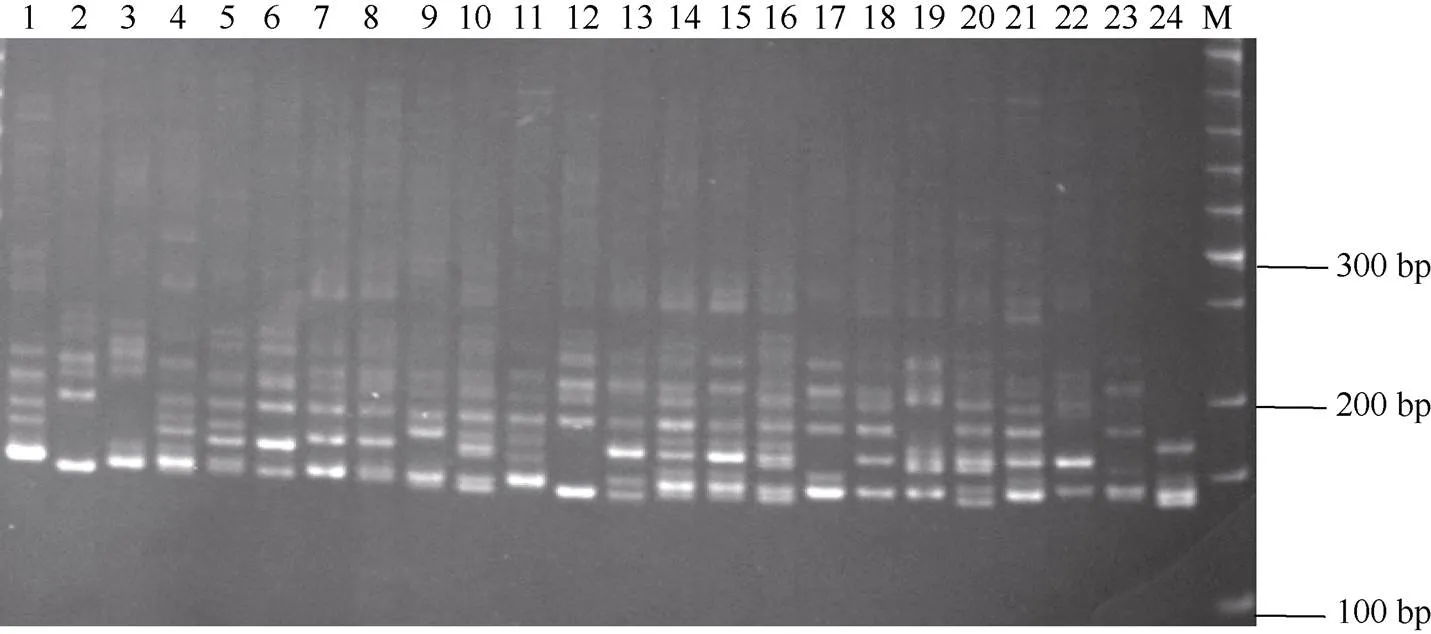


3 讨论
3.1 甘蔗栽培种基因组SSR位点的特征分析
3.2 甘蔗栽培种基因组SSR的多态性分析和应用
