菊叶香藜转录组单核苷酸多态性(SNP)信息挖掘及功能注释分析
2019-05-14付苏宏张勇群郝豆豆
付苏宏,雷 鸣,张勇群*,施 静,郝豆豆
(1. 西藏自治区人民政府驻成都办事处医院,四川 成都 610000;2. 西藏大学,西藏 拉萨 850000)
【研究意义】菊叶香藜(Dysphaniaschraderiana),具有强烈的气味因此又称之为臭菜,为藜科(Chenopodiaceae)刺藜属一年生草本植物,广泛分布于我国高海拔地区如西藏、内蒙古、甘肃、青海、陕西等省份。菊叶香藜被用作蒙药(蒙文名称:乌努日特-诺衣乐)[1],具有平喘解痉、清热解毒、止痛、止痒等功效[2]。此外,菊叶香藜富含植物精油,表现出良好的生物活性,对植物螨虫、昆虫、细菌均具有显著的抑制活性[3-5],菊叶香藜中最具有潜在天然药物开发价值的是其中的活性化合物。此外,由于菊叶香藜对极端环境具有较好的耐受性,也有研究报道称菊叶香藜可以改善其生存的生态环境[6]。然而,菊叶香藜的研究主要局限在传统民族药物方面,近几年才逐渐开展植物学、植物化学以及生物活性研究,在遗传学与基因组学方面的研究更是相当匮乏。单核苷酸多态性(single nucleotide polymorphism, SNP)指的是基因组DNA上的单个碱基发生变异且变异频率不小于1 %所引起的DNA序列多态性[7]。SNP可以作为DNA分子标记,具有位点丰富、分布广泛、高遗传稳定性、易于检测和分型等优点,现已成为最常用的分子标记之一,可广泛用于构建高密度遗传图谱、植物分子辅助育种、品种鉴定、个体性状遗传等方面的研究[8-11]。因此,通过对菊叶香藜SNP分子标记的开发,可以为鉴定活性物质高产性状以及极端环境耐受性状的亚型提供参考依据。【前人研究进展】菊叶香藜不是模式植物,目前还未获得其基因组数据以及其他近缘属植物的基因组测序数据,NCBI数据库中也尚无菊叶香藜的SNP信息登录,因此大大限制了菊叶香藜SNP分子标记的开发。【本研究切入点】由于基因测序技术发展迅猛,非模式植物可以进行转录组测序,其转录组数据是开发SNP分子标记的良好资源,并且这些基于转录组SNP分子标记具有信息量大、通用性好的优点[12]。【拟解决的关键问题】本研究利用菊叶香藜花和叶组织的转录组测序数据对其基因内部单核苷酸多态性(genic SNP)位点进行搜索,分析这些SNP的基本特征,并对SNP-unigene进行功能注释,为菊叶香藜的SNP分子标记的开发及其功能研究奠定基础。
1 材料与方法
1.1 试验材料与测序数据
菊叶香藜的花和叶组织采集于西藏拉萨市(N29°38′,E91°10′),采集后立即进行液氮速冻,然后送上海美吉生物医药科技有限公司于IlluminaHiseq4000平台上进行转录组测序,花和叶组织分别获得24 187 800和27 832 004条干净数据(Clean reads),所有的Clean reads通过组装后共获得40 142条Unigene (4.72 Gb)。菊叶香藜花和叶组织的转录组测序数据已上传至NCBI数据库,登录号分别为SRX3145241和SRX3145242。
1.2 菊叶香藜转录组SNP位点检测
以组装好的转录本为模板序列,将原始序列与其进行比对,利用Samtools (http://samtools.sourceforge.net/)[13]和VarScan v.2.2.7 (http://varscan.sourceforge.net/)[14]软件寻找候选的SNP位点。
1.3 SNP-unigene序列功能注释分析
对含有SNP的unigene序列(SNP-unigene)进行功能注释,探究SNP-unigene的生物学功能,注释所选数据库有基因本体数据库(gene ontology database, GO)、蛋白直系同源簇数据库(Clusters of Orthologous Groups of proteins, COG)、京都基因和基因组百科全书数据库(kyoto encyclopedia of genes and genomes, KEGG)数据库,期望值E设定为1e-5[15]。
2 结果与分析
2.1 菊叶香藜转录组SNP鉴定
使用Samtools软件和VarScan v.2.2.7软件对菊叶香藜花和叶的转录组序列进行SNP位点检测,在花和叶转录组中分别鉴定到889个和673个SNP位点。菊叶香藜花转录组中的889个SNP位点中转换(Transition)占有560个,颠换(Transversion)占有329个;6种核苷酸变异中属于转换的A/G和C/T发生频率最高,所占比例均为31.50 %,属于颠换的A/T、A/C、T/G和C/G的比例则分别为15.00 %、6.07 %、7.76 %和8.21 % (图1A);889个SNP中分布在密码子的第3位(Third codon)的比例最高,占花转录组SNP总数的40.16 %,然后依次为3端非编码区(3-UTR,15.52 %)、密码子第1位(First codon,12.60 %)、密码子第2位(Second codon,12.26 %)和5端非翻译区(5-UTR,5.62 %),有13.84 %分布位置无法确定(图1B)。菊叶香藜叶转录组中SNP变异类型与分布位置趋势与花组织转录组SNP类似,但数量较少(图1)。

图1 菊叶香藜转录组中genic SNP概况Fig.1 Summary of genic SNP within transcriptome ofD.schraderiana
2.2 SNP-unigene序列注释
从菊叶香藜花和叶组织中鉴定出的SNP位点总共位于643条SNP-unigene上,为进一步探索这些SNP-unigene的生物学功能,将这643条SNP-unigene进行GO注释、COG注释和KEGG注释,结果显示,643条SNP-unigene中总共有440 (68.43 %)条SNP-unigene注释到了GO数据库、COG数据库和KEGG数据库。在有注释信息的440条SNP-unigene中,注释到KEGG数据库的有232条,注释到COG数据库的有370条,注释到GO数据库的有343条;同时注释到GO数据库、COG数据库和KEGG数据库的总共有179条(40.7 %);同时注释到KEGG数据库和COG数据库197条(44.8 %),同时注释到COG数据库和GO数据库285条(64.8 %),同时注释到GO数据库和KEGG数据库201条(45.7 %);只注释到KEGG数据库的有13条(64.8 %),只注释到COG数据库的有67条(15.2 %),只注释到GO数据库的有35条(8.0 %) (图2)。
2.3 SNP-unigene序列GO分类
GO分类可分为3个方面:生物过程(Biological Process,BP)、分子功能(Molecular Function,MF)及细胞组分(Cellular Component,CC),而在该3个大分支下又可分为更加细致的功能,通过对SNP-unigene进行GO注释有助于理解基因背后所代表的生物学意义。使用blast2go软件将643条SNP-unigene与GO数据库比对,其中总共有342条SNP-unigene (53.19 %)注释到GO条目中,分布于分子功能、细胞组分和生物学过程的level2 GO条目分别有397、477和756个。在生物过程中,结合活性(162条)和催化活性(168条)中所涉及的SNP-unigene较多;在细胞组分中,细胞(358条)中所涉及的SNP-unigene较多;在生物学过程中,代谢过程(379条)和细胞过程(165条)中所涉及的SNP-unigene较多(图3)。
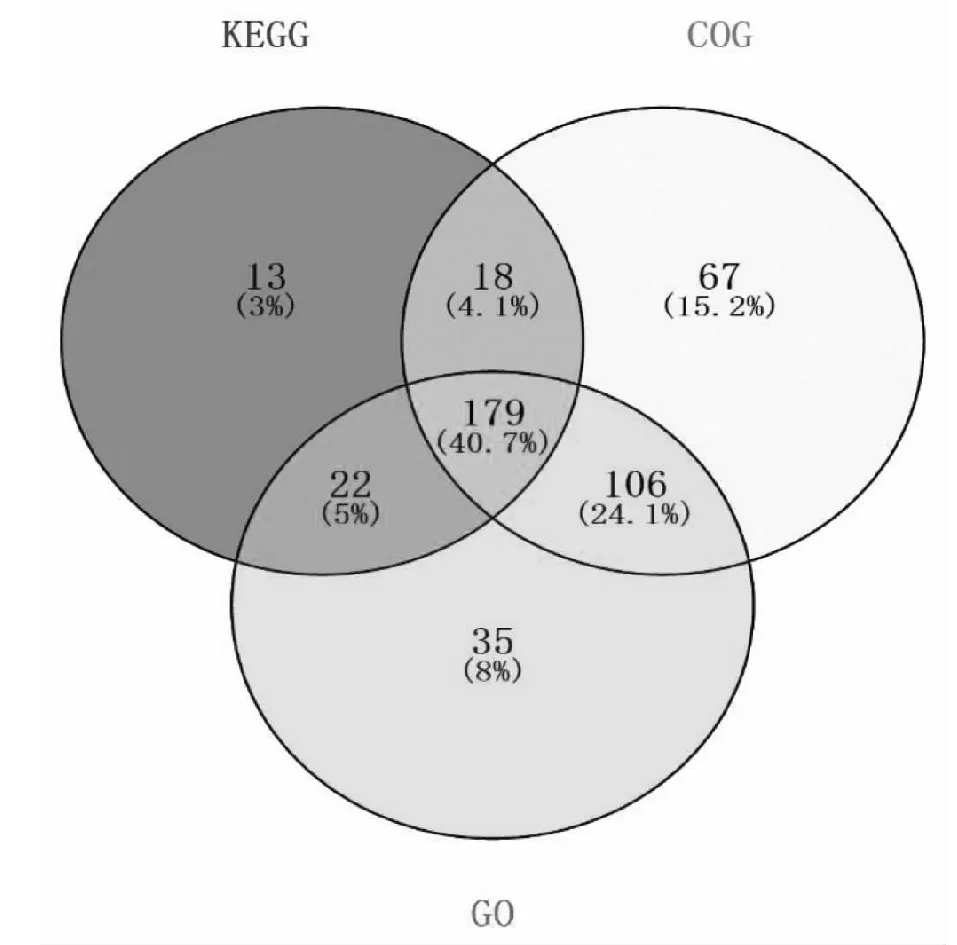
图2 SNP-unigene注释信息统计Venn图Fig.2 Venn diagram of annotation information of SNP-unigene
2.4 SNP-unigene序列COG分类
将SNP-unigene与COG数据库的比对,可以获取序列的功能注释、归类以及蛋白进化等信息。COG注释根据功能可分为25类(图3,A-Z表示,除X),总共有370条(57.54 %)SNP-unigene在COG
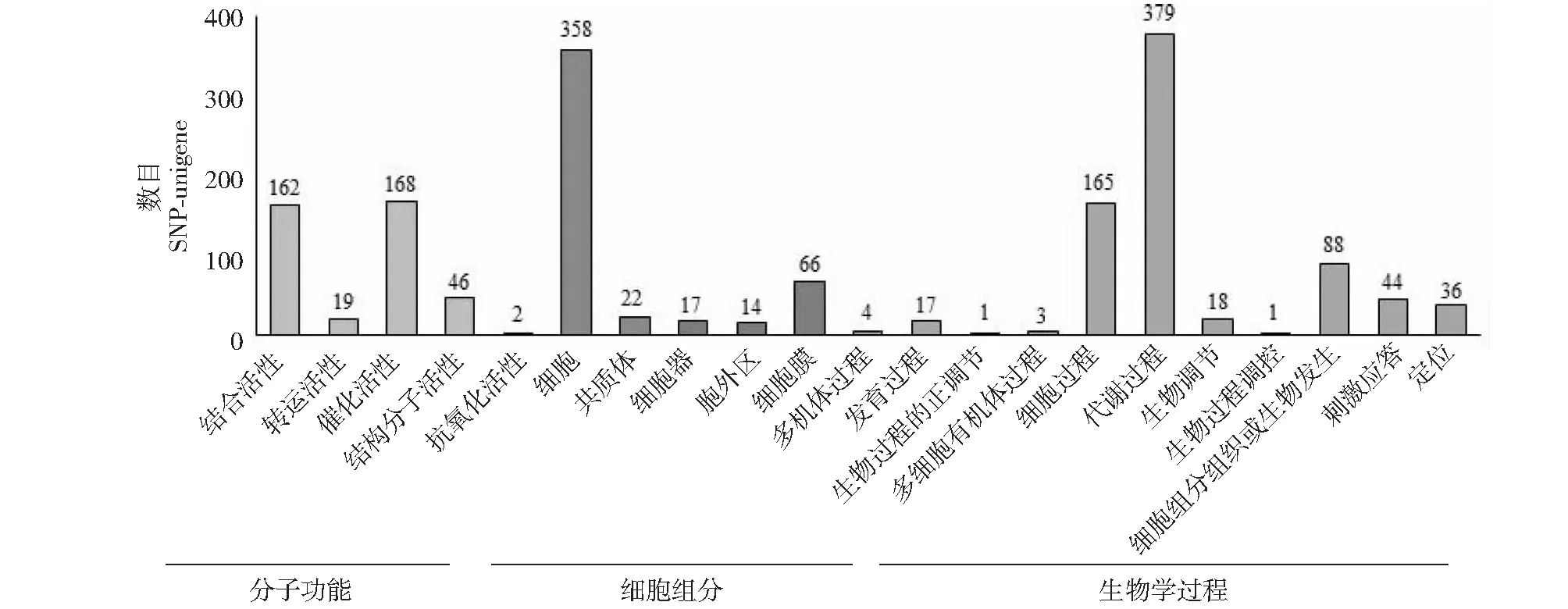
图3 GO功能分类统计Fig.3 GO functional classifications
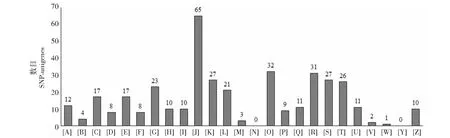
A:RNA加工和修饰;B:染色质结构和动力学;C:能量产生和转换;D:细胞周期控制、细胞分裂和染色体分离;E:氨基酸转运和新陈代谢;F:核酸转运和代谢;G:碳运输和新陈代谢;H:辅酶运输和代谢;I:脂类转运和代谢;J:翻译,核糖体结构和合成;K:转录;L:复制,重组和修复;M:细胞壁膜核膜的合成;N:细胞机动性;O:翻译后修饰,蛋白翻转和分子伴侣;P:无机离子转运和代谢;Q:二级代谢生物加工,转运和分解代谢;R:一般预测功能组;S:未知功能;T:信号传导机制;U:胞内的交换,分泌和膜泡输送;V:防御机制;W:胞外结构;Y:核结构;Z:细胞骨架图4 COG分类统计Fig.4 COG functional classification
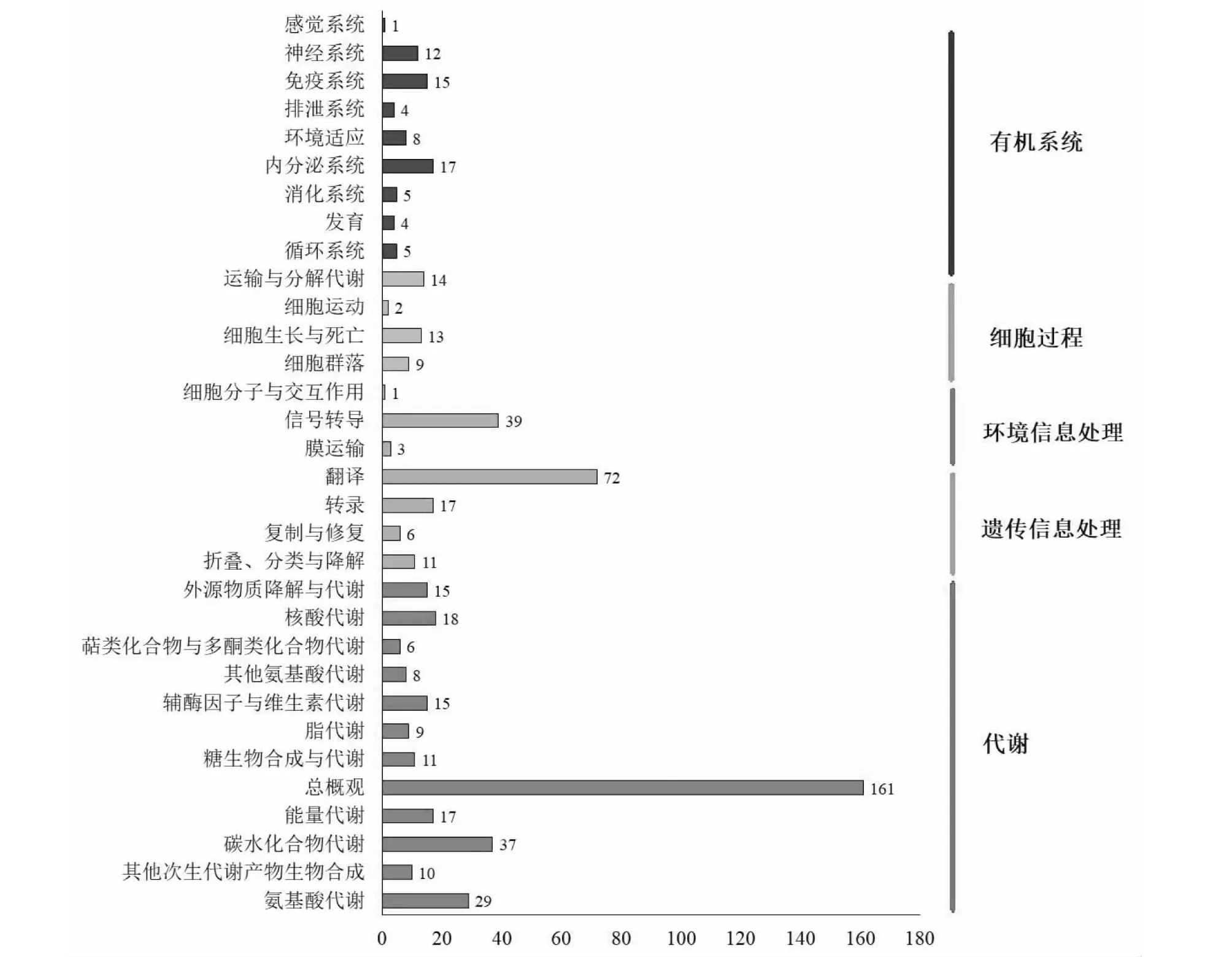
图5 KEGG注释统计Fig.5 Summary of SNP-unigene with mainly KEGG annotation
数据库中找到了相对应的注释信息,得到385个COG注释结果。通过对每一类功能的数量进行统计发现,除了“细胞机动性”(N)和“核结构”(Y)两个功能没有注释有SNP-unigene以外,其余的23个COG功能均注释有SNP-unigene,由此可见,被注释的370条SNP-unigene功能种类比较全面,覆盖了大多数(23/25)的生命活动过程及功能。在注释有SNP-unigene的COG功能分类中,“翻译,核糖体结构和合成”是数量最多的一大类,包括65个SNP-unigene;然后依次为“翻译后修饰,蛋白翻转和分子伴侣”、“一般预测功能组”、“转录”,包含的SNP-unigene分别为32、31、27条(图4)。
2.5 SNP-unigene序列KEGG分析
KEGG数据库可以将基因组的信息与功能信息联系起来,更加系统地分析和破译基因的功能。将unigene与KEGG数据库进行比对,可以获得与unigene相对应的KO编号,根据KO编号则可进一步获得该unigene可能参与的具体生物学通路。对643条SNP-unigene进行KEGG通路注释,其中总共有232条SNP-unigene注释到241条KEGG代谢通路中。将这232条SNP-unigene根据所参与的KEGG代谢通路划分为5个分支:代谢(A, Metabolism)、遗传信息处理(B, Genetic Information Processing)、环境信息处理(C, Environmental Information Processing)、细胞过程(D, Cellular Processes)、有机系统(E, Organismal Systems),其五大类的KEGG通路统计情况如图5所示。
筛选注释基因比例(占所有KEGG注释unigene)大于1 %的71条KEGG通路列于表1。从表1可知,注释到“代谢途径”的SNP-unigene最多,可达到78条,占所有KEGG注释SNP-unigene的33.62 %,然后依次为“核糖体”、“次级代谢产物的生物合成”、“微生物在不同环境中的代谢”、“嘌呤代谢”以及“RNA运输”,分别注释有48、36、19、13和13条SNP-unigene。通过KEGG的注释分析,可以着重研究这些具有代表性的生物学进程,探索菊叶香藜中这些生物学进程所涉及的功能。
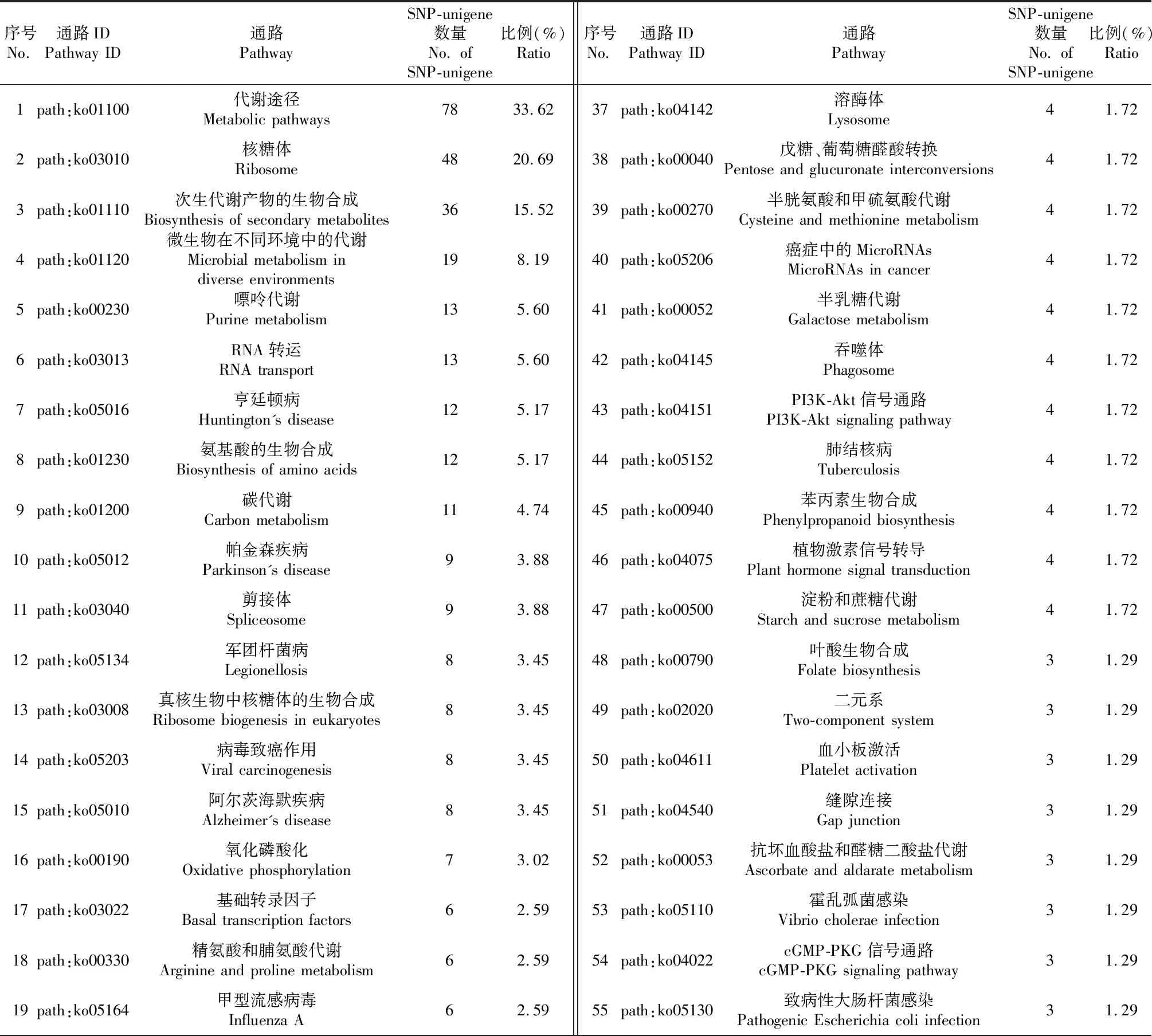
表1 主要KEGG pathway注释统计
续表1 Continued table 1
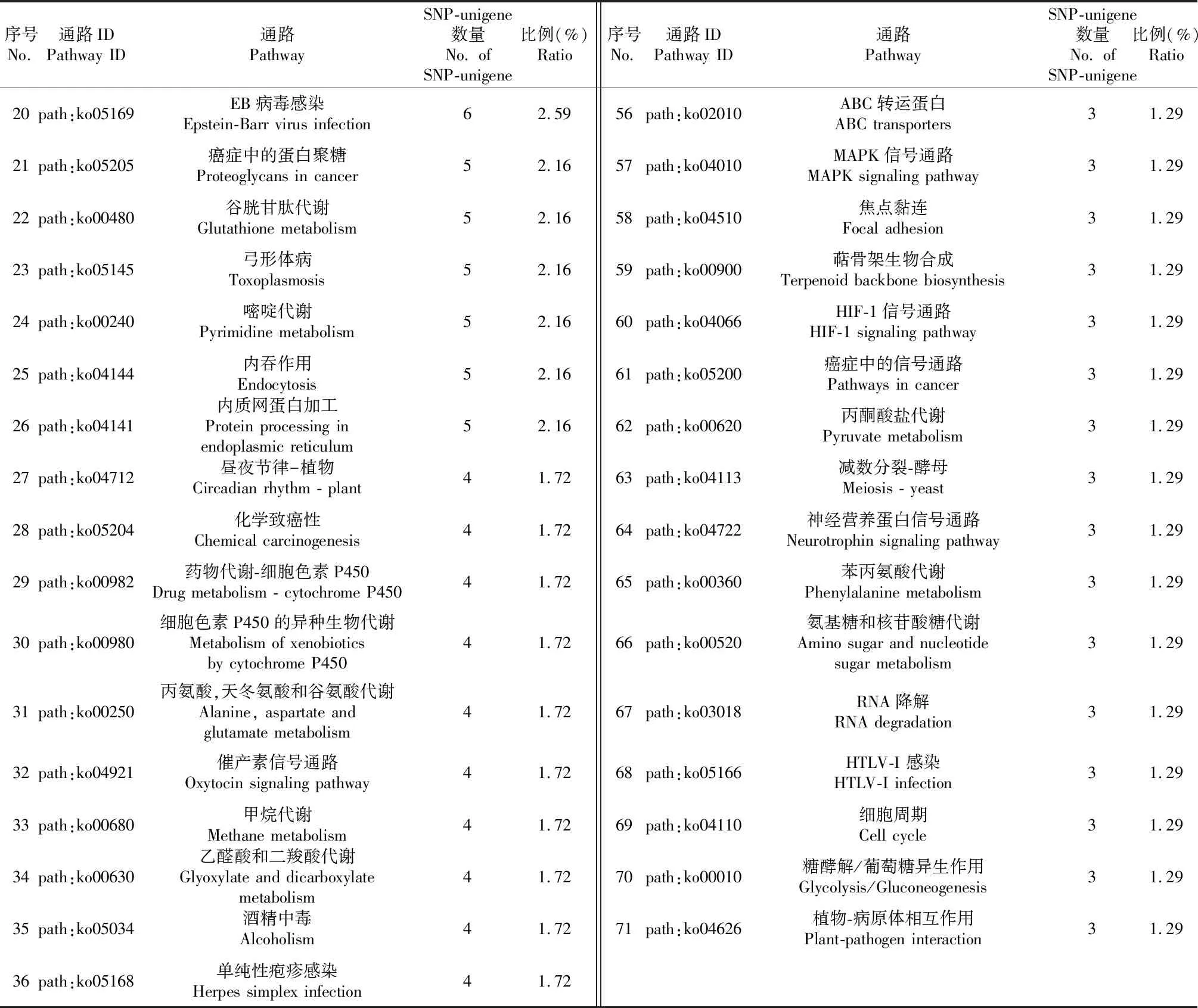
序号No.通路IDPathway ID通路PathwaySNP-unigene数量No. of SNP-unigene比例(%)Ratio 序号No.通路IDPathway ID通路PathwaySNP-unigene数量No. of SNP-unigene比例(%)Ratio20path:ko05169EB病毒感染Epstein-Barr virus infection62.5956path:ko02010ABC转运蛋白ABC transporters31.2921path:ko05205癌症中的蛋白聚糖Proteoglycans in cancer52.1657path:ko04010MAPK信号通路MAPK signaling pathway31.2922path:ko00480谷胱甘肽代谢Glutathione metabolism52.1658path:ko04510焦点黏连Focal adhesion31.2923path:ko05145弓形体病Toxoplasmosis52.1659path:ko00900萜骨架生物合成Terpenoid backbone biosynthesis31.2924path:ko00240嘧啶代谢Pyrimidine metabolism52.1660path:ko04066HIF-1信号通路HIF-1 signaling pathway31.2925path:ko04144内吞作用Endocytosis52.1661path:ko05200癌症中的信号通路Pathways in cancer31.2926path:ko04141内质网蛋白加工Protein processing in endoplasmic reticulum52.1662path:ko00620丙酮酸盐代谢Pyruvate metabolism31.2927path:ko04712昼夜节律-植物Circadian rhythm - plant41.7263path:ko04113减数分裂-酵母Meiosis - yeast31.2928path:ko05204化学致癌性Chemical carcinogenesis41.7264path:ko04722神经营养蛋白信号通路Neurotrophin signaling pathway31.2929path:ko00982药物代谢-细胞色素P450Drug metabolism - cytochrome P45041.7265path:ko00360苯丙氨酸代谢Phenylalanine metabolism31.2930path:ko00980细胞色素P450的异种生物代谢Metabolism of xenobiotics by cytochrome P45041.7266path:ko00520氨基糖和核苷酸糖代谢Amino sugar and nucleotide sugar metabolism31.2931path:ko00250丙氨酸,天冬氨酸和谷氨酸代谢Alanine, aspartate and glutamate metabolism41.7267path:ko03018RNA降解RNA degradation31.2932path:ko04921催产素信号通路Oxytocin signaling pathway41.7268path:ko05166HTLV-I 感染HTLV-I infection31.2933path:ko00680甲烷代谢Methane metabolism41.7269path:ko04110细胞周期Cell cycle31.2934path:ko00630乙醛酸和二羧酸代谢Glyoxylate and dicarboxylate metabolism41.7270path:ko00010糖酵解/葡萄糖异生作用Glycolysis/Gluconeogenesis31.2935path:ko05034酒精中毒Alcoholism41.7271path:ko04626植物-病原体相互作用Plant-pathogen interaction31.2936path:ko05168单纯性疱疹感染Herpes simplex infection41.72
3 讨 论
随着新一代测序技术的成熟以及成本的降低,对于非模式生物来说,转录组序列是开发SNP分子标记最有效的策略之一[16]。目前,已经有大量的非模式生物通过转录组序列进行了SNP分子标记的研究和开发,在松萝凤梨叶片转录组数据中总共挖掘到了69 570个基因内部SNP,发生频率为1/579 bp[17];同时,在龙眼转录组数据中挖掘并验证了60个SNP分子标记并进行了基因型鉴定[18];通过对盾叶薯蓣转录组数据进行挖掘,得到了124 692个SNP位点,每条unigene中平均含有3.36个SNP位点[19]。大量研究表明,挖掘转录组数据中的SNP信息是非模式生物开发SNP分子标记可行且有效的方法。
本次研究中,菊叶香藜SNP位点处碱基变异类型主要为C/T和A/G,以转换为主,花组织和叶组织中转换和颠换的比值分别为1.70和1.68,均远远大于理论值的0.5。生物中SNP位点碱基变异的转换和颠换的比值往往会大于理论值的0.5,这种现象称为“转换偏差”[20],而这一现象也恰恰说明了SNP位点的碱基突变并不是随机发生的,可能与环境选择和进化有关。除了有少量的SNP分布位置不确定以外,位于非编码区的SNP在花和叶转录组序列中分别占21.15 %和21.10 %,而位于编码区的SNP在花和叶转录组序列中则分别占到了67.27 %和67.46 %。由此可见,菊叶香藜的SNP位点大多位于编码区内,此类SNP位点处的碱基变异可能会引起氨基酸序列的变化,进一步引起菊叶香藜生物性状的变化,因此,开发此类的SNP标记具有重要意义。
挖掘菊叶香藜转录组数据获得了大量的SNP信息,通过对这些SNP-unigene进行功能注释则有利于探索菊叶香藜在哪些生物和代谢途径中可能存在个体性状差异,从而将这些SNP和其表型关联起来进行更深入的研究。在菊叶香藜SNP-unigene功能注释中,菊叶香藜SNP-unigene涉及较多的功能主要与代谢、核糖体、次生代谢产物的生物合成相关,可针对这些SNP-unigene设计引物进行SNP验证并做表型关联研究,所以针对这类功能的SNP分子标记进行开发研究,可以为以后分子标记辅助育种等工作奠定坚实的基础。
4 结 论
本研究挖掘获得了菊叶香藜转录组测序数据中的SNP信息,并获取了SNP-unigene的GO功能注释和KEGG通路注释信息,本研究成果将积极地促进菊叶香藜的遗传图谱构建、遗传育种、遗传多样性分析等方面的研究。
