分子信标探针技术用于PCR检测诺卡氏菌SecA1基因
2017-06-28王颜颜夏茂宁明春艳刘涛华康颖倩
王颜颜,夏茂宁,明春艳,黄 劲,刘涛华,周 兵,康颖倩
分子信标探针技术用于PCR检测诺卡氏菌SecA1基因
王颜颜1,夏茂宁1,明春艳1,黄 劲2,刘涛华1,周 兵1,康颖倩1
目的 将诺卡氏菌属细菌的一段特异性DNA设计成分子信标探针,用于该细菌的PCR检测。方法 将诺卡氏菌属细菌、戈登氏菌属细菌及红球菌属细菌菌株分别接种于脑心浸液琼脂培养基分离培养,观察其生长情况,提取菌株DNA作为扩增模板;设计诺卡氏菌属细菌基于secA1基因的特异性分子信标探针,在实时荧光定量PCR反应译体系中加入分子信标探针,PCR产物进行荧光信号检测。结果 诺卡氏菌secA1基因经实时荧光定量PCR扩增后可产生阳性荧光信号,红球菌属细菌及戈登氏菌属细菌的secA1基因、阴性对照实验组及空白对照组经实时荧光定量PCR扩增后不产生荧光信号,为阴性。结论secA1作为看家基因,是用来进行种水平的鉴定及系统进化研究非常理想的靶分子,而分子信标探针技术可以准确、快速、灵敏的进行诺卡氏菌secA1基因检测。
诺卡氏菌属;SecA1基因;分子信标探针;实时荧光定量PCR
诺卡氏菌属(Nocardia)为需氧放线菌,是土壤中腐生菌,不是人和动物体内的正常菌群。诺卡氏菌感染既可发生在健康人体外伤后,又可引起免疫缺陷患者致死性感染,当吸入或者由外伤进入人体后,可引起肺部及皮下组织的感染,也可播散至全身,引起各种内脏器官发生感染[1-3]。诺卡氏菌属中对人或动物致病的的病原体至少有25个种,从人体内分离出来的诺卡菌重星形诺卡氏菌占90%,巴西诺卡氏菌占7%,而豚鼠诺卡菌占3%[4]。有报道称,星形诺卡氏菌是报道最多的引起动物疾病的病原需氧放线菌,其中最常见的动物疾病为牛乳腺炎,也曾被报道可引起鱼类感染[5]。在临床上除非是已经怀疑有相应感染可能性,需氧放线菌的感染通常会被漏诊或误诊[6],目前常规使用的诺卡氏菌属细菌的分离培养、生理生化反应及经典的PCR技术等检测方法操作繁琐、耗时长,不适于临床上诺卡氏菌属细菌的快速鉴别诊断。因此,建立诺卡氏菌属细菌的快速、可靠、特异、灵敏且操作简单的检测方法十分必要。
特异性基因是多数细菌存在的最直接的标志,有多项研究表明,在以核苷酸序列为基础的放线菌分类及鉴别研究中,16S rRNA 序列难以区分某些种,而看家基因secA1的序列具有足够的保守性以及可变性,可作为区分不同种的靶分子。Fischer[7]等扩增了分枝杆菌属29 个种的47 株ATCC 标准菌和59 株临床分离菌的secA1基因序列,首次发现分泌相关基因可以用于区分分枝杆菌属内的不同菌种。 Conville[8]等分析比较了诺卡氏菌属的29 个种的16S rRNA 以及secA1基因序列,发现secA1基因能更好更准确地区分和鉴别诺卡氏菌属内的菌种。同样地,国内学者康颖倩[9]等对戈登氏属的23 个模式种分别进行了secA1基因和16S rRNA 系统进化关系的比较分析,而后得出结论,secA1作为看家基因用来进行种水平的鉴定以系统进化的研究,是非常理想的靶标,同时还发现[9-10],对于放线菌目中的几种主要相关菌属如诺卡氏菌属、戈登氏菌属、分枝杆菌属、链霉菌属等细菌的secA1基因翻译出来的氨基酸序列,不同属别的菌种各有其特异性的基序。本研究利用分子信标探针技术对诺卡氏菌进行secA1基因检测。
1 材料与方法
1.1 实验菌株与试剂
1.1.1 诺卡氏菌属菌株CMCC(F)D4C、红球菌属菌株CMCC(F)D4B及戈登氏菌属菌株IFM10866各一株,菌株由中国科学院微生物研究所及日本千叶大学真菌医学研究所惠赠。
1.1.2 脑心浸出液固体培养基;洗涤液: 50 mmol/L Tris,pH7.7,25 mmol/L EDTA,0.1% PVP;裂解液:50 mmol /L Tris,pH8,25 mmol /L EDTA,20% SDS,1.2% PVP;抽提液:10 mmol/L Tris,pH8,1 mmol/L EDTA;3 mol/L NaAc,1.2%PVP;1×TAE缓冲液;PCR试剂;DNA Marker;琼脂糖。
1.2 仪器 ABI StepOnePlus 实时荧光定量PCR仪为赛默飞世尔科技(中国)有限公司产品。
1.3 分子信标探针的设计与合成
1.3.1 按照一般杂交探针设计的要求,根据GenBank上公布的诺卡氏菌菌株secA1基因序列,采用Primer 软件设计分子信标探针,并将选取的19 bp的DNA片段与诺卡氏菌属菌株基因组DNA序列进行同源比较,确定是一段无同源片段的特异性DNA。再按照分子信标探针设计的要求,在19 bp 特异性DNA片段的5′和3′端分别添加互补的且与目的DNA无关的6个碱基臂,5′端为CCTAGA,3′端为TCTAGG,形成一个发卡结构(如图1),19 bp特异性DNA构成发夹的噜扑环,6个互补碱基配对形成发夹的茎。在发夹结构的5′端标记荧光素FAM,3′端标记淬灭剂DABCYL。
1.3.2 分子信标探针由立菲生物技术有限公司合成,将合成的探针用高效液相色谱(High Performance Liquid Chromatography,HPLC)纯化:A液(0.1 mol/L醋酸三乙酰)30%-80%,B液(75%乙腈+A液)20%-70%,梯度洗脱25 min,再用B液(100%)洗5 min,流速1 mL/min,收集相应的峰,获得纯度较高的分子信标探针。
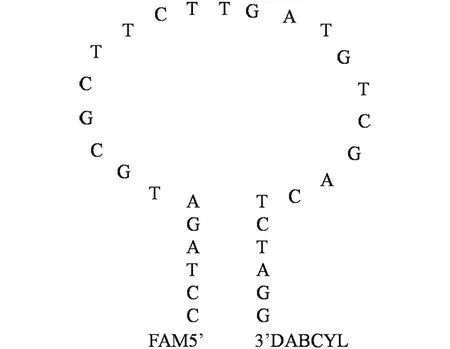
图1 分子信标探针示意图Fig.1 The sketch map of molecular beacon probe
1.4 模板制备 阴性对照和阳性对照的模板按下列方法处理[11]:各细菌于37 ℃温箱培养5-7 d后:①挑取0.05 g湿菌体,加入35 μL裂解液及15 μL 20%SDS溶液后混匀;②微波90 s;③快速加入459 μL抽提液,混匀;④加入500 μL酚氯仿抽提液,混匀,13 400 g离心10 min,尽量吸取上清液;⑤重复步骤④一次;⑥取上清液加入800 μL无水乙醇及80 μL 3M 乙酸钠(pH4.8-5.2)后,室温静置10 min;⑦13 400 g离心10 min后去上清,用70%的乙醇(150~200 μL)洗1-2次;⑧离心去上清,于37 ℃~55 ℃温箱烘干;⑨加30~50 μL 1×TAE溶解后放入-20 ℃备用。

M:DL2 000;1-3:诺卡氏菌CMCC(F)D4C;4:红球菌属菌株CMCC(F)D4Bs;5:戈登氏菌属菌株IFM10866M:DL2000;1-3:Nocardia CMCC(F)D4C;4:GordinaCMCC(F)D4B;5:RhodococcusIFM10866图2 3株实验菌株secA1基因扩增琼脂糖凝胶电泳Fig.2 Three strains agaroseg electrophoresis of amplification production of secA1 gene
1.5 实时荧光定量PCR的扩增 对上一步骤中已扩增出特异性secA1基因片段所对应细菌DNA模板进行实时荧光定量PCR扩增,采用20 μL体系:PCR Mix 10 μL,引物各1 μL,DNA模板1 μL,分子信标探针1 μL,双蒸水6 μL,设空白对照。按常规PCR条件进行扩增:95 ℃ 5 min后,95 ℃ 1 min,63 ℃ 1 min,72 ℃ 1 min,35个循环,72 ℃ 10 min。每个模板重复3次实验操作。
2 结 果
2.1 分子信标探针对PCR扩增的影响 分别以含有诺卡氏菌特异性DNA的菌株作为PCR扩增模板,设计两个实验,一是在PCR反应液中加入分子信标探针,另一实验在PCR反应液中不含探针,同时进行PCR扩增。琼脂糖凝胶电泳结果表明(图3),在PCR反应液中加入探针及不含探针的扩增条带基本位于同一位置,扩增产物的量也基本一致,这表明在PCR扩增液中加入分子信标探针不会影响PCR的扩增反应。
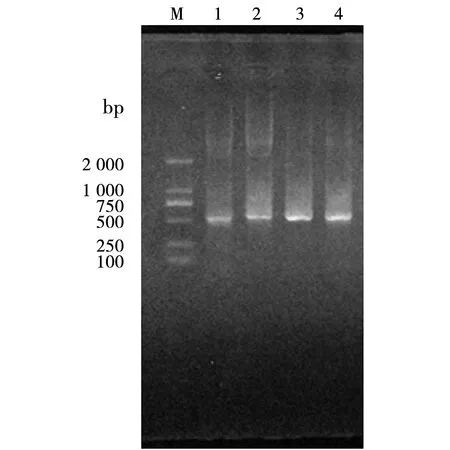
M:DL2000;1-2:诺卡氏菌CMCC(F)D4C;3-4:诺卡氏菌CMCC(F)D4C+分子信标探针M:DL2000;1-2:Nocardia CMCC(F)D4C;3-4:Nocardia CMCC(F)D4C+ molecular beacon probe图3 分子信标探针对PCR扩增的影响Fig.3 Effect of molecular beacon probe on PCR amplification
2.2 分子信标探针的特异性 本研究所设计的分子信标探针用于检测诺卡氏菌属细菌secA1基因,经实时荧光PCR检测后发现:仅诺卡氏菌属细菌产生阳性信号,另一实验组红球菌属细菌及戈登氏菌属细菌的secA1基因、阴性对照实验组及空白对照组经实时荧光定量PCR扩增后不产生荧光信号,为阴性(见图4)。说明本研究设计的用于检测诺卡氏菌属细菌secA1基因的分子信标探针能快速、特异、灵敏地检测诺卡氏菌,可与其他需氧放线菌不同属细菌进行鉴别区分。
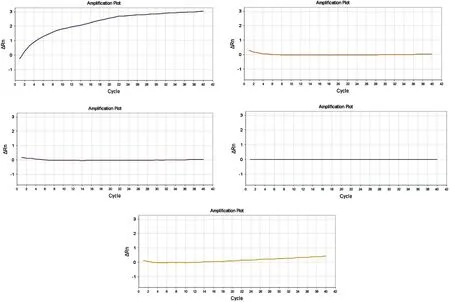
A:诺卡氏菌CMCC(F)D4C+分子信标探针;B:戈登氏菌IFM10866+分子信标探针;C:马红球菌CMCC(F)D4B+分子信标探针;D:阴性对照;E:空白对照A:Nocardia CMCC(F)D4C+ molecular beacon probe;B:Gordina CMCC(F)D4B+ molecular beacon probe;C:Rhodococcus IFM1086+ molecular beacon probe;D:Negative control;E:Blank control图4 不同实验组的实时荧光定量PCR结果Fig.4 Result of RT-PCR in different experimental groups
3 讨 论
分子信标探针的原理是荧光共振能量转移,当目的DNA不存时,分子信标探针两端的碱基臂可互补配对形成稳定的发夹结构,此时FAM 和DABCYL距离较近,在紫外光激发下,FAM吸收的能量通过荧光共振能量转移并传递给与它靠近的DABCYL,最终DABCYL再将能量以热量的形式散失,因此无荧光产生;当存在诺卡氏菌属细菌目的DNA时,分子信标探针的噜扑环可与目的DNA互补结合,发生比两臂杂交更长也更稳定的配对,从而使发夹结构打开,FAM 和DABCYL随之分开,在紫外光激发下FAM吸收能量,而FAM与DABCYL相距较远,FAM吸收的能量不能转移给DABCYL,因此产生荧光信号。
据报道,影响分子信标探针的构型变化的参数主要包括臂长、臂序列%GC含量、噜扑环长度及溶液盐浓度,尤其是二价阳离子如Mg2+对两臂形成的杂交茎有较强的稳定作用[12],所以在本研究中常规PCR扩增时,Mg2+是反应体系中重要的成分之一,但在进行实时荧光定量PCR扩增时未加入Mg2+的原因之一;一般探针设计的要求是噜扑环长度至少应该为臂长的两倍才能保证探针与目的DNA杂交后荧光素与淬灭剂分开,所以在设计本研究的分子信标探针时,噜扑环长度为19个碱基,而臂长仅为6个碱基,从而保证了本研究中进行探针特异性检测时荧光素与淬灭剂可以分开;另外,臂序列GC含量一般要求是在50%以上,这有利于形成较稳定的发夹结构,鉴于此,在设计本研究的分子信标探针时,噜扑环中碱基数量GC为6个,GC含量为50%,完全符合分子信标探针设计的一般要求[13]。
目前设计分子信标探针时可选用的荧光染料很多,而分子信标探针依赖的是供体与受体之间的共振能量转移,为了荧光能被有效淬灭,供体的发射光谱应完全覆盖受体的激发光谱,而本研究中所选用的FAM与DABCYL完全符合这一要求[14],FAM的荧光效率较高,DABCYL吸收供体能量后以热量散失,减少了干扰,因此FAM-DABCYL是较好的供体-受体对。
据文献报道,分子信标探针与目的DNA杂交后,未杂交的探针会有残余荧光,这可能是由于荧光素和淬灭剂与探针碱基之间的接头过长或杂交茎末端瞬间解开造成的荧光本底,从而影响了分子信标探针检测的灵敏性,因此降低荧光本底是在进行实时荧光定量PCR扩增时应该解决的首要问题[15]。但在本研究中,即使在PCR扩增循环数较低情况下,所测得的荧光强度仍然高于本底的荧光强度,并不影响对诺卡氏菌属细菌快速、准确的鉴定。
本课题的下一步研究将会持续完善诊断鉴别分子信标探针功能,后续将研发相关的试剂盒并投入临床检验测试,收集临床上疑似诺卡氏菌感染的病例标本验证所研发试剂盒的性能。
[1] Li S, Song XY, Zhao YY, et al. Clinical analysis of pulmonaryNocardiosisin patients with autoimmune disease[J].Medicine(Baltimore),2015,94(39):1-8.DOI: 10.1097/MD.0000000000001561
[2] Tang H, Mao T, Gong Y, et al.Nocardialbrain abscess in an immunocompromised old patient: a case report and review of literature[J]. Int J Clin Exp Med, 2014, 7(5): 1480-1482.
[3] Adjamian N,Kikam A,Wessell KR, et al.Nocardiabrain abscess and CD4+lymohocytopenia in previously healthy individual[J].Case Reports Immunol, 2015:374956.DOI: 10.1155/2015/374956
[4] Wang DL. Medical mycology-guide to laboratory examination[M]. Beijing: People’s Medical Publishing House,2005,472-475.(in Chinese)
王端礼.医学真菌学-实验室检验指南[M].北京:人民卫生出版社, 2005,472-475.
[5] Mu LL,Tu YH, Kang YQ, et al.SecA1 gene sequences for aerobic actinomycetes[J]. Chin J Zoonoses,2016,32(4):349-355.DOI:10.3969/j.issn.1002-2694.2016.04.007 (in Chinese)
牟丽丽,涂云华,康颖倩,等.几类主要病原需氧放线菌菌属的SecA1基因分析研究[J].中国人兽共患病学报,2016,32(4):349-355.
[6] McNeil MM, Brown JM. The medically important aerobicActinomycetes:epidemiology and microbiology [J].Clin Microbiol Rev,1994, 7(3):357-417.
[7] Zelazny AM, Calhoun LB, Li L, et al. Identification ofMycobacteriumspecies bysecA1 sequences[J]. J Clin Microbiol, 2005, 43(3): 1051-1058.
[8] Conville PS, Zelazny AM, Witebsky FG. Analysis ofsecA1 gene sequences for identification ofNocardiaspecies[J]. J Clin Microbiol, 2006, 44(8): 2760-2766.
[9] Kang Y, Takeda K, Mikami Y, et al. Phylogenetic studies ofGordoniaspecies based ongyrBandsecA1 gene analyses[J]. Mycopathologia. 2009,167 (2): 95-105. DOI:10.1007/sl1046-00809151-y.
[10] Takeda K, Kang Y, Mikami Y, et al. Phylogenetic studies ofNocardiaspecies based ongyrBgene analyses [J]. J Med Microbiol,2010, 59(2): 167-171. DOI 10.1099/jmm.0.011346-0
[11] Xu P, Li WJ, Xu LH, et al. A microeave-based method for genomic DNA extraction from actinomycetes[J].Microbiol China,2003,30(4):82-84. (in Chinese)
徐平,李文均,徐丽华,等.微波法快速提取放线菌基因组DNA[J].微生物学通报,2003, 30(4):82-84.
[12] Qiu J, Brown W,et al. Combination probes with intercalating anchors and proximal fluorophores for DNA and RNA detection[J]. Nucleic Acids Res,2016,30,44(17):e138. DOI: 10.1093/nar/gkw579
[13] Jiang YX, Fang XH, Wan LJ, et al.Progress in the study and application of a novel fluorescent probe-molecular beacon[J].Chin J Analytical Chem, 2004, 32(4): 668-672. (in Chinese)
江雅新, 方晓红, 万立骏, 等.一种新型荧光探针—分子信标的研究及应用进展[J].分析化学评述与进展,2004, 32(5):668-672.
[14] Fields GB.Using fluorogenic peptide substrates to assay matrix metalloproteinases[J].Methods Mol Biol,2010,622(622):393-433.DOI:10.1007 /978-1-60327-299-5_24
[15] Watanabe S, Hagihara K, Hashimoto M, et al. Microbead-based ligase detection reaction assay using a molecular beacon probe for the detection of low-abundance point mutations[J].Anal Chem. 2014,86(1):900-906.DOI:10.1021/ac403531x
PCR detection of theNocardiageneSecA1 using molecular beacon probe
WANG Yan-yan1, XIA Mao-ning1, MING Chun-yan1, HUANG Jing2,LIU Tao-hua1, ZHOU Bing1, KANG Ying-qian1
(1.DepartmentofMicrobiology,GuizhouMedicalUniversity,Guiyang550025,China;2.DepartmentofBiochemistryandMolecularBiology,GuizhouMedicalUniversity,Guiyang550025,China)
Molecular beacon probe was designed based on a specific DNA sequence ofNocardiato PCR detection of this bacterium. The strains ofNocardia、GordinaandRhodococcuswere inoculated in Brain Heart Infusion Agar medium separately, then the growth condition was observed, DNA was extracted as a template; the molecular beacon probe was designed based on the partialsecA1 gene sequences ofNocardiastrains, and the probe was added into the reaction system of real-time fluorescence quantitative PCR (RT-PCR),and the fluorescence signal was tested at the end of PCR.Showed that the amplifiedsecA1 gene ofNocardiacould produce positive fluorescence signal in RT-PCR, but those ofGordoniaandRhodococcuswith control groups showed negative results because of no fluorescence signal. In conclusion as a housekeeping gene,secA1 is an ideal target molecule to identify the actinomycetes strains on the species level in the systematic evolution research, and the technique of fluorescence molecular beacon probe is accurate, rapid and sensitive for detecting theNocardiastrains withsecA1 gene.
Nocardia;SecA1gene;molecular beacon probe; RT-PCR
Kang Ying-qian,Email: joycekangtokyo@qq.com
10.3969/j.issn.1002-2694.2017.06.007
国家自然基金(No.31260029),贵州省国际科技合作项目(黔科合外G字[2014]7006),贵阳市人民政府-贵州医科大学科学技术联合基金“贵阳市科技局创新团队”项目(GY2015-18)和贵州省科技计划项目(黔科合[2010]3154号)联合资助
康颖倩,Email:joycekangtokoyo@qq.com
1.贵州医科大学微生物学教研室,贵阳 550025; 2.贵州医科大学生化与分子生物学教研室,贵阳 550025
R379.1
A
1002-2694(2017)06-0508-05
2016-11-26 编辑:梁小洁
Supported by the National Natural Science Fund of China(No. 31260029), the International Science and Technology Cooperation Project of Guizhou(No.[2014]7006), the Technology Innovation Team Project of Guiyang(No.GY2015-18),and the Science and Technology Plan Project of Guizhou(No. [2010]3154)
