鹅细小病毒HN株的分离鉴定及全基因组序列分析
2017-01-04贺冬梅谭树义陈朝喜叶保国林哲敏曹宗喜
贺冬梅,谭树义,陈朝喜,叶保国,张 艳,林哲敏,曹宗喜*
(1.海南省农业科学院畜牧兽医研究所,海南海口 571100;2.西南民族大学生命科学与技术学院,四川成都 610041)
鹅细小病毒HN株的分离鉴定及全基因组序列分析
贺冬梅1,2,谭树义1,陈朝喜2,叶保国1,张 艳1,林哲敏1,曹宗喜1*
(1.海南省农业科学院畜牧兽医研究所,海南海口 571100;2.西南民族大学生命科学与技术学院,四川成都 610041)
为研究鹅细小病毒(GPV)基因遗传变异特征,采集海南某养鹅场疑似鹅细小病毒感染的病料,将其处理后接种番鸭胚成功分离到一株病毒,经PCR鉴定为鹅细小病毒,命名为HN株,并获得了其全基因组序列,将该序列与GenBank数据库中登录的16条鹅和番鸭细小病毒基因序列进行了比对分析。结果显示,该株病毒基因组全长为5 106 bp,由ITR、NS、VP构成,其中ITR为444 bp,NS1为1 844 bp,VP1为2 199 bp;HN株与SHFX1201株的NS1基因和VP1同源性最高,分别达到99.8%和99.7%,与番鸭细小病毒株FM的NS1基因同源性最低,为82.7%;与90-0215株VP1同源性最低,为80.1%。HN株的遗传进化树可以看出,GPV可以分成明显的2个基因亚群,HN株与鹅细小病毒匈牙利株(B)、欧洲疫苗株(VG32/1)和台湾株(82-0321V、82-0321、06-0329)均处在第I亚群,且与安徽分离株Y株以及SHFX1201株同源性最接近,番鸭源匈牙利株FM单独处于第Ⅱ亚群。本研究丰富了GPV的数据资料,为研究GPV分类地位以及遗传进化关系提供了依据,同时也为研究GPV流行趋势和疫苗的开发奠定了基础。
鹅细小病毒;分离鉴定;全基因组;IRT;NS1;VP1
鹅细小病毒病又称小鹅瘟(Derzsy′s disease or Gosling plaque),是由鹅细小病毒(Goose parvovirus,GPV)所引起的雏鹅急性或亚急性的败血性传染病。临床上以精神委靡,离群独偶,鼻孔流出浆液性鼻液,患鹅频频摇头,拉灰黄色或黄绿色稀粪,神经紊乱,小肠中后段黏膜坏死脱落与纤维素性渗出物凝固形成栓子,形如腊肠状为特征。本病常呈败血经过,发病率和病死率很高,对养鹅业危害极大[1-2]。GPV为单链线性DNA病毒,其基因组约5 kb,在基因组的两端均含有相同的末端倒置重复序列(inverted terminal repeat,ITR)可折叠形成U形双链发夹结构,其主要功能是病毒的复制起点并提供顺式包装信号,中间有2个开放阅读框,左侧编码非结构蛋白NS1和NS2,主要参与病毒的复制和转录调节;右侧编码衣壳蛋白VP1、VP2和VP3,VP3是GPV的免疫保护抗原,能刺激机体产生中和抗体[3-4]。
本研究通过在海南疑似鹅细小病感染的病料中分离到1株疑似鹅细小病毒,进行分离鉴定后确定为鹅细小病毒,且命名为HN株,并对其全基因组序列进行分析,丰富了我国鹅细小病毒基因组数据资料,为研究该病的流行、基因遗传进化特征及指导小鹅瘟的防控奠定基础。
1 材料与方法
1.1 材料
1.1.1 样品 由本实验室采集并保存的海南某养鹅场疑似GPV感染雏鹅组织(肝脏、脾脏等组织样品)。
1.1.2 试剂 琼脂糖、胶回收纯化试剂盒、pMD18-T载体、大肠埃希菌DH5α感受态细胞、LATaqDNA聚合酶、DNA Maker DL 2 000等为宝生物工程(大连)有限公司产品;DNA提取试剂盒为生工生物工程(上海)股份有限公司产品。
1.2 方法
1.2.1 引物设计与合成 根据GenBank中GPV标准株的全基因组序列设计7对引物,引物均由宝生物工程(大连)有限公司合成(表1)。
1.2.2 样品处理 采集发病雏鹅的肝脏和脾脏剪碎研磨,按1∶3加入灭菌生理盐水,反复冻融3次,5 000 r/min离心10 min取上清液,用0.22 μm微孔滤器过滤除菌,置-70 ℃保存备用[5]。
1.2.3 病毒分离鉴定 将冻存疑似GPV样品用前加入终浓度为10 000 IU的青霉素和链霉素4 ℃感作1 h,接种10日龄健康鹅胚(0.3 mL/枚),弃去24 h 内死亡的鹅胚,收取24 h后死亡的鹅胚尿囊液,盲传3代,置-20 ℃保存,并将培养物进行PCR检测[6-7]。
1.2.4 PCR鉴定 按照DNA抽提试剂盒说明进行DNA提取,PCR反应体系为LATaq10 μL,上、下游引物各1 μL,DNA 2 μL,灭菌双蒸水6 μL,直接进行PCR扩增。PCR反应条件:94℃ 3 min;94℃ 30 s,52 ℃ 30 s,72 ℃ 30 s,共30个循环;72 ℃延伸10 min。
1.2.5 全基因组的扩增、克隆及序列测定 按照DNA抽提试剂盒说明进行基因组DNA提取,将提取物进行PCR,反应体系:LATaq10 μL,上、下游引物各1 μL,DNA 2 μL,灭菌双蒸水6 μL,直接进行PCR扩增。PCR扩增反应条件:94 ℃ 3 min;94 ℃ 30 s,52 ℃ 30 s,72℃ 150 s,共30个循环;72 ℃延伸10 min。取PCR产物200 μL,在10 g/L琼脂糖凝胶上电泳。将PCR产物经胶回收试剂盒纯化后与pMD18-T载体连接,转化大肠埃希菌DH5α感受态细胞,经氨苄西林筛选和菌液PCR鉴定后,取阳性菌液送上海英骏生物技术公司进行测序[8-9]。用Lasergene 7.1软件进行序列拼接,并将拼接后的全基因序列向GenBank提交,进行序列分析。
1.2.6 鹅细小病毒参考毒株 本分析中使用的细小病毒参考株见表2。
表1 本研究所用引物
Table 1 Primers used in this study

引物名称Primername引物序列(5'~3')Primersequence(5'-3')扩增长度/bpAmplificationlengthFRTATCAACAACCAYTGGGGAATTCTGCTGCTGTCTACCTCAT470A1FA1RTCATTGGAGGGTTCGTTCGTGCATGCGCGTGGTCAACCTAACAGCCGGAAA202A2FA2RGCATGCCGCGCGGTCAGCCCAATATTCGTCCTGGTTGAACTGATT585A3FA3RGTCGGAGAGATGGCACTTTCTGTGGTGGCAGGTCCGTAGAGC1028A4FA4RCTTCTTCAAATAATAGACAAGTGTAGACATATCTGCTTTGAGTC1137A5FA5RATGAACATGGGTGGTATGATTACTAGAATGCACTCCGGTCAT1297A6FA6RAACCATTGGGGAATCAGACCGCATGCGCGTGGTCAACCTAACAGCCGGAAA1574
表2 GPV全基因参考毒株
Table 2 Reference strains of GPV full genomes used in this study

毒株Virus宿主Host分离时间Yearofisolation登录号GenBank№来源GeographicoriginGPV-YG鹅Goose2001AF416726中国ChinaHG5/82鹅Goose2003AY506546中国ChinaFM番鸭Muscovyduck1980U22967匈牙利HungaryHBZF07鹅Goose2007EU022755中国ChinaLN-1/06鹅Goose2007EU253479中国(辽宁)China(Liaoning)82-0321V鹅Goose1982EU583389中国(台湾)China(Taiwan)82-0321鹅Goose1982EU583390中国(台湾)China(Taiwan)06-0329鹅Goose2006EU583391中国(台湾)China(Taiwan)VG32/1鹅Goose-EU583392欧洲EuropeB鹅Goose1960U25749匈牙利HungaryGDaGPV鹅Goose1978HQ891825中国(福建)China(Fujian)SH鹅Goose2009JF333590中国(上海)China(Shanghai)Y番鸭Muscovyduck2011KC178571中国(安徽)China(Anhui)E鹅Goose2012KC184133中国(安徽)China(Anhui)SHFX1201天鹅swan2012KC478066中国ChinaYZ99-6鹅Goose1999KC996730中国China90-0215鸭Duck2003AY382891中国ChinaGD鹅Goose2003AY512830中国(广东)China(Guangdong)SCh鹅Goose2007EU088101中国(四川)China(Sichuan)
2 结果
2.1 GPV分离鉴定
将疑似GPV感染的病料进行DNA提取后,以GPV的F/R为引物进行PCR检测(图1),然后将检测为阳性样品接种鹅胚,死亡鹅胚头部和背部出血(图2),取鹅胚培养物进行PCR检测(图3)。由图1、图3可知,检测样品有约470 bp的目的条带出现,与预期大小一致。

M.DNA标准DL 2 000;1~3.病料处理样品;4.阴性对照
M.DNA Marker DL 2 000;1-3.PCR products of treated samples;4.Negative control
图1 病料PCR扩增结果
Fig.1 PCR amplification results of samples

图2 接毒后死亡的鹅胚
2.2 全基因组特征
GPV HN株经PCR扩增后获得其全基因组序列,各片段大小分别为0.2、0.5、1.0、1.1、1.2、1.5 kb,结果与预期的片段大小一致(图4)。获得GPV HN株基因组全长为5 106 bp,并由ITR、NS、VP构成,其中NS1为1 844 bp,VP1为2 199 bp,符合GPV的结构特征,且与2011年在安徽分离的Y株以及2012年在天鹅上分离到的SHFX1201株的全基因组同源性最高,分别为99.3%和99.6%。该株病毒有左右两侧两个开放阅读框,左侧编码非结构蛋白(NS1和NS2),右侧编码结构蛋白(VP1、VP2和VP3)。其结构蛋白基因NS1(537-2 420 nt)主要编码627个氨基酸,NS2编码451个氨基酸;其衣壳蛋白基因VP1(2 439-4 637 nt)编码732个氨基酸,
VP2(2 874-4 637 nt)基因编码587个氨基酸,VP3(3 033-4 637 nt)基因编码534个氨基酸,在其右侧的ITR前有1个Poly(A)的尾。

M.DNA标准DL 2 000;1~5.培养物样品;6.阴性对照
M.DNA Marker DL 2 000; 1-5.PCR products of culture samples;6.Negative control
图3 培养物PCR扩增结果
Fig.3 PCR amplification results of culture samples

M.DNA 标准 DL 2 000;1~6.基因组扩增产物
M.DNA Marker DL 2 000; 1-6.PCR products of complete genomes
图4 HN株全基因组PCR扩增结果
Fig.4 PCR amplification result of complete genome of HN strain
2.3 GPV非结构蛋白遗传进化分析
利用Lasergene 7.1软件将拼接好的HN株的非结构蛋白基因与GenBank登录的其他鹅细小病毒非结构蛋白基因进行比较分析(表3)。从表中可知HN株与HBZF07株、82-0321株、06-0329株、B株、Y株、E株、SHFX1201株和YZ99-6株NS1基因的核苷酸同源性为99%~99.8%,推导其氨基酸的同源性为99.2%~100%。试验株与SHFX1201的同源性最高达到99.8%,与番鸭细小病毒株FM的NS1基因同源性最低为82.7%。从图5的HN株NS1基因的遗传进化树可以看出,GPV在NS1基因可以分成明显的2个基因亚群,HN株与鹅细小病毒匈牙利株(B)、欧洲疫苗株(VG32/1)和台湾株(82-0321V、82-0321、06-0329)均处在第I亚群,番鸭源匈牙利株FM 单独处于第Ⅱ亚群。HN株与安徽分离株Y以及SHFX1201株同源性最接近。

图5 HN株的NS1基因遗传进化树
2.4 GPV结构蛋白遗传进化分析
通过与其他株的对比(表4)可知试验株与82-0321株、06-0329株、Y株、E株、SHFX1201株和YZ99-6株基因的同源性为99.2%~99.7%,推导氨基酸的同源性为99.0%~99.6%,其中与SHFX1201株同源性最高,与90-0215株同源性最低。从遗传进化树(图6)可以看出HN株与分离株Y株以及SHFX1201株同源性最近。且与标准株B株和E株等处于同一基因亚群,90-0215株与FM株处于单独的一基因亚群。
表3 HN株与其他毒株的NS1基因核苷酸和推导氨基酸序列的同源性比较
Table 3 Homology comparison of nucleotide and amino acid sequences of NS1 gene of HN strain with other strains

1234567891011121314151617194.394.382.799.694.497.399.599.198.399.094.294.799.799.599.899.6298.199.881.194.399.994.294.293.794.494.399.699.494.394.194.394.2398.199.781.294.399.994.294.293.794.494.399.799.594.394.294.494.3490.889.689.682.981.282.482.982.782.982.980.981.382.782.782.982.85100.098.198.190.894.497.799.999.498.799.494.294.799.799.799.899.8698.299.899.889.898.294.394.393.894.594.499.799.594.494.294.494.3799.297.397.390.199.297.597.797.397.898.094.194.697.497.597.697.6899.897.997.990.699.898.199.099.498.799.494.194.699.699.699.799.7999.297.397.390.099.297.598.49998.398.993.694.199.299.299.399.31099.497.897.890.499.497.998.699.298.699.094.394.798.598.598.598.51199.897.997.990.699.898.199.099.799.099.294.294.899.299.299.299.21297.699.299.289.297.699.496.897.596.897.397.599.394.294.194.394.21398.499.499.490.098.499.597.698.297.698.198.698.994.794.594.794.51499.897.997.990.899.898.199.099.799.099.299.797.598.299.699.899.61599.797.897.890.499.797.998.999.598.999.099.597.398.199.599.699.716100.098.198.190.8100.098.299.299.899.299.499.897.698.499.899.799.71799.898.298.290.699.898.499.099.799.099.299.797.898.299.799.599.8
注(Note):1.HN;2.AF416726 GPV-YG;3.AY506546 HG5/82;4.BDU22967 MDPV FM;5.EU022755 HBZF07;6.EU253479 LN-1/06;7.EU583389 82-0321V;8.EU583390 82-0321;9.EU583391 06-0329;10.EU583392 VG32/1;11.GPU25749 B;12.HQ891825 GDaGPV;13.JF333590 SH;14.KC178571 Y;15.KC184133 E;16.KC478066 SHFX1201;17.KC996730 YZ99-6.
3 讨论
鹅细小病毒病是一种传染性极强的传染病,既能传染鹅也能传染番鸭,对于养殖业造成了极大的危害,因此GPV的分离鉴定、遗传进化分析以及疫苗的研制成为重点。目前,GPV的分离主要通过接种鹅胚、番鸭胚或鹅胚成纤维细胞进行。周彩显等[10]通过接种10日龄鹅胚成功分离到6株GPV;邱娜等[11]通过采用不同代次的鹅胚成纤维细胞连续培养YN株GPV的方法,获得了高滴度的鹅细小病毒细胞适应株,本试验通过尿囊腔接种番鸭胚,成功分离到1株疑似GPV,通过PCR验证为GPV,并命名为HN株。匈牙利强毒株B株、安徽Y株、中国大陆SH株、GDaGPV株在ITR区均为444 bp;欧洲VG32/1株和安徽E株都为443 bp;中国台湾地方分离株06-0329、82-0321、82-0321V在ITR区分别为418、416、381 bp;安徽Y株和E株分别为444 bp和443 bp;HN株的ITR区与安徽Y株相同,各株NS1基因区和VP1基因区相对较稳定。从以上目前分离到的不同毒株来看,GPV的结构的不同对其致病力是有影响的[12],作为GPV的免疫保护抗原,能够刺激机体产生中和抗体的VP1基因具有相对较高的保守性,其常常作为GPV分子生物学诊断以及遗传进化分析的依据[13],这都有待于进一步的研究。
表4 HN株与其他毒株的VP1基因核苷酸和氨基酸序列的同源性比较
Table 4 Homology comparison of nucleotide and amino acid sequences of VP1 gene of HN strain with other strains

1234567891011121314151617195.880.195.980.395.996.699.399.296.096.595.796.199.699.299.799.5298.079.599.679.799.793.995.595.393.593.799.899.495.895.695.795.7387.085.979.598.179.580.580.280.180.881.179.579.980.380.580.280.3498.099.285.779.799.893.995.795.593.593.799.599.295.995.795.895.8586.685.897.485.579.780.880.580.481.181.579.780.080.580.880.580.6698.199.385.899.385.793.995.795.593.593.799.699.395.995.795.895.8798.497.087.097.086.997.196.896.596.696.993.794.296.896.796.896.9899.398.187.098.186.998.298.899.696.296.695.596.099.499.399.599.6999.097.587.097.586.997.798.299.296.096.495.395.799.399.199.499.41097.596.587.496.587.396.697.797.797.898.793.493.996.396.596.396.31197.796.387.496.387.696.597.897.897.798.593.594.196.796.996.796.71297.799.785.798.985.599.096.797.897.396.296.099.495.795.595.695.61398.099.586.298.986.199.097.398.197.596.796.699.596.196.096.096.01499.698.187.298.187.098.298.899.799.598.098.197.898.199.599.899.61599.698.187.098.186.998.298.699.699.397.898.097.898.199.999.599.51699.698.187.298.187.098.298.899.799.598.098.197.898.1100.099.999.71799.398.187.098.186.898.298.599.799.297.797.897.898.199.799.699.7
注(Note):1.HN;2.AF416726 GPV-YG;3.AY382891 90-0215;4.AY512830 GD;5.BDU22967 MDPV FM;6.EU088101 SCh;7.EU583389 82-0321V;8.EU583390 82-0321;9.EU583391 06-0329;10.EU583392 VG32/1;11.GPU25749 B ;12.HQ891825 GDaGPV;13.JF333590 SH;14.KC178571 Y;15.KC184133 E;16.KC478066 SHFX1201;17.KC996730 YZ99-6.
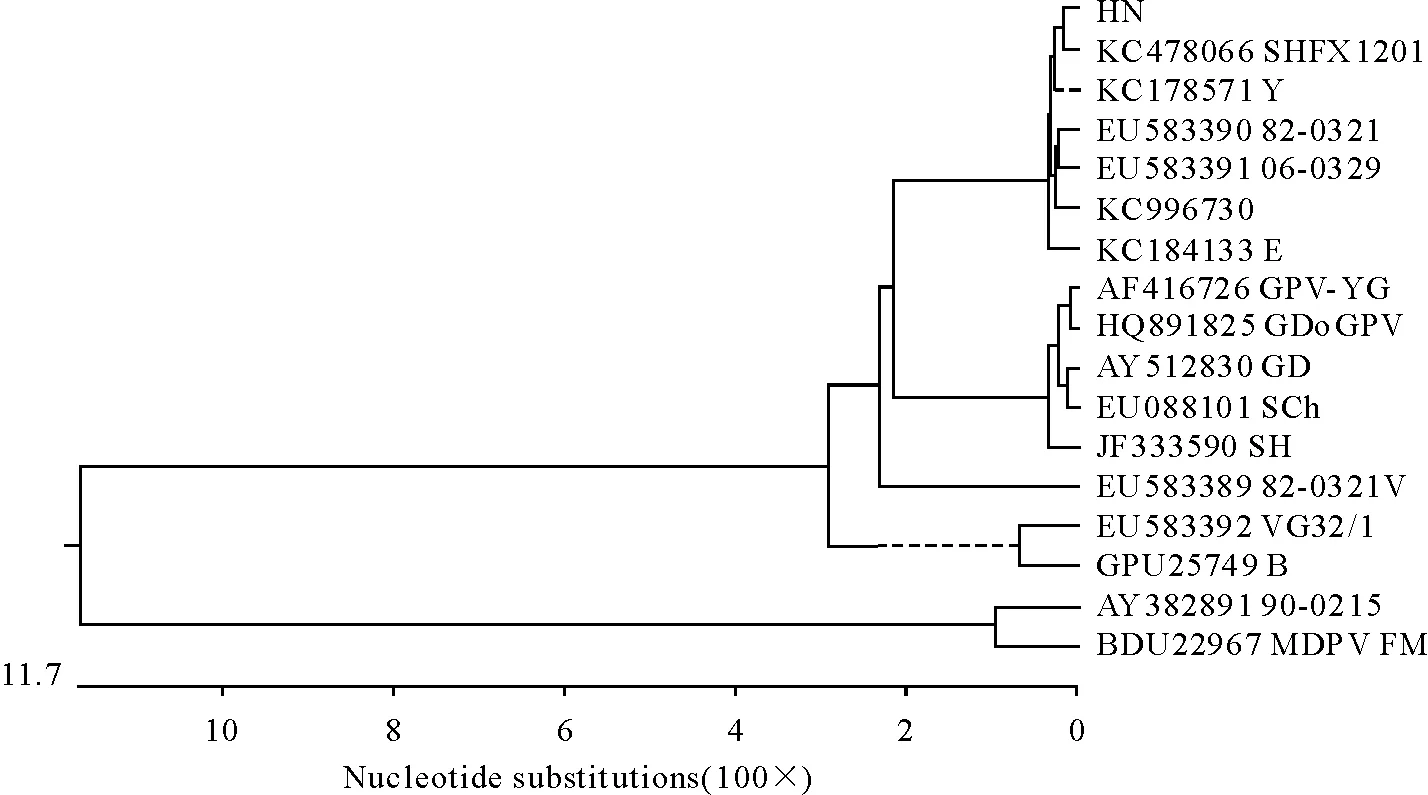
图6 HN株的VP1基因遗传进化树
本研究通过将GPV HN株与GenBank上的各GPV的非结构蛋白和结构蛋白相对比,发现NS1基因和VP1基因的遗传较稳定[14],但是HN株与两株国内南方分离株Y株和SHFX1201株同源性最高并且属于同基因亚群,造成这一现象的原因可能是近年来养鹅业和养鸭业的不断扩大,海南省从南方各地区引进鹅苗和番鸭苗,导致鹅群和番鸭群携带的GPV进入海南省而出现毒株同源性高。
海南省从20世纪70年代以来,在海口、三亚、保亭、昌江、陵水、澄迈、儋州等地先后有鹅细小病毒流行的报道,但是对鹅细小病毒的研究只是局限在诊断、防控与部分基因片段的研究,王德富等[15]对海南地区的小鹅瘟病做了诊断与防控的相关报道;曹宗喜等[16]对海南分离的3株鹅细小病毒VP3基因进行了分析。本文首次对鹅细小病毒海南分离株进行全基因组分析,丰富了我国鹅细小病毒基因组数据资料,可为海南省鹅细小病毒的防控提供理论依据。
[1] 杨焕章,王 凯,陈绍红,等.鹅细小病毒VP3亚单位疫苗的制备及其免疫效果[J].动物医学进展,2013,34(10):41-45.
[2] 张 颖,鲁 承,赵文婧,等.鹅细小病毒YBLJ株VP3基因的真核表达[J].动物医学进展,2012,33(12):23-25.
[3] 胡自立,刘海玲,张光明,等.鹅细小病毒SS/10株VP3蛋白的原核表达及鉴定[J].动物医学进展,2012,33(5):35-38.
[4] 孟繁兴,董 浩,胡桂学.鹅细小病毒分子生物学的研究新进展[J].黑龙江畜牧兽医,2015(12上):65-67.
[5] 陈翠腾,万春和,傅秋玲,等.樱桃谷肉鸭源鹅细小病毒的分离与鉴定[J].中国家禽,2015,37(23):47-49.
[6] 王 郑,徐 萍,李甜甜,等.5株鹅细小病毒分离鉴定及VP3基因遗传进化分析[J].国外畜牧学:猪与禽,2015,35(12):74-76.
[7] 王建业,黄 钰,龚建森,等.番鸭细小病毒鹅胚化弱毒株FZ91-30的全基因组克隆及序列分析[J].中国家禽,2015,37(22):46-48.
[8] 孟繁兴,董 浩,胡振林,等.鹅细小病毒吉林株的分离鉴定及其全基因序列分析[J].中国兽医杂志,2015,51(12):33-35,38.
[9] 赵瑞宏,沈学怀,戴 银,等.抗番鸭细小病毒卵黄抗体的制备[J].安徽农业科学,2015,43(29):62-63.
[10] 周彩显,吴 昊,于 晶,等.6株鹅细小病毒的分离及其VP3基因序列分析[J].中国兽药杂志,2016,50(3):1-5.
[11] 邱 娜,刘 明,白小飞,等.鹅细小病毒鹅胚成纤维细胞适应病毒株的培育及序列分析[J].中国预防兽医学报,2014,36(11):898-900.
[12] Shien J,Wang Y,Chen C,et al .Identification of sequence changes in live attenuated goose parvovirus vaccine strains developed in Asia and Europe [J].Avian Pathol,2008,37(5):499-505.
[13] Liu H M,Wang H,Tian X J,et al.Complete genome sequence of goose parvovirus Y strain isolated from Muscovy ducks in China[J].Virus Gen,2014,48(1):199-202.
[14] Zhang Y,Li Y,Liu M,et al .Development and evaluation of a VP3-ELISA for the detection of goose and Muscovy duck parvovirus antibodies[J]. J Virol Meth,2010,163(2):405-409.
[15] 王德富.小鹅瘟的诊断报告[J].畜牧兽医科技信息,2010(2):84.
[16] 曹宗喜,叶保国,林哲敏,等.三株鹅细小病毒VP3基因的克隆和序列研究[J].中国家禽,2013,35(13):16-18.
Isolation,Identification and Complete Genomic Sequence Analysis of Goose Parvovirus HN Strain
HE Dong-mei1,2,TAN Shu-yi1,CHEN Chao-xi2,YE Bao-guo1,ZHANG Yan1,LIN Zhe-min1,CAO Zong-xi1
(1.InstituteofAnimalHusbandryandVeterinaryMedicine,HainanAcademyofAgriculturalSciences,Haikou,Hainan,571100,China; 2.CollegeofLifeScienceandTechnology,SouthwestUniversityforNationalities,Chengdu,Sichuan,610041,China)
To investigate the variation status of goose parvovirus,the samples(liver,spleen and so on) of suspected GPV infection were collected from a goose farm of Hainan province.A virus strain was isolated by inoculation of goose embryo after treatment,and named as HN strain after PCR. The complete genomic sequence was obtained,and compared with the sequences of sixteen strains of goose parvoviruses and Muscovy duck parvovirus in GenBank.The results showed that the full-genome contains the inverted terminal repeat (ITR) and two major open reading frames (ORF).ITR at the 5′ and 3′ ends of the genome has 444 nucleotides (nt). The left ORF contains 1884 nts in length,the right ORF contains 1 884 nts in length. The nucleotide sequences of HN strain showed the highest homology with NS1 gene and VP1 gene of SHFX1201 strain,with 99.8% and 99.7%,respectively.And the homologies with the NS1 gene of FM strain and VP1 gene of 90-0215 strain were lower,with 82.7% and 80.1%,respectively.Phylogenetic tree of HN strains demonstrated that GPV can be obviously divided into two gene subsets.HN strain and B strain from Hungary and VG32/1 strain from European are located in the subgroup I,and the high homology with Y strain isolated from Anhui and SHFX1201 strain,the Muscovy duck parvovirus of FM strain isolated from Hungary located in the subgroup II.This study provided a basis for the study of the relationship between GPV classification status and genetic evolution,and laid the foundation for the study of GPV epidemic trends and vaccine development.
Goose parvovirus; isolation and identification;complete genome; IRT; NS1; VP1
2016-05-23
海南省属科研院所技术开发研究专项(KYYS-2015-13);海南省自然科学基金项目(20153085);现代农业产业技术体系专项资金项目(CARS-43)
贺冬梅(1988-),女,四川简阳人,硕士研究生,主要从事动物病毒学研究。*通讯作者
S852.659.2
A
1007-5038(2016)12-0013-06