一种基于计算机的单核苷酸多态性位点检测方法
2014-12-07贾奇男易青青贺建峰
马 磊,贾奇男,张 俊,易青青,贺建峰,张 琪
(昆明理工大学信息工程与自动化学院生物医学工程研究所,云南昆明650500)
单核苷酸多态性(single nucleotide polymorphism,SNP)是指在染色体基因组水平上单个核苷酸的变异引起DNA序列多态性,它包括单碱基的转换,颠换、插入及缺失等形式,SNP位点的检出可以在一定程度上预测个体在自然选择过程中的演变程度。目前,SNP位点检测方法主要有单链构象多态性分析(single strand conformation polymorphism,SSCP)[1],变性梯度凝胶电泳(denatured gradient gel electrophoresis,DGGE)[2],及其他一些常见方法[3-6]。但是这些方法均具有耗时长,过程繁琐和技术难度大,费用高等缺点,从而制约了SNP的研究和发展。近年来,出现了一些利用生物信息技术等简单易行的方法,进行SNP位点筛选分析的研究[7],但仍存在应用范围较小等问题。
本研究提出了一种基于计算机的SNP位点检测方法,并以HBV的演化为例,应用所提出的方法,通过特征信息的提取,剔除冗余的非疾病基因或非疾病风险基因,采用基于最优风险与预防模式的数学算法,研究个体被HBV感染的风险性及被感染后疾病的演变程度,结论可为临床诊断提供参考。
1 方法
1.1 基于信息熵的特征属性值项的提取
在信息增益的DNA序列中,特征信息选择的衡量标准是判断该位置上的特征基因为目标属性带来的信息量,信息越多,该特征基因就越重要。对一个特征基因而言,特定位置上它存在和不存在时信息量将发生明显变化,而前后信息量的差值就是这个特征基因给该位置带来的信息量,也称为熵[8]。
假如有变量X,其可能的取值有n种,取到每一种的概率为pi,那么X的熵值计算公式1为:
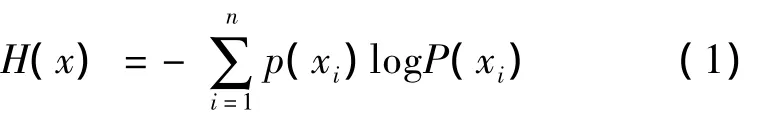
X可能的变化越多,X所携带的信息量越大,熵也就越大。所以特征信息T给聚类C或者分类C带来的信息增益为公式2:
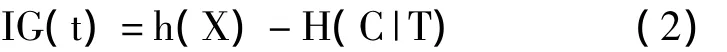
这里包含两种情况:一种是特征T出现,标记为t,一种是特征T不出现,标记为t'。所以计算见公式3所示。
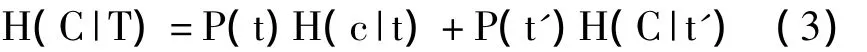
由公式2可知特征与类别的信息增益公式。
该方法可在WEKA工作台上执行和实现[9]。
由于基因信息数据量大以及生物学数据本身的复杂性,导致了对这类数据进行信息挖掘非常困难。因此在进行特征基因选择之前,首先将数据源进行格式处理。数据源映射图如图1所示。sequences是样本号对应的每条序列,attribute1至attribute3221表示每条序列对应位置上的碱基,class表示每条序列对应的类属性。
1.2 最优风险与预防模式
最优风险与预防模式算法MORE[10]的核心思想是采用局部支持度(supp)[11]来挖掘频繁模式,其次应用流行病理学中常用的相对风险值(RR)[12]度量指标产生最优风险与预防模式。这里简要介绍局部支持度(lsupp)、相对风险值与比值比的概念及计算方法。

由于在实际的医疗数据集中,数据量很大且正反类事例严重不平衡,因此采用局部支持度作为风险模式的支持度,即滤除非患病序列样本。可以表示为样本中同时出现模式P和a的概率与样本中只出现a的概率的比值。其计算如公式4所示:
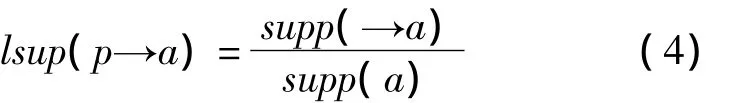
式中supp(p→a)表示模式P的支持度,即同时出现模式P和a的概率。局部支持度满足反单调性,它的含义为:一个超集的支持度小于或等于它的任一子集的支持度。最优风险与预防模式满足反单调性原则就是最优风险与预防模式能够被挖掘的原因。在本研究中,如果一个模式的局部支持度大于给定的阈值,则这个模式就是频繁的。
相对风险值表示某种疾病在特定人群中发生率是在非特定人群中发生率的多少倍。RR存在阈值的选取,例如阈值为1,则RR=1,表明此模式既不是风险模式也不是预防模式,此模式中的属性对患者患病没有影响;RR大于1,则说明此模式为风险模式,此模式中的属性为风险因子;RR小于1,表示此模式为预防模式,此模式中的属性为预防因子。相对风险值(relative risk,RR)的计算如公式5所示。

表1 模式产生的可能性与输出结果Table 1 The results and possibility of patterns
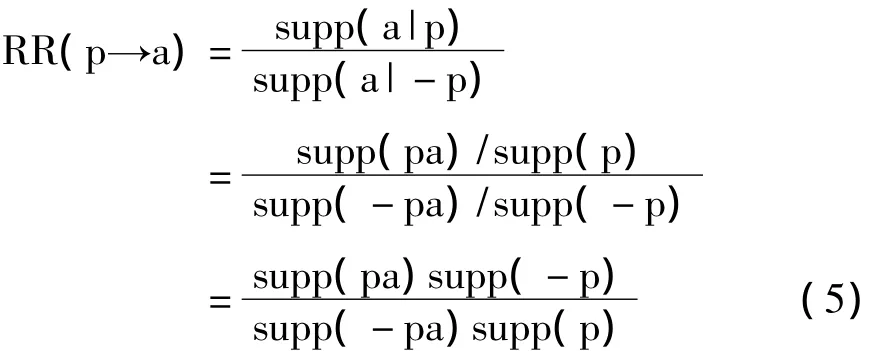
在表1中,-p表示模式P没有发生的所有记录。supp(-p)不在P模式下的所有记录,-pa是包括a但不在P模式下的所有记录。supp(p,a)表示supp(p→a)。
1.3 基于最优风险和预防模式集的特征属性值项差异分析
由于通过最优风险与预防模式挖掘出的疾病相关模式或无关模式有时相互矛盾。例如,如果某些模式中的特征属性值项同时出现在最优风险和最优预防模式中,有可能会给临床诊断带来偏差。针对该问题,假设最优风险和预防模式中每个特征属性值项是相互独立的,然后利用最优风险模式中的特征属性值项形成风险集,利用最优预防模式的特征属性值项产生预防集,并统计风险集和预防集中的特征属性值项的频率,去除相等频率值的公共特征属性值项。同时分别生成最优风险频率项集和最优预防频率项集。最优风险频率项集中的特征属性值项为风险因子,表示患者携带该基因并有患病风险。最优预防频率项集中的特征属性值项为预防因子,表示患者携带该基因不具有患病风险。最后对每一个特征属性值项的频率归一化处理,得该特征属性值项的权重值,总权重为100。
2 结果
2.1 实验数据源
本研究使用的数据是从 Genbank[13]下载。样本集包括FJ349205-FJ349241,EU859930-EU859937和EU859939-EU859956共40条序列,包括9例急性乙型肝炎患者,22例慢性乙型肝炎患者,9例肝硬化患者。在乙型肝炎病毒序列中,实验的目的是挖掘HBV的SNP位点即单突变位点,因此这里将所有HBV序列的每一个垂直列映射为特征属性进行数据处理。HBV序列数据类型分为3类,即急性(acute)、慢性(chronic carrier)和肝硬化(cirrhosis)。
2.2 最优风险和预防模式的生成
如前所述,将其应用到乙型肝炎转化关系研究中。由于实验数据源HBV序列经过多条序列比对后长为3 221 bp,则映射为3 221个垂直列。为了尽可能获得更多的最优风险与预防模式,对模式长度和相对风险阈值进行了多次选取实验,最后选定了一个最佳方案即在乙型肝炎转化关系研究中,设置模式长度为6,特征属性的阈值为0.20,相对风险阈值为1。在此条件下,实验共返回400个最优风险与预防模式,分别为36个最优风险模式和364个最优预防模式。限于篇幅,这里只列举了部分具有代表性的最优风险模式(表2)和最优预防模式(表3)。

表2 在局部支持度为0.05,模式长度为6,特征属性选取阈值为0.20,相对风险阈值为1的条件下由HBV序列生成的部分最优风险模式Table 2 lsupp=0.05,pattern length=6,threshold of feature property=0.20,RR threshold=1,selected optimal risk patterns are generated from HBV sequence
针对表2和表3的部分实验结果,以最优风险模式中的Pattern 1为例解释说明。模式中length=1表示模式长度为1,说明此模式包括1个特征属性值项,RR=3.6667表示相对风险值为3.6667。
2.3 乙肝病毒序列的差异分析
在此实验中,假设模式中每一个特征属性值项是相互独立的,HBV序列的特征属性值项的最优风险与预防权重如表4和表5所示,表示为最优风险集和最优预防集中各特征属性值项的权重。每个特征属性值项的权重来自它们在最优风险与预防集中的百分比。它可以了解某个特征属性值项对患者患某种疾病的风险性与预防性。
表4中,特征属性值项attribute2531=T出现在最优风险频率集中,attribute2531对应在HBV序列中的位置为第2531位碱基位点。风险权重为10.4427,是最优风险集中最大的风险权重,表明attribute2531在HBV序列第2531位碱基为T时发生急性乙肝转慢性乙肝的可能性在所有特征属性值项中最大,这些是导致此处发生碱基突变的决定因素。attribute2567=T出现在最优预防集中,且预防权重为8.9241,表明attribute2567在 HBV序列第2567位碱基为T时不发生急性乙肝转慢性乙肝的可能性很大,是此处防止碱基突变的决定因素。如果某一属性同时出现在最优风险集与最优预防集中,说明此处为决定急性转慢性的候选SNPs位点。
根据上述表述,基于表4中最优风险权重集,实验共检测出18处急性转慢性候选SNPs位点,其中4 处属于碱基替换突变(nt1655,nt1753,nt1762,nt1764),1处属于碱基缺失突变(nt1896),这5处均已在前期的文献中报道过[14-15]。其余13处是新发现的候选SNP位点(其中nt2576为替换突变,其他12处为缺失突变)。基于表5中最优风险权重集,在实验中共检测出3处慢性乙型肝炎转肝硬化候选SNPs位点,其中2处属于碱基替换突变(nt2159、nt902),1处属于碱基缺失突变(nt507)。
3 讨论
近年来SNP的检测方法已被广泛研究,国内外专家学者也相应提出了多种方法检测SNP,但是大多需要依赖昂贵的仪器或专业人员的技术支持,且操作相对复杂。本研究针对40条HBV病毒序列数据(9条急性乙型肝炎序列,22条慢性乙型肝炎序列,9条肝硬化序列)提出了一种基于最优风险与预防模式算法来研究HBV病毒序列的SNP位点检测问题。实验共检测出18处急性乙型肝炎转慢性乙型肝炎候选SNPs位点,其中5处已有报道,另外13处是新发现的候选SNP位点。同时检测出3处慢性乙型肝炎转肝硬化候选SNPs位点。该方法成本低廉,操作简便,并能在庞大的基因数据中选出SNP位点,可以对乙型肝炎的临床诊断和生物医学研究起到有益的参考和借鉴作用,有可能成为适用于临床针对肝炎及其他疾病的SNPs检测方法。

optimal risk and preventive pattern(supp=0.43,info gain>0.15,pattern length=7)property in risk pattern property in preventi ve pattern risk factor loci weight preventive factor loci weight attribute2531=T nt2531 10.4427 attribute2567=T nt2567 8.9241 attribute1896=G nt1896 9.2698 attribute1773=T nt1773 8.0144 attribute1588=A nt1588 7.3401 attribute3090=A nt3090 7.0107 attribute2689=T nt2689 6.8104 attribute1421=G nt1421 5.756 attribute1320=C nt1320 6.5834 attribute1657=C nt1657 5.458 attribute1655=T nt1655 6.3942 attribute49=G nt49 5.3795 attribute1762=T nt1762 6.3564 attribute1764=G nt1764 5.0502 attribute681=C nt681 5.1457 attribute2576=T nt2576 4.5483 attribute3073=T nt3073 4.7673 attribute1999=A nt1999 4.3444 attribute3094=C nt3094 4.5781 attribute1334=C nt1334 4.313 attribute1764=A nt1764 4.0484 attribute917=G nt917 4.313 attribute1753=T nt1753 4.0484 attribute3129=A nt3129 4.2033 attribute3094=C nt3094 3.3674 attribute1762=T nt1762 4.1248 attribute2819=A nt2819 3.1025 attribute600=C nt600 4.1248 attribute2687=G nt2687 2.9512 attribute2260=A nt2260 3.9837 attribute2576=G nt2576 2.6485 attribute8=A nt8 3.3407 attribute2588=C nt2588 2.4593 attribute2717=T nt2717 3.2465 attribute681=C nt681 2.1188 attribute9=A nt9 3.2152 attribute1655=C nt1655 3.2152 attribute513=C nt513 3.074 attribute1753=A nt1753 2.3683 attribute896=T nt896 1.9918
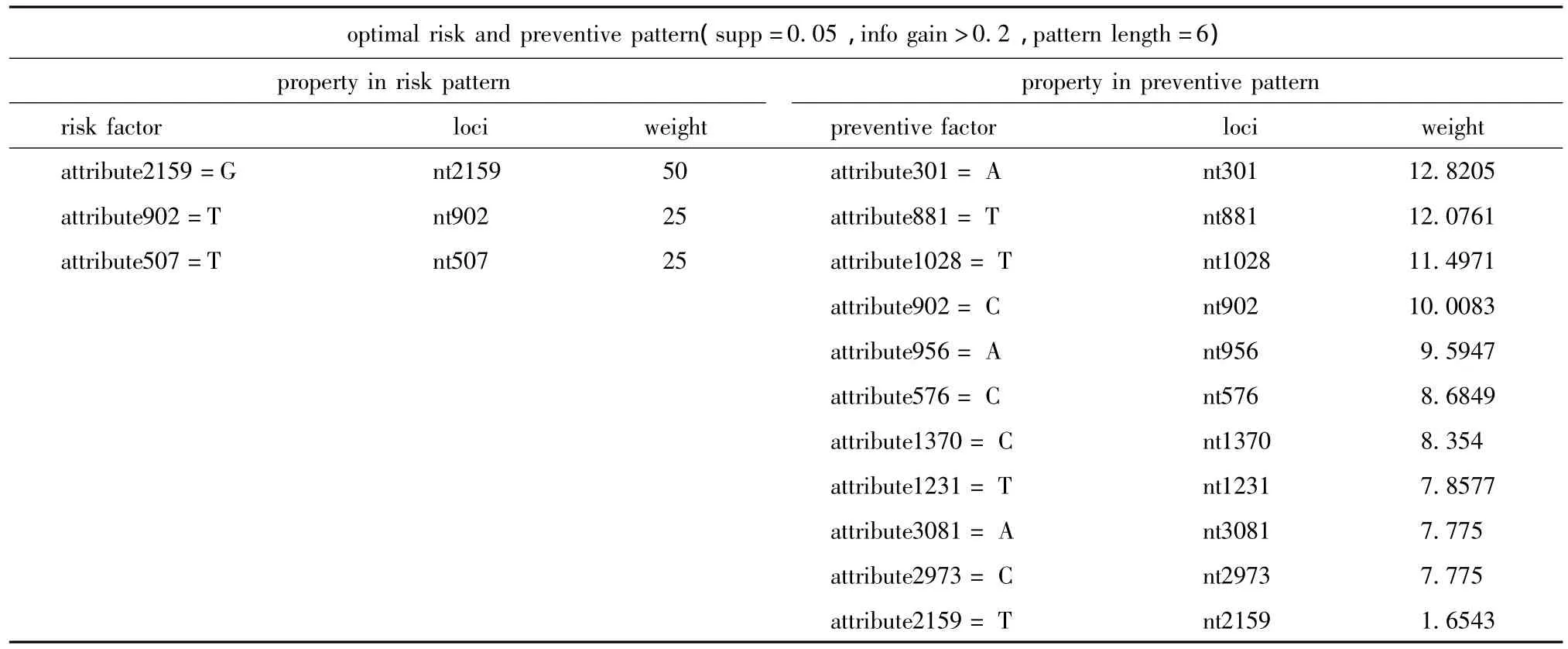
表5 慢性乙型肝炎与肝硬化数据生成的最优风险集与预防集中的每个特征属性的频率并按照降序排列,每一个位置对应的是HBV序列中的碱基位置Table 5 The descending ranking of frequency of each feature p?roperty in the optimal risk and preventive sets generated from the data of chronic HBV carrier and cirrhosis
[1]Kubo KS,Stuart RM,Freitas-Astúa J,et al.Evaluation of the genetic variability of orchid fleck virus by single-strand conformational polymorphism analysis and nucleotide sequencing of a fragment from the nucleocapsid gene[J].Arch Virol,2009,154:1009 -1014.
[2]Drabovich AP,Krylov SN.Identification of base pairs in single-nucleotide polymorphisms by MutS protein-mediated capillary electrophoresis[J].Anal Chem,2006,78:2035-2038.
[3]Jiang HH,Huang SY,Zhou DH,et al.Genetic characterization of Toxoplasma gondii from pigs from different localities in China by PCR-RFLP[J].Parasites & Vectors,2013,6:227.
[4]Dunnen JT,Antonarakis SE.Mutation nomenclature extensions and suggestions to describe complex mutations:A discussion[J].Human Mutat,2000,15:7 -12.
[5] Sapolskya RJ,Hsie L,Berno A,et al.High-throughput polymorphism screening and genotyping with high-density oligonucleotide arrays[J].Elsevier,1999,14:187 - 192.
[6] Gross E,Arnold N,Goette J,et al.A comparison of BRCAI mutation analysis by direct sequencing,SSCP and DHPLC[J].Human Genet,1999,105:72 -78.
[7]陈客宏,曾灵,顾玮等.利用生物信息学技术挑选TLR2基因标签单核苷酸多态性位点[J].基础医学与临床,2010,30:242-245.
[8] Cover TM,Thomas JA.Elements of information theory[M].Wiley-interscience,2006.
[9]Hall M,Frank E,Holmes G,et al.The WEKA data mining software:an update[J].SIGKDD Explor Newsl,2009,11:10-18.
[10]Li JY,Fu AW,He HX,et al.Efficient discovery of risk patterns in medical data[J].Artif Intell Med,2009,45:77-89.
[11]Ohsaki M,Kitaguchi S,Okamoto K,et al.Evaluation of rule interestingness measures with a clinical dataset on hepatitis.In:Boulicaut J-FF.,Esposito F,Giannotti F,Pedreschi D,editors.Proceedings of the eighth European conference on principles and practice of knowledge discovery in databases[M],New York:Springer-Verlag,2004:362-373.
[12]Triola MM,Triola MF.Biostatistics for the biological and health sciences[M],Boston:Addison-Wesley,2006.
[13]Benson DA,Cavanaugh M,Clark K,et al.GenBank[J].Nucleic Acids Res,2013,41:36 -42.
[14]Yuan JM,Ambinder A,Fan Y,et al.Prospective evaluation of hepatitis B 1762T/1764A mutations on hepatocellular carcinoma development in Shanghai,China[J].Cancer Epidemiol Biomarkers& Prevention,2009,18:590-594.
[15]Wang Y,Liu H,Zhou Q,et al.Analysis of point mutation in site 1896 of HBV precore and its detection in the tissues and serum of HCC patients[J].World JGastroenterol,2000,6:395-397.
