北京大气降水中细菌气溶胶的多样性研究
2014-04-26梁宗敏杜鹏瑞王亚玲李梓铭中国科学院大学资源与环境学院北京100049
梁宗敏,杜 睿,杜鹏瑞,王亚玲,李梓铭 (中国科学院大学资源与环境学院,北京 100049)
北京大气降水中细菌气溶胶的多样性研究
梁宗敏,杜 睿*,杜鹏瑞,王亚玲,李梓铭 (中国科学院大学资源与环境学院,北京 100049)
利用16S rRNA基因测序分类学技术,分别以北京市区2011及2012年不同月份的降水样品中细菌的基因组DNA为模板,通过克隆、测序构建基因组文库,研究了北京市大气降水中细菌的群落结构组成及多样性变化.系统发育分析结果表明,变形菌门(Proteobacteria) (α-,β-,γ-)是北京市降水样品中细菌的优势菌群(75.6%~100%),另外包括拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、异常球菌门(Deinococcus-Thermus)、蓝藻门(Cyanobacteria)、硝化螺菌门(Nitrospira)、厚壁菌门(Firmicutes)共7个主要门类的细菌,以及未定菌(TM7).多样性指数分析结果显示,不同的降水样品,细菌群落结构组成及多样性均存在着差异性,冬季 12月份雪水样品细菌群落结构多样性明显高于其他季节的样品,细菌群落多样性(Shannon, H)特点是,冬季>秋季>夏季.
降水;细菌;群落结构;多样性;16S rRNA
大气水环境代表着一种极端的生存环境,低温、低压、高辐射、相对较低的pH值以及含有有机物与无机物相混合的复杂混合物[1].然而细菌不仅可以在这种环境下生存[2-3],而且还可以调节云与降水的理化性质[4].大气中的细菌通常吸附在尘埃粒子表面而悬浮在大气中. 湿清除是大气气溶胶粒子的主要清除机制之一,它是指大气气溶胶粒子参与云滴的形成,并在云滴增长和发展为降水的过程中,被云、降水收集而随降水下落至地面,包括云内清除和云下清除两个过程[5].降水过程能够明显冲刷和净化大气中的细菌等微生物气溶胶[6-7].
大气生物气溶胶与城市空气污染、城市环境质量以及人类健康密切相关[8-9].城市生态系统中,大气中微生物状况是城市环境综合因素的集中表现,是评价城市空气质量的重要指标[10].近年来,国际大气科学重新开始关注生物气溶胶,尤其是细菌作为生物冰核在气候变化、降水形成与分布过程中的作用及贡献.已有的研究表明:细菌、真菌、病毒以及藻类等生物气溶胶粒子可作为云凝结核(CCN)和冰核(IN),参与大气云物理和化学过程,影响大气降水、大气化学以及微生物的地球化学循环[11-14].云中的异质核化过程是影响大气降水形成的关键因素之一,而已经发现的冰核细菌(可以在-10℃以上较温暖的温度条件下催化液滴产生冰核的细菌[15])如Pseudomonas syringae,是被公认的冰核活性最强的菌种[16],研究发现它可在-2℃冻结[17].
为能更有效地反映大气中的微生物群落结构,了解降水中微生物的群落变化机制,尤其是了解细菌的群落结构与多样性的变化特点非常必要.已有国内外学者通过培养的方法获得的大气微生物气溶胶中细菌的群落结构组成与多样性
[18-19].然而,大气中可培养细菌的量占总量的比值仅为 1%左右,对于细菌等微生物多样性的认知,存在很大程度的不足[20].近些年,分子生物学技术在研究环境样品微生物的多样性及群落结构中,已发展成为一种成熟且有效的方法[21-23].本研究是基于16S rRNA基因的分子生物学技术手段,通过多样性分析与系统发育分析,对北京城市环境中2011年和2012年不同月份降水中细菌的群落结构组成与多样性进行的了研究,旨在描述北京市降水中细菌的群落结构、物种多样性以及不同季节的变化特征.
1 材料与方法
1.1 采样点
大气降水样品于2011年(8、12月)与2012年(5、6、7、8、9月)分别采集于北京市中国科学院大学教学楼二楼楼顶(39°54′N,116°14′E,高度约 10m)与北京市中国科学院大气物理研究所实验楼楼顶(39°55′N, 116°26′E,高度约 40m),两者分别地处北京市西五环与北四环,附近为科研楼以及居民生活区.
1.2 样品
1.2.1 样品采集 2011年8月份采集的样品(标记为B18)、12月(B112,雪),为保证每次实验所需的雨水量,同时使用多个 3L玻璃烧杯收集雨水,杯口处加置可以增加接水面积的不锈钢漏斗.每月多次收集雨水,每次收集时间不超过 24h,所接雨水尽快移至 4℃冰箱保存直至累积接雨量到1.5L左右开始过滤.
2012年5~9月的样品(分别标记为B5, B6, B7, B8, B9)采集使用的是长沙湘蓝科学仪器有限公司生产的降水自动采样器(APS-2B).除在7、8月份的雨季是24h内降水过程的雨水累积样品,其他月份的雨水样品通常是每月中多天次降水的累积样品.以上实验器材等用品使用前均经过严格的高压蒸汽灭菌处理.
1.2.2 样品处理 使用真空抽滤泵(津腾公司)与硝化纤维素滤膜(孔径 0.22μm,直径 47mm,美国 Millipore公司生产)进行雨水过滤,过滤雨水量为 1L.过滤之后的膜置于-20℃保存,雨水原液、滤液各取100mL于4℃保存.
1.3 样品的处理
1.3.1 DNA的提取 在超净台中,把每个滤膜剪成碎片,分别装入 2mL的离心管中,使用 FastDNA SPIN试剂盒(MP Biomedicals公司,美国),按照操作说明提取DNA.提取的DNA用1%的琼脂糖凝胶电泳(TBE缓冲液)检测,然后-20℃保藏.
1.3.2 PCR扩增 以上述提取的总 DNA作为扩增模板.采用细菌16S rRNA基因片段上的通用引物 27f(5'-AGAGTTTGATCCTGGCTCAG-3')和1492r(5'-GGTTACCTTGTTACGACTT-3')[24]对降水样品进行PCR扩增.PCR仪为美国Bio-Rad公司生产50μL的反应体系:200μM d NTP (Takara), 0.2μmol/L引物(Invitrogen),10×buffer (Takara), 2.5U Taq DNA聚合酶(Takar-a), 8% (W/V)BSA (美国).反应程序:95℃预变性5min;95℃变性1min;55℃退火1min;72℃延伸1.5min,35个循环; 72℃延伸
10min.
1.3.3 克隆与测序 PCR产物使用 GeneJET PCR试剂盒(Fermentas,美国)按照操作说明进行纯化,然后克隆;PCR纯化产物与 pGEM-T Easy Vector (Promega,美国)在T4连接酶及缓冲液的作用下连接,4℃静置 16h;连接体系按照操作说明转化到大肠杆菌 DH-5α(Takara)中,LB培养液中摇菌培养1.5h,然后把菌液涂布到含有100μg/mL氨苄青霉素的LB平板上;筛选白色菌落采用M13f (5'-GTTTTCCCAGTCACGAC-3')和 M13r(5'-CAGGAAACAGCTATGAC-3')引物做菌落PCR,然后随机挑选出 100个左右的阳性菌落,交由上海美吉生物医药科技有限公司进行测序.
1.4 系统发育分析
1.4.1 系统发育多样性分析 利用 Mallard软件(http://www.bioinformatics-toolkit.org/Mall ard)对测序所获得的16S rRNA序列片段进行嵌合体检测,去除嵌合体.利用 Mothur软件(http://www. mothur.org/wiki/OTU-based_approa-ches)按照不小于97%的相似性划分成一个OTU(Operational Taxonomic Units)的原则,对样品序列进行 OTU的划分.利用RDP在线分类程序对处理好的16S rRNA序列片段进行初步的系统分类.把处理之后有效的16S rRNA序列片段提交到GeneBank (http://www.ncbi.nlm. nih.gov/),获取序列号.最后,使用Mothur生成稀有度曲线以及群落系统发育树. 1.4.2 物种多样性指数分析 物种多样性指数(Shannon’s diversity indices, H),物种丰度(Species richness, Chao 1),稀有度曲线(Rare-faction curves)与覆盖率(Coverage)分别采用不同的Mothur程序(http://www.mothur.org/wiki/Shannon; http://www.mothur.org/wiki/Chao;http://www. mothur.org/wiki/Rarefaction.sin;http://www.mothur. org/wiki/Coverage)分析.
1.5 核酸序列提交号
将处理后的有效序列提交至GenBank数据库中,获得的登录号为KF010653-KF010774.
2 结果与讨论
2.1 16S rRNA序列多样性指数分析
对472个克隆序列进行16S rRNA基因序列分析,分别来自 7 个不同样品的克隆文库:B5(69个克隆),B6(57 个克隆),B7(82 个克隆),B8(67 个克隆),B9(93个克隆),B18(59个克隆),B112(45个克隆).以序列相似性大于或者等于97%划分为同一个 OTU 的标准进行多样性分析,共有 113 个OTUs (表1).
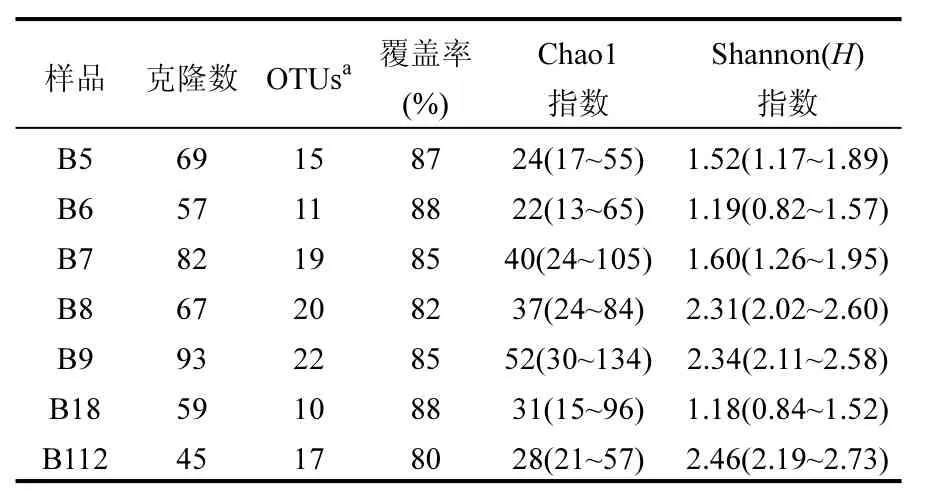
表1 降水中细菌16S rRNA基因文库生物多样性指数与物种丰度评估Table 1 Biodiversity indices and richness estimators in the clone libraries based on 16S rRNA gene of the precipitation samplers
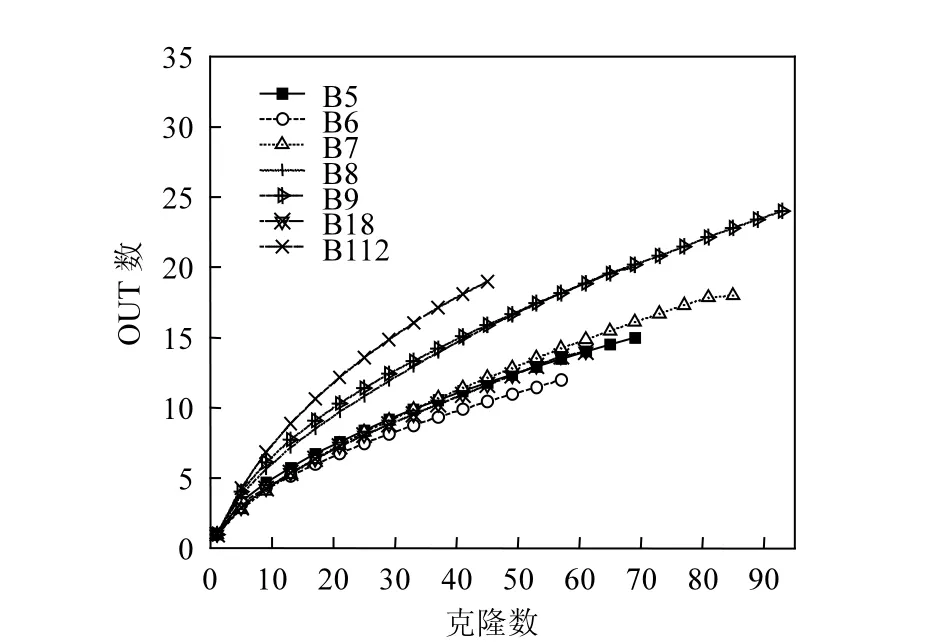
图1 克隆文库中基于OTUs的稀有度曲线Fig.1 Rarefaction curves of observed OTUs richness in the clone libraries
多样性分析包括:稀有度曲线,群落系统发育树,物种丰度,多样性指数以及覆盖率.从7个不同样品划分的OTUs与预期OTUs来看,样品中细菌具有很高的多样性.覆盖率(80%~88%)表明样品测序结果已包含了主要的细菌优势种群,这同基于物种丰度计算出来的稀有度曲线是一致的(图 1).另外,不同月份降水中的细菌群落多样性存在一定的差异.群落多样性指数(H)以及物种丰度指数(Chao l)表明,2012年5~9月降水中的细菌群落多样性有逐渐增加的趋势;2011年8月份降水中细菌群落多样性比同年 12月份的要低;2011年与2012年同期8月份降水中的物种多样性存在一定的差异.
2.2 降水样品中细菌群落结构分析
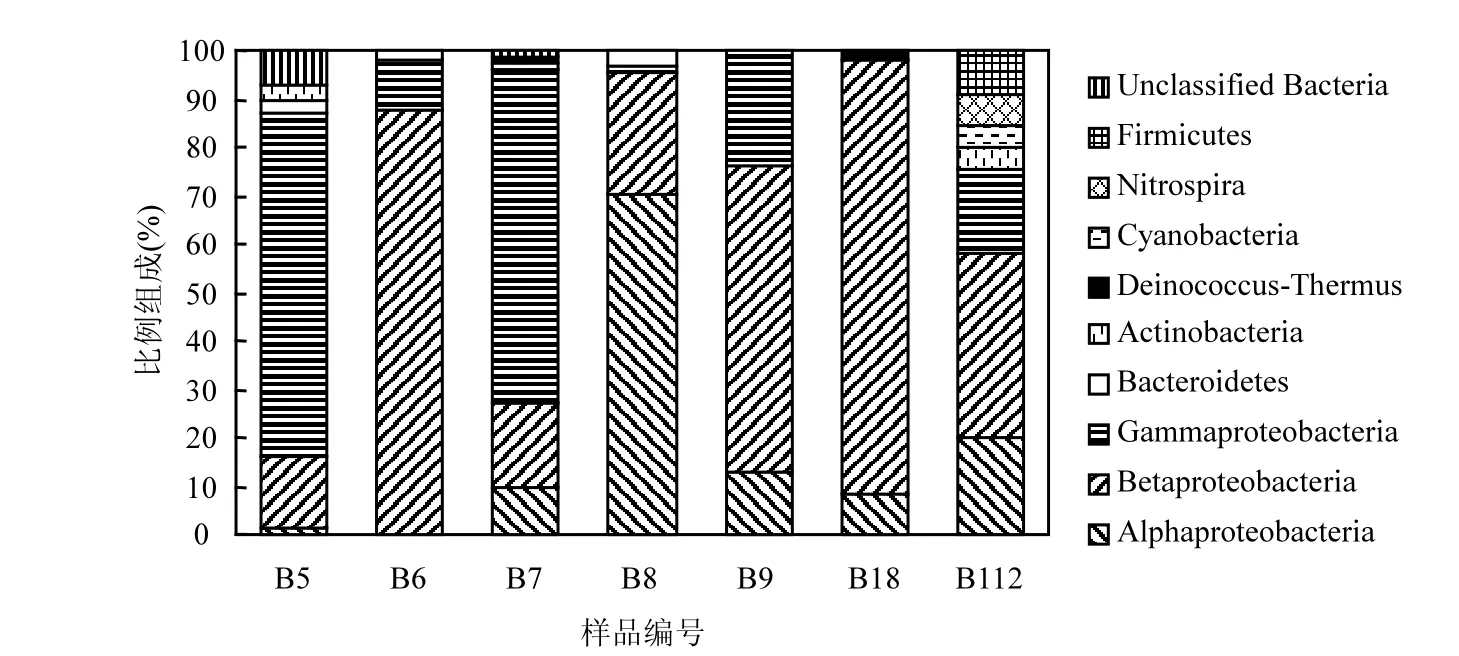
图2 细菌群落结构比例组成Fig.2 Proportion of bacteria community structure
各样品细菌群落结构比例如图2所示.变形菌门(革兰氏阴性,G-)是优势菌群,在 B5,B6,B7, B8,B9,B18,B112中各占比例分别为87.0%,98.2%, 96.3%,97%,100%,98.3%,75.6%;不同时期样品中变形菌纲(α-,β-,γ-)的比例各不相同,不同年份同时期的 B8(α-70.1%,β-25.4%,γ-1.5%)与 B18 (α-8.5%,β- 89.8%)细菌的比例组成也存在很大的差异.细菌群落结构分析结果表明,革兰氏阴性菌(G-)明显要多于革兰氏阳性菌(G+),这与国内外学者在不同环境中的研究结果一致,通过可培养的方法获得细菌 G+均多于 G-[18-19],而通过非培养的方法所获得的细菌则 G-多于 G+[25-27],这也符合 G-细菌可生活在低气温,甚至嗜冷环境中
[18]以及对辐射有抗性[28]的结论.
北京市大气降水中的细菌具有很高的多样性与丰富的群落结构组成,本研究发现了 8个主要门类的细菌:变形菌门、拟杆菌门、放线菌门、异常球菌门、蓝藻门、硝化螺菌门、厚壁菌门以及未定菌门.变形菌门主要包括α变形菌纲,β变形菌纲,γ变形菌纲以及未分类的变形菌纲,其中β变形菌纲和γ变形菌纲属于优势菌亚群,这与国内外研究一致[25,29],以上研究表明,变形菌门类微生物适合在大气水环境中生存.Fang等[19]和Gonzalez等[30]通过可培养的方法检测北京市大气中的细菌,所得到的只有变形菌门、厚壁菌门和放线菌门,说明可培养方法用于了解环境样品细菌的多样性,其灵敏度要低于分子生物学方法.另外,降水中存在一定量的未知菌(TM7,1.2%~7.2%),表明即使通过分子生物学方法,降水中仍有一部分细菌未被我们所认知.
目分类水平的细菌群落结构组成见表 2,α变形菌纲中微生物主要有柄杆菌目(Caulobacterales),根瘤菌目(Rhizobiales),红杆菌目(Rhodobacterales),螺 菌 目 (Rhodosperillales) 鞘 脂 单 胞 菌 目(Sphingomonadales),其中根瘤菌目(Rhizobiales)是α变形菌纲中的优势菌群.β变形菌纲中含有的微生物优势菌群是伯克氏菌目(Burkholderiales),同时,还有一定量未分类的微生物类群.γ变形菌纲中的微生物主要由潜在病原菌组成,除B18样品外均发现了假单胞菌目(Pseudomonadales).根瘤菌目常生活在土壤环境中,土壤是大气微生物气溶胶的一个重要源,降水样品中存在丰富的根瘤菌(例如 B8),可能是受到某一次人为或者自然活动的影响,同时也表明了大气中微生物的群落结构组成易受人为或自然活动的影响.假单胞菌目是寄生在植物上潜在的病原菌,大气中普遍存在该类细菌,说明植被区域是大气微生物来源的稳健类型之一[5].
2.3 细菌群落结构与多样性的季节性变化
聚类树聚类在同一个分支上的样品相似性较高,因此样品 B5、B6、B7、B8、B9、B18、B112之间聚类分析结果(图 3)表明:同一季节内降水中微生物的群落结构相似性较高,如夏季样品B5、B6、B7;秋季样品B8、B9;冬季样品B112单独聚类在一个分支,而且群落结构相似性相比较秋天来看比夏天高;另外,2011年秋季样品B18也是单独聚类在一个分支,位置处在同年冬季样品B112与次年秋季样品B8、B9之间.这些现象表明降水中细菌的群落结构与月份、季节以及年份相关.这也验证了把大气降水样品按照季节性划分来分析细菌的群落结构组成与多样性的可行性与正确性.同一季节内的微生物群落结构相似性较高,像夏季的B5、B6、B7;秋季的B8、B9.不同季节的细菌群落差异性显著,像冬季(B12),秋季(B8,B9)与夏季(B5,B6,B7),它们的聚类结果完全在不同的分支结构上.另外,相同月份不同年份的样品分析结果也存在差异性,B8,B18多样性指数与物种丰度指数均差别较大,而且聚类结果相似性较差,表明不同年份即使是相同时期的降水,其中的微生物群落结构及多样性也会因气象因素等条件变化而产生很大的差异,气象条件能直接影响细菌的群落结构组成与多样性[17].分子生物学分析结果表明,北京市各月份降水中的细菌存在差异性.总体而言,按照季节性划分七个月份的样品,细菌多样性与物种丰度的趋势为冬季(B112)>秋季(B8、B18、B9)>夏季(B5、B6、B7)(表2),这与2010年Fahlgren等[26]采集海边空气样品得到的细菌多样性与群落结构研究结果一致.但Fang等[19]与Kaarkainen等[31]通过直接采集培养大气中的细菌的研究结果不一致,其多样性与物种丰度则在夏秋季最高.造成结果不同的原因可能是,由于样品的来源不同,后者收集的样品直接取自于大气样品,而非来自于大气降水;夏秋季雨水相对较多,湿沉降作用对大气冲刷明显,造成夏秋雨水中细菌的多样性及物种丰度较冬季低;另外,已有研究表明大气中的细菌能够降解并利用大气中的有机物,如甲酸、乙酸等[32],从我们同期观测到的数据也显示此类低分子量的有机酸浓度是冬季>夏秋季(数据略),大量的观测结果显示有机气溶胶粒子是大气云凝结核与冰核的重要组成成分[32],在提供给细菌附着点的同时也能够满足细菌代谢的营养需求,大气中有机污染物可为细菌等微生物的生长与代谢提供所需的物质
[33].夏季降水中发现了包括未定菌门在内的 5个门类的细菌,秋季降水中发现了 3个门类的细菌,冬季降水中发现了5个门类的细菌,其中蓝藻门、硝化螺菌门、厚壁菌门是冬季降水中独有的.可能原因是冬季降水较少,大气中悬浮的固体颗粒物悬浮时间较长,有助于微生物附着与生存.季节内各月份细菌群落多样性变化不大.Maron等
[34]研究认为,大气中细菌群落结构的季节性变化主要是由气候以及大气的改变所触发的.
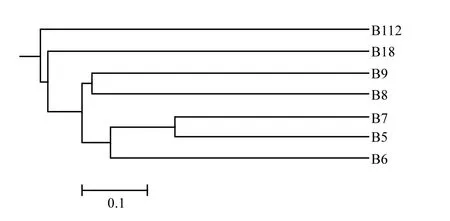
图3 聚类分析不同降水中细菌群落的相似性Fig.3 Cluster analysis of the bacteria community from the different precipitation
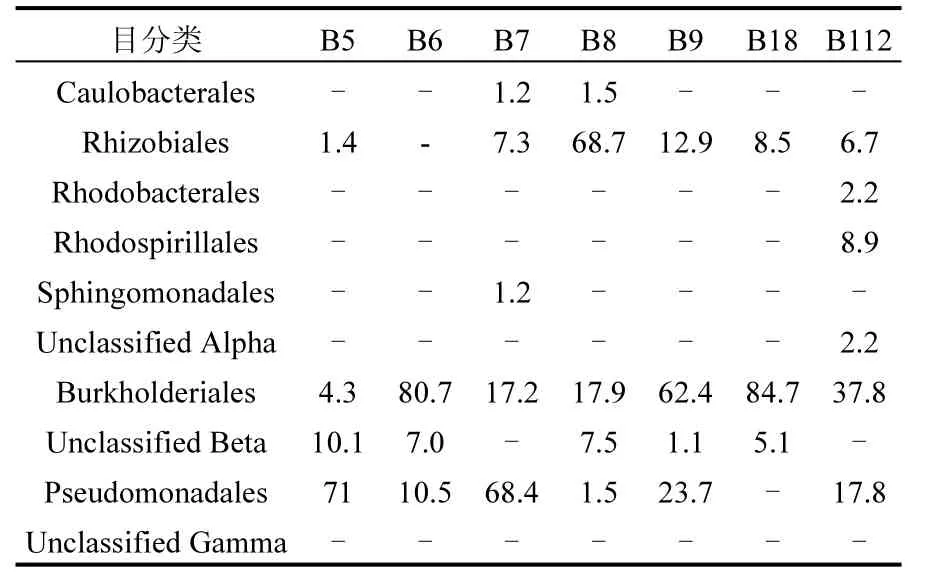
表2 样品中变形菌纲目分类的组成(%)Table 2 The taxonomic distribution of proteobacteria (Alpha-,Beta-,Gamma-) for each samples(%)
气候在改变细菌群落结构组成与多样性的同时,具有冰核活性的细菌对云与降水的形成也具有实质性的影响,因此微生物至少可能在区域性地带影响大气水循环与气候[13-14,35].本研究中经对比分析发现样品 B112含有假单胞菌属,已经证实具有很高冰核活性(Ice Nuclei Activity, INA)的丁香假单胞菌种[24,36]就是该属菌种,一定程度上或许可以推测,大气降水中可能存在着具有冰核活性的细菌而且能够在云与降水过程中起到作用[37],这也是我们接下来的工作重点,利用分子生物学方法来揭示大气降水中的冰核活性菌的存在与否.
3 结论
3.1 北京市大气降水中的细菌群落具有很高的多样性,共发现了8个门类的细菌,革兰氏阴性菌比例大于革兰氏阳性菌,其中优势菌群是变形菌门,β变形菌纲和γ变形菌纲属于优势菌亚群.此外,大气降水中还有一定量未被认知的细菌.
3.2 北京市大气降水中的细菌群落结构多样性,不同季节、不同年份均有所差异.整体而言,北京市大气降水中的细菌多样性具有季节性变化,冬季细菌多样性最高,秋季次之,夏季最低.
3.3 北京市大气降水中细菌群落的聚类分析结果表明,同一季节内大气降水的细菌群落结构相似性较高;季节性的气候差异性越明显,细菌群落间的相似性越低.
[1] Amato P, Menager M, Sancelme M, et al. Microbial population in cloud water at the Puy de Dome: Implications for the chemistry of clouds [J]. Atmos. Environ., 2005,39(2):4143-4153.
[2] Bauer H, Kasper-Giebl A, Loflund M, et al. The contribution of bacteria and fungal spores to the organic carbon content of cloud water, precipitation and aesosols [J]. Atmospheric Research, 2002,64:109-119.
[3] Sattler B, Puxbaum H, Psenner R. Bacerial growth in supercooled cloud droplets [J]. Geophysical Research Letters, 2001,28:239-242.
[4] Cochet N, Widehem P. Ice crystallization by Pseudomonas syringae [J]. Applied Microbiology and Biotechnology, 2000, 54:153-161.
[5] Sun J, Ariya P A. Atmospheric organic and bio-aerosols as cloud condensation nuclei (CCN): A review [J]. Atmos. Environ., 2006, 40:795-820.
[6] Hu Q X, Chen Z S, Xu G Q, et al. Study on particle chart of airborne microbe in Beijing area [J]. Environmental Monitoring in China, 1991,7(1):9-11.
[7] Marchisio V F, Cassinelli C, Tulloi V, et al. Outdoor airborne dematophytes and related fungi: A survey in Turi [J]. Mycoses, 1992,5:251-257.
[8] Peccia J, Hospodsky D, Bibby K. New directions: a revolution in DNA sequencing now allows meaningful integration of biology with aerosol science [J]. Atmos. Environ., 2011,45:1896-1897.
[9] Lee T, Grinshpun S A, Martuzevicius D, et al. Relationship between indoor and outdoor bioaerosols collected with a button inhalable aerosol sampler in urban homes [J]. Indoor Air, 2005, 16:37-47.
[10] Wright J, Greene V, Paulus H. Viable microorganisms in an urban atmosphere [J]. Journal of Air Pollution Control Associate, 1969,19:337-339.
[11] Bauer H, Giebl H, Hitzenberger R, et al. Airborne bacteria as cloud condensation nuclei [J]. J. Geophys. Res., 2003,108: AAC2/1-AAC2/5.
[12] Franc G D, Demott P J. Cloud activation of airborne Erwinia carotovora cells [J]. J. Appl. Meteor., 1998,37:1293-1300.
[13] Andreae M O, Rosenfeld D. Aerosol-cloud-precipitation interactions. Part 1. The nature and sources of cloud-active aerosols [J]. Earth Sci., 2008,89:13-41.
[14] Prenni A J, Petters M D, et al. Relative roles of biogenic emissions and Saharan dust as ice nuclei in the Amazon basin [J]. Nat. Geosci., 2009,(2):401-404.
[15] Maki L R, Willoughby K J. Bacteria as biogenic sources of freezing nuclei [J]. Appl. Meteor., 1978,17:1049-1053.
[16] Li J, Martha PI, Lee T C. Effects of ice nucleation active bacteria on the freezing of some model systems [J]. Int. J. Food Sci. Technol., 1997,32:41-49.
[17] Maki L R, Galyan E L, Chang-Chien M M, et al. Ice nucleation induced by Pseudomonas syringae [J]. Appl. Microbiol., 1974, 28:456-459.
[18] Amato P, Parazols M, Sancelme M, et al. Microorganisms isolated from the water phase of tropospheric clouds at the Puy de Dôme: major groups and growth abilities at low temperatures [J]. FEMS Microbiol. Ecol., 2007,59:242-254.
[19] Fang Z G, Ouyang Z Y, Zheng H, et al. Culturable airborne bacteria in outdoor environments in Beijing, China [J]. Microb. Ecol., 2007,54: 487-496.
[20] Lighthart B. Mini-review of the concentration variations found in the alfresco atmospheric bacterial populations [J]. Aerobiologia, 2000,16:7-16.
[21] Nocker A, Burr M, Camper A K. Genotypic microbial community profiling: a critical review [J]. Microbial Ecology, 2007,54: 276-289.
[22] 郑艳玲,侯立军,陆 敏,等.崇明东滩夏冬季表层沉积物细菌多样性研究 [J]. 中国环境科学, 2012,32(2):300-310.
[23] 姜睿玲,杨统一,唐玉斌,等.多环芳烃污染对桑园土壤微生物结构及种群多样性的影响 [J]. 中国环境科学, 2012,32(9):1655-1661.
[24] Weisburg W G, Barns S M, Pelletier D A, et al. 16S ribosomal DNA amplification for phylogenetic study [J]. J. Bacteriol., 1991,173:697-703.
[25] Despres V R, Nowoisky J F, Klose M, et al. Characterization of primary biogenic aerosol particles in urban, rural, and high-alpine air by DNA sequence and restriction fragment analysis of ribosomal RNA genes [J]. Biogeosciences, 2007,4:1127-1141.
[26] Fahlgren C, Bratbak G, Sandaa R A, et al. Diversity of airborne bacteria in samples collected using different devices for aerosol collection [J]. Aerobiologia, 2010,27:107-120.
[27] Fierer N, Liu Z Z, Rodriguez-Hernandez M, et al. Short-term temporal variability in airborne bacterial and fungal populations [J]. Appl. Environ. Microbiol., 2008,4:200-207.
[28] Christner B C, Mosley-Thompson E, Thompson L G, et al. Bacterial recovery from ancient glacial ice [J]. Environmental Microbiology, 2003,5:433-436.
[29] Maron P A, Lejon D P H, Carvalho E, et al. Assessing genetic structure and diversity of airborne bacterial communities by DNA fingerprinting and 16S rDNA clone library [J]. Atmos. Environ., 2005,39:3687-3695.
[30] Gonzalez-Toril E, Amils R, Delmas R J, et al. Bacterial diversity of autotrophic enriched cultures from remote, glacial Antarctic, Alpine and Andean aerosol, snow and soil samples [J]. Biogeosciences, 2009,6:33-44.
[31] Kaarkainen P, Meklin T, Rintala H, et al. Seasonal variation in airborne microbial concentrations and diversity in landfill, urban, and rural sites [J]. Clean, 2008,36:556-563.
[32] Herlihy L J, Galloway J N, Mills A L. Bacterial utilization of formic and acetic acid in the rainwater [J]. Atmospheric Environment, 1987,21:2397-2402.
[33] Ariya P A, Nepotchatykh O, Ignatova O, et al. Microbiological degradation of atmospheric organic compounds [J]. Geophysical Research Letter, 2002,29:1231-1232.
[34] Maron P A, Mougel C, Lejon D P H, et al. Temporal variability of airborne bacterial community structure in an urban area [J]. Atmos. Environ., 2006,40:8074-8080.
[35] Poschl U, Martin S T, Sinha B, et al. Rainforest aerosols as biogenic nuclei of clouds and precipitation in the Amazon [J]. Science, 2010,329:1513-1516.
[36] 王亚玲,杜 睿,梁宗敏,等.冰核细菌Pseudomonas syringae是否可以影响大气的冰核核化过程 [J]. 科学通报, 2012,57:2413-2418.
[37] Morris C E, Georgakopoulos D G, Sands D C. Ice nucleation active bacteria and their potential role in precipitation [J]. Phys., 2004,121:87-103.
致谢:感谢中国科学院大气物理研究所在样品采集阶段提供的帮助与支持.
《中国环境科学》被Ei收录
根据Ei 总部2013 年颁布的期刊收录情况,《中国环境科学》已被Ei 数据库作为源期刊收录,详见http://www.chinaeidata.com/periodical.htm
《中国环境科学》编辑部
2013-03-14
Phylogenetic diversity of bacteria areasols in precipitation of Beijing Area.
LIANG Zong-min, DU Rui*, DU Peng-rui,
WANG Ya-ling, LI Zi-ming (College of Resource and Environment, University of Chinese Academy of Science, Beijing 100049, China). China Environmental Science, 2014,34(2):317~323
Atmosphere bioaerosols was always closely linked with the health of human beings and flora and fauna, however, recent studies suggested that the bio-aerosols may impact the environment and climate change directly by acting as cloud condensation nuclei (CCN) and/or ice nucleation (IN). Unfortunately, there was still little knowledge about the composition of microbial community of the biological ice nucleis in the precipitation, especially, in China mainland. At present study, the precipitation samplers were taken from the Beijing city area. The sampling was carried out in different months in 2011 and 2012. The bacterial diversity was analyzed using the 16S rRNA gene sequencing based approaches. Seven clone libraries were established resulting from the different rain-water sampling months. The results indicated that Proteobacteria (75.6%~100%) were dominant in the precipitation including Alphaproteobacteria, Betaproteobacteria,and Gammaproteobacteria. While other bacterial communities such as Bacteroidetes, Actinobacteria, Deinococcus-Thermus, Cyanobacteria, Nitrospira and Firmicutes were involved as well. Furthermore, a small proportion of undetermined bacteria (TM7, 1.2%~7.2%) were also found. In addition, microbial diversity in the snow water samplers was obviously much more than that in the rain-water samplers, which suggested that some biological ice nuclei may present and influence in the formation of precipitation. Moreover, results showed that Pseudomonas, in which some strains with high effective ice nucleation activity, was also found in the samplers. Therefore, further concerns should be done for the climatic effect of these microbes.
precipitation;bacteria;community;diversity;16S rRNA
X513
:A
:1000-6923(2014)02-0317-07
梁宗敏(1986-),男,河南周口鹿邑县人,中国科学院大学硕士研究生,主要从事大气生物气溶胶的多样性研究.
2013-06-21
国家自然科学基金(41175135)
* 责任作者, 副教授, ruidu@gucas.ac.cn
