中国蛇岛蝮蛇毒腺cDNA 文库ESTs 序列测定及生物信息学分析
2012-12-23郭春梅孙明忠郑体花任一鑫刘淑清
郭春梅,孙明忠,郑体花,任一鑫,刘淑清*
1大连医科大学生物技术系;2大连医科大学生物化学与分子生物学教研室;3大连医科大学寄生虫教研室,大连116044
蛇毒(snake venom,SV)具有抗凝止血、镇痛、影响血小板聚集、诱导出血或溶血、诱导细胞凋亡、刺激水肿形成、抗病毒、抗肿瘤、抗菌消炎、抗HIV 等多种生物学活性[1-10],具有很好的临床应用前景和开发价值。蛇毒中主要的活性及毒性成分是蛋白(酶),获得蛇毒蛋白进行功能研究尤为重要。
中国蛇岛蝮蛇(Gloydius shedaoensis shedaoensis,GSS)是我国独有珍稀蛇种,为国家二级保护物种。蛇毒价格昂贵且来源稀少,而且过度采集蛇毒会严重影响GSS 的生存;蛇毒中的大多活性蛋白(酶)含量都很低,采用传统纯化方法很难得到足够量的目的活性蛋白(酶)[3,4,11],阻碍了GSS 蛇毒活性蛋白(酶)的开发利用。从相应cDNA 文库中筛选cDNA克隆进行表达序列标签(EST)分析,采用基因工程手段大量表达和获得蛇毒目的活性蛋白(酶)进行深入研究是一种可行的方法。
EST 是通过随机选取cDNA 克隆进行单向测序获取的特定组织在特定时期表达基因的一段序列。EST 是基因的“窗口”,可特异性地代表生物体某组织某一时间的一个表达基因,故被称为基因表达序列标签。对生物进行大规模cDNA 测序不仅可以获得已知的有重要价值的基因,还可以发现许多新的未知基因。EST 分析可以用于寻找功能基因、分离与鉴定新基因、基因表达谱研究、构建遗传图谱、物理图谱等[12-18]。
本研究在我们前期成功构建的GSSG-cDNA 文库基础上,对211 个ESTs 进行了测序和功能分析,发现84 个克隆为已知功能基因,29 个克隆为未知功能基因,98 个克隆为新基因,从而为进一步对GSS 活性蛋白基因的克隆、表达和功能研究奠定了一定实验基础。
1 材料与方法
1.1 实验材料与试剂
GSSG-cDNA 文库由本实验室制备并保存;DNA Marker、Rnase A 酶为宝生物工程(大连)有限公司产品;胰蛋白胨、酵母提取物为OXOID 公司产品;三羟甲基氨基甲烷(Tris)购于GE Health Care 公司;十二烷基磺酸钠(SDS)、EB、苯酚、琼脂糖购于Sigma公司;其他试剂均为国产分析纯。
1.2 cDNA 文库的构建
由本研究组采用SMART 技术构建中国蛇岛蝮蛇毒腺的cDNA 文库[19]。
1.3 cDNA 克隆的序列测定
1.3.1 cDNA 文库菌液的扩增
cDNA 文库进行梯度实验,选出最佳稀释浓度后,在LB 固体培养基(34 μg/mL 氯霉素)上涂板,每个平板的理论细菌个数为960,37 ℃倒置培养16 h;然后挑取单个菌落至3 mL LB 液体培养基中(34 μg/mL 氯霉素),37 ℃震荡培养16 h;然后在10000 rpm 离心1 min,弃上清,细胞沉淀尽可能干燥,–20℃保存。
1.3.2 cDNA 文库质粒的提取
采用碱裂解法提取质粒:(1)将细菌沉淀悬浮于200 μL 预冷至4 ℃的溶液Ⅰ中(1M Tris-HCl,0.5 M EDTA,20% glucose,pH 8.0),剧烈震荡,室温放置5 min;(2)加入400 μL 溶液Ⅱ(0.4 M NaOH,2%SDS),快速轻颠倒5 次混匀,室温静置5 min;(3)加入300 μL 溶液Ⅲ(29% KAC,11% CH3COOH,pH 4.8),轻颠倒数次混匀,冰上放置5 min 后在4 ℃、1000 rpm 下离心5 min;(4)小心吸出上清,加入与上清液等体积的苯酚-氯仿混合液抽提,颠倒混匀,冰上放置5 min 后在4 ℃、10000 rpm 下离心2 min;(5)小心吸出上层水相,加入2 倍体积无水乙醇,混匀,室温放置5 min 后离心(5 min,4 ℃,10000 rpm);(6)弃上清,倾去液体,加入1 mL 0 ℃70%乙醇洗涤沉淀,吹打后离心(2 min,4 ℃,10000 rpm);(7)弃上清,乙醇挥发干净后,每个质粒加入30 μL TE(1M Tris-HCl,0.5 M EDTA,20 μg/mL RNase,pH 8.0)缓冲液溶解,-20 ℃保存。取0.5 μL 样品,采用1%琼脂糖凝胶电泳鉴定质粒提取结果。
1.3.3 cDNA 克隆的序列测定
随机挑选216 个cDNA 单克隆进行测序。设计通用引物M13F 序列5'-GTAAAACGACGGCCAGT-3',对单克隆进行5'端单向测序。反应体系总体积为20 μL:8 μL DYEnamic ET Dye Terminator reagent premix (Amersham Pharmacia,USA),1 μL 5 μmol/L M13 F,质粒模板0.2~2 μg。PCR 测序反应条件为:95 ℃20 s,50 ℃15 s,60 ℃1 min,进行30 个循环,测序用MegaBACETM1000 DNA 测序仪(Amersham Pharmacia,USA)完成。
1.3.4 ESTs 序列分析
获得的cDNA 质粒序列用Chromas 剔除pDNRLIB 载体序列以及包含的接头序列,并去除冗余序列,得到每个克隆的EST 序列信息;然后通过Stackpack 软件将获得的序列进行拼接,获得unigenes,由重叠群(contigs)及单一序列(singlets)组成,通过NCBInr 数据库进行Blast X 检索和序列比对及功能分析,根据蛋白相似性推测基因功能并分类。
2 结果与讨论
2.1 cDNA 文库质量分析
我们前期以pDNR-LIB 为载体,构建了我国第一个GSSG-cDNA 文库,其原始文库库容量为8 ×107cfu/mL。随机从文库中挑选单菌落进行菌液PCR 鉴定结果表明:大多数插入片段均大于1000 bp,表明所构建的GSSG-cDNA 文库质量很高[19]。
2.2 cDNA 文库质粒鉴定
用碱裂解法提取文库质粒,经1%琼脂糖凝胶电泳检测,图1 显示了部分cDNAs 的质粒提取电泳图。cDNA 文库质量直接影响其使用价值。评价一个cDNA 文库质量,主要是看该cDNA 文库的库容量和重组cDNA 片段的序列完整性[20]。电泳结果显示大多数重组质粒分子量大于3000 bp,进一步表明我们构建的GSSG-cDNA 文库是完整高质量的,获得全长序列的可能性大,可满足EST 研究要求。为今后利用该文库进行大规模基因筛选、获得有价值的功能基因奠定了基础。电泳图上发现提取的质粒伴有不同程度开环现象,但并不影响后续的序列分析。
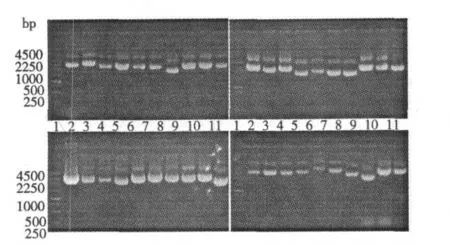
图1 GSSG-cDNA 文库质粒琼脂糖凝胶电泳(1%)Fig.1 Agarose gel electrophoresis (1%)of GSSG-cDNAs
2.3 ESTs 序列分析
随机选取GSSG-cDNA 文库中的216 个单克隆进行5'单向测序,获得有效序列211 条,即211 个ESTs,测序成功率为97.7%。
EST 包含了完整基因上能够特异性标记基因的部分序列,通常包含了基因足够的信息区,从而可与其它基因相区分。将ESTs 序列与数据库中存储的基因/蛋白序列进行比对和相似性分析,可确定出哪些EST 为已知基因,哪些EST 为未知基因[21,22]。211 条高质量GSSG-ESTs 经Blast X 检索NCBInr 数据库比对、查询和注释(E≤10-6)表明:(1)84 条ESTs 与数据库中有序列注释的已知功能蛋白具有一定同源性,为已知功能基因,占39.8%;(2)29 条ESTs 与有序列注释的未知功能蛋白具有同源性,为未知功能基因,占13.7%;(3)98 条ESTs 是数据库中无注释信息的序列、仅有很低同源性或在数据库中未搜索到序列,即新基因,占46.5%,说明蛇岛蝮蛇具有自己的特异基因(图2,表1)。
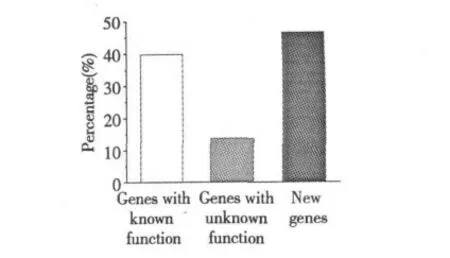
图2 GSSG-cDNA 文库ESTs 比对结果Fig.2 The blasting results of ESTs of GSSG-cDNA library
组织中一定数量的ESTs 可代表该组织中基因表达情况,随机测序所获得的同一基因的EST 数目在一定程度上代表该基因在该组织中的表达丰度。一个基因的表达次数越多,能够测到的相应EST 也越多,因此通过EST 分析可以了解基因表达情况和表达丰度[16]。一般把表达频率<2 的基因视为低表达丰度基因;表达频率≥2 且<5 的基因视为中表达丰度基因;表达频率≥5 的基因视为高表达丰度基因[16,21,22]。GSSG 的211 个ESTs 代表130 个unigenes,包括16 个singlets 和114 个contigs。文库平均冗余率12.3%,其中高表达丰度基因7 个,占全部unigenes 的5. 4%;中表达丰度基因9 个,占6.9%;低表达丰度基因114 个,占87.7% (图3,表1),GSSG-cDNA 文库大多数基因呈低丰度表达,这样可避免重复测序,获得单一基因的几率大大增加,避免人力物力的浪费。
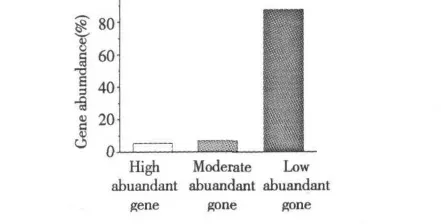
图3 GSSG-cDNAs 基因表达丰度分布Fig.3 The gene expression abundances of GSSG-cDNA

表1 GSSG-cDNA 文库部分cDNA Blast 检索结果Table 1 Blasting results of GSSG-cDNAs

elongation factor-1 alpha Pelodiscus sinensis 86% 2 7 Splicing factor SLU7 Monodelphis domestica 82% 1 8 ribosomal protein L23a Salmo salar 75% 1 9 calnexin Gallus gallus 83% 1 10 phosphatase 2 Taeniopygia guttata 90% 1 11 zinc finger Sus scrofa 73% 1 12 adenylosuccinate synthetase Gallus gallus 85% 1 13 Bax inhibitor-1 Canis familiaris 76% 1 14 cyclin C Gallus gallus 89% 1 15 ribosomal protein L5 Gallus gallus 83% 1 16 GTP-binding protein SAR1b-like Callithrix jacchus 86% 1 17 Adapter-related protein complex 3 delta 1 Taeniopygia guttata 85% 1 18 lysosomal acid lipase Macaca mulatta 60% 1 18 PSEN2 gene for presenilin 2 Homo sapiens 78% 1 20 transketolase Gallus gallus 83% 1 21 NNU 95048 12S ribosomal RNA gene Gloydius intermedius shedaoensis 94% 1 22 membrane protein 1 Monodelphis domestica 80% 1 23 cell division cycle and apoptosis regulator 1 Ailuropoda melanoleuca 86% 1 24 alpha actin Atractaspis microlepidota microlepidota 97% 1 25 dehydrogenase Ornithorhynchus anatinus 82% 1 26 Rho GTPase activating protein 5 Taeniopygia guttata 85% 1 27 member RAS oncogene family Gallus gallus 78% 1 28 arginine-rich Monodelphis domestica 62% 1 28 cellular nucleic acid binding protein Gallus gallus 78% 1 30 cysteine dioxygenase Gallus gallus 78% 1 31 ubiquitin-fold modifier 1 Gallus gallus 86% 1 32 Ribosomal protein S6 kinase Gallus gallus 86% 1 33 tetraspan NET-6 Monodelphis domestica 75% 1 34 gamma-actin mRNA Trichosurus vulpecula 84% 1 35 programmed cell death 4 Ailuropoda melanoleuca 80% 1 36 ribonuclease UK114-like Ailuropoda melanoleuca 80% 1 37 tRNA selenocysteine-associated protein 1 Taeniopygia guttata 81% 1 38 Ubiquitin-conjugating enzyme E2 G2 Rana catesbeiana 83% 1 39 serine palmitoyltransferase Gallus gallus 71% 1 40 eukaryotic translation initiation factor 1A Taeniopygia guttata 88% 1 41 constitutive photomorphogenic protein Gallus gallus 90% 1 42 predicted protein Postia placenta Mad-698-R 90% 1 43 KIAA0987 protein (LOC100015377) Monodelphis domestica 70% 1 44 RP23-157G2 from chromosome 5 Mus musculus BAC 85% 1 45 hypothetical protein LOC100227078 Taeniopygia guttata 84% 1 46 E33DIFJ02GS9NC microsatellite sequence Agkistrodon contortrix 84% 1 47 E33DIFJ02F7Q1J microsatellite sequence Agkistrodon contortrix 80% 1 6

1-41:已知功能基因;42-55:未知功能基因;56-61:新基因,其余92 条ESTs 在数据库中未能搜索到同源蛋白,没有列出。1-41:genes with known function;42-55:genes with unknown function;56-61:new genes,the rest 92 ESTs were not listed due to had no significant matches to any protein sequences in the public database
在Blast X 分析中,我们获得了一部分已知功能基因,这些基因具有重要的生物学功能;因此,可以根据获得的序列来设计引物,克隆、表达目的活性蛋白组分,进一步研究其生物学活性。例如我们得到一个GSSG 基因,其EST 序列为648 bp(图4 A),编码216 个氨基酸(图4 B),氨基酸序列比对发现其与非洲家蛇和水游蛇的转铁蛋白同源性高达84%(图4 C)。但转铁蛋白在蛇毒中扮演的角色以及具体生物学活性,目前尚不清楚。
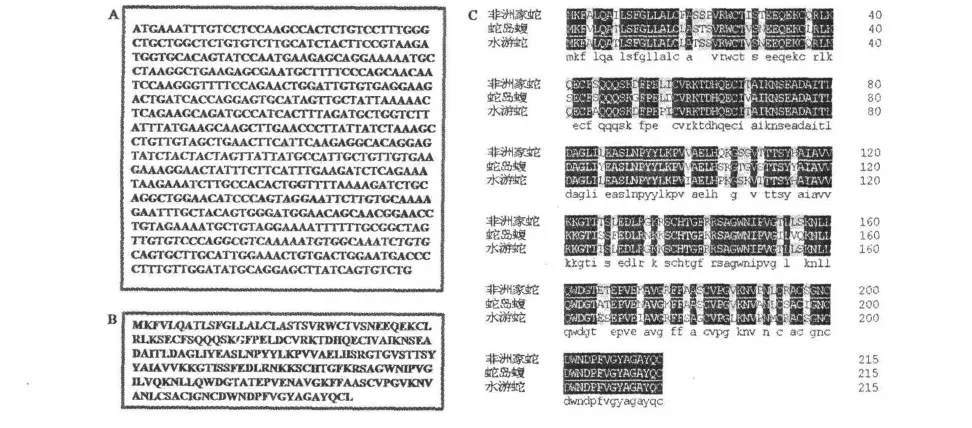
图4 蛇岛蝮蛇转铁蛋白序列分析Fig.4 The sequence analysis of GSS-transferrin
此外有近60%的GSSG 基因为新基因或未知功能基因,说明在GSS 中存在自身含有的特异基因/蛋白,提示我们去搞清楚:1)它们是什么基因/蛋白,2)它们在GSS 中起什么作用,3)它们有什么具体应用和生物功能,亟须进一步研究去揭示,是今后研究的重点。例如,本研究发现一个GSSG 基因,其全长序列为384 bp:
ATGGGGCAGGATGAAATTAGCAACTTCACTGA CTTAGCCTTGCAATATTATAACCAGCAGGAAGAG TCCCTGTTTATACCTTTGAAGAACACAACCGTGA CGGTCAAGAAAGCCATTGGATCCATATTGGGC CTGCCGATGATTGTGGAAAGGACCAACTGCACC AAAAAAGATGTGAAACGACATTTTAGGAGTC CCTTTGATTCTGAAGAAGAACCCCGGAAATATTG TATGCCGCTTCCGGACCCTGAGCAACTGAACT GCAAATTTCACATTTTTAAAGATGAAAGGACTT ACAGCGAAGCTATACTGGGCCATGAATGCGA GCCAATTAAAATAATCGATAAGGAGATTCTT AATGTTGAAGATACAAAATCACCATAT,编码128 个氨基酸。序列比对发现其与抗辐射不动杆菌的一个假设蛋白有8%序列同源性,但这个GSSG 基因具体是什么基因,有什么样的生物学功能,在GSS 中起什么作用,需要和值得进一步研究。
近年来,随着激光显微切割、cDNA 文库标准化、DNA 自动测序技术和cDNA 芯片技术的快速发展,使得随机挑取cDNA 单克隆进行大规模测序得以顺利进行,并为克隆新基因、寻找组织特异性基因等提供了快速有效的方法[21,22]。用EST 代替基因组测序,可使研究费用大大降低,同时效率大大提高,因为EST 能大大缩小基因筛选范围,提高分离基因的效率。在生物体内表达基因只占整个基因组的3%~5%,而EST 反映的是表达基因的编码部分,故通过EST 可直接获得基因表达信息。一旦发现有价值的EST,可通过PCR 技术获得全长cDNA序列,并克隆、表达目的基因。由于EST 在分析研究功能基因组方面具有的明显优越性,已被广泛用于新基因的克隆、基因表达谱分析、基因差异表达以及基因组序列的功能注释等诸多研究领域。
总之,本实验在我们成功构建中国蛇岛蝮蛇毒腺全长cDNA 文库基础上,获得了211 条ESTs 序列,其中84 个为已知功能基因,29 个为未知功能基因,98 个为新基因。尽管本次所测ESTs 序列不太多,但已经提示所得结果可能代表整个蛇毒基因的表达情况,其中低丰度表达基因占总GSSG 基因的近90%,说明采用我们所构建的GSSG-cDNA 文库,将来进行GSS 基因大规模筛选和鉴定时,可避免重复测序,获得单一基因的几率很大,便于发现更多的、有价值的功能基因以开展后续研究,可有效避免人力物力的浪费。
1 Liu SQ(刘淑清),Guo CM(郭春梅),Xu YF(徐跃飞),et al.Effect of metal ions on the biological activity and structural information of snake venom proteins. Chem J Chin Univ(高等学校化学学报),2009,30:1773-1778.
2 Liu S,Sun MZ,Sun C,et al.A novel serine protease from the snake venom of Agkistrodon blomhoffii ussurensis. Toxicon,2008,52:760-768.
3 Liu S,Sun MZ,Greenaway FT.A novel plasminogen activator from Agkistrodon blomhoffii Ussurensis venom(ABUSV-PA):Purification and characterization. Biochem Biophys Res Commun,2006,348:1279-1287.
4 Sun MZ,Liu S,Greenaway FT.Characterization of a fibrinolytic enzyme(ussurenase)from Agkistrodon blomhoffii ussurensis snake venom:Insights into the effects of Ca2+on function and structure.Biochim Biophys Acta,2006,1764:1340-1348.
5 Sun MZ,Liu S,Yang F,et al.A novel phospholipase A2from Agkistrodon blomhoffii ussurensis venom:purification,proteomic,functional and structural characterizations.Biochimie,2009,91:558-567.
6 Liu S,Zhang C,Xu Y,et al. Electrospray ionization mass spectrometry as a critical tool for revealing new properties of snake venom phospholipase A2. Rapid Commun Mass Spectrom,2009,23:1158-1166.
7 Ma C(马驰),Liu SQ(刘淑清),Zong JW(宗军卫),et al.Proteomic analysis of Gloydius shedaoensis shedaoensis venom by two-dimensional electrophoresis combined to high performance liquid chromatography-electro-spray ionization tandem mass spectrometry:a preliminary study.Chin J Zool(动物学杂志),2009,44:70-77.
8 Rodrigues RS,da Silva JF,Boldrini Franca J,et al.Structural and functional properties of Bp-LAAO,a new L-amino acid oxidase isolated from Bothrops pauloensis snake venom. Biochimie,2009,91:490-501.
9 Doley R,Kini RM. Protein complexes in snake venom. Cell Mol Life Sci,2009,66:2851-2871.
10 Lomonte B,Escolano J,Fernandez J,et al. Snake venomics and antivenomics of the arboreal neotropical pitvipers Bothriechis lateralis and Bothriechis schlegelii. J Proteome Res,2008,7:2445-2457.
11 Yu RM(余瑞铭),Xu YL(许云禄).The research progress of snake venom plasminogen activator.Strait Pharm J(海峡药学),2008,20:6-8.
12 Chen L,Zhao LP,Gao QK. Generation and analysis of expressed sequence tags from the tender shoots Cdna library of tea plant(Camellia sinensis).Plant Sci,2005,168:359-363.
13 Wang YC,Yang CP,Liu GF,et al.Generation and analysis of expressed sequence tags from a cDNA Library of Tamarix androssowii.Plant Sci,2006,170:28-36.
14 Jae SK,Hyoungseok L,Sung M,et al.Construction and characterization of subtractive stage-specific expressed sequence tag(EST)libraries of the pinewood nematode Bursaphelenchus xylophilus.Genomics,2009,94:70-77.
15 Bausher M,Shatters R,Chaparro J,et al. An expressed sequence tag(EST)set from Citrus sinensis L. Osbeck whole seedling and the implications of further perennial source investigations.Plant Sci,2003,165:415-422.
16 White JA,Todd J,Newman T,et al.A new set of arabidopsis expressed sequence rags from developing seeds,the metabolic pathway from to seed oil. Plant Physiol,2000,124:1582-1594.
17 Jaroslav M,Stephan G,Won KC,et al.Construction,database integration,and application of an Oenothera EST library.Genomics,2006,88:372-380.
18 Zhang JP,Liu TS,Fu JJ,et al.Construction and application of EST library from Setaria italica in response to dehydration stress.Genomics,2007,90:121-131.
19 Liu SQ(刘淑清),Guo CM(郭春梅),Hou ZJ(侯志杰),et al. Construction of a cDNA Library for Chinese Gloydius shedaoensis shedaoensis venom Gland. Chin J Zool(动物学杂志),2010,45:136-140.
20 Meng YP(孟玉平),Cao QF(曹秋芬),Sun HF(孙海峰),et al. Construction and partial ESTs analysis of a cDNA library for fruit-bearing shoot in Ziziphue jujuba.Acta Agric Boreali-Sin(华北农学报),2009,24:102-106.
21 Yu FC(于风池). The technology of EST(Expressed sequence tag)and its applications. Chin Agric Sci Bull(中国农学通报),2005,21:54-58。
22 Wan HW(万海伟),Du LX(杜立新).Application of EST in the study of genomics. Biotechnol Bull(生物技术通报),2004,1:34-38.
