转录组和蛋白质组关联分析解析巴西蕉幼苗响应低温的分子机制
2024-04-24林蔚吴水金李跃森
林蔚,吴水金,李跃森
转录组和蛋白质组关联分析解析巴西蕉幼苗响应低温的分子机制
林蔚,吴水金,李跃森
福建省农业科学院亚热带农业研究所,福建漳州 363005
【目的】低温是导致香蕉减产的主要自然灾害之一。基于转录组与蛋白质组关联分析参与香蕉抗寒的相关基因、蛋白及信号、代谢通路调控网络,探讨香蕉抗寒的分子机制。【方法】以巴西蕉为试材,在7 ℃低温下处理1 d(Cold1)和3 d(Cold3),以28 ℃培养的巴西蕉为对照(CK),基于笔者课题组前期获得的蛋白质组数据,利用转录组测序技术检测巴西蕉在低温胁迫下基因调控网络的变化,同时与蛋白质组学数据进行关联分析,共同解析巴西蕉响应低温胁迫的分子机制。【结果】转录组分析结果显示,Cold1 vs CK、Cold3 vs CK和Cold1 vs Cold3三个对比组分别鉴定出11 370、15 460和9 619个差异表达基因,对这些基因的KEGG富集分析发现,差异表达基因在光合作用信号、谷胱甘肽代谢、-亚麻酸代谢途径和苯丙素生物合成等多个低温胁迫关键信号代谢通路中富集。对部分差异表达基因进行实时荧光定量(qRT-PCR)分析,其中DREB、MAPK和MYB等低温调控关键基因的表达量在低温处理后显著上升,所选20个基因的表达变化趋势与RNA-seq基本相符,证实了RNA-seq的准确性。转录组与蛋白质组关联分析结果显示,共鉴定到6 211个与转录本相对应的蛋白,转录组与蛋白质呈现正相关关系,共有105个转录本及其对应的蛋白表达量共同上调,有218个转录本及其对应的蛋白表达量共同下调。GO富集分析显示,差异表达基因和蛋白在光响应、叶绿体、氧化还原酶活性等功能大量富集。此外,对差异表达基因和蛋白KEGG通路的关联分析发现,低温处理抑制了苯丙素生物合成和光合作用信号途径相关基因及蛋白的表达,促进了-亚麻酸代谢和谷胱甘肽途径的表达。【结论】运用转录组结合蛋白质组学技术绘制了基因和蛋白质水平上的香蕉抗寒调控网络,并发现香蕉响应低温的信号通路主要涉及光合作用信号、谷胱甘肽代谢、-亚麻酸代谢途径和苯丙素生物合成等途径。
香蕉;低温胁迫;转录组;蛋白质组;关联分析
0 引言
【研究意义】巴西蕉(Cavendish cv. Baxi)属于芭蕉科芭蕉属,是中国南方四大水果之一[1]。香蕉喜高温高湿,因此,我国的香蕉主要产区多位于南亚热带地区,如福建、广西、云南等省(区)[2]。低温是影响果树分布、产量、品质的主要环境因素之一,而大多数香蕉主栽品种对于低温胁迫十分敏感,耐冷性普遍较差,当气温低于10 ℃时生长就会受阻,当温度下降到5 ℃时植株就会呈现叶片发黄的冷害反应,温度低于2 ℃则会致死[3-4]。因此,深入探究香蕉苗期抗寒分子基础及响应机制,可以为后续香蕉的抗寒育种提供理论基础。【前人研究进展】低温胁迫对植物各个器官的生长发育都会造成严重影响,能够抑制根的生长,降低根分生组织细胞延伸率[5];此外,随着温度的降低,根系对水分的吸收能力也会受到影响,进而影响植物的水分状况及光合作用强度[6-7]。叶片对低温的响应也不仅仅是表观的褐化和卷曲,还表现在叶绿体超微结构的改变,进而影响叶绿素的合成及光合作用功能[8]。植物对低温的感知主要依赖于质膜和内质网的G-蛋白信号调节器,通过激活Ca2+通道,改变植物中的Ca2+浓度来接收低温信号[9]。植物对低温信号的传导及响应主要是通过冷胁迫反应原件结合因子(C-repeat/dehydration- responsive element binding factor,CBF)途径实现,在低温胁迫中,CBF被激活,进而调控下游抗寒及冷驯化相关基因(cold-regulated or cold-responsive gene,COR)[10]。香蕉对低温敏感,其抗寒分子机制研究较多,耿小慧[11]对巴西蕉钙离子通道CNGC基因家族进行全基因组鉴定,共发现17个CNGC家族成员,且大部分CNGC家族成员的表达在低温胁迫下呈现下调趋势。刘嘉鹏[12]利用转录组分析了褪黑素和低温联合处理后的巴西蕉幼苗,发现褪黑素主要通过影响植物激素信号传导、MAPK信号途径和苯丙烷生物合成等途径相关基因的表达来提高香蕉的抗寒性。Gao等[13]利用酵母双杂验证大蕉MaICE1与MaMAPK3互作,且沉默MaMAPK3后,会显著降低大蕉MaICE1的表达,从而使其抗寒性下降。Lin等[14]利用Western blot检测发现低温处理1 d后,巴西蕉叶片中的泛素化程度上升,结合泛素修饰组分析结果,发现泛素化修饰在巴西蕉响应低温胁迫中发挥重要作用。【本研究切入点】组学技术在分析植物基因或蛋白整体表达上具有巨大的优势,但单一的组学对深入剖析生物学现象的机制、机理具有一定的局限性。因此,有必要利用多组学关联分析探究机理,得到更完整的表达信息。笔者课题组在前期研究中,已利用非标定量法蛋白质组学技术对巴西蕉幼苗在低温胁迫下的蛋白表达情况进行了分析。【拟解决的关键问题】以主栽品种巴西蕉幼苗为试材,在7 ℃低温分别处理1 d、3 d后取叶片进行转录组测序。深入研究巴西蕉低温胁迫下的基因表达模式,并且与前期获得的巴西蕉低温胁迫蛋白质组学数据关联分析,以全面地了解巴西蕉响应低温的调控网络和表达模式。
1 材料与方法
1.1 材料
试验以巴西蕉为材料,于2022年在福建省农业科学院亚热带农业研究所进行。选择培养至生长状态基本一致的“五叶一心”香蕉幼苗,并在培养箱中以12 h白天/12 h黑夜(28 ℃)、光强240 μmol∙m-2∙s-1,湿度70%的条件下缓苗一周,以28 ℃培养的香蕉幼苗为对照组,以7 ℃处理1 d、3 d为处理组(分别记为Cold1、Cold3),每个时间点重复采集5株香蕉幼苗的第一和第二片幼叶,切碎后混合,3次生物学重复。所有样品在液氮中快速冷冻,并在-80 ℃下储存。
1.2 转录组测序
利用MJZol total RNA提取试剂盒(上海美吉生物医药科技有限公司)对7 ℃处理1 d、3 d及对照组的香蕉叶片共9个样品(每组3个重复)中的总RNA进行抽提,1%琼脂糖凝胶电泳检测组织总RNA完整性(RNA integrity,RIN),RIN≥8。委托上海美吉生物医药科技有限公司基于Illumina平台建库测序并对原始数据去冗余过滤。
1.3 数据分析
1.3.1转录组数据分析 利用HiSat2软件将质控后的原始数据(clean data)以香蕉基因组(NCBI,ASM31385v2)为参考基因组进行比对,使用RSEM软件对基因和转录本的表达水平进行分析,使用R语言软件的DESeq2包分析基因差异表达显著性(筛选标准:FDR<0.05,|Log2FC|≥1)。在GO和KEGG数据库中对差异表达基因进行富集分析,基于FPKM值计算基因表达量,筛选巴西蕉响应低温胁迫的差异表达基因(DEG)。使用R语言软件的ggplot2包绘制基因统计柱状图、火山图、气泡图、GO及KEGG富集图。
1.3.2 转录组与蛋白质组关联分析 基于转录组数据和前期获得的蛋白质组数据(登录号:PXD041481,蛋白提取、分解、检测等方法详见文献[14]),进行两组学间的鉴定、定量比较分析。将差异表达的基因和蛋白(FDR<0.05、|Log2FC|≥1)作为分析目标,统计分析转录组数据和蛋白质组数据之间的相关性,使用R语言软件的ggplot2包绘制基因、蛋白的相关性分析、韦恩图、GO和KEGG富集,了解低温胁迫下各差异表达基因和蛋白的富集情况。
1.4 qRT-PCR验证
为验证转录组测序的准确性,本研究选择了20个差异表达的低温相关基因进行荧光定量PCR验证。基于基因组数据库获得的基因全长信息,使用Primer 5.0设计荧光定量特异引物(表1),内参基因为,qRT-PCR试剂盒(湖南艾科瑞生物工程有限公司)反应体系包括10 μL 2×SYBR Green Pro Tap HS premix,1 μL上、下游引物,1 μL稀释后的cDNA模板(100 ng∙μL-1),无菌水补充至20 μL,反应步骤:95 ℃变性30 s,95 ℃退火5 s,60 ℃延伸20 s,40个循环。以2-△△CT[15]计算差异表达基因的相对表达量。
2 结果
2.1 数据质控及序列比对分析
对巴西蕉低温处理及对照共9个样本进行转录组测序(表2),对获得的原始数据(raw reads)过滤去冗余后,得到总数为63.1 Gb的clean reads,各样品clean data均达到6.35 Gb以上,错误率仅为0.02%左右,Q20碱基百分比均在97.06%以上,Q30碱基百分比在91.83%以上,GC含量均在50%左右。以上指标数据说明巴西蕉叶片测序质量良好,可进行下一步分析。转录组所用参考基因为小果野蕉基因组(NCBI,ASM31385v2),将过滤后的clean data与基因组数据进行对比分析(表3),结果显示序列对比率均高于88%,此外还统计了多方比对(在参考序列上有多个比对位置的clean reads)和唯一比对(在参考序列上有唯一比对位置的clean reads数目)。
2.2 基因差异表达分析
进一步对CK、Cold1和Cold3组间的DEGs数量进行统计分析。结果显示,Cold1 vs CK的DEGs共有11 370个,其中6 231个DEGs上调(54.8%),5 139个DEGs下调(45.2%);Cold3 vs CK的DEGs共有15 460个,其中7 889个DEGs上调(51%),7 571个DEGs下调(49%);Cold1 vs Cold3的DEGs共有9 619个,其中4 640个DEGs上调(48.2%),4 979个DEGs下调(51.8%)(图1)。

表1 qRT-PCR引物序列信息
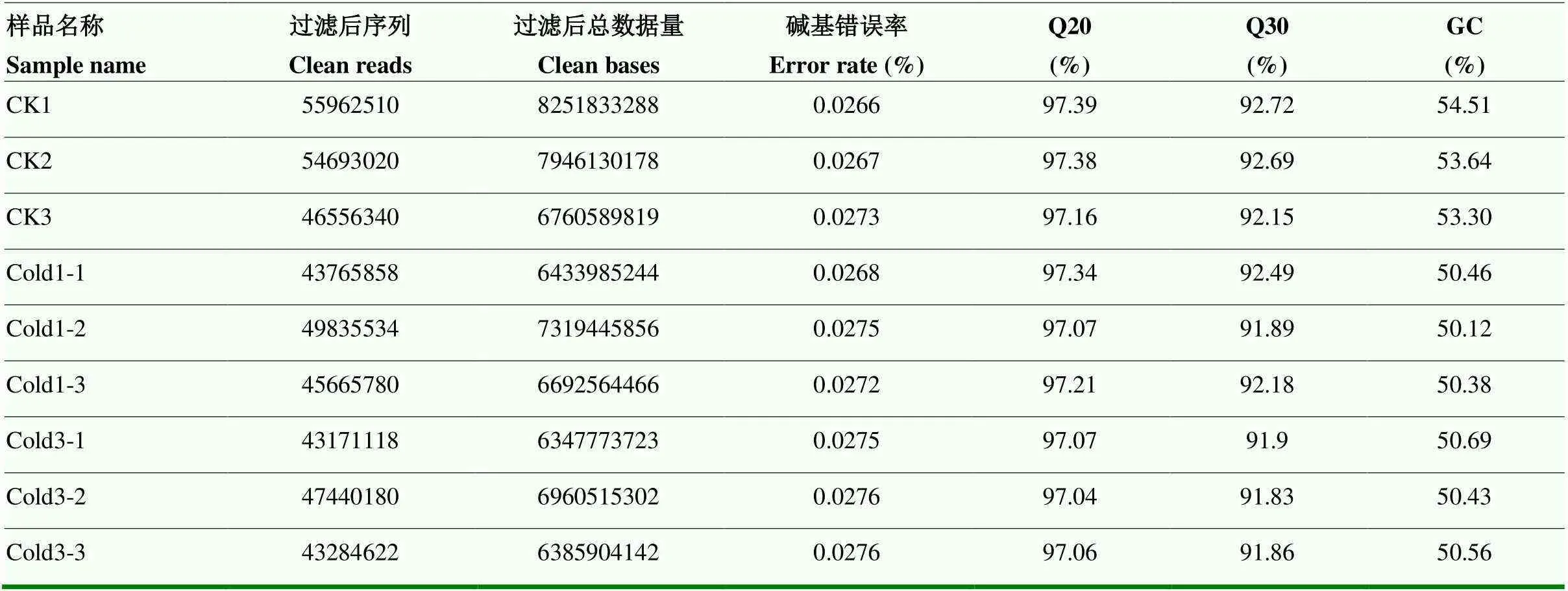
表2 转录组测序数据质控分析
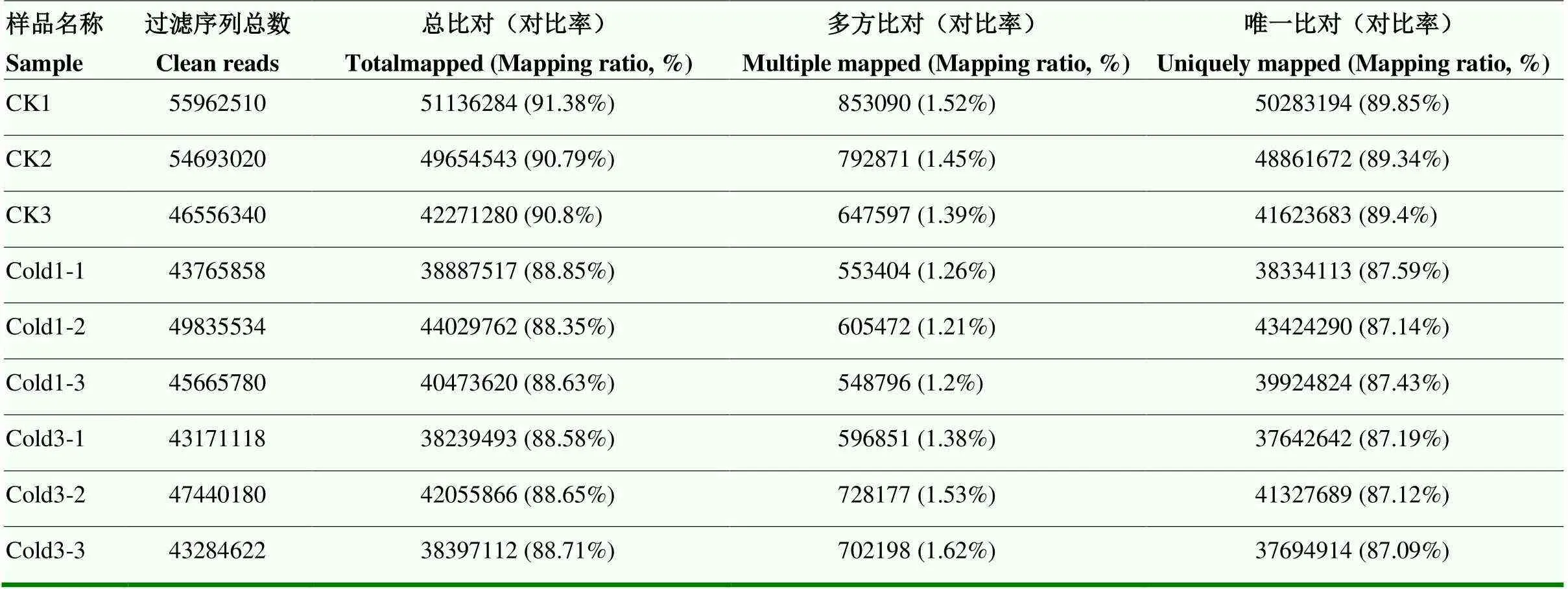
表3 基因组对比结果

图1 差异表达基因统计图
2.3 差异表达基因GO、COG富集分析
对Cold1 vs CK、Cold3 vs CK和Cold1 vs Cold3三个对比组的DEGs进行GO聚类分析,分别有11 370、15 460和9 619个DEGs在GO不同功能节点上富集,分布在分子功能、细胞组分和生物过程3个大类中(图2)。其中生物调节、代谢过程和细胞过程在生物过程组分中是排名前3的亚类;细胞器、细胞膜部分和细胞部分在细胞组分中是排名前3的亚类;转录调节活性、催化活性和结合活性在分子功能组分是排名前3的亚类。以上结果说明巴西蕉幼苗在低温胁迫下发生了复杂的新陈代谢和酶促反应。将3个对比组的DEGs与COG数据库进行对比分析,预测基因功能并分类统计(表4),结果表明转录类所占比例最大,其次是翻译后修饰和信号传导机制。
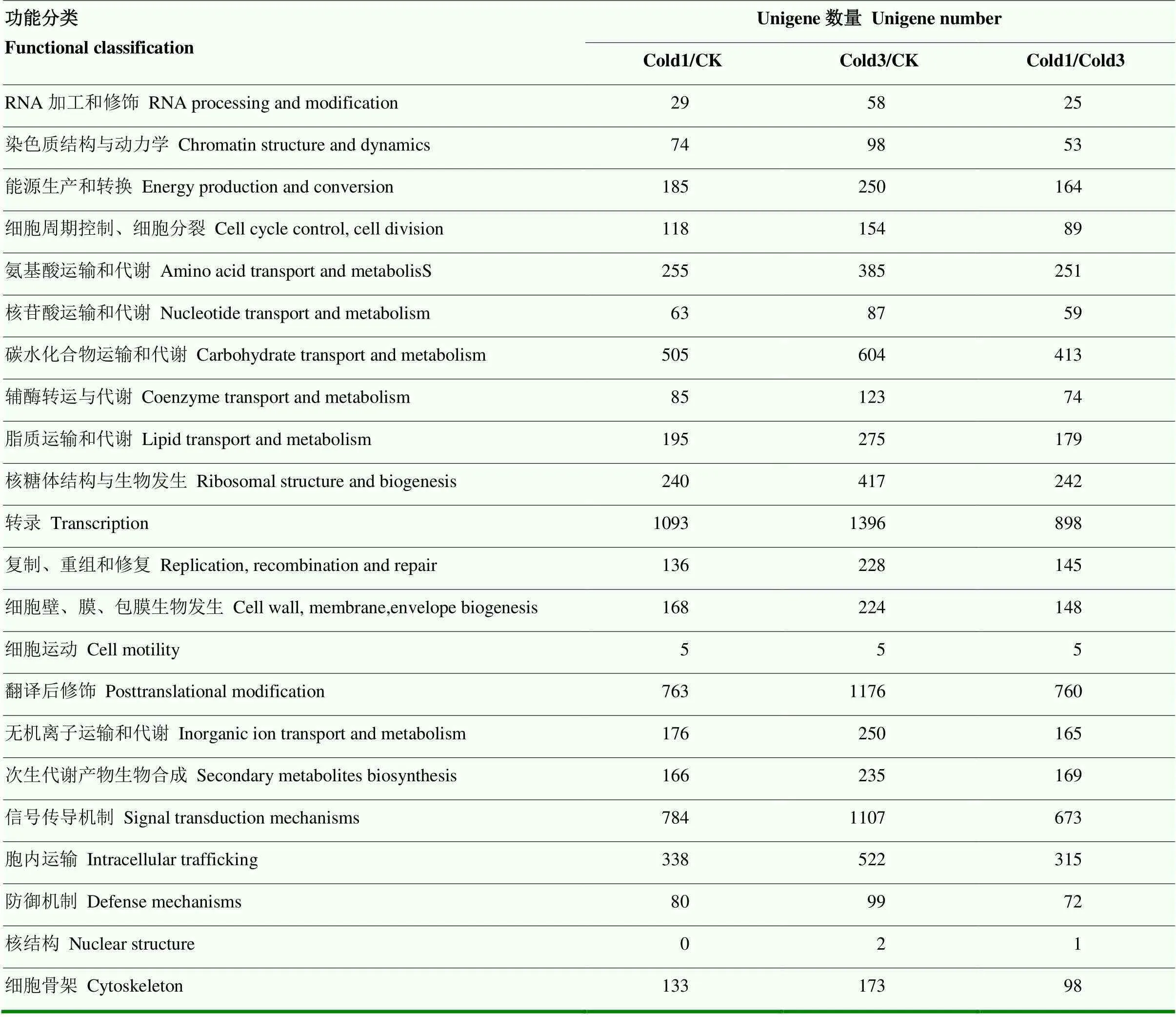
表4 差异表达基因的COG注释分类情况
2.4 差异表达基因KEGG富集分析
为了进一步探索巴西蕉在低温胁迫下的代谢途径变化,将低温处理组与对照组的DEGs及KEGG数据库进行对比分析(表5),同时利用气泡图展示DEGs在KEGG途径中的富集情况(图3)。结果显示,在Cold1 vs CK、Cold3 vs CK和Cold1 vs Cold3这3个对比组中,DEGs主要富集在光合作用、植物病原体相互作用、植物激素信号传导、MAPK信号通路、苯丙素生物合成、谷胱甘肽代谢和-亚麻酸代谢等途径,其中许多代谢通路与低温胁迫有关。
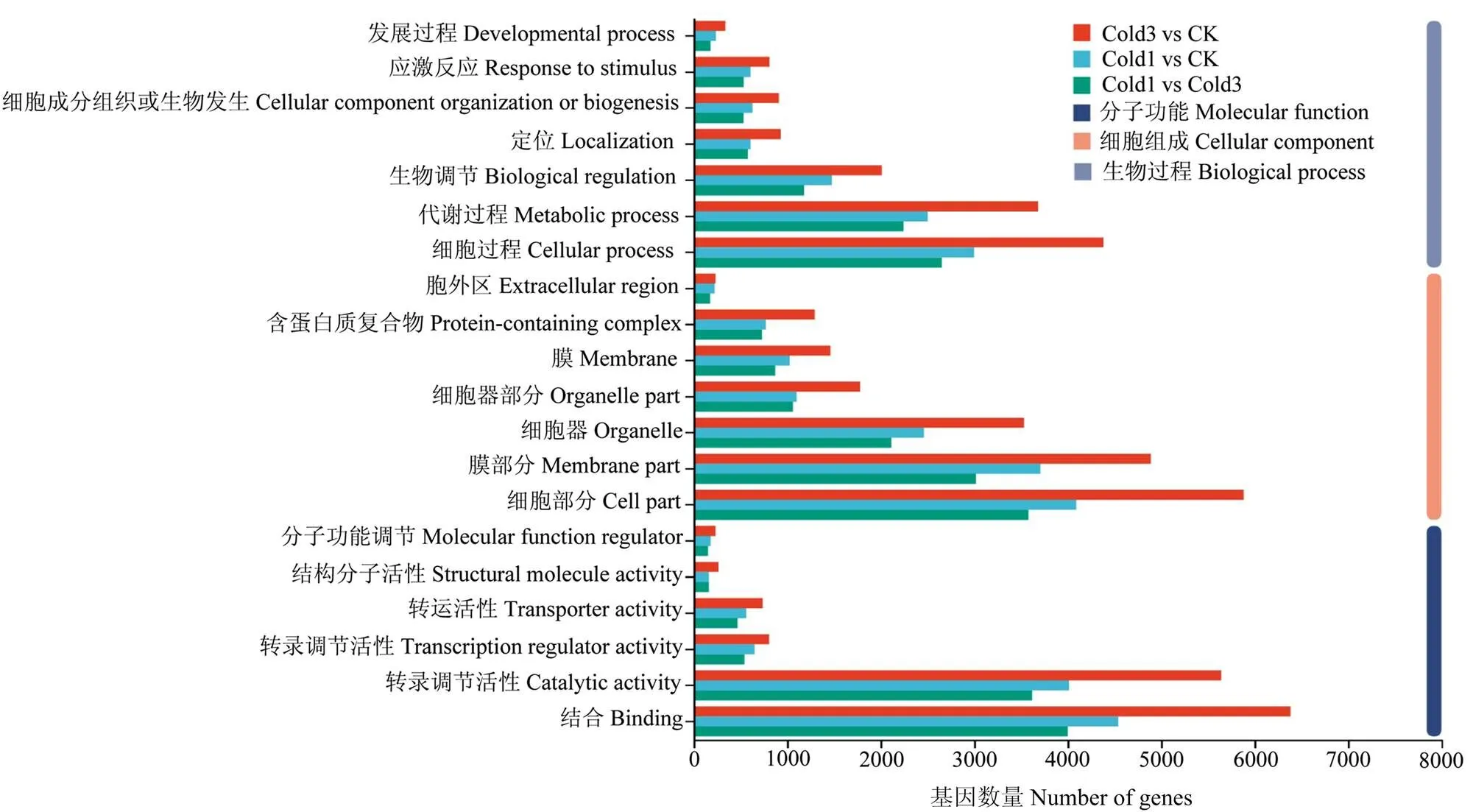
图2 差异表达基因GO功能分类
2.5 转录组qRT-PCR验证
为了验证转录组数据的准确性,对20个低温胁迫相关DEGs进行qRT-PCR验证,结果表明这20个基因在香蕉低温胁迫中的变化趋势与转录组的测序结果基本相符(图4),说明转录组数据较为可靠。
2.6 转录组与蛋白质组关联分析
前期研究中,课题组利用蛋白质组测序对巴西蕉叶片在低温胁迫中的蛋白质丰度变化进行了分析,共鉴定到6 223个蛋白,为了统计鉴定到的蛋白与mRNA之间的交叉情况,将蛋白ID与转录本ID进行比对,共鉴定到6 211个与转录本ID相对应的蛋白。转录组与蛋白组之间除了一对一的相互关系外,还存在其他更为复杂的调控关系,统计分析基因及其对应的蛋白表达量并绘制散点图(图5-A),结果显示转录组和蛋白质组之间呈正相关关系(=0.17)。以Log2FC>1且<0.05为显著差异表达上调,|Log2FC|≥1且<0.05为显著差异表达下调作为筛选条件,比较转录组和蛋白质组中差异表达的转录本和蛋白个数分布情况(图5-B)。结果显示,巴西蕉在低温胁迫时,叶片转录组中表达上调的基因数量多于下调的基因数量,而在蛋白质组中表达上调的蛋白质数量与表达下调的蛋白质数量相差不大。为了直观比较DEPs与DEGs的交叉情况,利用韦恩图对交叉情况进行分析(图5-C),结果显示有105个转录本及其对应的蛋白表达量共同上调,有218个转录本及其对应的蛋白表达量共同下调。
为了比较分析各差异表达基因和蛋白的功能,通过GO功能富集分析对与DEGs表达趋势相同的DEPs进行分析,如图6所示,当DEGs和DEPs共同上调时,其功能主要富集在一元羟酸代谢过程、线粒体、叶绿体、质体、氧化还原酶活性和离子结合等;当DEGs和DEPs共同下调时(图7),其功能主要富集在对光的响应、叶绿体、叶绿素、质体、蛋白质结构特异性结合以及氧化还原酶活性等;为了关联分析DEGs-DEPs在各信号和代谢通路上的联系,通过值来比较这些KEGG通路在不同基因和蛋白分类下的富集程度,采用热图的形式可视化展示结果(图8)。结合图3筛选的低温胁迫相关代谢通路进行对比分析,发现苯丙素生物合成(map00940)、光合作用信号途径(map00195,map00196)这两个通路的相关基因和蛋白的表达量在低温处理后均下调;而-亚麻酸代谢(map00592)的相关基因和蛋白的表达量在低温处理后均上调;另外2种代谢通路的DEGs和其对应蛋白的表达变化不一致,如谷胱甘肽途径(map00480)的相关基因表达量在低温处理后产生上调或下调的变化,而其相关蛋白的表达量普遍升高。

A: Cold1 vs CK; B: Cold3 vs CK; C: Cold1 vs Cold3

表5 部分差异表达Unigene的代谢通路
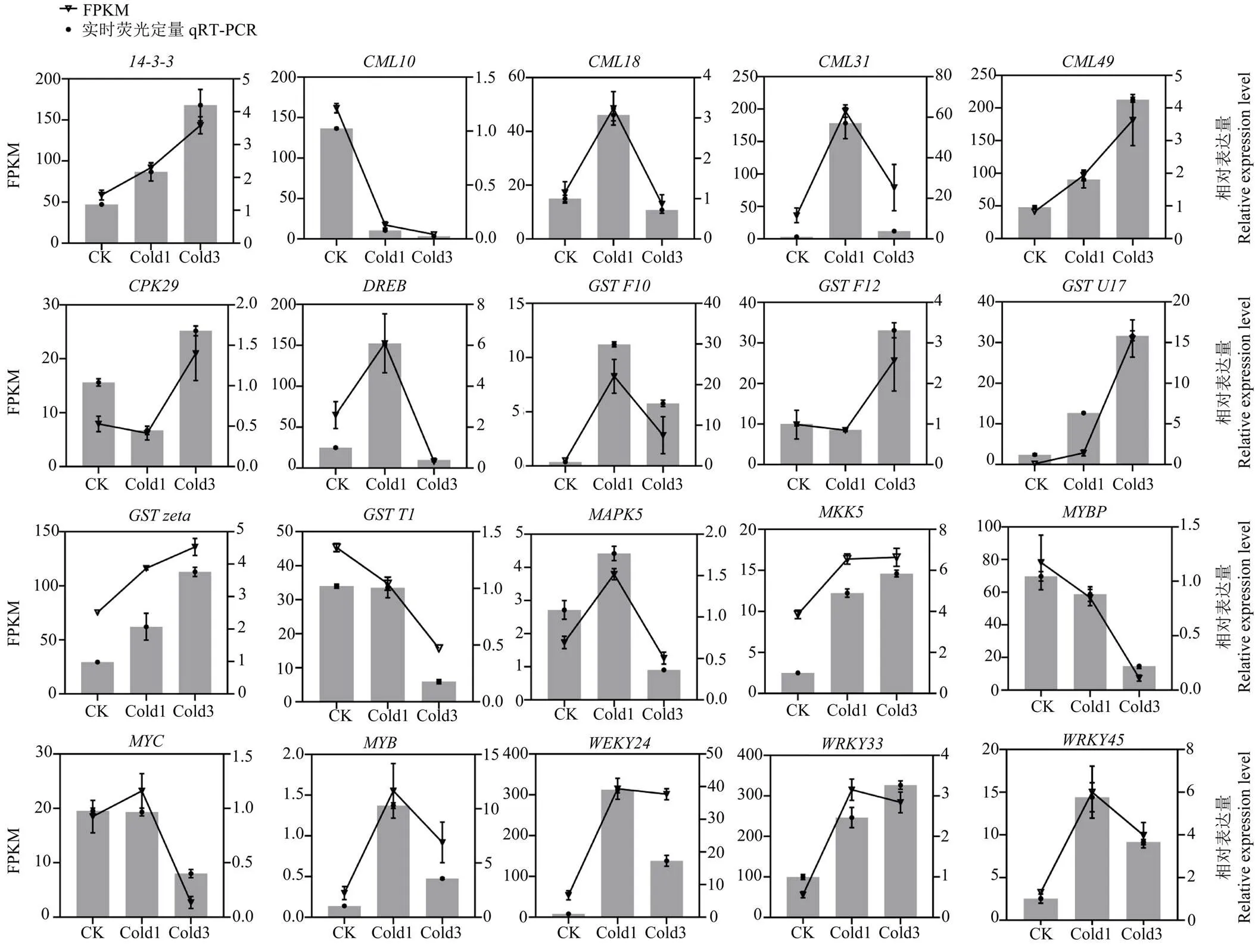
图4 qRT-PCR验证结果

A:转录本与其对应蛋白表达量相关性分析;B:差异表达转录本与蛋白质分布统计图;C:差异表达蛋白与转录本比较分析韦恩图
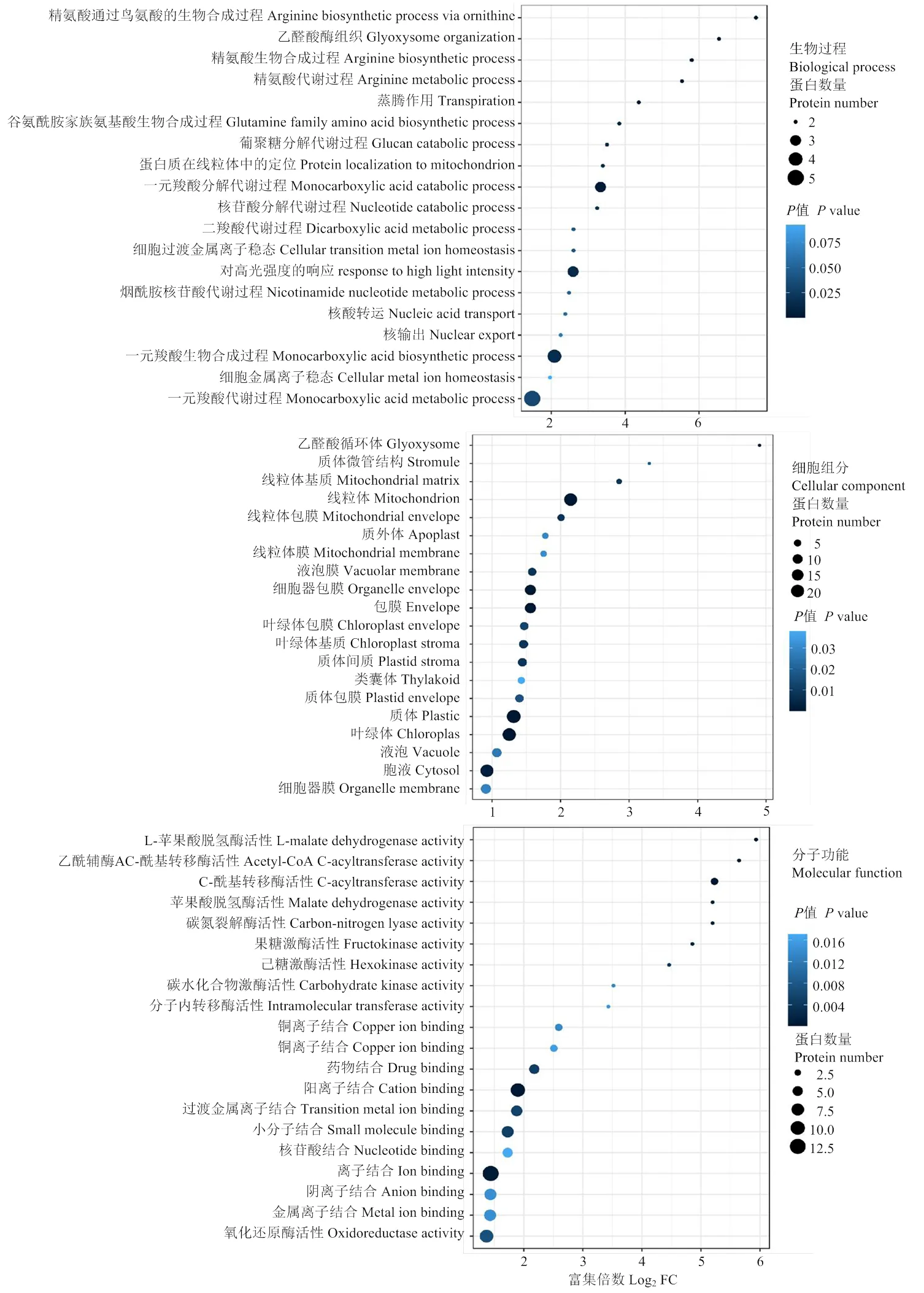
图6 与DEGs共同上调的DEPs的GO富集分析
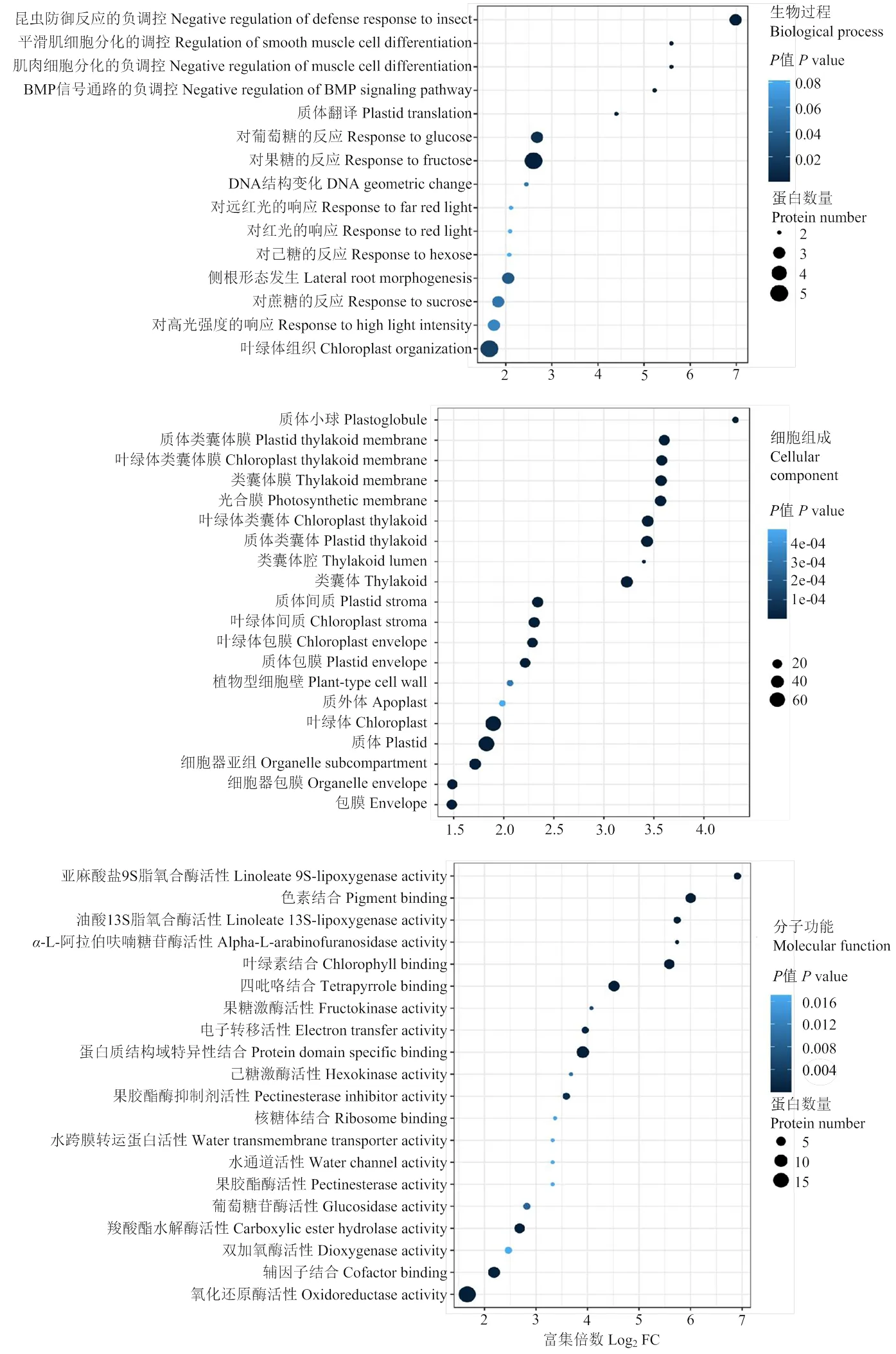
图7 与DEGs共同下调的DEPs的GO富集分析
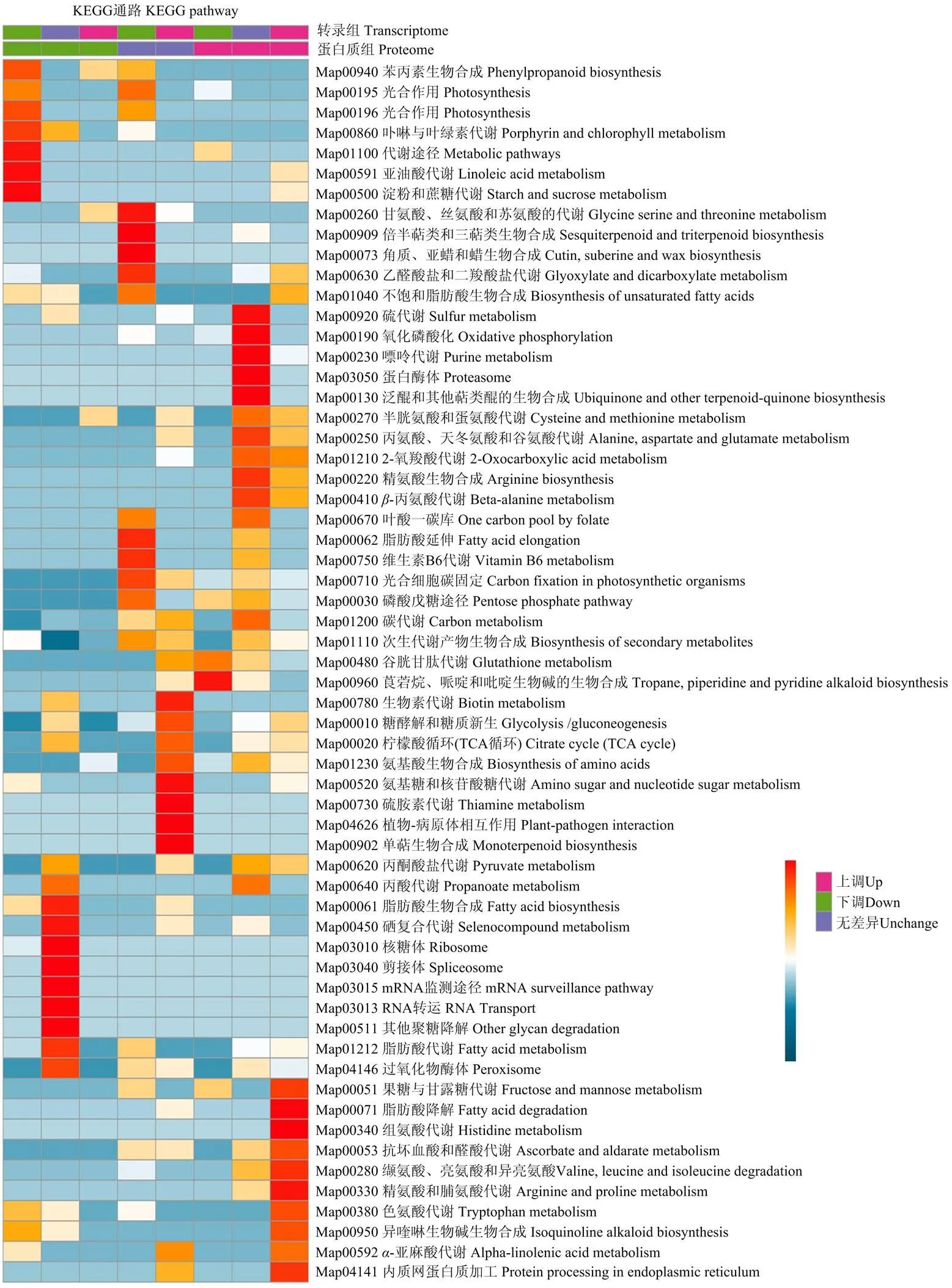
图8 基于KEGG通路的DEGs-DEPs关联分析
3 讨论
3.1 低温胁迫影响香蕉叶片基因与蛋白表达
蛋白质合成、降解等代谢过程在植物响应低温胁迫中起着重要作用[16],本研究中,鉴定到了大量的DEGs和DEPs,表明低温处理使巴西蕉叶片中大量基因和蛋白的表达水平产生了显著变化。分析结果显示,DEGs和DEPs表达一致的共有323对,另外有148对呈负相关关系,此外还有大量的基因表达无显著差异而蛋白表达差异显著或蛋白表达无显著差异而基因表达差异显著的情况,其原因可能是细胞中的蛋白质丰度受多种因素的影响和控制,如蛋白质的翻译水平、半衰期、合成速率和数量,都会导致蛋白质丰度与mRNA的显著差异;此外,翻译效率、技术平台成熟度和测序方法之间的差异可能导致相关性较低[17-18]。
3.2 低温胁迫影响光合作用信号
光合作用信号途径与冷胁迫之间存在着复杂的互作关系[19-20]。在低温条件下,生物体内的光合作用电子传递和碳固定的效率会降低,从而抑制其吸收光的能力[20]。此外,光合作用电子传递能力的下降也是导致光抑制和氧自由基增加的主要原因[21]。本研究中,低温处理后,当DEGs和DEPs共同下调时,其功能在光响应、叶绿素和叶绿体方面大量富集,且光合作用信号途径相关基因和蛋白的表达量都下降,光合生物中的碳固定相关基因表达量下降,说明低温处理抑制了香蕉叶片的光合作用,进而对香蕉的正常生长产生影响。
3.3 低温胁迫对谷胱甘肽代谢的影响
在植物中,谷胱甘肽巯基转移酶基因超家族(GSTs)根据序列相似性、免疫学活性和结构构象被分为11个亚类,其中Tau、Phi、DHAR和Lambda 4个亚类为植物中特有[22],能够有效缓解植物在非生物胁迫中如低温胁迫、盐胁迫造成的氧化损伤[23-25]。而不同植物物种响应低温胁迫的GST亚类也不同,如拟南芥中的Tau[26]、茄属植物中的Phi[27]、水稻中的Zeta[28]等。在本研究中,通过关联分析鉴定到了许多差异表达的GST基因和蛋白,主要为Tau亚类(LOC103997659,LOC103997954)、Phi亚类(LOC103971959,LOC103981216,LOC103982403)、Zeta亚类(LOC103989353,LOC103995971)和Theta亚类(LOC103973270,LOC103973269)。本研究发现,冷处理3 d的谷胱甘肽代谢途径较1 d的更活跃,而其他4种信号代谢通路上两个处理的差异基因表达情况基本相同,说明谷胱甘肽代谢途径在香蕉响应低温胁迫有一定的滞后性,随着低温时间的增加而逐渐活跃。关联分析的KEGG富集结果显示(图8),低温处理后激活了谷胱甘肽代谢途径(map00480)相关蛋白的表达,说明谷胱甘肽代谢途径在香蕉响应低温胁迫中发挥重要作用。
3.4 低温胁迫对α-亚麻酸代谢的影响
-亚麻酸是多元不饱和脂肪酸,与植物抗氧化能力密切相关,是脂质代谢的重要组成部分,对植物抵御低温胁迫起着重要的作用[29-30]。植物在应对非生物胁迫时,通过释放-亚麻酸代谢来维持细胞膜的流动性,同时在膜脂和膜蛋白的相互作用下抵御不良环境造成的影响[31]。本研究中,当DEGs和DEPs共同下调时,在亚麻酸脂氧合酶方面富集,说明低温处理对亚麻酸属不饱和脂肪酸的催化能力降低,增加了其在香蕉叶片中的含量;同时,低温处理后有许多差异表达基因和蛋白在-亚麻酸代谢途径中富集(map00592),且无论是基因还是其对应的蛋白表达量均上调,说明低温诱导了-亚麻酸的合成及其代谢途径。-亚麻酸还与茉莉酸(JA)生物合成密切相关,通过脂质代谢途径诱导JA的合成[32],研究表明,JA能够减轻番茄低温诱导的氧化胁迫[33]。本研究通过关联分析鉴定到3个参与JA生物合成途径的差异表达基因和蛋白,包括2个3-酮酰基-CoA硫解酶(LOC103973111,LOC103985063)和4-双酸辅酶A连接酶(LOC103994510),且这3个差异基因和蛋白的表达量在低温处理后均上调,说明香蕉在遭受低温胁迫后,通过对-亚麻酸及JA合成途径的调节改变叶片中的脂质组成成分及相关植物激素的含量,以抵御低温环境。
3.5 低温胁迫对苯丙素生物合成的影响
苯丙素类物质是植物体内重要的代谢产物,当植物受到非生物胁迫时,在体内快速积累黄酮与类黄酮化合物,清除植物体内自由基,缓解胁迫对植物造成的损害[34]。PAL是酚类化合物合成途径中的第一个关键酶,促进L-苯丙氨酸催化转化为反式肉桂酸,而后与4CL共同作用合成4-香豆酰辅酶A,在植物冷胁迫中发挥重要作用[35-37],而CHI主要通过催化查尔酮转化为黄烷酮来提高植株的抗逆性[38]。本研究对苯丙氨酸解氨酶、4-香豆酸:辅酶A连接酶和查尔酮异构酶这几个苯丙素代谢途径关键酶[39]进行关联分析。其中,在低温处理后,PAL(LOC103985827)和4CL(LOC103994510)基因及其对应的蛋白表达量均上升,还鉴定到两个显著上调的CHI蛋白,但其对应的基因表达一个下调(LOC103981836),另一个无显著差异(LOC103972228),推测低温诱导并增加了这些苯丙素代谢途径关键酶的活性,在香蕉抵御低温胁迫中发挥作用。
4 结论
低温处理改变了巴西蕉叶片中基因的表达水平,GST基因家族、MAPK基因家族和DREB等低温调控关键基因的表达量在低温处理后显著上调。结合蛋白质组学进行关联分析发现,低温处理后巴西蕉叶片中的DEPs和DEGs呈正相关关系,其基因功能主要富集于线粒体、光的响应和氧化还原酶活性等。此外,对筛选出的低温胁迫关键信号通路进行关联分析发现,苯丙素生物合成和光合作用信号途径这两个通路的相关基因和蛋白表达量在低温处理后均下调,而-亚麻酸代谢的相关基因和蛋白的表达量在低温处理后均上调,推测这些通路中的基因和蛋白表达量的变化是造成巴西蕉对低温敏感的重要原因。
[1] 吴烁凡, 何维弟, 李春雨, 董涛, 毕方铖, 盛鸥, 邓贵明, 胡春华, 窦同心, 高慧君, 刘思文, 易干军, 姚振, 杨乔松. 香蕉抗寒分子机制研究进展. 果树学报, 2022, 39(3): 483-494.
WU S F, HE W D, LI C Y, DONG T, BI F C, SHENG O, DENG G M, HU C H, DOU T X, GAO H J, LIU S W, YI G J, YAO Z, YANG Q S. Research progress in molecular mechanism of cold resistance in banana. Journal of Fruit Science, 2022, 39(3): 483-494. (in Chinese)
[2] 谢江辉. 新中国果树科学研究70年: 香蕉. 果树学报, 2019, 36(10): 1429-1440.
XIE J H. Fruit scientific research in New China in the past 70 years: banana. Journal of Fruit Science, 2019, 36(10): 1429-1440. (in Chinese)
[3] 王伟英, 邹晖, 林江波, 戴艺民. 香蕉抗寒技术研究进展. 东南园艺, 2018, 6(6): 61-66.
WANG W Y, ZOU H, LIN J B, DAI Y M. Research progress on techniques involved in cold resistance of banana. Southeast Horticulture, 2018, 6(6): 61-66. (in Chinese)
[4] 王安邦, 金志强, 刘菊华, 贾彩红, 张建斌, 苗红霞, 徐碧玉. 香蕉寒害研究现状及展望. 生物技术通报, 2014(8): 28-33.
WANG A B, JIN Z Q, LIU J H, JIA C H, ZHANG J B, MIAO H X, XU B Y. The current situation and prospects of banana chilling stress. Biotechnology Bulletin, 2014(8): 28-33. (in Chinese)
[5] PLOHOVSKA S G, YEMETS A I, BLUME Y B. Influence of cold on organization of actin filaments of different types of root cells in. Cytology and Genetics, 2016, 50(5): 318-323.
[6] SONAWANE B V, KOTEYEVA N K, JOHNSON D M, COUSINS A B. Differences in leaf anatomy determines temperature response of leaf hydraulic and mesophyll CO2conductance in phylogenetically related C4 and C3 grass species. The New Phytologist, 2021, 230(5): 1802-1814.
[7] SACK L, STREETER C M, HOLBROOK N M. Hydraulic analysis of water flow through leaves of sugar maple and red oak. Plant Physiology, 2004, 134(4): 1824-1833.
[8] ZHENG G H, PAN D M, NIU X Q, WU H W, ZHANG J B. Changes in cell Ca2+distribution in loquat leaves and its effects on cold tolerance. Horticultural Science and Technology, 2014, 32(5): 607-613.
[9] MA Y, DAI X Y, XU Y Y, LUO W, ZHENG X M, ZENG D L, PAN Y J, LIN X L, LIU H H, ZHANG D J, XIAO J, GUO X Y, XU S J, NIU Y D, JIN J B, ZHANG H, XU X, LI L G, WANG W, QIAN Q, GE S, CHONG K. COLD1 confers chilling tolerance in rice. Cell, 2015, 160(6): 1209-1221.
[10] LI M, DUAN X Y, GAO G, LIU T, QI H Y. CmABF1 and CmCBF4 cooperatively regulate putrescine synthesis to improve cold tolerance of melon seedlings. Horticulture Research, 2022, 9: uhac002.
[11] 耿小惠. 钙离子通道促进剂及抑制剂对香蕉抗寒性的影响及相关基因MaCNGC家族成员鉴定与表达分析[D]. 福州: 福建农林大学, 2022.
GENG X H. Effect of calcium channel promoters and inhibitors on cold resistance in banana and identification and expression analysis of MaCNGC family members [D]. Fuzhou: Fujian Agriculture and Forestry University, 2022. (in Chinese)
[12] 刘嘉鹏. 褪黑素对香蕉抗寒性的影响及其机制研究[D]. 福州: 福建农林大学, 2022.
LIU J P. Effects of melatonin on cold resistance of banana and study of its mechanism [D]. Fuzhou: Fujian Agriculture and Forestry University, 2022. (in Chinese)
[13] GAO J, DOU T X, HE W D, SHENG O, BI F C, DENG G M, GAO H J, DONG T, LI C Y, ZHANG S, YI G J, HU C H, YANG Q S. MaMAPK3-MaICE1-MaPOD P7 pathway, a positive regulator of cold tolerance in banana. BMC Plant Biology, 2021, 21(1): 97.
[14] LIN W, WU S J, WEI M. Ubiquitylome analysis reveals the involvement of ubiquitination in the cold responses of banana seedling leaves. Journal of Proteomics, 2023, 288: 104994.
[15] SCHEFE J H, LEHMANN K E, BUSCHMANN I R, UNGER T, FUNKE-KAISER H. Quantitative real-time RT-PCR data analysis: Current concepts and the novel “gene expression’s CT difference” formula. Journal of Molecular Medicine, 2006, 84(11): 901-910.
[16] JANMOHAMMADI M, ZOLLA L, RINALDUCCI S. Low temperature tolerance in plants: Changes at the protein level. Phytochemistry, 2015, 117: 76-89.
[17] GHAZALPOUR A, BENNETT B, PETYUK V A, OROZCO L, HAGOPIAN R, MUNGRUE I N, FARBER C R, SINSHEIMER J, KANG H M, FURLOTTE N,. Comparative analysis of proteome and transcriptome variation in mouse. PLoS Genetics, 2011, 7(6): e1001393.
[18] RODRIGUES R S, BOLDRINI-FRANÇA J, FONSECA F P P, DE LA TORRE P, HENRIQUE-SILVA F, SANZ L, CALVETE J J, RODRIGUES V M. Combined snake venomics and venom gland transcriptomic analysis of. Journal of Proteomics, 2012, 75(9): 2707-2720.
[19] SAVITCH L V, ALLARD G, SEKI M, ROBERT L S, TINKER N A, HUNER N P A, SHINOZAKI K, SINGH J. The effect of overexpressionof twoCBF/DREB1-like transcription factors on photosynthetic capacity and freezing tolerance in. Plant & Cell Physiology, 2005, 46(9): 1525-1539.
[20] HUNER N P A, ÖQUIST G, SARHAN F. Energy balance and acclimation to light and cold. Trends in Plant Science, 1998, 3(6): 224-230.
[21] ASADA K. Production and Action of Active Oxygen Species in Photosynthetic Tissues. Boca Raton: CRC Press, 2020: 77-104.
[22] VIJAYAKUMAR H, THAMILARASAN S K, SHANMUGAM A, NATARAJAN S, JUNG H J, PARK J I, KIM H, CHUNG M Y, NOU I S. Glutathione transferases superfamily: Cold-inducible expression of distinctgenes in. International Journal of Molecular Sciences, 2016, 17(8): 1211.
[23] XU H Y, YU C. Transcriptomic analysis reveals crucial biological pathways associated with cold response inin Guizhou Province, China. Scientia Horticulturae, 2022, 295: 110883.
[24] SOUSA B, LOPES J, LEAL A, MARTINS M, SOARES C, AZENHA M, FIDALGO F, TEIXEIRA J. Specific glutathione-S- transferases ensure an efficient detoxification of diclofenac inL. plants. Plant Physiology and Biochemistry, 2021, 168: 263-271.
[25] SUBRAMANIAN S, SOULEIMANOV A, SMITH D L. Thuricin17 production and proteome differences inNEB17 cell-free supernatant under NaCl stress. Frontiers in Sustainable Food Systems, 2021, 5: 630628.
[26] SAPPL P G, CARROLL A J, CLIFTON R, LISTER R, WHELAN J, HARVEY MILLAR A, SINGH K B. Theglutathione transferase gene family displays complex stress regulation and co-silencing multiple genes results in altered metabolic sensitivity to oxidative stress. The Plant Journal, 2009, 58(1): 53-68.
[27] SEPPÄNEN M M, CARDI T, BORG HyökkI M, PEHU E. Characterization and expression of cold-induced glutathione S- transferase in freezing tolerant, sensitiveand their interspecific somatic hybrids. Plant Science, 2000, 153(2): 125-133.
[28] TSUCHIYA T, TAKESAWA T, KANZAKI H, NAKAMURA I. Genomic structure and differential expression of two tandem-arrangedgenes in rice. Gene, 2004, 335: 141-149.
[29] HONG M J, JANG Y E, KIM D G, KIM J M, LEE M K, KIM J B, EOM S H, HA B K, LYU J I, KWON S J. Selection of mutants with high linolenic acid contents and characterization of fatty acid desaturase 2 and 3 genes during seed development in soybean (max). Journal of the Science of Food and Agriculture, 2019, 99(12): 5384-5391.
[30] 李国斌, 党林学, 郑国强, 董小云, 魏家萍, 崔俊美, 李辉, 王莹, 田海燕, 刘自刚. 甘蓝型冬油菜亚麻酸代谢参与应答低温胁迫的转录组学分析. 分子植物育种, 2023. https://kns.cnki.net/kcms2/ detail/46.1068.s.20230615.1429.008.html.
LI G B, DANG L X, ZHENG G Q, DONG X Y, WEI J P, CUI J M, LI H, WANG Y, TIAN H Y, LIU Z G. Analysis of the linolenic acid metabolism involving low temperature stress-response in leaves of brassica napus based on RNA-seq. Molecular Plant Breeding, 2023. https://kns.cnki.net/kcms2/detail/46.1068.s.20230615.1429.008.html. (in Chinese)
[31] KOH I. Acclimative response to temperature stress in higher plants: approaches of gene engineering for temperature tolerance. Annual Review of Plant Biology, 2002, 53: 225-245.
[32] OLSSON M, NILSSON K, LILJENBERG C, HENDRY G A F. Drought stress in seedlings: Lipid metabolism and Lipid peroxidation during recovery from drought inand. Physiologia Plantarum, 1996, 96(4): 577-584.
[33] DING F, WANG C, XU N, WANG M L, ZHANG S X. Jasmonic acid-regulated putrescine biosynthesis attenuates cold-induced oxidative stress in tomato plants. Scientia Horticulturae, 2021, 288: 110373.
[34] JANNATIZADEH A. Exogenous melatonin applying confers chilling tolerance in pomegranate fruit during cold storage. Scientia Horticulturae, 2019, 246: 544-549.
[35] CHENG Y D, LIU L Q, ZHAO G Q, SHEN C G, YAN H B, GUAN J F, YANG K. The effects of modified atmosphere packaging on core browning and the expression patterns of PPO and PAL genes in ‘Yali’ pears during cold storage. Lwt-Food Science and Technology, 2015.
[36] SANCHEZ-BALLESTA M T, LAFUENTE M T, ZACARIAS L, GRANELL A. Involvement of phenylalanine ammonia-lyase in the response of Fortune mandarin fruits to cold temperature. Physiologia Plantarum, 2000, 108(4): 382-389.
[37] VOGT T. Phenylpropanoid biosynthesis. Molecular Plant, 2010, 3(1): 2-20.
[38] CHRISTIE P J, ALFENITO M R, WALBOT V. Impact of low- temperature stress on general phenylpropanoid and anthocyanin pathways: Enhancement of transcript abundance and anthocyanin pigmentation in maize seedlings. Planta, 1994, 194(4): 541-549.
[39] JANAS K M, CVIKROVÁ M, PAŁĄGIEWICZ A, EDER J. Alterations in phenylpropanoid content in soybean roots during low temperature acclimation. Plant Physiology and Biochemistry, 2000, 38: 587-593.
Transcriptome and Proteome Association Analysis to Revealthe Molecular Mechanism of Baxi Banana Seedlings in Response to Low Temperature
LIN Wei, WU ShuiJin, LI YueSen
Subtropical Agriculture Research Institute, Fujian Academy of Agricultural Sciences, Zhangzhou 363005, Fujian
【Objective】 Low temperature is a significant natural disaster that affects banana production. In this study, based on transcriptome and proteome association analysis, the regulatory network of genes, proteins, signals and metabolic pathways involved in banana cold resistance was investigated. The aim was to explore the molecular mechanism of banana cold resistance.【Methods】‘Baxi’ banana (Lour) was treated at 7 ℃ for 1 and 3 d, and a control group was treated at 28 ℃. Based on the proteome data obtained in the previous study, the transcriptome sequencing technology was used to detect changes in the gene regulatory network of banana under cold stress. Simultaneously, the correlation analysis was conducted with proteomics to analyze the molecular mechanism of banana response to cold stress. 【Result】 Transcriptome analysis revealed that 11 370, 15 460 and 9 619 differentially expressed genes were identified in the three comparison groups of Cold1 vs CK, Cold3 vs CK and Cold1 vs Cold3, respectively. KEGG enrichment analysis of these genes revealed that the differentially expressed genes were enriched in several key signaling metabolic pathways, such as photosynthesis signal, glutathione metabolism,-linolenic acid metabolic pathway and phenylpropanoid biosynthesis under low temperature stress. Moreover, there were significant differences in the enrichment degree of glutathione metabolic pathway between Cold1 d vs CK and Cold3 d vs CK. Real-time quantitative PCR analysis (qRT-PCR) was performed on several differentially expressed genes. Among them, the expression levels of key low-temperature regulatory genes, such as DREB, MAPK and MYB, were significantly increased after the low-temperature treatment. The expression trend of the selected 20 genes was essentially consistent with that of RNA-seq, confirming the accuracy of RNA-seq. The results of the transcriptome and proteome association analysis showed a positive correlation between the transcriptome and proteomics. A total of 6 211 proteins corresponding to transcripts were identified. Among these, 105 transcripts and their proteins were up-regulated, while 218 transcripts and their proteins were down-regulated. GO enrichment analysis showed that the differentially expressed genes and proteins were enriched in functions, such as photoresponse, chloroplast and oxidoreductase activity. Furthermore, the correlation analysis of differentially expressed genes and protein KEGG pathway revealed that the low temperature treatment suppressed the expression of genes and proteins related to phenylpropanoid biosynthesis and photosynthesis signaling pathway, while promoting the expression of proteins associated with-linolenic acid metabolism and the glutathione pathway. 【Conclusion】 The transcriptome and proteomics were used to map the regulatory network of banana cold resistance at the gene and protein levels. It was found that the signal pathway of banana response to low temperature mainly involved photosynthesis signal, glutathione metabolism,-linolenic acid metabolism and phenylpropanol biosynthesis.
Lour; low temperature stress; transcriptome; proteome; comparative analysis

10.3864/j.issn.0578-1752.2024.08.012
2023-09-18;
2023-12-15
福建省公益类科研院所专项(2022R1030006,2021R1030006)、福建省自然科学基金(2023J01374)
通信作者林蔚,E-mail:Linw102@163.com
(责任编辑 赵伶俐)
