籼粳杂种不育的遗传分析和候选基因鉴定
2024-04-24许娜唐颖徐正进孙健徐铨
许娜,唐颖,徐正进,孙健,徐铨
籼粳杂种不育的遗传分析和候选基因鉴定
许娜,唐颖,徐正进,孙健,徐铨
沈阳农业大学水稻研究所,沈阳 110866
【目的】籼粳亚种间杂交F1的育性问题严重阻碍了亚种间杂种优势的利用,探究籼粳杂种不育的遗传机理,挖掘新的籼粳杂种不育调控基因,为促进籼粳杂种结实率遗传改良提供理论依据。【方法】粳稻品种Sasanishiki和籼稻品种Habataki杂交后,采用单粒传法自交10代,获得包含95个株系的稳定遗传重组自交系(RIL),基于Illumina平台,对双亲和RIL进行高通量测序,在全基因组水平分析RIL中Habataki血缘的分布与比例,进而鉴定偏分离区域作为潜在的籼粳杂种不育位点。同时,剖析“全球3000份水稻核心种质资源重测序计划”中典型籼粳稻基因组变异数据,对群体水平进一步验证并趋近目标育性基因区域。最终通过序列比对锁定籼粳杂种不育候选基因。应用CRISPR基因编辑技术进行定点基因敲除,对目标基因进行功能验证。【结果】Habataki和Sasanishiki的杂交F1在穗数、每穗粒数和千粒重上体现出明显的杂种优势,但其结实率显著降低,I2-KI镜检发现F1花粉育性显著降低。RIL的全基因组高通量测序在第1、3、5、6、7和12染色体上检测到明显的偏分离,即该区域的基因型趋向于籼稻Habataki。通过序列比对,进一步确定已知育性基因、和为第3、6和12染色体上偏分离区域的目标基因。应用CRISPR基因编辑技术敲除Habataki中的多拷贝,成功改善了其与Sasanishiki杂交F1的花粉育性和结实率,说明该基因可独立行使功能。同时,在第1染色体的偏分离区域发现Habataki和Sasanishiki之间存在复杂的结构性变异,Sasanishiki基因组中一段24.7 kb包含4个预测基因的片段在Habataki中被一段64.8 kb包含10个预测基因的片段取代,此结构性变异可能参与籼粳杂种育性的调控。【结论】检测到多个籼粳杂种不育相关位点,应用CRISPR基因编辑技术敲除Habataki中的多拷贝成功改善其杂种F1育性,确定其为水稻亚种间育性改良的目标基因。同时锁定了第1染色体区域的目标基因。
水稻;籼粳杂种不育;高通量测序;基因编辑;候选基因
0 引言
【研究意义】水稻籼粳亚种间杂种优势突出,可作为提高水稻单产的重要途径,但是籼粳间杂种育性较低,限制了籼粳杂种优势的育种应用。因此,阐明水稻籼粳杂种不育的遗传机制,挖掘新的籼粳杂种育性调控基因,深入解析育性调控的分子机理,将为籼粳杂交育种与杂种优势利用取得新突破提供理论基础和种质资源。【前人研究进展】水稻是世界重要粮食作物,为全球约一半人口提供主食,其中90%以上集中在亚洲。亚洲栽培稻分为籼(/)和粳(/)2个亚种,在长期自然选择和人工选择过程中,籼粳稻基因组不断分化,生态适应性等生物学特性产生显著差异,农艺性状各有利弊[1-4]。籼稻广泛分布在印度、中国、东南亚,约占10%的粳稻集中分布在中国、日本、韩国等[5]。中国是世界上唯一同时大面积种植籼稻和粳稻的国家,约占2/3的籼稻主要分布于纬度和海拔较低地区,粳稻主要分布于纬度和海拔较高地区,中部地区籼粳交错。我国籼粳并重,促使相关遗传基础研究成为得天独厚且成果卓著的优势研究领域[6-8]。杂交水稻技术的开发和应用为水稻增产作出了巨大贡献[9-10]。在过去的几十年里,仅在中国,杂交水稻每年就种植约1 700万hm2(约占水稻总种植面积的60%),粮食产量比自交系品种提高了约20%[10]。然而,目前杂交水稻品种主要由同一亚种的品系之间的杂交培育而成,大多数是籼稻品系,由于亲本系的遗传多样性狭窄,杂交产量已达到平稳水平,导致杂种优势较低。相比之下,粳稻和籼稻品种之间的杂交种具有更强的抗逆性,因此,具有进一步提高产量潜力的巨大前景。在籼粳稻杂交中,多个籼粳杂种不育基因座的累积遗传效应导致花粉和小穗的受精率非常低,严重影响杂交种结实率,因此,籼粳杂种不育是阻碍利用籼粳间杂种优势获得更高产量的主要障碍,克服籼粳杂种不育仍然是高产水稻育种的主要挑战。遗传学研究已发现大约50个与杂种不育相关的基因位点,提出了2种解释水稻杂种不育模型,单位点孢子-配子体相互作用模型和重复配子-致死模型[11-12]。前者指孢子体细胞中单个基因位点的不同等位基因之间的遗传相互作用会导致携带特定等位基因的配子致死;后者指2个基因座的不同等位基因之间的上位相互作用导致杂种不育。迄今为止,已经成功克隆的籼粳杂种不育基因位点主要有[13]、[14]、[15]、[16]、/[17]和[18]。【本研究切入点】籼粳杂种不育是受多个基因控制,依靠某一种或几种不育基因位点的等位基因相互作用很难完全解释籼粳不育遗传机理,更多籼粳亚种杂种不育位点的挖掘将深入育性调控的分子遗传机理研究,尤其是发掘到可独立提升杂种育性的关键基因位点,将为水稻籼粳杂种优势利用作出贡献。【拟解决的关键问题】本研究围绕籼粳杂种不育遗传分析和调控基因位点挖掘这一关键科学问题,以籼粳交重组自交系为试验试材,应用高通量测序和CRISPR基因编辑等方法,探索籼粳杂种不育的遗传机理,挖掘新的籼粳不育调控位点,为亚种间杂交育性的遗传改良提供理论依据。
1 材料与方法
1.1 供试水稻材料
以粳稻品种笹锦Sasanishiki(Sasa)和籼稻品种Habataki(Haba)为双亲杂交获得F1植株,采用单粒传法套袋自交10代,获得包含95个株系的稳定遗传重组自交系(RIL)。亲本、F1植株和重组自交系于2022年春季4月23日播种,5月23日种植于沈阳农业大学水稻所试验田(41°N,123°E),每个株系按照3行×10株规模种成小区,株距为10 cm,行距为30 cm,每穴单苗插植,常规水肥管理。
1.2 表型鉴定
抽穗后45 d,每小区取中部5株收获,充分晒干后,考察单株穗数。取长势均匀的5穗,统计每穗粒数和结实率。随机取100粒饱满籽粒称重统计千粒重。最后运用Microsoft Excel 2021计算各材料每株的各性状平均值、标准差,并对各性状进行两尾等方差检验,使用GraphPad Prism 8进行作图。用1% I2-KI溶液进行花粉育性的鉴定。
1.3 DNA提取与全基因组测序和遗传图谱构建
插秧后3周,取幼嫩叶片,采用CTAB法提取DNA,送北京百迈克生物科技有限公司进行高通量测序分析,重组自交系的测序共获得207.50 Gb数据,平均深度为5.46×。使用Lep-MAP3(ref)软件的OrderMarkers2模块,计算标记间的遗传距离和各个染色体的图距,主要参数设置为:identical Limit=0.01,min Error=1e-5,sex Averaged=0,informative Mask=123,use Kosambi=1,其他参数使用默认值。利用划bin策略进行图谱构建,得到2 241个bins。
1.4 RNA提取和qRT-PCR
使用TRIzol试剂(Invitrogen,USA)从Sasa、Haba及其杂交种的水稻组织(花药、幼穗、叶、茎和根)中提取总RNA。RNA样品用DNaseⅠ(Promega,USA)处理。使用Superscript Ⅲ逆转录酶(Invitrogen,USA)和寡聚dT或-特异性引物从2—3 μg总RNA合成第一链cDNA。以1()为内参,对进行qRT-PCR分析,试验设置3次生物学重复,并用双尾Student’s检验分析其显著性。SYBR Green QPCR混合物(Bio-Rad)用于qRT-PCR。使用Bio-rad CFX Connect实时PCR系统进行反应:95 ℃ 3 min;95 ℃ 10 s,60 ℃15 s,72 ℃ 20 s,42个循环。()在Sasa中的表达模式参考自水稻表达谱数据库(http://ricexpro.dna. affrc.go.jp)。
1.5 CRISPR/Cas9基因编辑
以籼稻品种Haba为遗传背景材料,进行重组载体转化。转化用的农杆菌为EHA105菌种,原核感受态细胞为大肠杆菌DH5α菌株。根据和序列存在多态性的区间,运用华南农业大学亚热带农业生物资源保护与利用国家重点实验室刘耀光院士团队开发的基因编辑工具包CRISPR-GE(http://skl.scau.edu.cn)进行靶位点设计,基因编辑靶点序列、引物合成及测序服务均由华大基因完成。参照Li等[19]方法进行基因编辑载体的构建和基因编辑植株的遗传转化和筛选。
1.6 籼粳亚种群体基因组分化分析
基于“全球3000份水稻核心种质资源重测序计划”(3K Rice Project)的数据库中样本背景信息,首先根据群体结构Structure信息去掉中间型组群-adm、-adm、aus(普遍具有广亲和性)、bus、admix。在剩下的籼稻亚种-1a、-1b、-2、-3中,将Structure模式图中体现为-2与aus血缘互渗的种质材料删掉,最后保留209份-1a、205份-1b、475份-3,粳稻亚种中保留319份-tmp与388份-trp的材料。由于现代水稻育种已将亚种间生殖隔离打破,目前主栽品种中很多具有混合的亚种血缘,因此,在筛选到的籼粳种质中,进一步选择农家品种将使其血统更加纯粹。最终在3K Rice Project数据库中筛选并下载424份典型籼稻农家品种(landrace-I)与238份典型粳稻农家品种(landrace-J)的相关数据。应用bcftools软件提取上述2个亚种入选种质的vcf文件。应用vcftools软件分别计算全部染色体、第1染色体的100 kb窗口与第1染色体目标QTL区域单独SNP的群体分化系数st,利用显著峰值判定在群体中基因组分歧强烈的SNP位点,作为生殖隔离的基因组响应位点,可视化用R的CMplot包。
2 结果
2.1 F1杂种优势
调查粳稻品种Sasa、籼稻品种Haba及其杂交F1,发现籼粳杂交F1展现出杂种优势(图1-A—B)。双亲株高没有显著差别,F1代株高显著高于双亲(图1-C)。Sasa穗数显著多于Haba,杂交F1显著多于Sasa(图1-D)。Haba每穗粒数显著多于Sasa,杂交F1每穗粒数超过200,显著多于Haba(图1-E)。Sasa和Haba之间千粒重没有显著差异,杂交F1千粒重显著高于双亲(图1-F)。Sasa和Haba之间结实率没有显著差异,达到90%,杂交F1结实率显著降低,仅达到20%(图1-G),不育籽粒并未受精,颖壳内空瘪,不育籽粒均匀分布在整个穗部。因此,F1籽粒结实率降低是限制发挥籼粳间杂种优势利用的主要问题。

A:株型;B:穗型;C:株高;D:穗数;E:每穗粒数;F:千粒重;G:结实率。不同字母表示差异显著(P<0.05)。下同
2.2 F1的花粉育性
Sasa和Haba杂交F1结实率显著降低,使用I2-KI镜检进行双亲和杂交F1花粉的育性检测。结果显示,Sasa和Haba的花粉表现出完全可育,可染色花粉比例没有显著差别,均达到90%(图2-A—B和图2-D),而杂交F1的可染色花粉比例显著降低,不足40%。不育花粉体积较小,呈圆球形或不规则形,空瘪无物,着色浅或不着色,大多为典型的空败花粉,少量为圆败花粉(图2-C—D)。因此,推测花粉育性降低是籼粳杂种F1结实率降低的原因之一。因为杂交F1结实率为20%,显著低于其花粉可染色比例,推测杂交F1存在一定比例的雌配子不育(图1-G和图2-C—D)。
2.3 F1杂种不育的遗传分析
为了阐明调控F1杂种不育的基因位点,对Sasa和Haba杂交后自交10代构建的包含95个株系的重组自交系(RIL)进行基于Illumina平台的全基因组高通量测序。重组自交系的测序共获得207.50 Gb数据,平均深度为5.46×,Haba测序得到22.94 Gb数据,测序深度为51.00×,Sasa测序得到23.24 Gb数据,测序深度为56.00×。比对双亲鉴定到1 947 668个SNP,过滤后得到1 591 495个高质量SNP进行遗传图谱构建,得到一个包含2 241个bins的遗传图谱(图3-A—B)。根据RIL的基因型,分析其Haba类型SNP在群体中的比例,绘制籼型血缘渗入比率示意图,发现虽然使用单粒传法构建RIL,但Haba血缘平均比例在全基因组中并不是维持在50%左右,其平均值为0.38,其中,第4、9、11和12染色体Haba血缘比例较低,第7染色体Haba血缘整体较高。在第1、3、6、7和12染色体检测到明显的偏分离,即该区域的基因型趋向于籼稻Haba,Haba血缘超过了平均值的2倍,超过了0.76,说明该区域在杂交后分离过程中有更多的籼稻Haba型配子保留下来(图3-C),暗示这些区域可能存在调控籼粳杂种育性的关键基因。

A:Sasa花粉I2-KI镜检;B:Haba花粉I2-KI镜检;C:F1(Sasa/Haba)花粉I2-KI镜检;D:Sasa、Haba和F1(Sasa/Haba)花粉可染色率
2.4 籼粳杂种不育基因序列比对
比对前人关于籼粳杂种不育调控位点的研究后发现第3、6和12染色体的偏分离峰与已克隆的关键籼粳杂种不育基因、和位置重合[14-16]。比对Sasa和Haba的位点的3个紧密相连的连锁基因开放阅读框(、和),发现Sasa和Haba为典型的粳稻和籼稻位点(图4);对比Sasa和Haba的位点,发现Sasa在位点只包含一个拷贝,但是Haba在该位点包含3个拷贝、和(图4)。比对(和)位点,发现位点在双亲之间存在丰富的多态性,并且位点的T缺失和2个SNP,导致移码突变并在外显子形成终止密码子。在本群体中并没有检测到已经克隆的籼粳杂种不育相关基因/、和[13, 17-18]。进一步比对这些基因在Sasa和Haba之间的多态性,结果显示,在Haba的位点并不存在与Kasalath相同的518 bp的转座子插入;比对(和)位点,发现Haba的位点序列与Sasa完全一致,这可能是本群体中没有检测到/和位点的原因。经比对发现Sasa和Haba在位点存在结构性变异,其中Sasa中一段11.2 kb包含和的片段被一段35.0 kb包含5个ORF(、、/、/和)的片段取代,但是在本群体中并没有检测到该位点的偏分离现象,暗示在Sasa和Haba的杂合背景下,的功能被其他育性恢复位点的遗传效应所补偿。
2.5 籼粳杂种不育位点Sc的基因编辑验证
Haba在位点存在多拷贝现象,其中,在第一个外显子中有一段2.0 kb的插入,并且缺失了第一个外显子的末端,导致其编码的DUF1618功能域缺失,此外,还在第一个内含子中有一段10.1 kb的大片段插入,因此,认为并不具备位点的功能,是一个无功能基因(图4-C)。籼稻Haba中的另外2个拷贝和均含有完整的拷贝,与的第一个外显子序列相似度分别为97.5%和97.8%,第二个外显子与完全一致(图5-A)。Sasa的单拷贝仅在花药中表达,而Haba的多拷贝使其在花药中表达量显著增加,而且在根、茎、叶和穗中均有较高表达(图5-B)。

A:重组自交系的遗传图谱;B:重组自交系中Haba血缘的渗入比例;C:已知籼粳杂种不育位点

A:DPL1和DPL2位点的序列比对;B:SaF和SaM位点的序列比对;C:Sc位点的结构性变异;D:S5位点的序列比对;E:RHS12位点的结构性变异;F:HSA1a和HSA1b位点的序列比对
虽然和序列相似,但其在第一个外显子中仍然存在细微差别,其中,一个G碱基的插入形成了中不存在的CRISPR基因编辑的靶点PAM序列CGG。以此PAM序列设计了一个只敲除拷贝,但是无法敲除拷贝的CRISPR载体,对Haba进行基因编辑。在T2代筛选到2个Cas free的纯合突变CR-1和CR-2,分别缺失了1个C碱基和5个碱基AGGCC,2个突变类型均引起移码突变,缺失了DUF1618功能域(图6-A)。对比CR-1和CR-2与Sasa的杂交F1植株和Sasa与Haba的杂交F1植株发现F1(Sasa/CR-1)和F1(Sasa/CR-2)的花粉可染色比例显著提高(图6-B)。表达量分析发现F1(Sasa/Haba)的表达量显著降低,暗示和会抑制的表达,但是F1(Sasa/CR-1)和F1(Sasa/ CR-2)的表达量显著高于F1(Sasa/Haba)的表达量(图6-C),暗示减少的拷贝数可以减弱其对的抑制。I2-KI镜检发现F1(Sasa/CR-1)和F1(Sasa/CR-2)的花粉育性显著高于F1(Sasa/Haba)(图6-D),并且其结实率也显著高于F1(Sasa/Haba)(图6-E)。
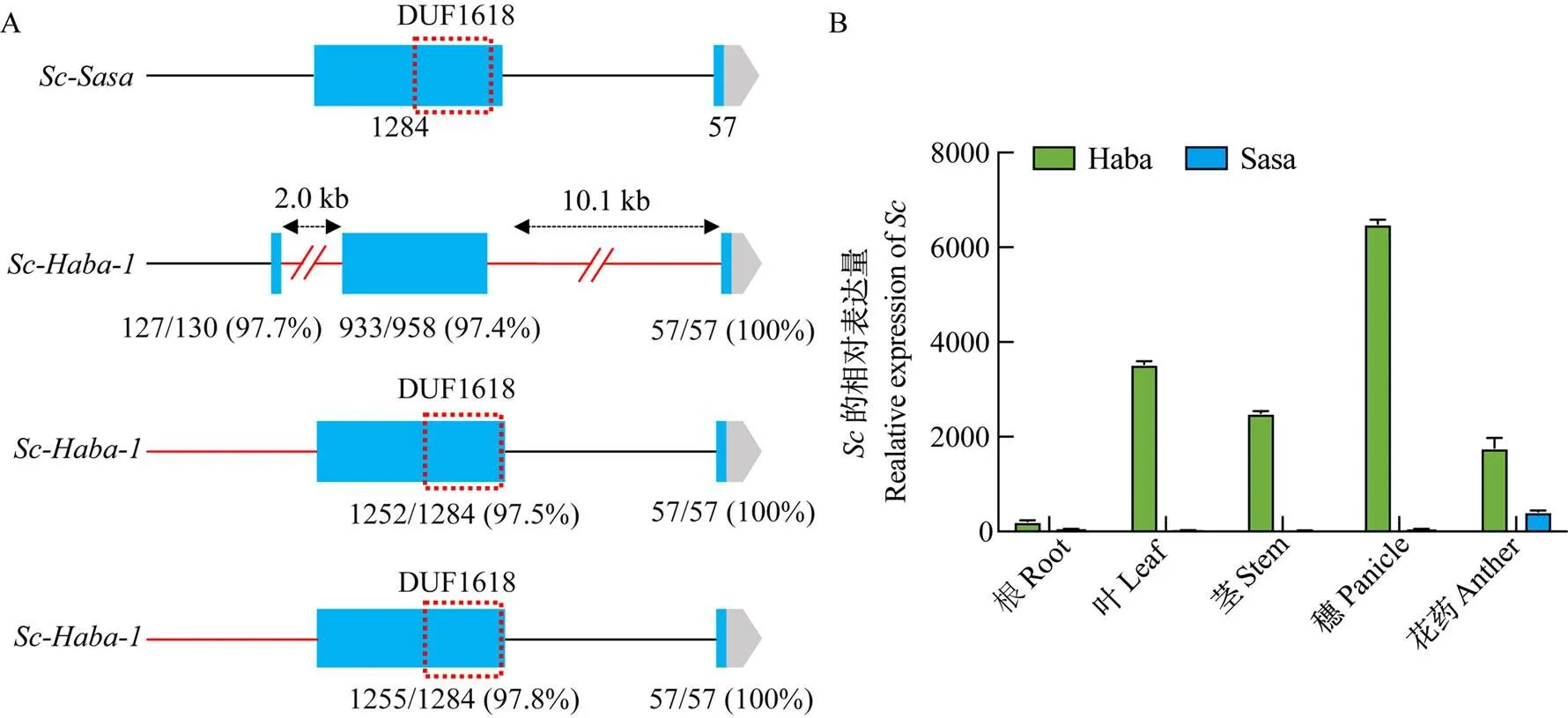
A:Haba中Sc位点的多拷贝;B:Sc在Sasa和Haba中的表达模式
2.6 籼粳杂种不育位点Sd的候选基因预测
进一步分析第1染色体3.1 Mb附近区域内的偏分离信号,该信号峰的区域作为亚种间杂交不育位点在台中65的近等基因系中被报道,然而,目标基因并没被成功地鉴定与克隆[20-21]。为了在群体水平中验证该位点是否具有亚种间生殖隔离的普遍性,本研究剖析了“全球3000份水稻核心种质资源重测序计划”中典型籼粳稻基因组变异数据。应用vcftools软件分析了入选典型籼粳稻群体间在第1染色体上每100 kb的分化系数st,发现亚种间st分化信号峰值出现在3.8 Mb物理位置,并辐射上下游1 Mb基因组区域,与RIL群体偏分离信号重叠(图7)。在群体水平上证实了该位点是亚种间生殖隔离的普遍性关键位点。因此,比对该基因组区域内Sasa和Haba的预测基因的序列差别,发现Sasa和Haba在该区间内存在复杂的基因组结构性变异,Sasa基因组中一段24.7 kb的片段在Haba中被一段64.8 kb的片段取代。Sasa基因组中24.7 kb的片段中包含、、和4个预测基因,而Haba的第1染色体64.8 kb片段中包含10个预测基因,且Sasa中的4个预测基因在Haba中无法比对到相似序列(图7)。综上所述,粳稻Sasa该区域内的4个预测基因和籼稻Haba该区域内的10个预测基因可能在其杂种育性调控上起重要作用。
3 讨论
3.1 籼粳杂种不育的遗传机理
根据配子败育的类型不同,籼粳杂种不育分为雌配子败育和雄配子败育,其中,利用台中65的近等基因系,对籼粳杂交花粉败育进行系统的遗传研究,先后定位了、、、和等5个籼粳杂种花粉不育位点[20-23],其中、和位点已经被成功克隆[13-14, 24]。为一个由2个相邻基因和组成的杂种不育复合基因位点,这两个基因分别编码一个F-盒蛋白和一个小的泛素样修饰物E3连接酶。在杂合子中,来自3个等位基因(籼型和,以及粳稻)的蛋白质之间的有害相互作用导致携带粳稻等位基因的花粉粒选择性致死,从而在分子水平上建立了双基因/三成分相互作用模型[13]。位点籼粳等位基因间的DNA序列存在复杂的结构变异,籼稻等位基因携带2—3个的同源拷贝,因此,籼粳杂种中的高表达抑制了花粉中的表达,进而造成携带的花粉败育[14]。近期研究发现华南农业大学刘自强团队克隆的位点[24]与南京农业大学万建民院士团队克隆的籼粳杂种不育基因[18]及华中农业大学欧阳亦聃教授团队报道的[25]为同一位点,该位点由紧密连锁的2个基因组成,可以分别比喻为“破坏者”和“守卫者”。“破坏者”对所有花粉产生伤害作用,引起花粉的败育;而“守卫者”阻止“破坏者”的伤害作用,因此,那些遗传了该基因的花粉,因受到保护能正常发育。和被定位到第1染色体和第5染色体上,目前还没有成功克隆[20, 22-23]。本研究在第1染色体上鉴定到的偏分离峰与定位区间重合,并且在“全球3000份水稻核心种质资源重测序计划”的群体水平上证实该位点是一个亚种间生殖隔离的普遍性位点。通过序列比对发现粳稻Sasa基因组中一段24.7 kb的片段在Haba中被一段64.8 kb的片段取代,进而提名了候选基因,为该基因的克隆与功能研究提供了关键线索。籼粳分化位点的分析也佐证了该区域为籼粳分化位点,暗示该区域可能调控籼粳杂种不育。/是一对同源基因,均编码植物特异小分子蛋白,对花粉萌发有重要作用。典型籼稻携带有功能的和无功能的,典型粳稻携带有功能的和无功能的,而籼粳杂种中携带2个功能缺失突变等位基因的花粉,不能萌发,这意味着在花粉萌发中扮演着一个重要的角色[17],本研究发现Haba并不含有典型的籼型等位基因。是一个复杂的基因座,调控雌配子败育,包含3个紧密连接的基因、和。在雌性孢子发生过程中,籼稻等位基因的杀手/伴侣基因和的共同作用导致内质网应激和胚胎囊的流产,其中,粳稻等位基因中含有非功能保护基因;籼稻等位基因的功能性拯救了这些效应[15]。基因座包含2个紧密连接的基因,和;粳稻和籼稻杂交种中基因的粳稻和籼稻等位基因之间的上位性相互作用导致雌配子致死[16]。本研究在第5染色体和第12染色体鉴定到的偏分离峰与和位置重合,佐证了和所引起的雌配子败育也是引起Sasa和Haba杂种结实率下降的原因。
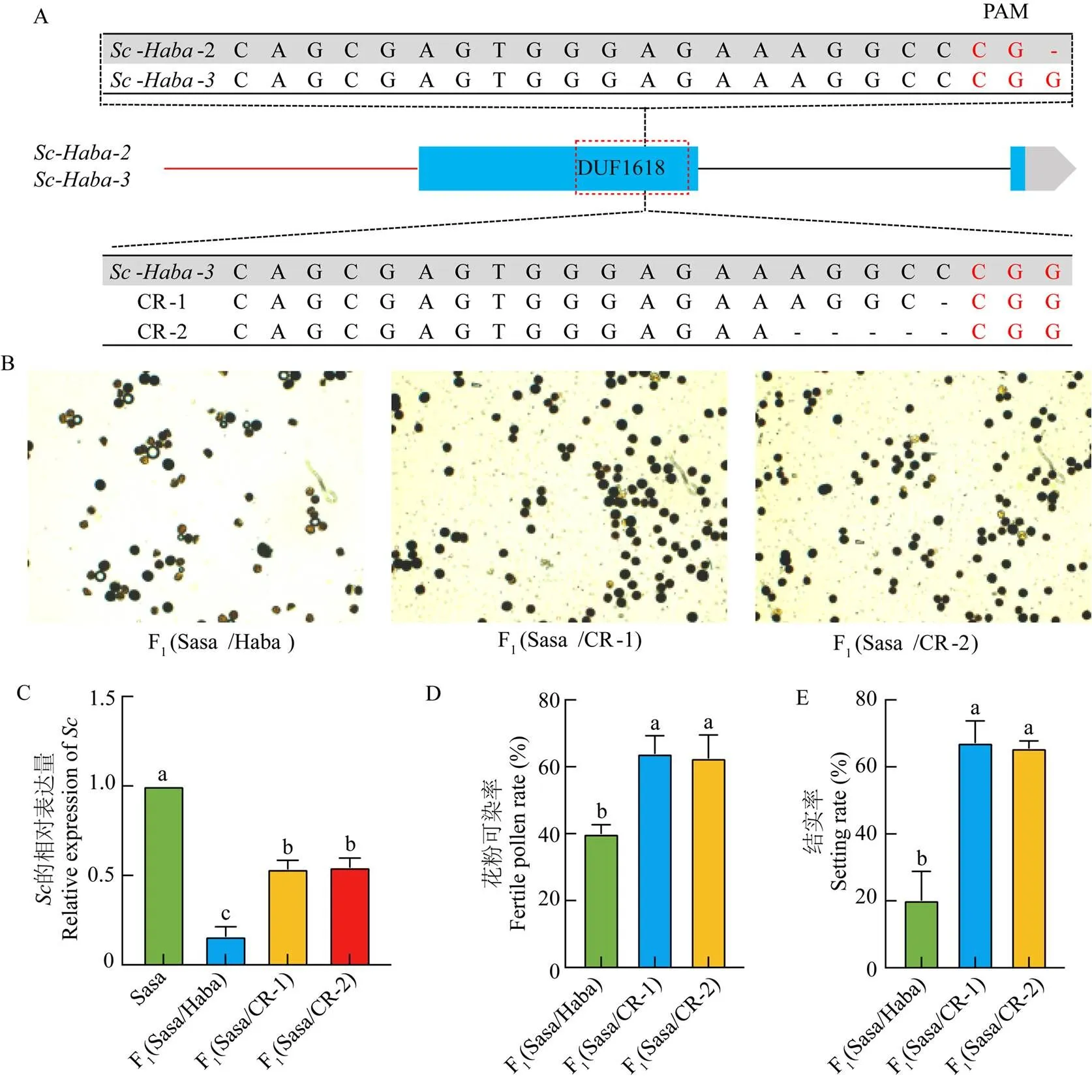
A:Sc-Haba-2与Sc-Haba-3靶点PAM序列的差异和基因编辑植株的突变序列;B:F1(Sasa/Haba)、F1(Sasa/CR-1)和F1(Sasa/CR-2)植株的花粉I2-KI镜检;C:Sasa、F1(Sasa/Haba)、F1(Sasa/CR-1)和F1(Sasa/CR-2)中Sc-Sasa的表达量;D:F1(Sasa/Haba)、F1(Sasa/CR-1)和F1(Sasa/CR-2)的花粉可染率;E:F1(Sasa/Haba)、F1(Sasa/CR-1)和F1(Sasa/CR-2)的结实率

A:Chr.1的100 kb窗口10 kb滑动的群体分化Fst区域,粉色区域表示3.8 Mb物理位置处Fst分化信号峰值所辐射的上下游1 Mb基因组区域;B:第1染色体中Haba基因型的渗入比例;C:候选区间内籼粳间的基因组结构性变异
3.2 结构性变异影响籼粳杂种育性
基因组结构变异,包括片段插入、缺失、反转、易位、重复和拷贝数变异,对各种生物过程、农艺性状和人类疾病具有显著的遗传影响[26]。在三代长读长测序技术推广以前,基因组结构性变异,尤其是基因拷贝数变异,很难通过图位克隆鉴定。拷贝数变异在植物基因组中广泛分布[27]。最近报道的泛基因组揭示了隐藏的拷贝数变异,并证明拷贝数变异调节重要的农艺性状[25, 28-29]。基因座的拷贝数变化有助于水稻的粒型多样性[29],基因座的额外拷贝数显著增加了每穗的粒数[30-31],的双拷贝可能是解释Koshihikari早花表型的原因候选者[26, 32]。基因拷贝数变异也参与水稻籼粳杂种育性的调控,的拷贝数变异在籼粳亚种之间分化,籼稻一般含有2—3个拷贝,而粳稻中只有一个拷贝[14]。利用RGP网站的RiceGAAS系统对座位定位区段进行预测,发现籼稻每个拷贝中还含有预测基因、、和,并且、、均为位点杂种不育功能所必需[33]。本研究还在和拷贝中发现了、、和预测基因,暗示位点可能存在超过10个预测基因参与的复杂遗传机理。//位点也存在籼粳间复杂的结构性变异,序列分析显示,在台中65中,定位区包含2个ORF(命名为和),而在籼稻G4中,除了与或同源的基因(分别命名为和)外,它还包含3个额外的ORF,命名为、和,而这额外的基因对籼粳杂种育性有重要的调控作用[18, 24-25]。本研究定位的位点区间同样在籼粳之间存在复杂的结构性变异,粳稻Sasa该区域含有4个预测基因,而籼稻Haba该区域含有10个预测基因,暗示这些ORF中可能存在调控籼粳杂交育性的关键基因。
3.3 籼粳杂种优势利用
作物杂种优势利用是大幅提高粮食产量的重要途径,一般来说品种间亲缘关系越远,杂交优势越明显。籼稻和粳稻亚种间能育成超级杂交稻可以比现有杂交水稻增产15%以上[34]。然而籼稻和粳稻之间存在严重的生殖隔离,是阻碍杂种优势利用的最大障碍之一。深入研究籼粳杂种不育遗传机理将为籼粳杂种优势的育种应用提供理论依据和种质资源。随着现代分子生物育种技术的发展,越来越多的杂种不育座位被鉴定、克隆,为籼粳稻杂种育性改良提供了潜在的靶点。应用CRISPR基因编辑技术可以在已知的杂交不育基因位点进行点突变或者大片段敲除,可改良籼粳杂种育性,从而加快广亲和系的培育,如,通过敲除座位的拷贝中的1个或2个,减少的基因表达量,从而恢复的正常表达,创制出等位基因[14];应用 CRISPR方法对基因座2个基因和进行编辑,快速创建等位基因也成功改良了籼粳杂种育性[35]。本研究也通过CRISPR基因编辑,应用的特异PAM序列将其敲除,改善了其与Sasa的杂种育性。重要的是该基因可独立行使功能来改善其杂种F1育性,可作为水稻亚种间育性改良的目标基因,具有较大的应用潜力。
4 结论
在第1、3、6、7和12染色体检测到可能存在调控籼粳杂种育性关键基因的偏分离区域。应用CRISPR基因编辑技术敲除Haba第3染色体上位点的多拷贝成功改善其籼粳杂种F1育性。同时发现第1染色体上位点籼粳间复杂结构性变异是调控亚种间生殖隔离的关键,并提名了目标候选基因。
[1] GARRIS A J, TAI T H, COBURN J, KRESOVICH S, McCOUCH S. Genetic structure and diversity inL.. Genetics, 2005, 169(3): 1631-1638.
[2] HUANG X H, WEI X H, SANG T, ZHAO Q, FENG Q, ZHAO Y, LI C Y, ZHU C R, LU T T, ZHANG Z W, LI M, FAN D L, GUO Y L, WANG A H, WANG L, DENG L W, LI W J, LU Y Q, WENG Q J, LIU K Y, HUANG T, ZHOU T Y, JING Y F, LI W, LIN Z, BUCKLER E S, QIAN Q, ZHANG Q F, LI J Y, HAN B. Genome-wide association studies of 14 agronomic traits in rice landraces. Nature Genetics, 2010, 42(11): 961-967.
[3] HUANG X H, ZHAO Y, WEI X H, LI C Y, WANG A H, ZHAO Q, LI W J, GUO Y L, DENG L W, ZHU C R, FAN D L, LU Y Q, WENG Q J, LIU K Y, ZHOU T Y, JING Y F, SI L Z, DONG G J, HUANG T, LU T T, FENG Q, QIAN Q, LI J Y, HAN B. Genome-wide association study of flowering time and grain yield traits in a worldwide collection of rice germplasm. Nature Genetics, 2012, 44(1): 32-39.
[4] YU H, LIN T, MENG X B, DU H L, ZHANG J K, LIU G F, CHEN M J, JING Y H, KOU L Q, LI X X, GAO Q, LIANG Y, LIU X D, FAN Z L, LIANG Y T, CHENG Z K, CHEN M S, TIAN Z X, WANG Y H, CHU C C, ZUO J R, WAN J M, QIAN Q, HAN B, ZUCCOLO A, WING R A, GAO C X, LIANG C Z, LI J Y. A route to de novo domestication of wild allotetraploid rice. Cell, 2021, 184(5): 1156-1170.
[5] WANG W S, MAULEON R, HU Z Q, CHEBOTAROV D, TAI S S, WU Z C, LI M, ZHENG T Q, FUENTES R R, ZHANG F, MANSUETO L, COPETTI D, SANCIANGCO M, PALIS K C, XU J L, SUN C, FU B Y, ZHANG H L, GAO Y M, ZHAO X Q, SHEN F, CUI X, YU H, LI Z C, CHEN M L, DETRAS J, ZHOU Y L, ZHANG X Y, ZHAO Y, KUDRNA D, WANG C C, LI R, JIA B, LU J Y, HE X C, DONG Z T, XU J B, LI Y H, WANG M, SHI J X, LI J, ZHANG D B, LEE S, HU W S, POLIAKOV A, DUBCHAK I, ULAT V J, BORJA F N, MENDOZA J R, ALI J, LI J, GAO Q, NIU Y C, YUE Z, NAREDO M E B, TALAG J, WANG X Q, LI J J, FANG X D, YIN Y, GLASZMANN J C, ZHANG J W, LI J Y, HAMILTON R S, WING R A, RUAN J, ZHANG G Y, WEI C C, ALEXANDROV N, MCNALLY K L, LI Z K, LEUNG H. Genomic variation in 3,010 diverse accessions of Asian cultivated rice. Nature, 2018, 557(7703): 43-49.
[6] WEI X, QIU J, YONG K C, FAN J J, ZHANG Q, HUA H, LIU J, WANG Q, OLSEN K M, HAN B, HUANG X H. A quantitative genomics map of rice provides genetic insights and guides breeding. Nature Genetics, 2021, 53(2): 243-253.
[7] LIU Y Q, WANG H R, JIANG Z M, WANG W, XU R N, WANG Q H, ZHANG Z H, LI A F, LIANG Y, OU S J, LIU X J, CAO S Y, TONG H N, WANG Y H, ZHOU F, LIAO H, HU B, CHU C C. Genomic basis of geographical adaptation to soil nitrogen in rice. Nature, 2021, 590(7847): 600-605.
[8] ZHOU G, CHEN Y, YAO W, ZHANG C J, XIE W B, HUA J P, XING Y Z, XIAO J H, ZHANG Q F. Genetic composition of yield heterosis in an elite rice hybrid. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39): 15847-15852.
[9] 袁隆平. 杂交水稻的育种战略设想. 杂交水稻, 1987(1): 1-3.
YUAN L P. The strategy of hybrid rice breeding. Hybrid Rice,1987(1): 1-3. (in Chinese)
[10] CHENG S H, ZHUANG J Y, FAN Y Y, DU J H, CAO L Y. Progress in research and development on hybrid rice: a super-domesticate in China. Annals of Botany, 2007, 100(5): 959-966.
[11] OKA H. Analysis of genes controlling f(1) sterility in rice by the use of isogenic lines. Genetics, 1974, 77(3): 521-534.
[12] SANO Y. The genic nature of gamete eliminator in rice. Genetics, 1990, 125(1): 183-191.
[13] LONG Y M, ZHAO L F, NIU B X, SU J, WU H, CHEN Y L, ZHANG Q Y, GUO J X, ZHUANG C X, MEI M T, XIA J X, WANG L, WU H B, LIU Y G. Hybrid male sterility in rice controlled by interaction between divergent alleles of two adjacent genes. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(48): 18871-18876.
[14] SHEN R X, WANG L, LIU X P, WU J, JIN W W, ZHAO X C, XIE X R, ZHU Q L, TANG H W, LI Q, CHEN L T, LIU Y G. Genomic structural variation-mediated allelic suppression causes hybrid male sterility in rice. Nature Communications, 2017, 8: 1310.
[15] YANG J Y, ZHAO X B, CHENG K, DU H Y, OUYANG Y D, CHEN J J, QIU S Q, HUANG J Y, JIANG Y H, JIANG L W, DING J H, WANG J, XU C G, LI X H, ZHANG Q F. A killer-protector system regulates both hybrid sterility and segregation distortion in rice. Science, 2012, 337(6100): 1336-1340.
[16] KUBO T, TAKASHI T, ASHIKARI M, YOSHIMURA A, KURATA N. Two tightly linked genes at thelocus cause both F1and F2hybrid sterility in rice. Molecular Plant, 2016, 9(2): 221-232.
[17] MIZUTA Y, HARUSHIMA Y, KURATA N. Rice pollen hybrid incompatibility caused by reciprocal gene loss of duplicated genes. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(47): 20417-20422.
[18] WANG C L, WANG J, LU J Y, XIONG Y H, ZHAO Z G, YU X W, ZHENG X M, LI J, LIN Q B, REN Y L, HU Y, HE X D, LI C, ZENG Y L, MIAO R, GUO M L, ZHANG B S, ZHU Y, ZHANG Y H, TANG W J, WANG Y L, HAO B Y, WANG Q M, CHENG S Q, HE X J, YAO B W, GAO J W, ZHU X F, YU H, WANG Y, SUN Y, ZHOU C L, DONG H, MA X D, GUO X P, LIU X, TIAN Y L, LIU S J, WANG C M, CHENG Z J, JIANG L, ZHOU J W, GUO H S, JIANG L W, TAO D Y, CHAI J J, ZHANG W, WANG H Y, WU C Y, WAN J M. A natural gene drive system confers reproductive isolation in rice. Cell, 2023, 186(17): 3577-3592.
[19] LI X K, WU L, WANG J H, SUN J, XIA X H, GENG X, WANG X H, XU Z J, XU Q. Genome sequencing of rice subspecies and genetic analysis of recombinant lines reveals regional yield- and quality- associated loci. BMC biology, 2018, 16(1): 102.
[20] LI W T, ZENG R Z, ZHANG Z M, DING X H, ZHANG G Q. Identification and fine mapping of, a new locus conferring the partial pollen sterility of intersubspecific F1hybrids in rice (L.). Theoretical and Applied Genetics, 2008, 116(7): 915-922.
[21] 张桂权, 卢永根, 张华, 杨进昌, 刘桂富. 栽培稻()杂种不育性的遗传研究: Ⅳ.F1花粉不育性的基因型. 遗传学报, 1994, 21(1): 34-41.
ZHANG G Q, LU Y G, ZHANG H, YANG J C, LIU G F. Genetic studies on the hybrid sterility in cultivated rice (): IV. Genotypes for F1pollen sterility. Acta Genetica Sinica, 1994, 21(1): 34-41. (in Chinese)
[22] LI W T, ZENG R Z, ZHANG Z M, DING X H, ZHANG G Q. Fine mapping of locusfor F1pollen sterility in rice (L.). Chinese Science Bulletin, 2006, 51(6): 675-680.
[23] ZHANG G Q. Prospects of utilization of inter-subspecific heterosis betweenandrice. Journal of Integrative Agriculture, 2020, 19(1): 1-10.
[24] WANG D Q, WANG H R, XU X M, WANG M, WANG Y H, CHEN H, PING F, ZHONG H H, MU Z K, XIE W T, LI X Y, FENG J B, ZHANG M L, FAN Z L, YANG T F, ZHAO J L, LIU B, RUAN Y, ZHANG G Q, LIU C L, LIU Z Q. Two complementary genes in a presence-absence variation contribute toreproductive isolation in rice. Nature Communications, 2023, 14(1): 4531.
[25] ZHOU P H, WANG Z J, ZHU X C, TANG Y, YE L, YU H H, LI Y T, ZHANG N K, LIU T, WANG T, WU Y Y, CAO D Y, CHEN Y, LI X, ZHANG Q L, XIAO J H, YU S B, ZHANG Q F, MI J M, OUYANG Y D. A minimal genome design to maximally guarantee fertile inter-subspecific hybrid rice. Molecular Plant, 2023, 16(4): 726-738.
[26] QIN P, LU H W, DU H L, WANG H, CHEN W L, CHEN Z, HE Q, OU S J, ZHANG H Y, LI X Z, LI X X, LI Y, LIAO Y, GAO Q, TU B, YUAN H, MA B T, WANG Y P, QIAN Y W, FAN S J, LI W T, WANG J, HE M, YIN J J, LI T, JIANG N, CHEN X W, LIANG C Z, LI S G. Pan-genome analysis of 33 genetically diverse rice accessions reveals hidden genomic variations. Cell, 2021, 184(13): 3542-3558.
[27] LYE Z N, PURUGGANAN M D. Copy number variation in domestication. Trends in Plant Science, 2019, 24(4): 352-365.
[28] SHANG L G, LI X X, HE H Y, YUAN Q L, SONG Y N, WEI Z R, LIN H, HU M, ZHAO F L, ZHANG C, LI Y H, GAO H S, WANG T Y, LIU X P, ZHANG H, ZHANG Y, CAO S M, YU X M, ZHANG B T, ZHANG Y, TAN Y Q, QIN M, AI C, YANG Y X, ZHANG B, HU Z Q, WANG H R, LV Y, WANG Y X, MA J, WANG Q, LU H W, WU Z, LIU S L, SUN Z Y, ZHANG H L, GUO L B, LI Z C, ZHOU Y F, LI J Y, ZHU Z F, XIONG G S, RUAN J, QIAN Q. A super pan-genomic landscape of rice. Cell Research, 2022, 32(10): 878-896.
[29] WANG Y, LI F C, ZHANG F, WU L A, XU N, SUN Q, CHEN H, YU Z W, LU J H, JIANG K, WANG X C, WEN S Y, ZHOU Y, ZHAO H, JIANG Q, WANG J H, JIA R Z, SUN J, TANG L, XU H, HU W, XU Z J, CHEN W F, GUO A P, XU Q. Time-ordering/genomes analysis indicates the importance of large structural variants in rice breeding. Plant Biotechnology Journal, 2023, 21(1): 202-218.
[30] WANG Y X, XIONG G S, HU J, JIANG L, YU H, XU J, FANG Y X, ZENG L J, XU E B, XU J, YE W J, MENG X B, LIU R F, CHEN H Q, JING Y H, WANG Y H, ZHU X D, LI J Y, QIAN Q. Copy number variation at thelocus contributes to grain size diversity in rice. Nature Genetics, 2015, 47(8): 944-948.
[31] WU Y, WANG Y, MI X F, SHAN J X, LI X M, XU J L, LIN H X. The QTLencodes, which increases grain number and yield by increasing cytokinin activity in rice panicle meristems. PLoS Genetics, 2016, 12(10): e1006386.
[32] FORNARA F, PARENICOVÁ L, FALASCA G, PELUCCHI N, MASIERO S, CIANNAMEA S, LOPEZ-DEE Z, ALTAMURA M M, COLOMBO L, KATER M M. Functional characterization of, a member of thesubfamily ofbox genes. Plant Physiology, 2004, 135(4): 2207-2219.
[33] 吴疆. 水稻座位蛋白复合体参与籼粳杂种雄性不育的研究[D]. 广州: 华南农业大学, 2017.
WU J. A protein complex of thelocus controlshybrid male sterility in rice[D]. Guangzhou: South China Agricultural University, 2017.(in chinese)
[34] 袁隆平. 超级杂交水稻育种研究新进展. 中国农村科技, 2010, Z1: 24-25.
YUAN L P. New progress in super hybrid breeding.China Rural Science & Technology, 2010, Z1: 24-25. (in Chinese)
[35] XIE Y Y, NIU B X, LONG Y M, LI G S, TANG J T, ZHANG Y L, REN D, LIU Y G, CHEN L T. Suppression or knockout ofovercomes the-mediated hybrid male sterility in rice. Journal of Integrative Plant Biology, 2017, 59(9): 669-679.
Genetic analysis and candidate gene identification on fertility and inheritance of hybrid sterility ofandcross
XU Na, TANG Ying, XU ZhengJin, SUN Jian, XU Quan
Rice Research Institute, Shenyang Agricultural University, Shenyang 110866
【Objective】The F1hybrid sterility between/and/severely hinders the utilization of hybrid advantage between subspecies. Exploring the genetic mechanism and identifying new regulatory genes for/hybrid sterility will provide theoretical basis for promoting genetic improvement of/hybrid seed setting rate.【Method】A series of stable genetic recombination inbred lines (RILs) containing 95 plant lines were derived from the cross betweenvariety Habataki andvariety Sasanishiki after 10 generations inbred using single seed descent method. High throughput sequencing was performed on both parents and RILs on the Illumina platform, and the distribution of Habataki pedigree in RILs was analyzed at the whole genome level. The segregation distortion regions were identified, and hybrid sterile related gene loci were screened within the segregation distortion regions, then identified candidate genes through sequence alignment comparison. The targeted gene was knockout to verify the function using CRISPR gene editing technology.【Result】The hybrid F1plants derived from the cross between Habataki and Sasanishiki showed significant heterosis in panicles, grains per panicle, and thousand grain weight, but its seed setting rate significantly decreased. I2-KI microscopy revealed a significant decrease in F1pollen fertility. High throughput sequencing of the entire genome of RILs revealed significant segregation distortion on chr.1, chr.3, chr.5, chr.6, chr.7, and chr.12, indicating that the genotype in this region tends towards the Habataki. Sequence alignment comparison revealed that,, andare target genes for the segregation distortion on chr.3, chr.6, and chr.12. The CRISPR gene editing mutants with a knock-outallele in Habataki successfully improved the pollen fertility and seed setting rate of F1hybrid with Sasanishiki. A complex structural variation was found between Sasanishiki and Habataki in the segregation distortion of chr.1. A 24.7 kb segment containing 4 predicted genes in the Sasanishiki genome was replaced by a 64.8 kb segment containing 10 predicted genes in Habataki, the structural variation may involve in controlling the hybrid sterility ofandcross. 【Conclusion】This study detected multiplehybrid infertility related loci, and successfully improved F1fertility by using CRISPR gene editing to knock out multiple copies ofin Habataki, locking in the target gene in theregion of chr.1.
rice; hybrid sterility; high throughput sequencing; gene editing;candidate genes

10.3864/j.issn.0578-1752.2024.08.001
2023-10-23;
2023-11-29
兴辽英才计划(XLYC2203171)
许娜,E-mail:xuna1109@163.com。通信作者徐铨,E-mail:kobexu34@syau.edu.cn。通信作者孙健,E-mail:sunjian811119@syau.edu.cn
(责任编辑 李莉)
