小麦非麸质致敏原α-淀粉酶抑制剂的表位定位及消减技术
2023-12-14张巧智王彦波傅玲琳
王 垚,张巧智,王彦波,傅玲琳
(浙江工商大学食品与生物工程学院,浙江食品质量安全工程研究院,浙江 杭州 310018)
当前,全球范围内食物过敏的发病率逐年上升,食物过敏已经成为严重的食品安全和公共卫生问题。在易导致过敏的食物中,小麦、牛奶、芝麻、鸡蛋、坚果、花生、鱼和贝类是最常见的8 大类过敏原,能够引起90%以上的食物过敏反应[1-2]。在欧美、澳大利亚等西方国家,食物过敏在儿童中的患病率约为8%,在成人中的患病率约为2%[3]。在我国,9 岁以下的儿童主要对牛奶、鸡蛋和水果蔬菜等过敏,10 岁以上的青年及成人主要对小麦过敏。在各类诱发过敏的原因中,食物过敏占了77%,其中占比最高的是小麦,占到了37%[4]。
小麦是世界卫生组织报道的八大常见过敏食物之一,其引发的过敏主要是由IgE介导的过敏反应,属I型超敏反应[5-9]。目前世界卫生组织过敏原命名小组委员会已公布的小麦致敏原高达28 种,分别命名为Tri a 12~Tri a 21、Tri a 25~Tri a 40。根据在不同溶剂(如水、盐、稀酸/碱和乙醇)中的溶解度不同,可将小麦蛋白分为球蛋白、清蛋白、麦谷蛋白和醇溶蛋白组分,其中小麦麸质蛋白由醇溶蛋白和麦谷蛋白组成[10]。
目前,针对小麦致敏原抗原表位的研究主要集中在麸质致敏原上,如Battais等[11]鉴定了γ-醇溶蛋白的抗原表位;Tanabe[12]鉴定了醇溶蛋白的表位PQQPF与QQPFP;Matsuo等[13]揭示了高分子质量麦谷蛋白的表位QQPGQ与QQSGQGQ等。除麸质致敏原外,小麦蛋白中还含有多种非麸质致敏原,主要包括非特异性脂转移蛋白、β-淀粉酶、3-磷酸甘油醛脱氢酶、硫醇还原酶同源物、四聚体α-淀粉酶抑制剂 CM3、二聚体α-淀粉酶抑制0.19、硫氧还原蛋白等[14]。研究发现,小麦非麸质蛋白是过敏性皮炎及面包师哮喘的主要致敏原,与70%~80%患者血清中的IgE有阳性反应[15-16]。此外,Battais等[17]也发现小麦非麸质蛋白是引起法国地区儿童过敏性皮炎的主要原因。类似地,Pastorello等[18]发现α-淀粉酶抑制剂CM3是引发日本地区人群小麦过敏性皮炎的主要蛋白。
在食物致敏反应进程中,致敏原的抗原特异性基团(即抗原表位,也称抗原决定簇)通过与特异性抗体IgE识别结合,激活下游效应细胞(如肥大细胞等)从而引发过敏反应[19],因此,致敏原的抗原表位在诱发食物过敏反应中具有十分重要的作用。表位是致敏反应的物质基础,被定义为氨基酸的短连续序列[20],根据结构的不同,可分为线性表位和构象表位。线性表位指由连续性排列的氨基酸残基所组成的短肽,并且可以和特定的抗体发生反应,构象表位是由正确折叠的蛋白空间分布上连续或相邻的氨基酸组成,分布于肽链不同部位或者不同肽链上[21-23]。根据抗体与抗原受体结合的不同可将表位分成B细胞表位和T细胞表位[24]。
表位定位是致敏原研究的基础。目前,致敏原表位定位的主要方法有蛋白质裂解、重叠肽技术、肽指纹图谱连用法、晶体复合物结构分析以及生物信息学分析等。其中,重叠肽技术[25]是将致敏原的一级结构分成多个连续重叠的肽段进行合成,并使用特定抗体进一步筛选出能够产生阳性反应的肽段,但是只适用于线性表位的定位。蛋白质裂解与肽指纹图谱联用法是对经酶或其他试剂切割后的肽段进一步用抗体去分析定位,这种方法对线性表位区域的定位不全面,花费较大且耗时较长。晶体复合物结构分析是通过解析致敏原与其抗体产生晶体复合物结构[26],是较为理想的直观确定蛋白构象表位的方法,但是该方法仅适用于单克隆抗体。近年来随着科技的进步,生物信息学技术广泛应用于抗原表位的定位,它主要根据目前已知抗原表位区域的序列以及其结构特点,分析形成抗原表位区域的特征和规律,从而推测出目标致敏原中可能形成抗原表位的部位,操作快速且验证方便,可在短时间内对多个致敏原的抗原表位进行预测鉴定。
在致敏原表位定位的基础上,有必要对其消减方法进行研究。采用消减技术有效降低小麦蛋白的致敏性有利于提高小麦过敏人群的生活质量,目前主要的消减技术有加热、微波、高压、超声等,但它们对小麦制品的营养物质破坏较大[27]。多酚是一类植物源天然成分,本身具有多种健康促进作用[28],存在于茶、蔬菜、豆类等食物中;研究发现多酚具有降敏潜力[29],酚类物质通过与致敏原非共价或共价相互作用[30-34],降低了抗原蛋白的IgE结合能力并减轻了肥大细胞脱颗粒程度,是一种有前景的致敏原消减手段,然而目前针对多酚与小麦致敏原互作构建低敏蛋白体系的研究报道较少。
鉴于此,本研究以小麦非麸质致敏原α-淀粉酶抑制剂为研究对象,通过生物信息学工具对其抗原表位进行预测,筛选出潜在致敏原表位,并对表位肽段进行合成,经间接竞争酶联免疫吸附实验(indirect competition enzyme-linked immunosorbent assay,ic-ELISA)对候选表位进行验证,从而得到α-淀粉酶抑制剂的表位,进一步通过茶多酚与α-淀粉酶抑制剂互作构建复合物对致敏原进行消减,探究其消减效果及消减机制,以期对小麦非麸质致敏原表位的定位及非麸质致敏原的消减方法提供参考依据。
1 材料与方法
1.1 材料与试剂
α-淀粉酶抑制剂 美国Sigma公司;小麦过敏血清重庆沃卡威生物技术有限公司;表没子儿茶素没食子酸酯(epigallocatechin gallate,EGCG,纯度≥98%)、表没食子酸儿茶素(epigallocatechin,EGC,纯度≥98%)上海阿拉丁生化科技股份有限公司;HRP标记的山羊抗人IgE 美国Abcam公司;3,3’,5,5’-四甲基联苯胺(3,3’,5,5’-tetramethylbenzidine,TMB)单组分底物溶液北京索莱宝科技有限公司;其他试剂均为分析纯。
1.2 仪器与设备
Milli-Q超纯水装置 美国Millipore公司;MOS-450圆二色谱仪 美国BioLogic公司;LRH-250A培养箱上海科学仪器有限公司;Spectra Max i3x酶标仪 美国Molecular Devices公司;LGJ-12真空冷冻干燥机 北京松原华兴科技有限公司;Vortex Mixer V8台式恒温振荡器 太仓实验有限公司;Legend Micro 21 R离心机美国Thermo Fisher Scientific公司;RF-5301PC荧光分光光度计、UV2600紫外-可见分光光度计 日本岛津公司。
1.3 方法
1.3.1 抗体血清池
收集5 名小麦过敏患者阳性血清,他们在摄入小麦后均有速发型超敏反应史,具体信息见表1。通过ic-ELISA证实过敏患者血清与α-淀粉酶抑制剂的强结合能力。将5 名小麦过敏患者阳性血清进行等体积混合后作为血清池,然后贮存在-80 ℃,待用。

表1 小麦过敏病人阳性血清信息Table 1 Information of sera from patients with wheat allergy included in this study
1.3.2 致敏原氨基酸序列
致敏原α-淀粉酶抑制剂的登录号从过敏原在线(http://www.allergenonline.org/)获得,为CAI84642。α-淀粉酶抑制剂的氨基酸序列从美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)获得。α-淀粉酶抑制剂含有119 个氨基酸。
1.3.3 线性表位预测
1.3.3.1 DNAStar Protean软件预测抗原表位
使用DNAStar中的Protean程序对α-淀粉酶抑制剂的抗原表位进行预测,其中亲水性分析选择Hoop-Woods、Kyte-Doolittle算法,溶剂可及性分析选择Emini-Surface Probability算法,抗原指数分析选择Jameson-Wolf算法,可塑性分析选择Karplus-Schultz算法[35-36]。
1.3.3.2 IMED、IEDB线上预测抗原表位
利用IMED(http://imed.med.ucm.es/Tools/antigenic.pl)、IEDB(http://www.iedb.org/)对α-淀粉酶抑制剂的抗原表位进行在线预测[37]。
1.3.4 多肽合成
委托南京金斯瑞生物有限公司采用标准Fmoc固相合成法合成了与预测的潜在表位相对应的多肽。经高效液相色谱鉴定,其纯度均为95%以上。同时用电喷雾电离-质谱仪对合成的多肽进行了分子质量鉴定。
1.3.5 ic-ELISA
1.3.5.1 抗原抗体最佳反应质量浓度测定
参照Fu Linglin等[38]的方法,并略有改动。通过棋盘法对抗原抗体最佳反应质量浓度进行测定,具体步骤为将α-淀粉酶抑制剂用包被缓冲液稀释至不同质量浓度1、5、10 μg/mL,取100 μL稀释后的样品加入96 孔酶标板中,每个稀释质量浓度3 个平行,同时设置等体积等质量浓度牛血清白蛋白作为阴性对照。将96 孔酶标板于4 ℃放置过夜。之后倒去酶标板中的包被缓冲液,在滤纸上将剩余液体拍干,每孔加入200 μL洗涤液,轻轻摇晃洗涤2 min,重复3 次。洗涤完成后,在酶标板中每孔加入100 μL封闭液,37 ℃条件下封闭2 h。封闭完成后,完成3 次洗涤步骤。在每个实验样品的相同稀释倍数下每孔依次加入100 μL不同稀释倍数的一抗,37 ℃条件下孵育1.5 h。一抗孵育完成后,洗涤3 次,在酶标板中每孔加入100 μL稀释5 000 倍的二抗,37 ℃条件下孵育1.5 h。二抗孵育完成后,洗涤3 次。在酶标板中每孔加入100 μL TMB底物显色,包上锡箔纸避光,37 ℃孵育30 min。酶标板显色后,每孔用移液枪快速加入50 μL终止液,停止显色反应。最后将酶标板置于酶标仪中,并测定其在450 nm波长下每孔的吸光度。
1.3.5.2 候选表位的验证
用磷酸盐缓冲液将合成的抗原表位多肽分别稀释至0.01、0.1、1、10、100、1 000 μg/mL,对表位多肽进行抑制率的测定,其中一抗是由5 位小麦过敏患者血清组成的血清池,操作方法同1.3.5.1节,在抗原抗体反应之前,先将各抗原表位与血清池(最佳质量浓度两倍)等体积混合并在37 ℃的培养箱中共孵育1 h,然后取100 μL加入到96 孔酶标板中同包被原反应1.5 h。设置对照组:α-淀粉酶抑制剂与血清池反应液。以对照组的OD450nm作为A0,加入多肽抑制时的OD450nm作为A,则抗体-抗原的结合率为A/A0,多肽对抗体-抗原结合的竞争抑制率为1-A/A0。
1.3.6α-淀粉酶抑制剂-茶多酚复合物制备
采用非共价互作的方法,将α-淀粉酶抑制剂稀释成1 μmol/L并与不同浓度的EGCG、EGC(0、5、10、20、25、30 μmol/L)等体积混合。将蛋白质-多酚溶液的混合物分别在37 ℃孵育过夜[39]。
1.3.7α-淀粉酶抑制剂-茶多酚复合物IgE结合能力的测定
利用ic-ELISA确定EGCG、EGC的添加对α-淀粉酶抑制剂致敏性的影响。方法同1.3.5.1节,需要注意的是在加入一抗时,血清池与复合物提前在37 ℃下孵育1 h。
1.3.8α-淀粉酶抑制剂-茶多酚复合物的内源荧光光谱表征
为评估与EGCG、EGC结合后对α-淀粉酶抑制剂结构的影响,测定了α-淀粉酶抑制剂-茶多酚互作物的内源荧光光谱。α-淀粉酶抑制剂终质量浓度为0.2 mg/mL,激发与发射狭缝的宽度均设置为2 nm,电压为400 V,使用280 nm的激发波长,在305 nm和450 nm波长范围扫描样品。测量3 次取平均值。
1.3.9α-淀粉酶抑制剂-茶多酚复合物的圆二色谱表征
圆二色谱法是基于左、右圆偏振光吸收不同而产生的椭圆偏振光。远紫外区的圆二色谱可用于分析与茶多酚非共价结合后α-淀粉酶抑制剂的二级结构、折叠和相互作用改变。圆二色谱中蛋白质α-螺旋的特征峰分别在208 nm和222 nm波长处有跃迁[40]。非共价复合物按上述方法制备(1.3.6节)。复合物中α-淀粉酶抑制剂的终浓度0.2 mg/mL,α-淀粉酶抑制剂与EGCG(EGC)的物质的量比分别为1∶0、1∶10、1∶20和1∶30。使用移液器吸取200 μL的复合物样品,将其置于0.1 cm光学长度的石英比色皿中,在190~260 nm范围内进行扫描。扫描速率为100 nm/min,带宽设置为1.0 nm。不同二级结构相对含量计算方法如Lang Yuxi等[41]所述,采用CD Pro软件(Bio-Logic Science Instruments)和SELCON3算法进行。
1.3.10α-淀粉酶抑制剂-茶多酚复合物的紫外光谱表征
紫外吸收光谱是一种用于测定蛋白分子氨基酸侧链基团发色特性的光谱技术[42],可用于蛋白质的结构表征,以及蛋白质大分子与小分子的相互作用解析等[43]。将α-淀粉酶抑制剂与茶多酚复合物样品稀释至适当质量浓度,使α-淀粉酶抑制剂的终质量浓度为0.2 mg/mL,置于紫外分光光度计中在190~350 nm波长范围内的紫外吸收区进行扫描,得到紫外吸收光谱,其中磷酸盐缓冲液作为背景溶剂当作基线进行扣除。
1.3.11α-淀粉酶抑制剂与EGCG、EGC的分子对接
为了阐明EGCG、EGC与α-淀粉酶抑制剂抗原表位及其附近残基之间的相互作用和结合能,进一步说明EGCG、EGC对α-淀粉酶抑制剂致敏性的影响,采用AutodockVina 1.2.2,一种计算机蛋白-配体对接软件[44]模拟α-淀粉酶抑制剂抗原表位与小分子间的相互作用。α-淀粉酶抑制剂的结构由从RCSB PDB蛋白质数据库(http://www.rcsb.org;PDB号:1HSS)下载,从PubChem化合物数据库(https://pubchem.ncbi.nlm.nih.gov/)中获得了EGCG、EGC的分子结构[45]。首先对蛋白和配体文件进行了准备,将所有蛋白质和分子文件都转换为PDBQT格式。PyMOL去除了受体结构中所有水分子,并添加了极性氢原子[46]。网格框居中以覆盖每个蛋白质的结构域并适应自由分子运动。热门、穷举次数设置为8。使用Discovery studio 2016(Accelrys Software,Inc San Diego,CA,USA)软件对数据进行分析。
1.4 数据分析
每组实验3 次平行,所有数据使用SPSS 16.0进行分析,并以表示。采用单因素方差分析和Duncan多极差检验进行统计学处理,P<0.05,差异显著。
2 结果与分析
2.1 致敏原线性表位的预测和选择
2.1.1 DNAStar Protean软件预测抗原表位
DNAStar Protean软件对抗原表位的预测主要集中在致敏蛋白氨基酸残基的理化性质。亲水性:通常位于蛋白表面的是亲水性氨基酸,而疏水性氨基酸被包埋于蛋白内部,所以抗体结合的位点一般都是亲水性氨基酸残基。溶剂可及性:表征抗原氨基酸接触溶剂分子的可能性。柔韧性:柔韧性较大的氨基酸残基更易形成抗原表位。二级结构:蛋白质的二级结构主要包括α-螺旋、β-折叠、无规卷曲和转角等,转角和无规卷曲一般分布在蛋白质的表面,与抗体更易结合,容易成为抗原表位。使用DNAStar Protean软件对α-淀粉酶抑制剂的二级结构、亲水性、抗原指数和溶剂可及性进行分析。如图1所示,α-淀粉酶抑制剂抗原表位预测存在的范围为:AA22~28、56~82、102~107、114~119。

图1 DNAStar对α-淀粉酶抑制剂抗原预测结果Fig.1 Prediction of the hydrophilicity,flexible regions,antigenic index,and surface probability of α-amylase inhibitor by DNAStar
2.1.2 IMED、IEDB线上预测抗原表位
利用IMED对α-淀粉酶抑制剂的线性表位进行了预测,结果如图2A所示。IMED预测可能的线性表位范围为:AA4~25、27~54、74~101、105~115。同时利用IEDB对致敏原的线性表位进行了预测,结果如图2B所示。IEDB主要根据氨基酸序列的溶剂可及性、抗原指数以及亲水性对抗原表位进行预测,最后选择得分>8对预测结果进行优化。α-淀粉酶抑制剂抗原表位预测存在的范围为:AA5~12、26~31、57~81、96~108。
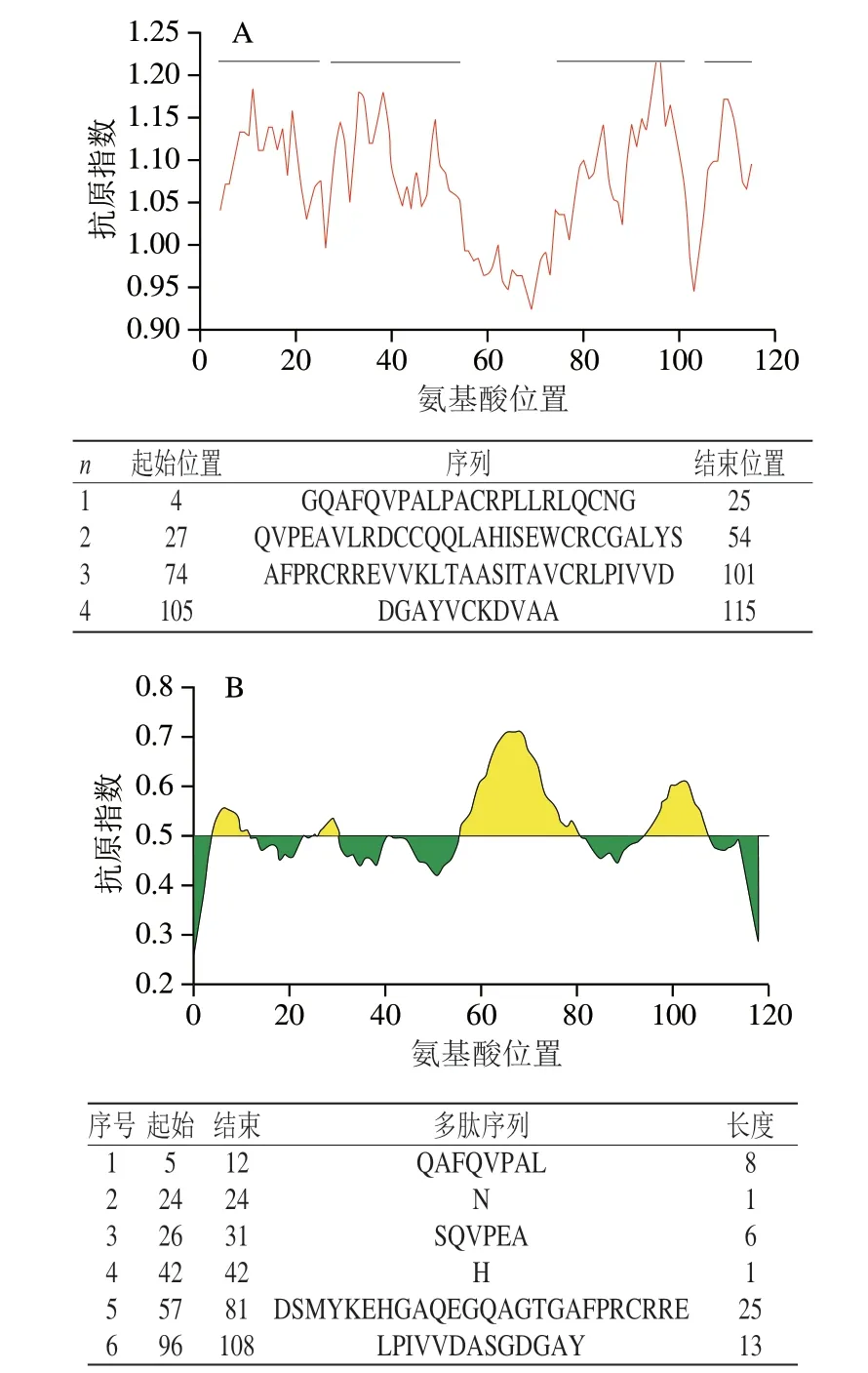
图2 IMED(A)及IEDB(B)对α-淀粉酶抑制剂抗原表位的预测Fig.2 Epitope prediction of α-amylase inhibitor by using IMED (A) and IEDB (B) databases
2.1.3α-淀粉酶抑制剂抗原表位综合预测
综合DNAstar Protean、IMED、IEDB 3 种方法对α-淀粉酶抑制剂氨基酸序列的预测结果,筛选柔韧性大、亲水性高、溶剂可及性强、可塑性好的肽段作为候选表位[37]。表2展示了不同方法预测可能的抗原表位结果。

表2 用3 种免疫信息学工具预测α-淀粉酶抑制剂的线性表位位置Table 2 Epitope sites in α-amylase inhibitor predicted by three immunoinformatic tools
根据表2中不同方法预测的重复区域,选择至少两种方法预测重复的肽段范围作为α-淀粉酶抑制剂最终候选的线性表位,结果如表3所示。最后选取6 条肽段进行Fmoc固相合成。
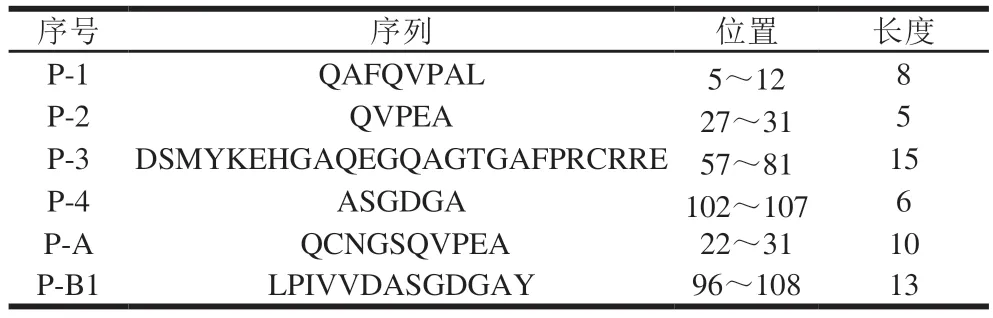
表3 生物信息学方法最终预测的α-淀粉酶抑制剂位点Table 3 Definitive α-amylase inhibitor sites predicted by bioinformatic methods
2.2 抗原抗体最佳质量浓度的确定
利用小麦过敏病人的混合血清池对α-淀粉酶抑制剂的致敏性进行鉴定。如图3A所示,患者血清中α-淀粉酶抑制剂的特异性IgE丰度较高,可以用于后续的实验。根据棋盘法的结果,确定了抗原抗体的最佳工作质量浓度,结果如图3B所示,抗原包被的最佳质量浓度为10 μg/mL,抗体血清(混合血清)的稀释倍数为40 倍。
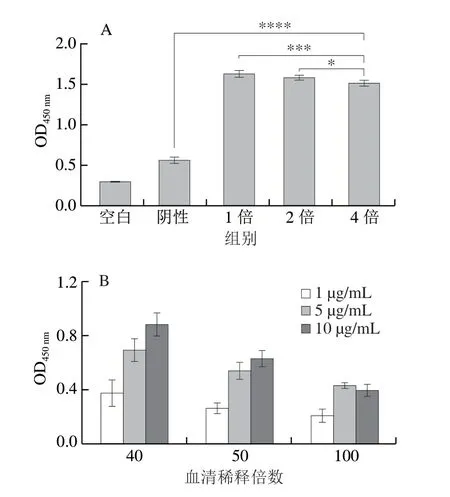
图3 小麦过敏病人血清的阳性反应核验(A)及抗原抗体最佳工作质量浓度的确认(B)Fig.3 Serum positive reaction against α-amylase inhibitor (A) and optimal concentrations of coating antigen and antibody (B)
2.3 线性表位的验证
利用最佳的包被抗原质量浓度和混合血清池稀释倍数,通过ic-ELISA测定各合成表位多肽对抗原-抗体结合反应的竞争抑制率。如图4所示,当多肽质量浓度在0.01~1 000 μg/mL范围内,P-1、P-2、P-3、P-A和P-B1这5 条多肽质量浓度对数值和竞争抑制率呈线性关系,且对抗体血清具有较强的亲和力。之后通过对表位多肽竞争抑制率曲线线性回归,计算出竞争抑制率为50%的IC50值,如表4所示,P-1、P-3、P-B1的IC50较低,说明这3 条多肽对小麦过敏血清的亲和力更好,因此从预测的6 条多肽里确定了P-1、P-2、P-3、P-B1为潜在的表位。P-4的抑制率曲线方程不呈线性关系,P-A的抑制率曲线方程虽然呈线性关系,但IC50相对较高。P-4竞争抑制率不呈线性关系的原因推测可能是多肽对小麦过敏血清的亲和力过弱[38,47]。
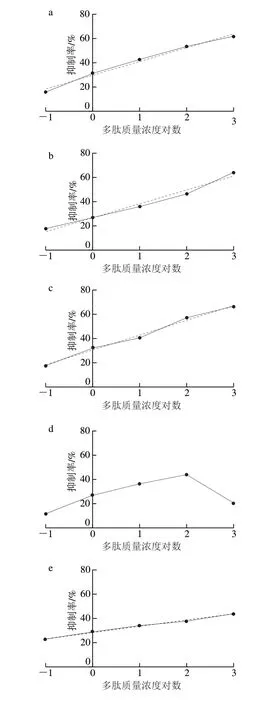

图4 合成表位多肽对抗原-抗体结合的抑制率曲线Fig.4 Inhibition rate curve of each synthesized epitope peptide against the antigen-antibody binding

表4 α-淀粉酶抑制剂表位多肽抑制率曲线回归方程Table 4 Regression equations for inhibition rates of epitope peptides against α-amylase inhibitor
2.4 α-淀粉酶抑制剂-EGCG/EGC复合物的IgE结合能力
在致敏原表位定位的基础上,有必要对其消减方法进行研究。已有研究表明,多酚类物质可以通过与致敏原蛋白发生非共价互作,改变致敏原构象结构及其抗原表位的可及性从而实现消减致敏性的目的[48]。本研究采用常见的多酚EGCG、EGC对小麦致敏原进行修饰,获得α-淀粉酶抑制剂-EGCG/EGC互作物,然后对多酚消减α-淀粉酶抑制剂的效果进行评价。
如图5所示,抑制率较低的样品,说明该浓度的多酚对α-淀粉酶抑制剂有较好的消减效果。随着EGCG浓度的增加,EGCG对α-淀粉酶抑制剂的致敏性消减能力逐步提高(图5A),其原因可能是EGCG通过改变蛋白的结构影响其表位的结构或可及性,从而抑制致敏原对特异性IgE的识别[49-50]。α-淀粉酶抑制剂-EGC复合物的IgE结合能力也呈相同趋势(图5B)。此外,发现相同浓度下EGC的消减效果较EGCG低,推测其原因可能是EGCG的分子质量较EGC大,与蛋白的亲和力高,可占据更多的残基结合位点,进而掩盖更多的IgE表位[51]。另有文献报道,分子质量越大的多酚,越能有效地沉淀蛋白质,从而改变蛋白质的结构,降低致敏性[31]。
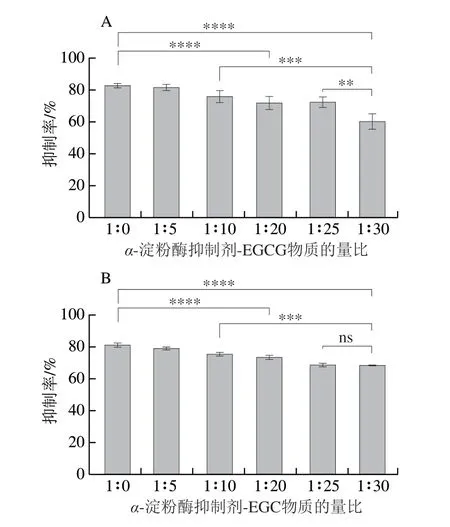
图5 α-淀粉酶抑制剂-EGCG/EGC复合物的IgE结合能力Fig.5 IgE-binding capacity of α-amylase inhibitor-EGCG/EGC complexes
2.5 α-淀粉酶抑制剂-EGCG/EGC复合物的内源荧光光谱表征
内源荧光光谱可以评价蛋白与多酚互作后对蛋白微环境的影响。蛋白质中含有色氨酸,当激发波长为280 nm时会产生荧光,荧光强度改变,说明蛋白质中暴露的色氨酸残基微环境发生了变化[52]。多酚添加后对α-淀粉酶抑制剂内源荧光光谱的影响如图6所示。所有样品中都观察到了α-淀粉酶抑制剂固有荧光的猝灭效应,且猝灭程度与多酚浓度呈正相关,说明多酚与α-淀粉酶抑制剂的相互作用影响了后者的荧光基团特性。此外,所有样品的最大荧光发射波长均在350 nm附近。α-淀粉酶抑制剂与EGCG/EGC结合后产生的荧光猝灭现象,推测原因可能是蛋白与多酚互作后改变了前者的构象结构,使α-淀粉酶抑制剂中暴露的色氨酸残基减少。同时也观察到同浓度的EGCG、EGC与α-淀粉酶抑制剂分别结合后,EGCG对α-淀粉酶抑制剂的荧光猝灭程度更大,原因可能是EGCG分子质量较EGC大,与α-淀粉酶抑制剂形成互作物后,使色氨酸处于更加包埋的状态。
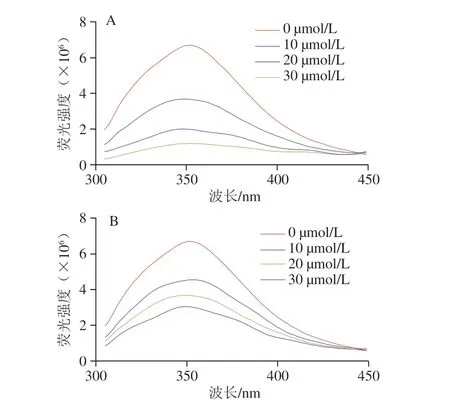
图6 α-淀粉酶抑制剂-EGCG复合物(A)与α-淀粉酶抑制剂-EGC复合物(B)的荧光光谱Fig.6 Fluorescence spectra of α-amylase inhibitor-EGCG complexes (A) and α-amylase inhibitor-EGC complexes (B)
2.6 α-淀粉酶抑制剂-EGCG/EGC复合物的圆二色谱表征
IgE结合能力发生改变的原因是抗原表位的结构发生变化[53]。圆二色谱可用于表征蛋白质的二级结构。如图7所示,多酚的加入影响了小麦α-淀粉酶抑制剂的二级结构。对于α-淀粉酶抑制剂-EGCG复合物,随着EGCG浓度的提高,蛋白无规卷曲的占比增加,α-螺旋的占比逐渐减小,β-转角占比变化不明显。而对于α-淀粉酶抑制剂-EGC复合物,其与α-淀粉酶抑制剂-EGCG复合物中蛋白的二级结构变化规律相似,即随着EGC浓度的升高,无规卷曲的占比增加,β-转角占比变化不明显,但β-折叠占比逐渐减小。总体而言,EGC、EGCG与小麦α-淀粉酶抑制剂复合后对后者的二级结构产生了一定的影响,主要表现在β-折叠、α-螺旋向无规卷曲转变。Rawel等[54]研究也发现绿原酸与牛血清白蛋白形成复合物后,蛋白质的无规卷曲占比增加,α-螺旋占比下降。Bohin等[55]发现无规卷曲是蛋白质中的开放结构,在蛋白质和表没食子儿茶素没食子酸酯的结合中起到重要作用。同时,致敏原结构的改变导致其结构稳定性下降,进而可能改变抗原表位的结构特性,最终影响抗原结合能力,改变蛋白致敏性[53]。Liu Fuguo等[49]研究发现EGCG/绿原酸与乳铁蛋白结合后致使后者的二级结构发生了改变,进而影响了表位结构并降低了蛋白的致敏能力。类似地,Xu Haoxie等[50]发现牛乳乳清蛋白和鸡蛋卵白蛋白均可与绿原酸相互作用,通过改变蛋白质的二级结构,破坏其构象或线性表位的结构/可及性,进而达到消减蛋白致敏性的效果。
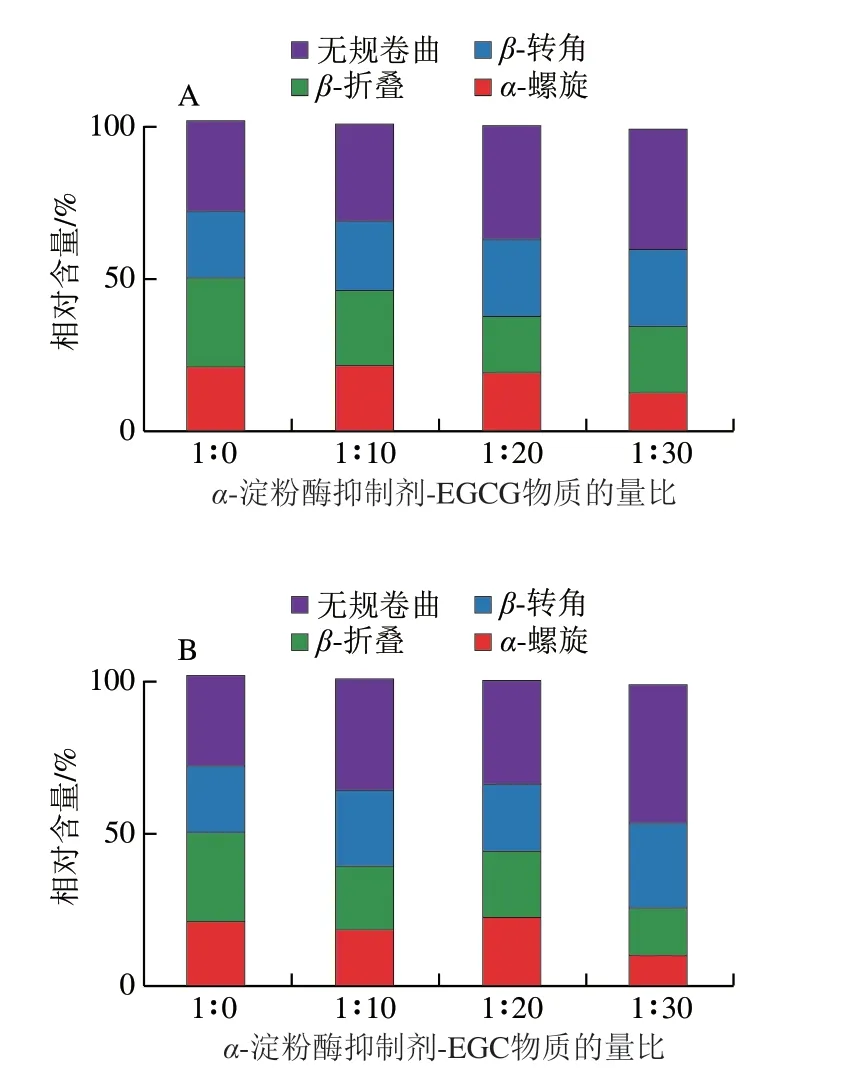
图7 α-淀粉酶抑制剂-EGCG复合物(A)与α-淀粉酶抑制剂-EGC复合物(B)二级结构的相对含量Fig.7 Relative contents of secondary structures in α-amylase inhibitor-EGCG complexes (A) and α-amylase inhibitor-EGC complexes (B)
2.7 α-淀粉酶抑制剂-EGCG/EGC复合物的紫外光谱表征
紫外吸收光谱反映了色氨酸、酪氨酸、苯丙氨酸残基所处的微环境变化,蛋白质结构的变化可以由吸收峰的强度与形状体现出来。如图8所示,与α-淀粉酶抑制剂对照相比,随着EGCG浓度的提高,α-淀粉酶抑制剂-EGCG复合物中蛋白吸收峰的强度持续降低,峰的强度改变说明了蛋白与小分子的相互作用。而随着EGC浓度的提高,α-淀粉酶抑制剂-EGC复合物中蛋白的吸收峰强度呈现先升高后降低的趋势,分析原因可能是EGC的加入改变了相邻蛋白间相互作用,随着EGC浓度的提高,使得更多的色氨酸和酪氨酸基团暴露到周围溶剂中,故紫外吸收强度先升高,但EGC浓度过高反而掩盖了部分色氨酸和酪氨酸基团,从而紫外吸收强度又出现降低[40]。此外,α-淀粉酶抑制剂和EGCG/EGC复合后的蛋白中也观察到最大吸收波长右移(红移)的现象,说明多酚与α-淀粉酶抑制剂相互作用使得蛋白质疏水氨基酸残基所处的微环境发生改变。
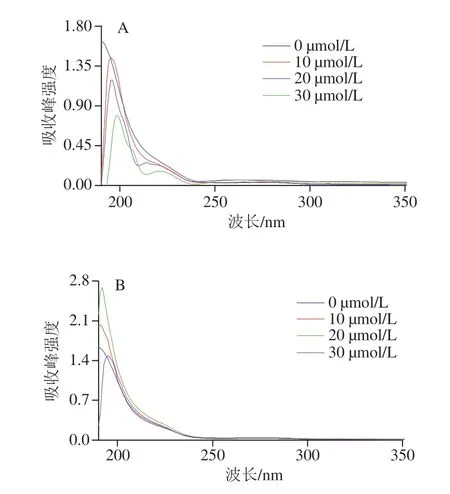
图8 α-淀粉酶抑制剂-EGCG复合物(A)与α-淀粉酶抑制剂-EGC复合物(B)的紫外吸收光谱Fig.8 UV absorption spectra of α-amylase inhibitor-EGCG complexes (A) and α-amylase inhibitor-EGC complexes (B)
2.8 α-淀粉酶抑制剂与EGCG/EGC的分子对接分析
通过Autodock分子对接软件进行结合位点扫描,发现α-淀粉酶抑制剂可与EGCG/EGC相结合,然后对α-淀粉酶抑制剂和EGCG/EGC小分子配体进行能量计算,选取蛋白质和EGCG/EGC配体的在最低能量下匹配的结果,从而获得理论的α-淀粉酶抑制剂-小分子复合物,通过软件模拟出可能的结合方式及其位点[44]。其中,α-淀粉酶抑制剂与EGCG结合的自由能ΔG为-7.46 kJ/mol,与EGC结合的自由能ΔG为-6.43 kJ/mol,说明α-淀粉酶抑制剂与EGCG的结合更牢固。类似的,Kanakis等[46]使用分子对接技术研究了几种不同儿茶素和β-乳球蛋白的复合,也发现EGCG与β-乳球蛋白的结合能力更强。使用Discovery studio 2016软件对蛋白配体-受体相互作用进行分析,得到分子对接2D与3D图,如图9、10所示。3D图直观展示了α-淀粉酶抑制剂和EGCG/EGC的结合位点,2D图展示了结合作用力及相互作用的氨基酸残基。由2D图可以看出,与EGC相比,EGCG与α-淀粉酶抑制剂氨基酸残基有更多的作用力,主要通过氢键、范德华力进行相互作用。3D图直观展现出发生相互作用的氨基酸残基具体位置,与EGCG作用的疏水性氨基酸残基主要有Gly 55、Val 104、Ile 103、Val 105、Ala 107、Ala 124,推测EGCG有可能作用在α-淀粉酶抑制剂的疏水空腔内。而与EGC作用的疏水性氨基酸残基较少,仅有Val 104、Val 105、Gly 55。
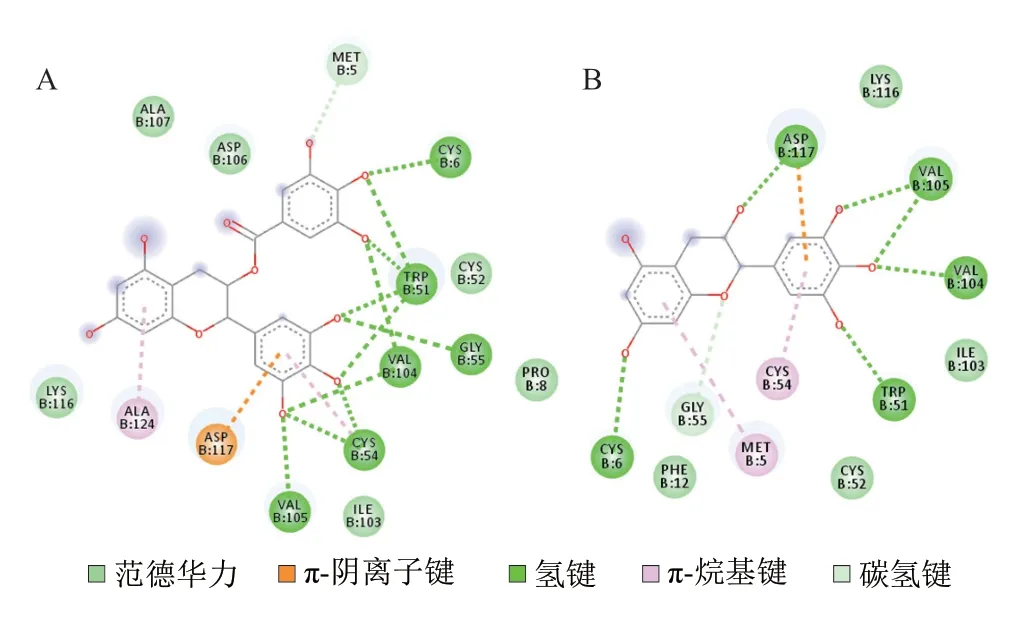
图9 α-淀粉酶抑制剂-EGCG(A)和α-淀粉酶抑制剂-EGC(B)的分子对接2D图Fig.9 2D diagrams of molecular docking between α-amylase inhibitor and EGCG (A) and EGC (B)
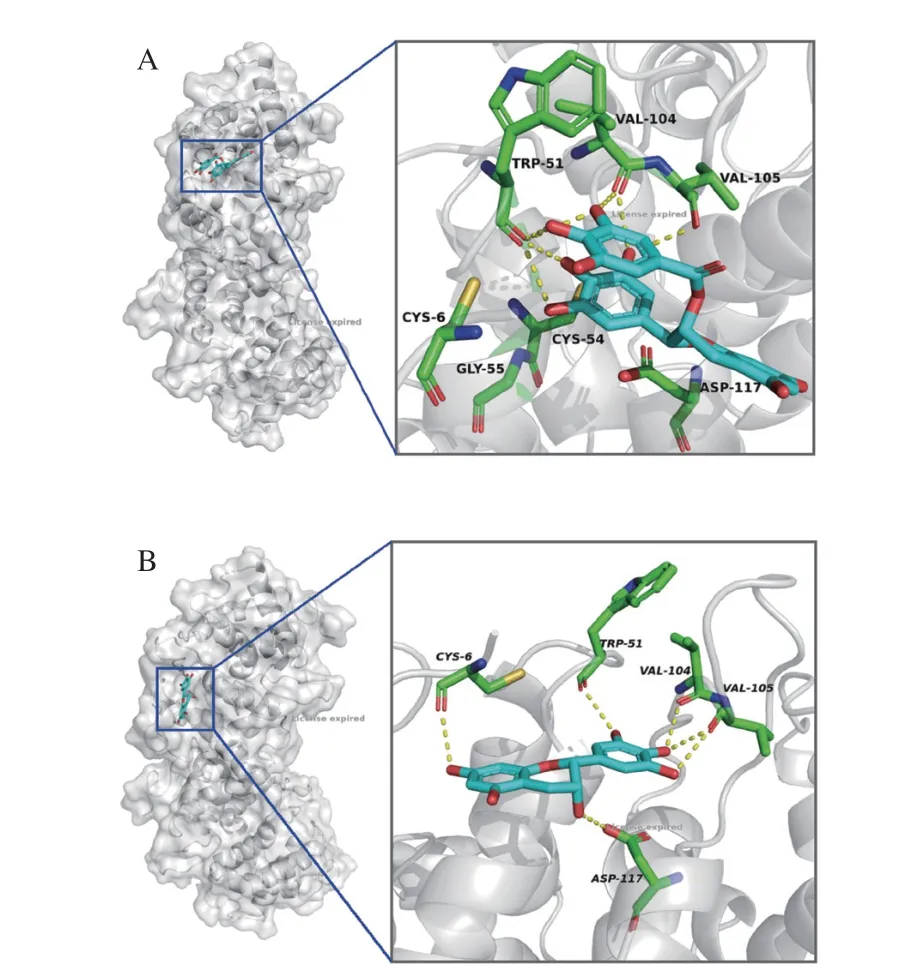
图10 α-淀粉酶抑制剂-EGCG(A)和α-淀粉酶抑制剂-EGC(B)的分子对接3D图Fig.10 3D diagram of molecular docking between α-amylase inhibitor and EGCG (A) and EGC (B)
对α-淀粉酶抑制剂-EGCG/EGC的结合位点与其筛选出的线性表位进行比对,如表5所示,其中抑制率效果较好的P-1、P-3、P-B1三条肽段有较多的结合位点。Chen等[56]指出阻断IgE结合表位序列中的一个氨基酸可能足以破坏IgE抗体序列特异性结合,同时多酚在IgE表位附近的结合理论上也会阻碍表位特异性IgE的空间可及性。这进一步证明了消减是多酚与α-淀粉酶抑制剂线性表位及其附近残基相互作用的结果。Plundrich等[51]研究发现苯甲酸可以消减Ara h 2的致敏性,并经过分子对接证明了Ara h 2表面共有14 个苯甲酸的潜在结合位点,苯甲酸通过氢键、盐桥与各种氨基酸相互作用,这也印证了多酚可以削弱表位特异性IgE对表位的线性识别。

表5 α-淀粉酶抑制剂-EGCG/EGC的结合位点与其线性表位比较Table 5 Comparison of the binding sites of α-amylase inhibitor-EGCG/EGC complexes with their linear epitopes
3 结论
本研究通过生物信息学预测对α-淀粉酶抑制剂线性表位进行预测,经综合分析后筛选得到6 条候选表位。通过ic-ELISA对候选表位进行验证,最后得到4 条线性表位。利用多酚与致敏原相互作用消减致敏,采用非共价的方法,制得α-淀粉酶抑制剂-EGCG、α-淀粉酶抑制剂-EGC复合物,利用ic-ELISA对消减效果进行研究,结果表明EGCG有较好的消减能力,且当α-淀粉酶抑制剂与EGCG物质的量比为1∶30时,复合物与IgE结合水平达到最低水平,结合能力下降22.7%。进一步通过紫外光谱、荧光光谱、圆二色谱对复合物的结构进行表征,结果表明当多酚加入后,复合物体系中α-淀粉酶抑制剂的结构发生改变,具体表现为蛋白疏水氨基酸残基所处的微环境发生改变,蛋白二级结构β-折叠、α-螺旋向无规卷曲转变。通过Autodock分子对接软件获取了最低能量匹配下的复合物,通过与其筛选出的线性表位进行比对,发现抑制率效果较好的P-1、P-3、P-B1与多酚有较多结合位点,这进一步证明了消减是多酚与α-淀粉酶抑制剂线性表位及其附近残基相互作用的结果。研究结果可为小麦非麸质致敏原表位鉴定、小麦非麸质致敏原的消减方法以及制备酚类低敏互作物提供参考依据。
