紫花苜蓿GPAT基因家族鉴定及在盐碱胁迫下的表达模式分析
2023-10-08高佳荷王佳妮石凤翎高翠萍
唐 芳, 梅 亭, 高佳荷, 王佳妮, 石凤翎, 高翠萍
(内蒙古农业大学草原与资源环境学院, 内蒙古 呼和浩特 010010)
植物脂质是生物膜的重要组成成分,在能量转换、碳储备、信号转导以及胁迫应答等方面发挥着不可估量的作用[1]。植物脂质种类繁多,包括甘油磷脂类、脂肪酸类、鞘脂类、糖脂类等[2],其生物合成受到基因和蛋白质网络的精确调控[3]。甘油-3-磷酸酰基转移酶(Glycerol-3-phosphate acyltransferase,GPAT)催化酰基从酰基辅酶A(Acylcoenzyme A,acyl-CoA) 或酰基载体蛋白(Acyl-carrierprotein,acyl-ACP)转移到甘油-3-磷酸(glycerin-3-phosphate,G3P) 的sn-1或sn-2位置,生成溶血磷脂酸(Lysophosphatidic acid,LPA)[4]。LPA是形成多种甘油脂的重要中间体[5],是催化甘油酯合成的第一步反应[6]。因此,GPAT是三酰甘油(Triacylglycerol,TAG)生物合成的限速酶[7],参与多种脂类的生物合成途径。植物中至少有3类GPAT基因参与甘油酯生物合成,分别定位在质体、线粒体和内质网[8]。GPAT除参与植物角质层及软木脂合成外,也响应植物应对非生物胁迫[7],在植物生长发育及抵御逆境胁迫中具有重要作用。
GPAT基因已在多种植物中进行了全基因组鉴定和功能分析,包括拟南芥(Arabidopsisthaliana)[7]、百合(Liliumregale)[9]、大麦(Hordeumvulgare)[10]、水稻(Oryzasativa)[11]、连翘(Forsythiasuspense)[12]和蒺藜苜蓿(Medicagotruncatula)[4]等。拟南芥的AtGPATs基因研究最为深入,目前已鉴定出10个AtGPATs基因,分别为质体ATS1、线粒体AtGPAT1,2,3和内质网AtGPAT4,5,6,7,8,9[7]。根据进化距离的远近程度将其分为3个亚组[7],分别是ATS1,AtGPAT9以及AtGPAT1,2,3,4,5,6,7,8。GPAT基因功能研究表明,ATS1参与膜脂质合成[13],AtGPAT1是花粉发育中的关键基因[14],但AtGPAT3功能尚待确定[13]。AtGPAT4,6,8均与植物角质层合成有关,其中AtGPAT4和AtGPAT8属于高度同源的功能冗余基因,是植物角质层合成过程中的关键酰基转移酶[15],AtGPAT6参与萼片和花瓣中角质层的合成[16]。AtGPAT5对根和种皮中软木脂的合成至关重要[17],AtGPAT7与AtGPAT5同源性高,功能相似,在组织受伤时被强烈诱导[18]。AtGPAT9与种子含油量呈正相关[13]。GPAT基因除直接参与油脂生物合成,还与植物响应非生物胁迫存在一定程度的正向调控关系[19]。隋娜等[20]将碱蓬(Suaedasalsa)SsGPAT基因转入拟南芥,显著提高了盐胁迫下拟南芥的发芽率和根长,进一步研究表明拟南芥中过表达的SsGPAT基因提高了PG不饱和脂肪酸含量,减轻了PSII和PSI系统的光抑制,最终增强宿主耐盐性。
紫花苜蓿(Medicagosativa)属于豆科苜蓿属多年生牧草,有“牧草之王”的美誉,是世界上种植面积最大,分布最广的人工牧草[21-22],蛋白含量高、适应性强[23],且具有改良土壤盐渍化[24-25]、丰富土壤有机质等功效[24,26]。多个紫花苜蓿基因家族已有研究,如与低木质素遗传改良有关的MsCCoAOMT基因家族[27],与种子贮藏、发育、光信号传导等有关的bZIP转录因子基因家族[28],与响应干旱胁迫有关的MsTCP转录因子基因家族[29],与铜转运有关的MsCOPT基因家族[30]等。但是,目前尚没有关于紫花苜蓿MsGPAT基因家族的报道。
我国盐碱地约有1 000 km2,土壤盐碱化造成土壤理化性质受到破坏,严重危害植物生长[31]。鉴于MsGPAT基因家族在植物生长发育与盐碱胁迫响应中的重要作用,本研究对紫花苜蓿MsGPAT基因家族进行鉴定和分析,并研究其在盐碱胁迫下的表达模式,为进一步研究GPAT基因在紫花苜蓿中的具体功能(如耐盐碱性)提供理论依据和候选基因。
1 材料与方法
1.1 MsGPAT基因鉴定
查询文献获取10个AtGPAT基因家族成员信息,从TAIR10拟南芥数据库(http://plants.ensembl.org)获取拟南芥蛋白质序列,利用紫花苜蓿基因组、蛋白质序列和注释信息(https://figshare.com/projects/whole_genome_sequencing_and_assembly_of_Medicago_sativa/66380[32])。鉴定MsGPAT基因家族成员。具体方法为:1、以拟南芥GPAT蛋白序列为靶序列,通过Blast比对紫花苜蓿蛋白质序列,获取紫花苜蓿GPAT候选蛋白序列;2、利用Pfam(http://pfam.xfam.org)的PF01553隐马尔可夫模型查找紫花苜蓿蛋白质序列获取候选蛋白序列。以上两种方法获取的序列取并集,并去除重复序列,利用NCBI CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)、Phhmer(https://www.ebi.ac.uk/Tools/hmmer/search/phmmer)、SMART(http://smart.embl-heidelberg.de/)在线结构域预测工具进行验证,保证所有MsGPAT结构域被识别为“Glycerol-3-phosphate acyltransferase”注释(NCBI CDD结果)或PF01553(Pfam结果)或PlsC结构域(SMART结果),再根据植物GPAT蛋白质氨基酸序列长度(237~621个氨基酸)进行筛选,最终将符合以上要求的蛋白序列归为MsGPAT基因家族成员。
1.2 蛋白质理化性质和染色体定位分析
利用ExPasy(https://web.expasy.org/protparam/)在线蛋白质分析工具预测氨基酸数、分子量、等电点和亲水性指数等信息。基因长度按照非冗余外显子长度之和计算。根据紫花苜蓿基因注释文件信息,提取MsGPAT基因在染色体上的位置,并利用在线工具MagGene2[33](http://mg2c.iask.in/mg2c_v2.1/index.html)绘制基因的染色体定位图。
1.3 基因结构、蛋白质基序和顺式启动元件分析
利用在线工具GSDS2.0(http://gsds.gao-lab.org/)分析MsGPAT家族成员的基因结构。通过MEME在线软件(https://meme-suite.org/meme/tools/meme)预测,设定最大预测值为10。利用TBTools[34]展示motif和基因结构。选取MsGPAT基因距离起始密码子上游的2 000 bp序列,利用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)的Search for care工具预测并分析启动子顺式作用元件。
1.4 系统发育树分析
通过MEGA7[35]软件的NJ法构建系统进化树,采用Mega7中的MUSCLE算法对拟南芥AtGPAT、蒺藜苜蓿MtGPAT与紫花苜蓿MsGPAT基因家族成员的蛋白质序列进行多序列比对,构建系统发育树,比较进化距离(Bootstrap测试值设定为1 000)。使用Evolview[36](https://evolgenius.info)在线进化树注释工具,对进化树进行注释。
1.5 共线性分析
构建紫花苜蓿MsGPAT基因家族数据库,利用Blast进行物种内序列比对,再利用MCScanX[37]进行共线性分析,并利用Circos软件[38]绘制共线性关系图。
1.6 基因表达分析
本研究基于杨紫贻[39]紫花苜蓿耐盐评价体系,因此胁迫浓度和胁迫后取样时间的选择延用其设计。以紫花苜蓿耐盐品种‘中苜1号’简称ZM1和敏盐品种‘WL323’为研究对象,对2周龄幼苗进行200 mmol·L-1NaCl单盐胁迫、50 mmol·L-1NaHCO3单碱胁迫以及200 mmol·L-1NaCl+50 mmol·L-1NaHCO3混合盐碱胁迫,并选择胁迫后0 h,6 h,12 h,24 h的根部(因根系盐胁迫下地下部分直接与盐碱溶液接触[40],首先受到影响[41])转录组测序结果进行分析。数据对原始FPKM值进行Log2(FPKM+1)变换已满足方差分析的条件,并利用Excel软件绘制差异基因表达模式图。
2 结果与分析
2.1 紫花苜蓿MsGPAT基因家族鉴定
MsGPAT基因家族成员在NCBI CDD数据库中均被注释为“Glycerol-3-phosphate acyltransferase”,在Pfam数据库中大部分被识别为pfam结构域(PF01553),在Smart数据库中大部分被识别为PlsC结构域。本研究中,将存在以上3种结构域中的任意一种蛋白质序列视为MsGPAT基因家族成员。Waschburger等人分析了450个GPATs蛋白质特性,认定GPATs蛋白质结构氨基酸长度在237~621个之间[3],故剔除不符合该长度范围内的蛋白质33个。最终从紫花苜蓿全基因组中鉴定出73个MsGPAT基因家族成员,按照其在染色体位置的先后顺序,分别命名为MsGPAT1-MsGPAT73。如表1所示,MsGPAT基因家族成员的ORF长度为720 bp~1 530 bp,编码239~509个氨基酸,其中蛋白质序列最长的是MsGPAT63,最短的是MsGPAT8。MsGPAT分子量介于26.8 kDa~57.9 kDa之间,理论等电点的变化相对较小,介于5.9~9.5之间,只有9个MsGPAT基因编码的蛋白等电点小于7。(表1)
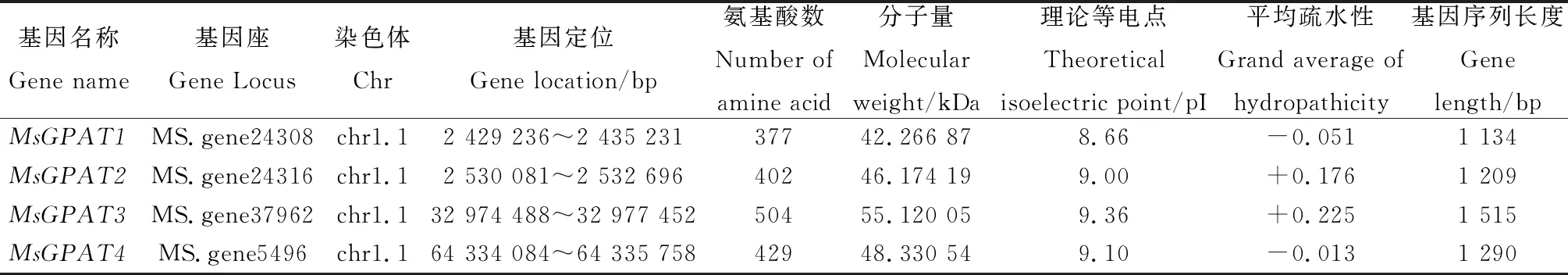
表1 紫花苜蓿MsGPAT基因家族成员信息
2.2 紫花苜蓿染色体定位分析
对MsGPAT基因家族成员进行染色体定位分析,结果表明MsGPATs的69个成员分布在紫花苜蓿26条染色体上,其余4个基因定位在scaffold上。如图1所示,Chr1.3,Chr7.4上分布的MsGPAT基因最多,均有5个成员;Chr2.2,Chr2.4,Chr4.1,Chr8.4上的含有的MsGPAT基因家族成员最少,仅有1个;其余20条染色体上分布着2~4个MsGPAT基因家族成员。此外,scaffold 6938,10775,10776和10777上分别分布着1个MsGPAT基因家族成员。紫花苜蓿MsGPAT基因家族成员在染色体上呈不均匀分布,多分布于各染色体的两端,且MsGPAT1和MsGPAT2,MsGPAT5和MsGPAT6,MsGPAT8至MsGPAT10,MsGPAT13和MsGPAT14、MsGPAT23和MsGPAT24,MsGPAT25和MsGPAT26呈小范围簇状排列。
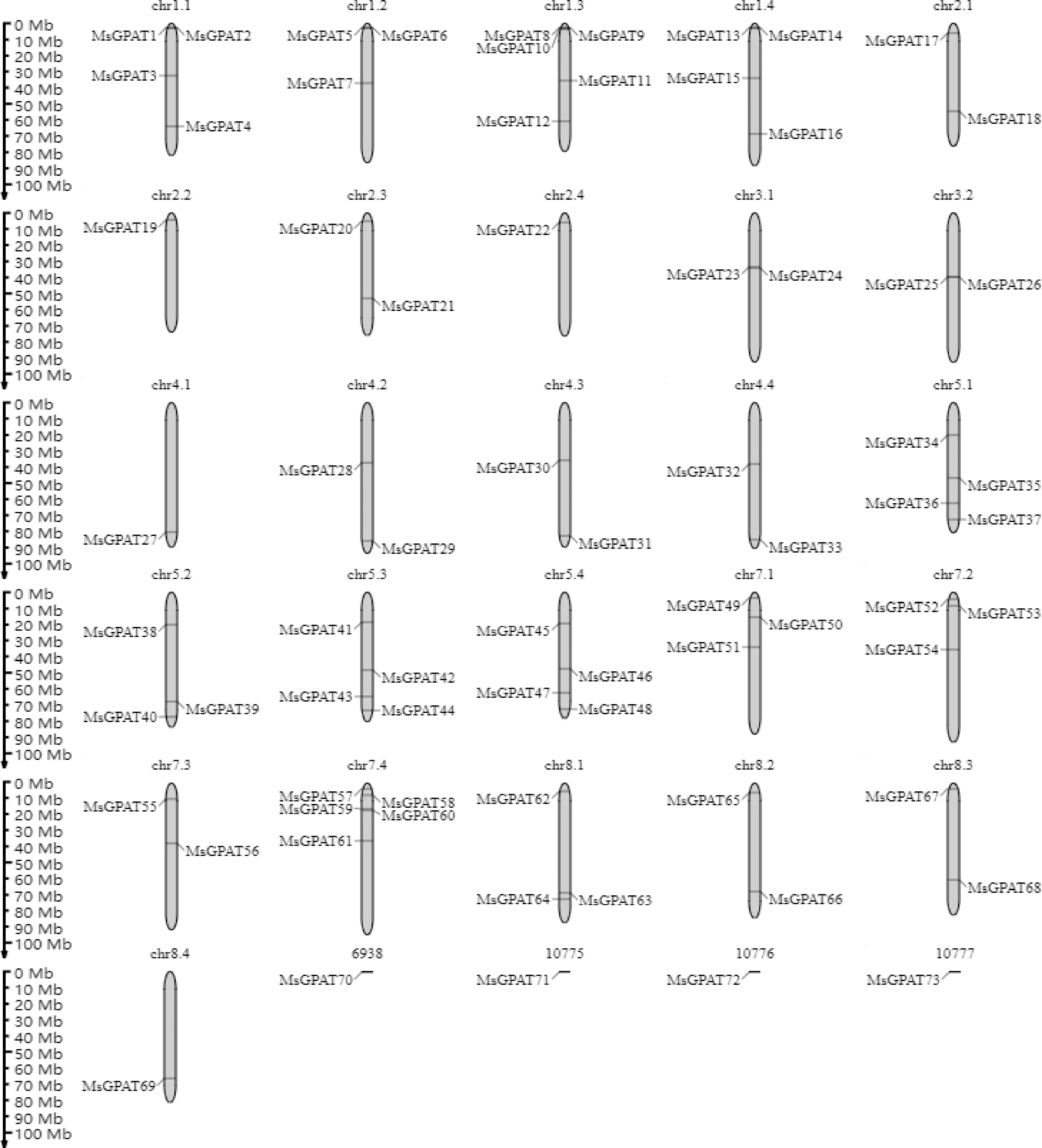
图1 紫花苜蓿MsGPAT基因的染色体定位分析
2.3 紫花苜蓿MsGPAT基因结构分析和保守基序分析
紫花苜蓿MsGPAT基因结构分析显示(图2),除MsGPAT15外,其余基因家族成员均含有内含子,内含子数在1~13个之间。不同成员基因结构存在较大差异,MsGPAT63和MsGPAT68基因结构最复杂,含有14个外显子;而MsGPAT15基因结构最简单,仅有1个外显子。其中,含有2~3个外显子的成员共有38个,占比52%;含有11~12个外显子的成员共有18个,占比25%;含有介于4~10个外显子的成员共14个,占比19%。

图2 紫花苜蓿MsGPAT基因结构和保守基序
利用Meme软件对MsGPAT基因编码蛋白的保守基序进行分析,共鉴定出10个保守基序(图2)。其中最保守的基序是Motif8,共有70个基因家族成员含有该基序,只有MsGPAT23,25,61不含有该基序。MsGPAT基因家族成员含有1~8个保守基序,其中含有全部8个基序的成员共30个,占比41%;仅含有1个保守基序的成员有MsGPAT34,38,41,45,49,52,57占比9%。
2.4 紫花苜蓿MsGPAT基因顺式启动元件分析
基因编码区上游区域存在多个顺式作用元件,具有调控相关基因表达的特性。本研究对73个MsGPAT基因的启动子区域进行顺式作用元件分析,以用于预测MsGPAT基因的潜在功能。如图3所示,通过对光反应元件(TCCC-motif,LAMP-element,GT1-motif)、生长素反应元件(G-Box和TGA-element)、胁迫反应元件(MYC,ABRE,MYB,W-box,GT1-motif,LTR和MBS)进行分析,MsGPAT基因成员的顺式作用元件差异较大,如MsGPAT71含有上述9种类型的顺式作用元件,而MsGPAT19则只有上述3种类型的顺式作用元件。在胁迫反应元件的种类上,大部分基因都有4个以上的胁迫应答元件,表明MsGPAT基因广泛参与植物响应逆境胁迫。
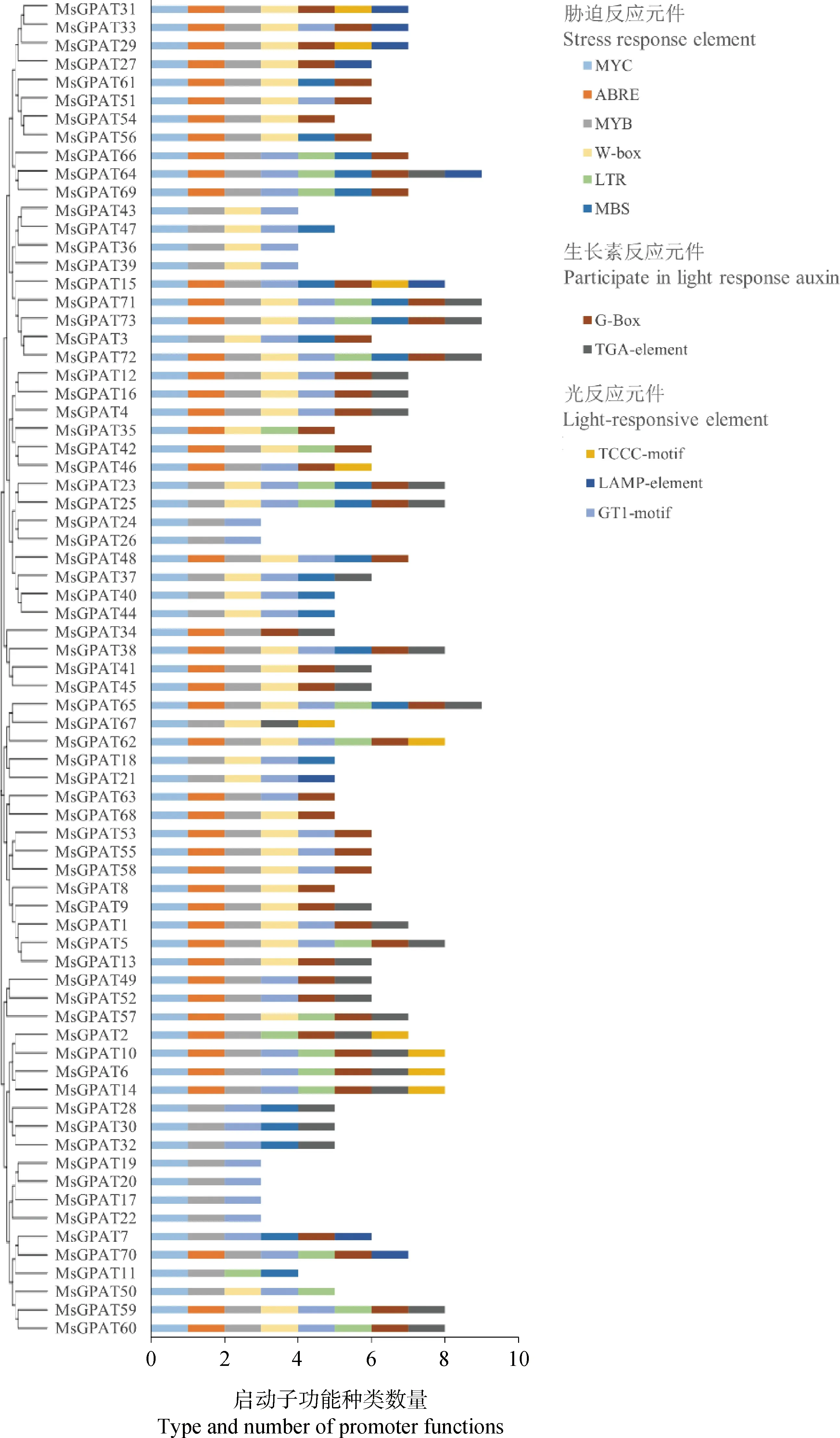
图3 紫花苜蓿MsGPAT顺式作用元件
2.5 紫花苜蓿MsGPAT基因家族系统发育分析
对10个拟南芥AtGPATs基因家族成员、24个蒺藜苜蓿MtGPATs基因家族成员和73个紫花苜蓿MsGPATs基因家族成员构建系统发育树(图4),结果表明73个紫花苜蓿成员分为3个亚家族,与拟南芥AtGPATs基因分组相同[4]。其中,24个MsGPATs基因、8个MtGPATs基因和拟南芥ATS1聚成第1亚族,表明该组基因与ATS1进化关系较近,与ATS1处于同一分支的MsGPAT34,38,45,41基因具有与拟南芥ATS1类似的12个外显子结构,均只含有1个motif。其他亚组MsGPATs基因外显子从2~11个不等。第2亚族中,15个MsGPATs基因,6个MtGPATs基因与AtGPAT9具有较近的进化距离。其中MsGPAT67,21,18与AtGPAT9基因结构类似,具有12个外显子,以及3个motif结构。第3亚族中,共包含34个MsGPATs基因、10个MtGPATs成员和拟南芥AtGPAT1-8。该亚族中MsGPATs基因均具有2~3个外显子,与拟南芥AtGPAT1-8的基因结构类似,并具有相似的motif结构。
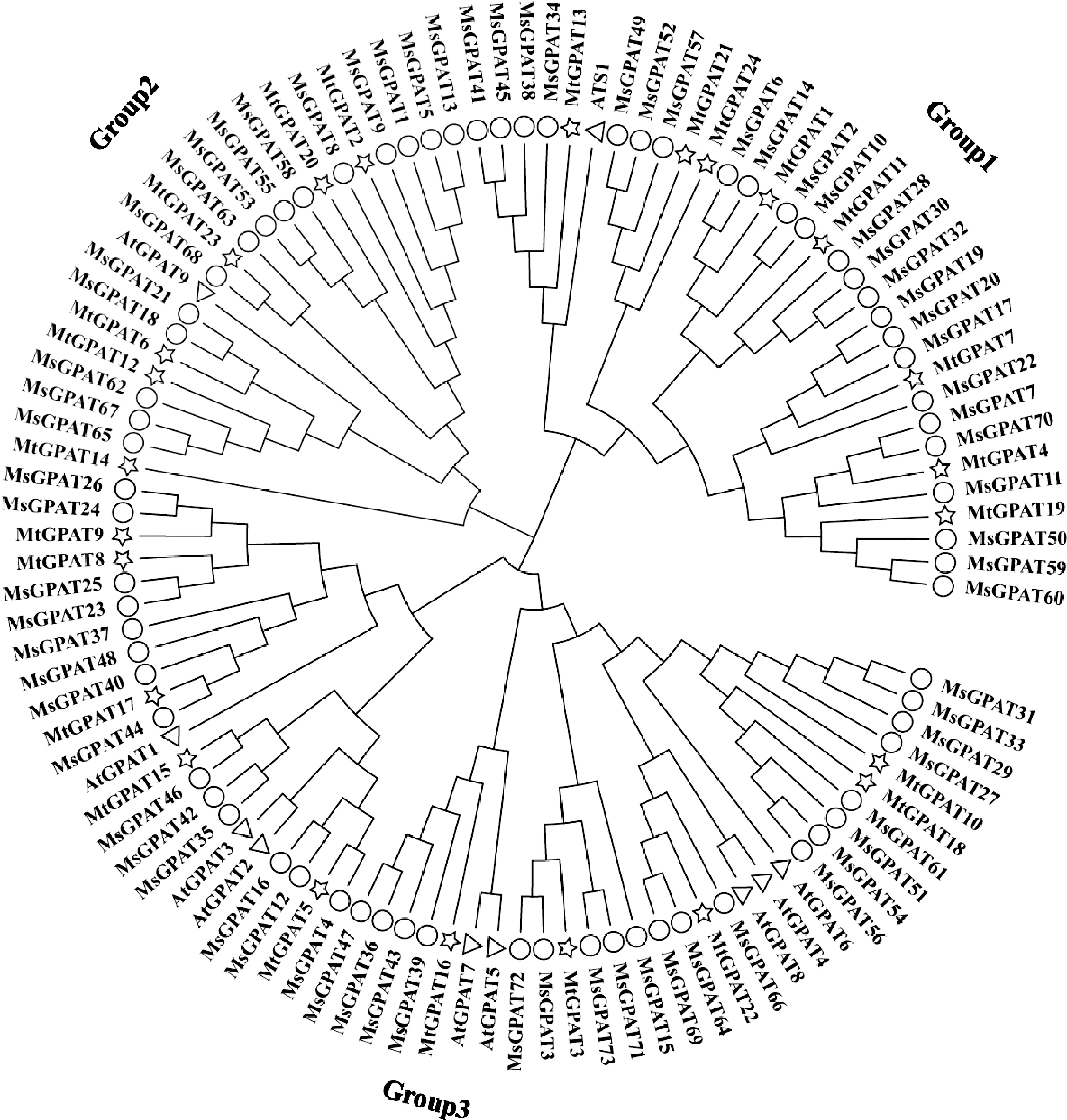
图4 紫花苜蓿MsGPAT、蒺藜苜蓿MtGPAT和拟南芥AtGPAT基因家族系统进化分析
2.6 紫花苜蓿MsGPAT基因家族共线性分析
紫花苜蓿MsGPAT基因家族大部分成员具有共线性关系,具有高度同源性(图5)。如同源染色体Chr1.1上的MsGPAT1与Chr1.2上的MsGPAT5,Chr1.3上的MsGPAT8与Chr1.4上的MsGPAT13均有共线性。此外,存在如Chr1.3的MsGPAT11与Chr7.1上的MsGPAT50和Chr7.4上的MsGPAT59的跨染色体大片段复制现象。
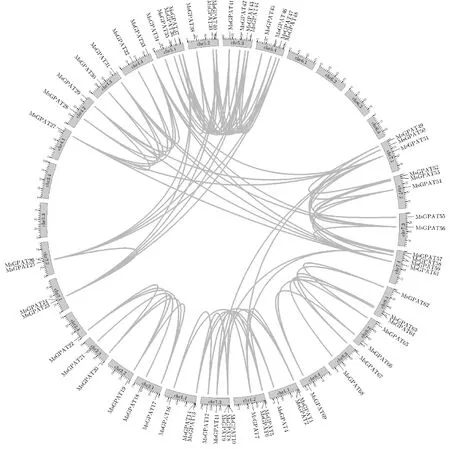
图5 MsGPAT基因家族共线性分析
2.7 紫花苜蓿MsGPAT基因盐碱胁迫下的表达模式分析
本研究中共筛选出19个响应盐碱胁迫的MsGPATs基因,其中共10个MsGPATs基因参与响应单盐胁迫(图6),5个MsGPATs基因参与响应单碱胁迫(图7),18个MsGPATs基因参与响应混合盐碱胁迫(图8)。
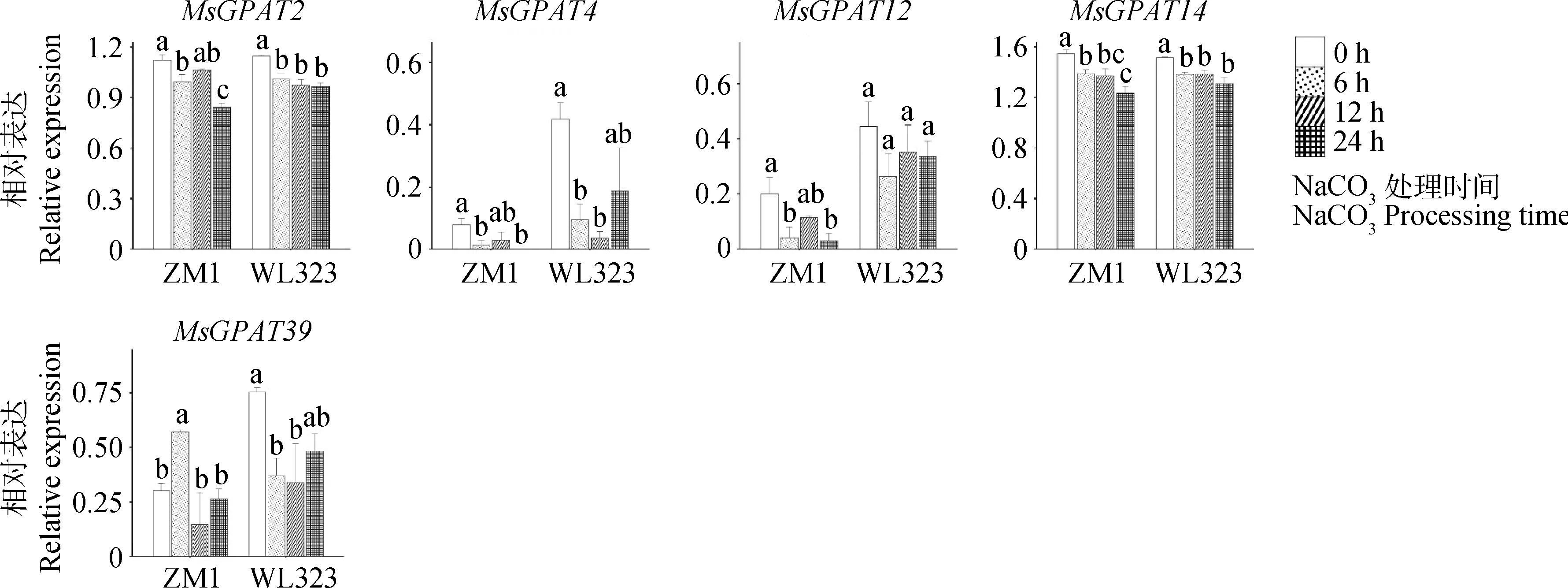
图7 ‘ZM1’和‘WL323’紫花苜蓿品种MsGPAT基因在碱胁迫下的表达模式
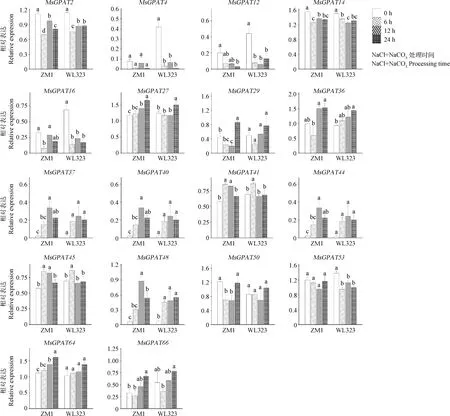
图8 ‘ZM1’和‘WL323’紫花苜蓿品种MsGPAT基因在混合盐碱胁迫下的表达模式
单盐胁迫下,两紫花苜蓿品种的大部分响应基因表现出大体相似的表达规律,MsGPAT36,37,40,44,48的相对表达量在盐胁迫后上调,在24 h表达量回落(图6),呈现出先增高后下降的趋势;而MsGPAT2,16随胁迫时间增长呈现先下降后增高的趋势,低表达量多出现在6~12 h。MsGPAT4,12中耐盐品种‘ZM1’和敏盐品种‘WL323’呈现不同的表达趋势,MsGPAT4在‘ZM1’中的表达量变化不大,而在‘WL323’中的表达量呈显著下降(P<0.05);MsGPAT12在‘ZM1’中的表达量变化不大,而在‘WL323’中的表达量呈显著下降(P<0.05),因此可能是调控两品种耐盐性差异的关键候选基因。
在单碱胁迫下,紫花苜蓿MsGPATs大部分基因相对表达量与0 h相比均有所下调(图7),且成员数量上也远小于响应单盐胁迫的基因成员。其中,MsGPAT2,4,12,14表达量随盐胁迫呈下降趋势(6~12 h)。而MsGPAT39在‘ZM1’中的表达量呈现先升高后下降的趋势,在与‘WL323’中的表达趋势相反(图7),可能是不同紫花苜蓿品种响应碱胁迫的特异基因。
混合盐碱胁迫下,共有18个MsGPATs基因家族成员的表达量在不同品种或不同胁迫时间下存在差异,其中的大部分成员的表达量随盐碱胁迫后上调(图8),表达量高峰在6~24 h之间均有分布。MsGPAT4,29,37,48,50,53,64在两品种间表达量存在显著差异(图8),如MsGPAT53在‘ZM1’中的表达量变化不大,而在‘WL323’中的表达量在胁迫后显著下降(P<0.05),MsGPAT64在‘ZM1’中的表达量随胁迫时间增长呈现显著上升(P<0.05)的趋势,而‘WL323’中的表达量在胁迫前后变化不大。因此MsGPAT4,29,37,48,50,53,64可能是调控苜蓿耐盐碱性的关键差异基因。
3 讨论
大部分植物GPAT基因的数量未超过30个,例如水稻[11]GPAT基因为26个,玉米(Zeamays)[42]为20个,大豆(Glycinemax)[3]为28个,蒺藜苜蓿为24个[4]。本研究共鉴定出73个紫花苜蓿MsGPAT基因家族成员,其数量约是蒺藜苜蓿的3倍,是目前已研究GPAT家族成员数量最多的植物,可能是由于紫花苜蓿基因组加倍造成的。在bZIP基因家族[28]研究中,紫花苜蓿共鉴定出138个bZIP基因,而蒺藜苜蓿鉴定出75个bZIP基因,紫花苜蓿的bZIP基因大约是蒺藜苜蓿的2倍,与GPAT基因家族数量的变化趋势一致。此外,也可能是不同的研究方法导致的成员数量有差异。水稻OsGPAT基因家族在较早的研究中为18个成员,而最新的研究中为26个[11]个成员。稍早的研究中,蒺藜苜蓿仅鉴定出12个MtGPAT基因[3],而最新的研究中共鉴定出24个[4],表明研究方法的更新使得研究者可以鉴定出更多的基因家族成员。
系统发育树中进化距离较近的MsGPAT家族成员的基因结构具有相似的外显子和内含子数目。除MsGPAT15没有内含子,其他MsGPATs基因均含有1个或更多数量的内含子或外显子,即内含子或外显子数量存在多种形式,表明MsGPATs基因结构在进化过程中的多样性。其他植物GPAT基因结构也有类似特征,如拟南芥ATS1和AtGPAT9基因均具有12个外显子,11个内含子,ATGPAT1-8基因具有2~4个外显子和1~3个内含子的形式。蒺藜苜蓿MtGPATs基因最少含有1个外显子,无内含子,最多含有14个外显子和13个内含子的多样化的基因结构。
紫花苜蓿MsGPAT基因家族成员系统发育树可分为3个亚组,这与在拟南芥[7]、蒺藜苜蓿[4]、水稻[11]、棉花(Gossypium)[43]和玉米[42]等植物中的GPAT基因家族的研究一致。拟南芥ATS1基因,是一种可溶性GPAT酶,参与叶片脂质合成途径[13],以酰基载体蛋白ACP为底物[8]。本研究中,共有24个MsGPATs基因与其聚类在一起,且MsGPAT34,38,41,45基因与拟南芥ATS1进化距离最为接近,可能具有生物膜脂质合成[13]的功能。拟南芥AtGPAT1-8,功能与花粉发育、逆境抵抗、角质层和软木脂合成有关。本研究中,共有34个MsGPATs成员与拟南芥AtGPAT1-8聚类在一起。其中MsGPAT17,23,24,25,26,37,40,44,48与AtGPAT1进化距离最为接近,可能具有相似的功能,与绒毡层磷脂合成和花粉发育有关。AtGPAT4,6,8均与植物角质层合成有关。MsGPAT27,29,31,33,51,54,56,61与AtGPAT6在同一分支,可能参与花器官角质层的合成[44]。MsGPAT36,39,43,47与AtGPAT5,7的进化距离最为接近,可能与根和种皮的软木脂合成有关[17]。AtGPAT9是参与生物TAG合成的重要GPAT酶,负责植物膜脂和油的合成[45]。过表达的AtGPAT9显著提升种子中甘油三酯的积累水平,并且与ER甘油酯生物合成的其他酶相互作用。本研究中,共有15个MsGPATs成员与AtGPAT9聚类在一起,且MsGPAT18,21,62,65,67与AtGPAT9处于同一分支,推测其可能具有与AtGPAT9相似的功能。前人研究表明,过表达的AtGPAT6、7可降低幼苗在盐胁迫下的死亡率,表明二者具有正向调控植物耐盐性的潜力[19],且AtGPAT2与AtGPAT6类似,与盐胁迫响应有关[20]。在本研究中,由于MsGPAT4,12,16与AtGPAT2聚类在一起,MsGPAT27,29与AtGPAT6聚类在一起,MsGPAT36,39与AtGPAT7聚类在一起,且在盐、碱和盐碱混合胁迫下有一定响应,因此推测以上基因在紫花苜蓿盐碱胁迫调控中可能具有重要作用,具有提高紫花苜蓿耐盐碱性的潜力。然而,目前没有对紫花苜蓿MsGPAT基因家族的具体功能进行研究的报道,因此推测的功能尚待研究验证。
4 结论
本研究表明,紫花苜蓿MsGPAT基因家族共有73个成员,大多数分布在26条染色体上,呈不均匀分布,另有4个成员分布在scoldfold上。系统发育分析显示紫花苜蓿基因分为3个亚组。基因结构分析显示该家族成员外显子数在1~14之间,其中含有2~3个外显子的成员和11~12个外显子的成员最多。蛋白质基序分析显示motif8是MsGPAT基因家族中最保守的基序。顺式作用元件分析显示MsGPAT基因家族成员大部分含有多种不同数量的光响应、生长素反应和胁迫应答元件。共线性分析表明MsGPAT基因家族大部分成员之间存在大片段复制现象。在18个响应盐碱胁迫的MsGPAT基因中,大部分成员积极响应单盐和混合盐碱胁迫,对单碱胁迫响应的基因较少。其中,MsGPAT4/12和MsGPAT39分别可能是调控紫花苜蓿耐盐性、碱性的关键差异基因。
