基于SSR标记的福建省闽楠代表性群体遗传多样性分析
2022-11-29冯一宁李因刚周鹏燕徐立安
冯一宁, 李因刚,祁 铭,周鹏燕,周 琦,董 乐,徐立安*
(1.南京林业大学,南方现代林业协同创新中心,江苏 南京 210037;2.浙江省林业科学研究院,浙江 杭州 310023)
闽楠(Phoebebournei)为樟科楠属高大常绿乔木[1],木材主要用于建筑、家具、造船等高档用材[2]。由于闽楠具有较高的经济价值,遭到生境破坏、过度采伐等人为影响,闽楠的野生资源逐渐减少,被列为国家二级珍稀濒危植物[3]。闽楠天然分布于福建、江西、湖南、湖北等省,主要分布在闽、赣两省[4]。野外调查显示有一定规模的闽楠天然种群现已难以找到,而在福建省内还偶有一定数量古树和较为集中的闽楠天然群落。
目前在林木遗传多样性研究中主要应用的分子标记有基于分子杂交技术的RFLP,以PCR技术为核心的SSR、RAPD、AFLP和ISSR等。其中,简单序列重复(simple sequence repeat,SSR)分子标记克服了RFLP对DNA检验出的多态性低、操作较烦琐,RAPD标记技术重复性和稳定性差且不能识别杂合位点,以及AFLP标记对基因组的反应条件和纯度要求高等缺点,且结果更加可靠[5-9]。
随着对闽楠研究的深入和分子标记技术的发展,有些学者已利用RAPD、ISSR等显性标记对闽楠开展了群体遗传多样性方面的研究[10-12],为闽楠的保护和利用提供了一定依据;而共显性SSR标记对闽楠的研究,目前仅有时小东等[13]对楠木进行的SSR标记开发,刘丹等[14]分析了部分优树及其子代中的优株遗传变异和遗传分化,黄雨芹等[15]探讨了基于SSR标记构建闽楠核心种质,而利用SSR标记对闽楠天然群体进行遗传多样性方面的研究鲜见报道。
本研究利用自主开发的18对SSR分子标记,对福建省内保存较好的3个代表性闽楠天然群体进行群体检测,研究其群体间及群体内的遗传变异水平及遗传结构特征,为闽楠天然群体的保护和利用提供进一步参考。
1 材料与方法
1.1 试验材料
本研究材料来源于南京林业大学林学院闽楠课题组2018年收集的3个闽楠天然群体,包括福建政和(ZH)群体(为华东最大,保存最好的闽楠古树群,有1 100余株,其中胸径85 cm以上的有240余株,最大单株胸径达到137 cm)、福建沙县罗卜岩(LBY)群体(含少量成年大树,更新较为旺盛,群落保存较好)和福建顺昌(SC)群体(为古树群,较为集中分布在500 m2范围内,人为干扰较大,幼苗更新很少)。3个群体的基本信息见表1。

表1 3个闽楠地理群体基本信息
从3个群体分别采集29、33和26份个体样品。福建罗卜岩保护区选取了仅存的大部分胸径30 cm以上的大树(占样本的少部分),其他样本胸径10~20 cm;福建顺昌群体采集了全部26株闽楠大树(近90%样本胸径40~95 cm);福建政和群体样本均是胸径大于45 cm的大树。罗卜岩和政和群体样本间距离为50 m以上。确定采样单株后,在每个单株上采集5~10片新萌的嫩叶,置于塑料自封袋内,注入10倍叶片质量的变色硅胶干燥带回实验室,存于-40 ℃冰箱内。在样品采集时对地理信息数据进行现场采集,并对群体内的所有采样单株进行胸径测定(表1)。
1.2 实验方法
采用改良的试剂盒法提取闽楠叶片基因组DNA,试剂盒为百泰克生物科技公司的植物材料DNA提取试剂盒,将DNA置于-20 ℃保存备用。
从实验室前期基于楠木转录组数据(EST)开发的SSR引物中筛选出18对多态性较高、稳定性好的SSR引物进行PCR扩增(表2)。引物由金斯瑞生物科技有限公司合成。10 μL SSR-PCR扩增反应体系包括:0.6 μL DNA,0.6μL F-Primer,0.6 μL R-Primer,0.4 μL Mg2+,0.5 μL DNTP,0.04 μLTaqDNA聚合酶,1 μL Buffer,6.26 μL ddH2O。反应程序为:94 ℃ 预变性2.5 min;94 ℃变性30 s,最佳退火温度(Tm)30 s,72 ℃ 延伸30 s,27~30个循环;72 ℃延伸20 min,10 ℃保存。引物筛选过程利用聚丙烯酰胺凝胶电泳(质量分数8%),以检测引物多态性。用于群体检测的PCR产物送于南京擎科生物公司进行毛细管荧光电泳检测。

表2 18对多态性SSR引物及其特征
1.3 数据分析
部分遗传参数通过Popgene 32软件完成,其中包括:观测等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、基因分化系数(Fst)、基因流(Nm)、Shannon多样性指数(I)、近交系数(F)、Hardy-Weinberg平衡卡方检验(HWE)、遗传距离、遗传相似系数。由Powermarker软件完成多态性信息含量(PIC)的测定;利用Fstat软件进行等位基因丰富度(AR)的计算。
通过Structure软件对群体遗传结构进行分析,依据贝叶斯聚类模型采用混合祖先模型(admixture ancestry model)分析群体遗传结构[16]。将群体分类成亚群数量的K值取1~3(不大于群体数),且每个K值重复运行5次,其中,基于马尔可夫链法将迭代次数和运行步数分别设为200 000和1 200 000。最佳的K值通过使用Structure Harvester软件参照Evanno等[17]的方法确定,通过Clumpp进行重复抽样分析,用Distruct 1.1图形化显示结果。分子方差分析(AMOVA)使用GenA-lEx 6.5软件完成,再利用FigTree 1.4.3软件实现聚类分析[17]。
2 结果与分析
2.1 SSR位点多态性
利用18对引物对闽楠3个群体共计88个体进行检测(部分产物电泳图见图1),18个SSR位点的遗传多样性参数见表3。由表3可知,共扩增出183个等位基因,每个位点检测出4~19个等位基因,平均Na为10.167个;有效等位基因数介于2.305(ph_EST-SSR13)和10.103(ph_EST-SSR8)之间,平均有效等位基因数(Ne)为4.972。Shannon多样性指数(I)为1.102~2.523,平均为1.771。在闽楠的物种水平上,平均期望杂合度(He=0.773)大于平均观测杂合度(Ho=0.451),除位点ph_EST-SSR7、ph_EST-SSR12和ph_EST-SSR17外,其余的期望杂合度均高于观测杂合度,说明大部分位点出现了纯合子偏多的现象。18个位点多态性信息含量(PIC值)平均为0.761,PIC值均大于0.5,表现出较高多态性。哈迪温伯格平衡检验结果显示各位点等位基因频率均极显著偏离了平衡(P<0.01)。

表3 18个SSR位点的遗传多样性参数
2.2 群体遗传多样性
闽楠3个天然群体的遗传多样性比较分析结果(表4)表明,群体水平上的观测等位基因数(Na)和有效等位基因数(Ne)均值分别为6.296和3.202。福建政和(ZH)群体的遗传多样性水平最高,观测等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)均最高,分别为6.944、0.521、0.641;福建罗卜岩(LBY)群体的遗传多样性水平最低,其Na、Ho、He分别为5.778、0.399、0.608;福建顺昌群体的有效等位基因数(Ne=3.379)和等位基因丰富度(AR为7.444)较高,但其他多样性指数均低于政和群体。因此,3个闽楠群体遗传多样性水平由大到小依次为:ZH>SC>LBY。总体来看,3个群体的遗传多样性水平差异不大。

表4 闽楠各个群体遗传多样性分析
F为近交系数,当F>0时,F的值越大,表明纯合子的比例越高;F的值越小,表明杂合子的比例越高。3个闽楠群体的F均值为0.280,其中ZH群体F值明显低于LBY和SC群体,说明LBY和SC群体近交程度高于ZH群体。
2.3 群体遗传分化和遗传结构
2.3.1 群体遗传分化
3个闽楠群体的基因分化系数(Fst)为0.197(表3),表明遗传变异虽然主要存在于群体内,还有一定变异(19.70%)存在于闽楠群体间。基于对闽楠群体的分子变异分析(AMOVA)结果(表5)也表明了3个闽楠群体以群体内的变异为主(72%),但有28%的变异存在于群体间。均表明群体间存在较明显的遗传分化。
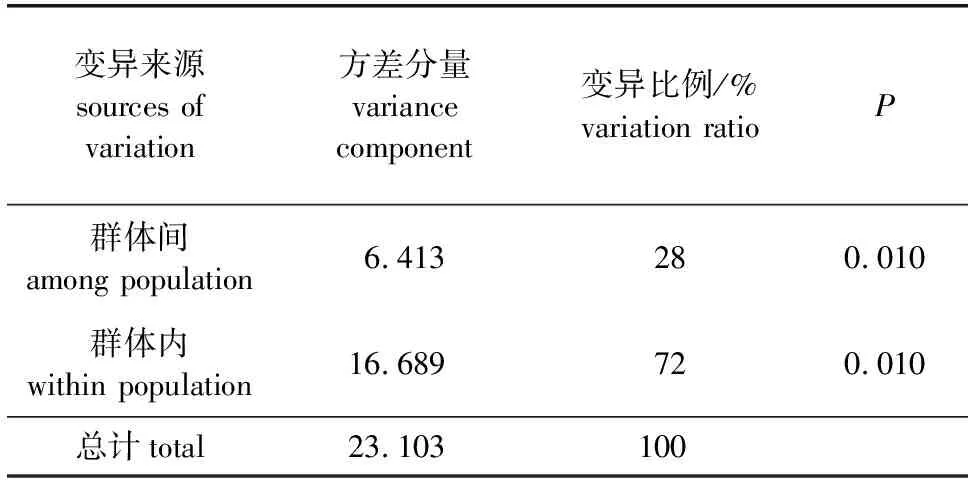
表5 3个闽楠群体的分子方差检验
2.3.2 群体遗传结构
采用Popgene软件计算得出3个闽楠群体Nei’s遗传相似系数和遗传距离。结果显示在3个群体中,遗传相似系数最大的是罗卜岩和顺昌群体间(0.499),说明2个群体亲缘关系较近,而福建政和与罗卜岩群体间的遗传相似系数最小(0.406),两个群体的亲缘关系较远。通过对比发现群体间的遗传距离与遗传相似系数结果一致,最近的遗传距离为罗卜岩和顺昌群体的0.696,最远的遗传距离为罗卜岩和政和群体的0.860。
基于Structure、Clumpp和Distruct 1.1 对3个闽楠群体遗传结构的分析表明(图2),当k=2时最能反映各群体的遗传分化情况。其中,福建政和群体与另外两个群体表现出了较明显的遗传分化,LBY和SC群体的大部分遗传信息表现为相似的祖先来源。
利用FigTree 1.4.3软件对闽楠3个群体88个单株进行聚类分析(图3)。由图3可知,88个个体可以归类为两大类群。第Ⅰ大类群主要包括福建罗卜岩群体LBY 2—LBY 33号,福建顺昌群体SC 1—SC 26号。第Ⅱ大类群包括福建罗卜岩群体LBY-1号,福建政和群体ZH 1—ZH 29号。因此遗传距离较近的LBY和SC被聚为一类,而ZH单独聚成一类,这与Structure聚类分析结果较一致。
3 讨 论
3.1 闽楠群体遗传多样性与近交程度
本研究基于闽楠转录组开发并筛选出的18对SSR引物的平均PIC值为0.761,平均有效等位基因数(Ne)为4.972,明显高于刘丹等[14]报道的10对SSR标记的PIC值(0.27),以及黄雨芹等[15]对237份闽楠分析获得的平均有效等位基因数(2.115),表明所筛选的引物具有高度多态性。本研究分析得到的闽楠3个群体平均期望杂合度(He)为0.629,稍低于栲树(Castanopsisfargesii)(0.654)[18],稍高于辽宁蒙古栎(Quercusmongolica)(0.624)[19],高于鹅掌楸(Liriodendronchinense)(0.558)[20],以及用7个SSR位点标记对237份闽楠材料进行研究的结果(0.442)[15],表明研究的3个闽楠群体具有较高水平的遗传多样性,其中保存较好的群体(政和和顺昌群体)高于人为干扰明显的(罗卜岩)群体。
进一步研究发现3个闽楠物种水平上,平均期望杂合度(He=0.773)明显大于平均观测杂合度(Ho=0.451),除引物ph_EST-SSR7、ph_EST-SSR12和ph_EST-SSR17外,其余的期望杂合度均高于观测杂合度,说明大部分表现出纯合子偏多的现象,可能由于闽楠为小群体的分布模式,容易引起近交,从而导致群体内纯合个体增加。本研究结果显示,3个群体的期望杂合度(He)均较高且较为接近,而各群体的观测杂合度(Ho)却有较大差异。政和(ZH)群体观测杂合度(Ho)为0.521,是3个群体中最高的,其近交程度最低。其原因为该群体是保存最为完好的古树群:群体分布面积大,群体内古树多、含各年龄层次的植株更新旺盛。顺昌(SC)群体也是位于村庄附近的古树群,但群体内植株较少,且集中分布在较小的面积内,导致近交程度较高。罗卜岩(LBY)群体观测杂合度(Ho)为0.399,是3个群体中最低的,其近交程度最高。罗卜岩群体是位于省级自然保护区内,分布面积较大,由于受保护前大多数古树被砍伐,现有群体中多为幼树,仅有少量零散分布的成年植株。因群体内能随机交配的成年大树有限,增加了近交的程度,导致其观测杂合度偏低。
3.2 闽楠群体遗传分化和遗传结构
一般而言,基因分化系数(Fst)为0~0.05,表明物种群体间的遗传分化水平很低;0.05
ZH群体与LBY、SC两个群体之间由于地理距离较远且有鹫峰山脉的阻隔,群体间难以进行基因交流,导致了遗传分化较大,结果使LBY和SC群体聚为一类,ZH单独为一类。
根据福建省内3个代表性闽楠群体的群落现状调查和遗传结构特征研究,笔者认为,由于历史上对闽楠的过度利用,使其资源尤其是大树(胸径30~40 cm及以上)资源急剧减少,较大规模、未受干扰的闽楠群体已很罕见。令人欣慰的是,现有较大的群体往往有较高的遗传多样性,目前保护较好的政和群体和罗卜岩群体内均更新良好,说明闽楠具有足够的更新能力;而罗卜岩群体的现状又表明,一些个体数量多但大树少的闽楠群体可能具有较高遗传多样性,由于近交的增加,可能导致群体的衰退。因此,应创造条件使群体内充分异交,以维持闽楠群体较高的遗传多样性。
