基于GEO数据库对儿童急性淋巴细胞白血病差异基因的筛选和生物信息学分析
2022-09-19虞莉莎张盈盈
虞莉莎,张盈盈
(浙江大学医学院附属金华医院 金华市中心医院检验科,浙江 金华 321000)
急性淋巴细胞白血病(acute lymphoblastic leukemia,ALL)是一种源于骨髓和淋巴细胞前体细胞的恶性克隆性疾病,是儿童最常见的恶性肿瘤之一。ALL的发病机制目前仍不完全清楚,遗传缺陷是ALL发病的重要原因,包括与细胞周期的进程调控相关的基因突变、染色体易位和数目改变等[1]。随着医疗技术的发展,虽然ALL患儿的5年生存率已达90%[2],但是仍然存在容易复发和严重的药物毒性反应等问题[3-5]。因此,寻找新的生物标志物对ALL的早期诊断,改善ALL患儿治疗效果至关重要。
生物信息学是使用生物算法和计算机软件对生物信息进行储存、检索和分析的一门新兴学科,其研究重点主要为基因组学和蛋白质组学2个方面。基因芯片是由大量DNA或寡核苷酸探针密集排列所形成的探针阵列,基本原理是通过杂交来捕捉信息。在肿瘤研究领域,通过生物信息学方法和基因芯片技术系统分析肿瘤相关基因及其调控机制是当前功能基因组学的一个重要研究方法[6]。越来越多的学者通过这一方法来分析各种肿瘤的差异表达基因,并研究这些基因在分子功能、细胞组成以及生物过程中所发挥的作用[7-8]。然而,单一芯片分析的假阳性率较高,因此,本研究从美国国立生物技术信息中心平台基因表达综合数据库(the Gene Expression Omnibus,GEO)下载多个芯片微阵列数据集,从而获取更可靠的健康儿童与ALL患儿之间的差异表达基因,进一步寻找可用于ALL早期诊断的生物标志物。
1 材料和方法
1.1 研究对象
通过GEO数据库检索含有新诊断ALL患儿和健康儿童样本的基因芯片,包括GSE67684、GSE71935、GSE116486、GSE26713、GSE41831、GSE8650和GSE9006(表1)。利用R语言Affy数据包对数据进行表达值背景矫正和表达谱数据归一化预处理,包括原始数据格式的转换,缺失值补充,用分位数法进行数据标准化。

表1 新诊断ALL患儿和健康儿童基因芯片基本信息
1.2 差异表达基因筛选
采用R语言Limma数据包筛选新诊断ALL患儿和健康儿童差异表达基因,筛选标准为P<0.05且|log2FC|≥2,并将探针名称按照制造商提供的批注文件与基因名称进行匹配,如果1个探针组对应多个基因,选择保留第1个,删除其他的冗余信息;如果多个探针对应1个基因,计算每个探针在各样本中的平均值,取平均值最大的探针。绘制差异表达基因火山图。利用网上工具VENNY2.1(https://bioinfogp.cnb.csic.es/tools/venny/index.html)筛选出共同差异表达基因,并用韦恩图进行展示。
1.3 基因功能富集和注释
基于筛选出的共同差异表达基因,通过DAVID在线软件(https://david.ncifcrf.gov),依据基因本体(Gene Ontology,GO)数据库对共同差异表达基因进行生物学功能注释;依据京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路数据库进行共同差异表达基因信号通路的富集。
1.4 蛋白质互作网络构建
利用STRING 11(https://string-db.org/)构建共同差异表达基因编码蛋白的互作网络,并用Cytoscape软件进行可视化分析,并用Cytoscape软件中的CytoHubba插件筛选出其中的关键基因(hub gene)。
2 结果
2.1 差异表达基因筛选结果
通过GPL570平台共筛选出741个差异表达基因,包括303个上调基因和438个下调基因(图1);通过GPL96平台筛选出352个差异表达基因,包括146个上调基因和206个下调基因(图2)。2个平台有交集的差异表达基因共245个(28.9%),包括88个上调基因和157个下调基因。

图1 GPL570平台ALL患儿与健康儿童差异表达基因火山图
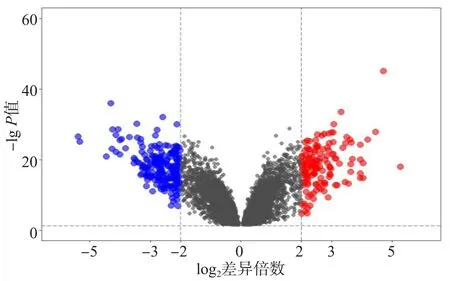
图2 GPL96平台ALL患儿与健康儿童差异表达基因火山图
2.2 差异表达基因功能富集及通路分析
对筛选出的245个共同差异表达基因进行GO和KEGG通路富集分析,结果显示,245个基因主要集中在免疫反应(生物学过程,图3),参与蛋白质结合(分子功能,图4),整合于细胞外空间(细胞组成,图5),富集在造血细胞谱系和细胞周期等信号通路(KEGG通路分析,图6)。

图3 ALL患儿与健康儿童差异表达基因GO富集(生物学过程)

图4 ALL患儿与健康儿童差异表达基因GO富集(分子功能)

图5 ALL患儿与健康儿童差异表达基因GO富集(细胞组成)

图6 ALL患儿与健康儿童差异表达基因KEGG通路分析结果
2.3 蛋白质互作网络的构建和关键基因的筛选
去除游离的蛋白后,共得到由2 0 0个节点(靶点蛋白)和1 514条边(蛋白质互作)构成的蛋白质互作网络(图7),再进一步筛选出蛋白质互作网络中的10个关键基因(hub genes),皆为上调基因,分别为CDK1、TOP2A、TYMS、MCM2、MCM4、TTK、CCNB2、BUB1B、KIF4A和MAD2L1(图8)。

图7 蛋白质互作网络关键基因簇分析
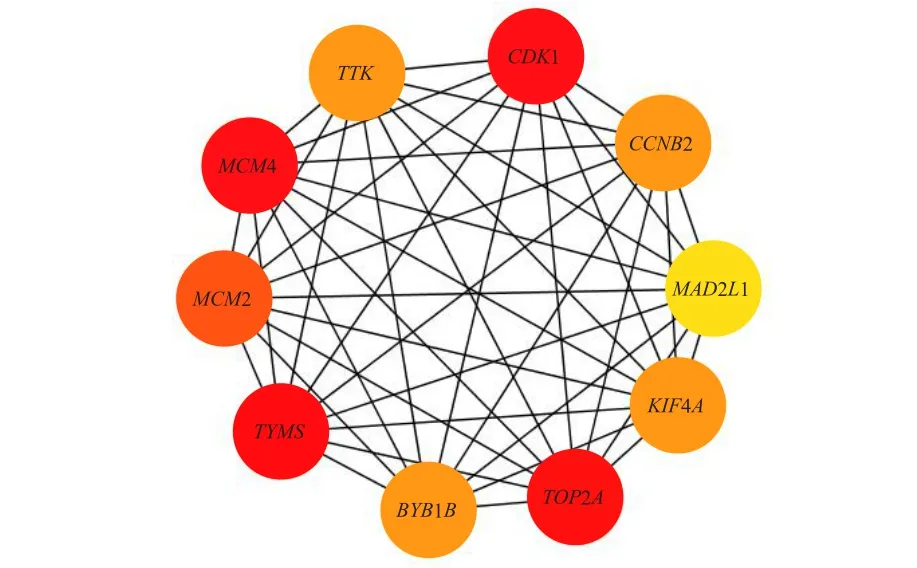
图8 关键基因
3 讨论
本研究从GEO数据库中选取新诊断ALL患儿的基因表达芯片,通过生物信息学分析筛选ALL患儿与健康儿童差异表达基因,进一步对这些差异表达基因进行功能注释和KEGG通路富集分析,结果显示,差异表达基因主要集中在免疫反应(生物学过程),整合在细胞外空间(细胞组成),参与蛋白质结合(分子功能),富集在造血细胞谱系、细胞周期等信号通路(KEGG通路分析)。免疫系统是机体监视和抵御内在和外来抗原入侵的防御系统,任何原因导致的免疫反应受损均可使机体对抗原的监控不力,从而出现一系列病理性变化,甚至发生恶性增殖。血液系统肿瘤患者均存在严重的免疫功能紊乱。细胞周期调控机制被破坏导致的细胞生长失控、分化受阻和凋亡异常存在于几乎所有肿瘤细胞。有研究发现,ALL发病机制与细胞周期进程调控相关的基因突变、染色体易位及数目改变等有关[1]。这些结果与ALL息息相关,提示本研究所筛选的差异表达基因参与了儿童ALL发生、发展的过程。
本研究通过STRING11对ALL患儿与健康儿童差异表达基因进行蛋白质互作网络分析,发现位于中心的关键基因分别为CDK1、TOP2A、TYMS、MCM2、MCM4、TTK、CCNB2、BUBIB、KIF4A和MAD2L1,其中CDK1、MCM2、MCM4、TTK、CCNB2、BUBIB和MAD2L1,皆富集在细胞周期通路上。CDK1是丝氨酸/苏氨酸蛋白激酶家族中的一员,负责控制细胞周期从G1期到S期和从G2期到M期的过渡[9],在多种恶性肿瘤中均可发现CDK1的失调[10]。蒋光洁等[11]发现,在T-ALL中CDK1表达上调。MCM2和MCM4是MCM蛋白家族成员,在DNA复制起始阶段表现出解旋酶活性,并参与细胞周期的控制[12]。高表达的MCM2可作为诊断乳腺癌、结直肠癌、肛门肿瘤等疾病的前瞻性生物标志物。有研究发现,MCM2的高表达可致ALL复发[13]。MCM4的异常表达是多种恶性肿瘤的预后指标[14-16]。BUB1B是纺锤体组装检查点(spindle assembly checkpoint,SAC)蛋白家族的成员,在有丝分裂期间确保染色体适当分离[17],BUB1B的异常表达通常会导致非整倍体和染色体不稳定,导致癌症发病率的增加。有研究发现,BUB1B的过表达与膀胱癌、肝细胞癌以及其他一些癌症的进展和复发有关[18-20]。TTK是丝氨酸/苏氨酸和酪氨酸双特异性蛋白激酶,对有丝分裂检查点和染色体附着的调控至关重要,因此TTK水平的升高可导致中心体增大和染色体不稳定,从而引起肿瘤的发生[21-23]。高水平的TTK对未分化甲状腺癌、三阴乳腺癌和肺癌具有诊断价值[24-26]。CCNB2是细胞周期蛋白家族的一员,可与CCNB1和CDK1形成复合物,调控哺乳动物细胞周期的G2/M期,在有丝分裂的启动中发挥重要作用[27]。有研究结果显示,CCNB2在肺癌、结直肠腺癌和卵巢癌等多种人类癌症中过表达[28-30]。MAD2L1是一个纺锤体检查点的组成部分,主要确保细胞分裂中期染色体的正确分离,其调控异常可导致染色体不稳定和非整倍体形成,乳腺癌、肺癌、肝癌等多种癌症中MAD2L1过表达[31-33]。
本研究筛选出的10个关键基因中,有3个没有富集在某一通路上,但既往研究结果显示其在多种肿瘤中异常表达。TOP2A是TOP2家族中的一员,在有丝分裂过程中对DNA复制、染色体凝聚等发挥重要作用,并在转录起始时发挥重要作用,其表达与多种癌症有关[34]。TYMS基因编码参与DNA复制和修复的胸苷酸合成酶[35]。高水平的TYMS已在乳腺癌、非小细胞肺癌和前列腺癌等多种癌症中被报道[36-38]。KIF4A是驱动蛋白超家族中的一员,参与有丝分裂过程中纺锤体的形成和变化、染色体的浓度和排列以及胞质的分裂[39]。有研究发现,KIF4A参与了DNA损伤修复,其异常表达可能影响同源重组酶RadS1及其调控因子BRCA2的表达,导致受损DNA修复失败[40-41]。DNA损伤可导致细胞异常增殖和分化,最终促进肿瘤的形成;KIF4A在肝癌、肺癌等许多癌症中过表达[42-43]。
本研究筛选出的10 个关键基因虽然在其他疾病中有相关研究,但在ALL发生、发展中所发挥的作用,尚未见报道,本研究的成果一定程度上能为儿童ALL发生、发展研究提供新的思路。
综上所述,本研究采用生物信息学方法分析已有的ALL基因芯片数据,筛选出潜在的ALL中表达显著的基因,通过建立蛋白质互作网络,筛选出10个关键基因,可能成为ALL辅助诊断的新的标志物。但本研究仅涉及生物信息学分析,筛选出的关键基因与ALL的相关性及相关发病机制仍需在临床样本中进行验证。
