基于NODE-UNet++和标记分水岭算法的红细胞图像分割
2022-09-07荣亚琪张丽娟崔金利盖梦野
荣亚琪,张丽娟,崔金利,苏 伟,盖梦野
(1.吉林农业大学 信息技术学院,吉林 长春 130118;2.长春工业大学 计算机科学与工程学院,吉林 长春 130012;3.长春中医药大学附属医院 医药影像科,吉林 长春 130000;4.长春中医药大学 医药信息学院,吉林 长春 130117)
1 引 言
红细胞是血液中最丰富的血细胞,其主要作用是运输氧气和一部分二氧化碳[1]。红细胞图像领域的研究大多集中在红细胞计数,通过检查红细胞的数量、形态来鉴别和诊断某些疾病。然而,由于图像中的红细胞通常会出现重叠和粘连等现象,因此很难对后续红细胞进行计数和识别。以往血液学家通常会避免选择重叠红细胞的区域,但在自动分割情况下,很难避免这种现象。自动分割方法必须处理重叠、粘连红细胞以产生真实的分割情形。为了更好地处理重叠、粘连细胞,近几十年来,国内外研究学者提出了许多细胞图像分割算法,这些算法主要分为传统算法和深度学习算法两大类。传统分割算法主要包括图论分割法[2-4]、水平集算法[5]、分水岭算法[6]、区域生长法等,深度学习算法主要有卷积神经网络(Convolutional Neural Networks,CNN)[7]、全卷积神经网络(Fully Convolutional Networks,FCN)[8]、U-Net神经网络[9]、U-Net++神经网络[10-11]等。
在传统细胞图像分割中,王卫星等人[12]利用图论与数学形态学结合的方法分割粘连细胞。该算法虽然简单,易于实现,但对于形状不规则、大小不均匀的细胞而言,误差率较高,分割效果不理想。Wei等人[13]结合区域生长算法和颜色空间变换分割重叠细胞。该算法分割平均准确率较高,但是当红细胞中没有明显的内部边缘时,准确率会降低。Miao等人[14]提出一种标记控制的分水岭算法用于分割红细胞,首先提取细胞的二值图像,然后获得前景、背景标记,最后对重建的梯度图像应用分水岭算法获得分割结果。该算法操作简单,计算时间少,但中等的图像质量会对分割算法准确度产生影响。由于噪声、图像质量等问题,一些传统分割算法不能表现出很好的鲁棒性,而深度学习算法利用大量的样本数据学习细胞图像特征,其分割准确率高于传统图像分割算法,因此可将传统算法与深度学习算法相结合实现语义分割[15-18]。Huang等人[19]提出将全卷积神经网络U-Net与改进的水平集方法相结合分割重叠细胞,该算法首先采用U-Net检测细胞核和细胞质,再利用改进的水平集能量函数结合距离图和新的形状先验项提取细胞轮廓以提高算法分割精度。Kowal等人[20]提出一种结合卷积神经网络和种子分水岭算法来分割细胞图像,使用CNN分类器预分割细胞核,种子分水岭处理重叠核。该算法优于经典的分水岭算法,分割结果较好,但计算时间复杂度较大。
本文综合U-Net++网络和神经常微分方程(Neural Ordinary Differential Equations,NODE)[21]各自的优点,提出了一种新的深度学习网络用于红细胞的初步分割,再利用标记分水岭(Marker Watershed,MW)算法提高血液涂片图像中红细胞的分割精度。本文主要贡献如下:
(1)结合U-Net++和NODE提出了一种新的语义分割体系结构,我们将它称之为NODEUNet++,它使用改进的编码器-解码器结构对红细胞初步分割。
(2)初步分割得到的概率灰度图输入到MW算法中得到红细胞最终分割图像。
实验结果表明,本文算法在分割精度方面比其他几种算法取得了较好的效果。
2 数据预处理
实验所采用的数据库由医学图像和信号处理研究中心以及伊斯法罕医学科学大学病理系提供[22]。数据集中包含148张清晰的血涂片图像,图像中含有少量的白细胞和大量红细胞。由于原始图像尺寸较大,图像分辨率为775×519,因此选择256×256像素大小的图像作为训练样本,将其中48张图像作为测试集,100张作为训练集和验证集。
LabelMe是麻省理工学院的计算机科学和人工智能实验室研发的在线注释工具,可用于执行图像标注[23]。我们将原始图像中的细胞经过LabelMe标注之后生成json文件,在标注时,将细胞与数字一一对应,红细胞对应数字1,背景为0。对json文件进行解析并转换分别得到它的mask图像和标签图像。图1是标注和解析的实例。

图1 图像标注和解析。(a)原始图像;(b)Mask图像;(c)标签图像。Fig.1 Image annotation and analysis.(a)Original image;(b)Mask image;(c)Label image.
由于训练样本较少,需要对红细胞图像执行数据增强以扩大样本集,主要是运用随机旋转、水平和竖直翻转等操作对100张训练样本进行变换,扩充为500张,在增加训练数据的同时也提高了模型泛化能力,防止网络模型过拟合。对扩充后的图像按照8∶2的比例分成训练样本和验证样本用于训练和验证NODE-UNet++网络模型,48张测试集图像用于评估训练好的网络模型。图2是数据增强的实例。

图2 数据增强实例Fig.2 Data enhancement
3 研究方法
为了提高红细胞分割准确度,我们提出了一种NODE-UNet++和MW算法相结合的红细胞分割模型。算法主要分为以下3个部分:(1)对血液涂片图像进行预处理、图像裁剪、标注数据集、数据增强;(2)利用深度卷积网络NODEUNet++对红细胞图像进行初步分割,使用标注好的训练集对模型进行训练,用训练好的模型对图像分割,得到概率灰度图;(3)采用MW算法进一步优化初步分割结果,得到红细胞的最终分割结果图。整个算法流程如图3所示。

图3 算法流程图Fig.3 Flow chart of our method for red blood cell image segmentation
3.1 神经常微分方程
常微分方程(Ordinary Differential Equations,ODE)是包含一个自变量t,未知函数h(t)和未知函数导数的等式,可表示为:

在数学中,我们通常的做法是解出未知函数h(t),然而在工程应用中更常用数值解,即不需要解出未知函数的通解,只需根据已知值逐渐趋近未知值即可。欧拉法是一种古老的求解微分方程数值的方法,即

但Δt每走一步都会产生误差,并且误差会不断积累。因此现在工程中针对求解微分方程提出一种适应性工具——ODE solver求解器,它不像欧拉法移动固定步长,相反它根据给定的误差容忍度选择合适的步长逼近未知值。
残差网络[24]是神经常微分方程的一个特殊形式,一个残差块包括直接映射部分和残差部分(图4),可表示为

图4 残差块结构图Fig.4 Structure of residual block

其中ht是t层的输出向量;Wt是t层的网络参数,F是Wt参数化后的神经网络,在残差网络中,F一般是由卷积操作构成。若Δt=1,那么残差网络可看作离散化的欧拉法。当Δt→0时,相当于用更多的层以更小的步骤一步步趋近,在这种极限状态下就变成了由神经网络指定的常微分方程,即神经常微分方程网络,ODE块结构如图5所示。


图5 ODE结构图Fig.5 Structure of ODE block
NODE是一个连续的神经网络,其可以用常微分方程求解器ODE solver求解。训练连续神经网络时,正向传递操作相对简单,难点在于ODE solver反向传播,其具体操作可在文献[21]中找到。
3.2 NODE-UNet++网络模型
所提出的初步分割网络模型是在U-Net++的基础上使用神经常微分方程,该网络架构如图6所示。该架构与U-Net++的区别主要有以下两点:(1)在编码器阶段,使用卷积块和ODE块一同来提取浅层特征图。(2)U-Net++在编码器和解码器之间加入了一系列的卷积层,并在各个卷积层之间加入了短连接。在U-Net网络特征提取过程中,使用长连接来组合编码器模块生成的浅层特征图和解码器模块生成的深层特征图,然而U-Net很难融合浅层和深层特征图,因此U-Net++使用跳跃连接来降低浅层特征图和深层特征图的语义差别。受U-Net++网络的影响,NODE-UNet++使用密集卷积块的跳跃连接来弥合编码器和解码器特征图之间的语义嫌隙。
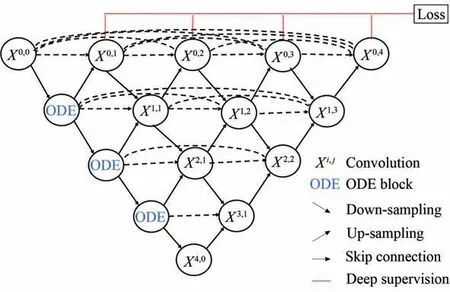
图6 NODE-UNet++网络结构图Fig.6 Architecture of NODE-UNet++
NODE-UNet++与U-Net++一样使用了深度监督,其在每个输出节点(X0,1,X0,2,X0,3,,X0,4)加入了1×1的卷积层和sigmoid函数。sigmoid函数取值范围为[0,1],它将输出值z映射到[0,1]区间,如式(5)所示:
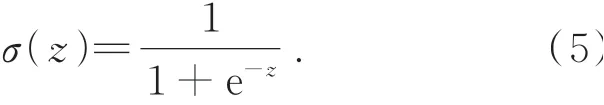
损失函数用来评估模型的预测值和真实值不一致的程度。在神经网络训练过程中,函数损失值越小,说明模型预测值越接近真实值,模型的鲁棒性越好。红细胞图像分割是二分类问题,因此,NODE-UNet++网络损失函数采用二元交叉熵损失,定义为:
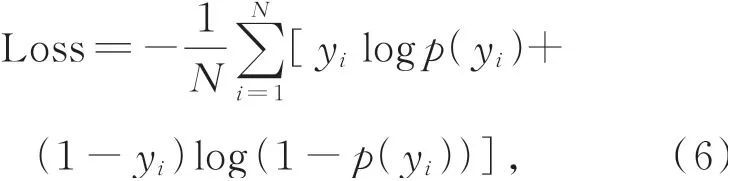
其中,N表示图像像素总数;y代表训练标签,当y为1或0时,表示像素i属于红细胞或非红细胞;p(y)代表输出y标签的概率。
3.3 标记分水岭(MW)算法
分水岭经典算法是Vincent等人于1991年提出,它是一种分割图像区域的方法,基本思想是将图像视为拓扑地形。图像中像素灰度值为0的点代表地面,像素灰度值最大的点代表地形的最高点,其中灰度值较大的点连接的线构成分水岭。由于红细胞图像存在不止一个极值点,直接对红细胞图像应用分水岭算法往往会导致过分割[25-26]。MW算法是在传统分水岭算法的基础上做出了改进,通过寻找一些前景标记和背景标记来更好地分割细胞图像,因此我们采用MW算法来分割红细胞。先提取红细胞梯度图像,对梯度图像进行处理,做好前景标记和背景标记之后,对梯度图像进行修正,最后应用分水岭算法分割红细胞。
首 先 使 用Sobel边 缘 算 子[27]来 获 得 红 细胞的梯度图像,然后运用形态学重建对红细胞梯度图像进行处理。通过Matlab中的strel函数创建一个半径为3的平面圆盘形的结构元素,在相同的结构元素下,利用imerode函数和imreconstruct函数得到基于开的重建图像。在此基础上,对重建的开闭图像利用imregionalmax函数计算局部极大值,腐蚀后获得前景标记。之后,对二值图像计算距离变换后再进行分水岭变换,得到的分水岭脊线图即为背景标记。利用imimposemin函数对红细胞梯度图像进行修正,则局部极小值只存在于标记位置。前景标记对应一个细胞,背景标记分布在图像背景上,形成一对一的关系,可以有效抑制过分割现象。最后对修正图像执行标记分水岭算法以获得红细胞最终的分割结果。
4 实验结果与分析
4.1 实验结果
实验利用大量256×256像素的图像测试和验证了所提方法的有效性和鲁棒性。将红细胞分割算法分为两个步骤。初步分割实验环境是Ubuntu16.04 LTS 64位操作系统,使用Keras库搭建神经网络,后端采用Tensorflow,调用Adam优化器对网络学习优化,学习率为1e-4,将训练集和验证集输入网络中训练100次,训练过程中每轮(epoch)的训练损失函数值和精度值、验证损失函数值和精度值如图7所示。图8是使用NODE-UNet++进行红细胞初步分割的3个案例。再次分割实验采用Matlab实现。图9是使用MW算法对红细胞再次分割的实验结果。

图7 NODE-UNet++训练过程中的损失函数值和精度Fig.7 Loss values and accuracy in the training process of NODE-UNet++

图8 NODE-UNet++分割效果图。(a)原始图像;(b)标签图像;(c)初始分割图像。Fig.8 Segmentation images of NODE-UNet++.(a)Original images;(b)Label images;(c)Presegmentation images.

图9 MW算法分割效果图。(a)前景标记图;(b)背景标记图;(c)最终结果图;(d)修正后的梯度图像;(e)分割的红细胞。Fig.9 Segmentation images of MW algorithm.(a)Foreground marker image;(b)Background marker image;(c)Final image;(d)Reconstruction map of gradient topographic;(e)Segmented RBCs.
4.2 对比分析
为了进一步验证本文算法,将文献[14]、U-Net++和MW算法相结合(MW-UNet++)的算法与本文算法进行比较分析。文献[14]是采用标记控制分水岭算法来分割红细胞,MWUNet++是先使用UNet++对红细胞进行初步分割,再采用标记分水岭算法分割粘连红细胞。实验结果如图10所示。由图10可知,NODEUNet++模型算法能较好地提取红细胞轮廓,但对于红细胞粘连、重叠等区域存在严重的欠分割现象;MW算法能很好地解决红细胞这一问题,但是它对微弱边缘等其他干扰因素过于敏感,往往会产生漏提取。综合两种方法的优点,NODE-UNet++与MW算法相结合能够提取出大部分大小不同、粘连、重叠的红细胞。

图10 3种算法分割效果图。(a)原始图像;(b)标签图像;(c)文献[14]算法;(d)MW-UNet++算法;(e)本文算法。Fig.10 Red blood cell segmentation results from three different algorithms.(a)Original image;(b)Label image;(c)Results using the algorithm in[14];(d)Results using MW-UNet++algorithm;(e)Results using our algorithm.
为了更好地评估本文算法的分割效果,对实验结果进行定性定量分析。目前,图像分割中通常使用一些评估方法来衡量算法的精度,本文使用Dice系数(Dice Similarly Coefficient,DSC)、平均像素准确率(Mean Pixel Accuracy,MPA)和平均交并比(Mean Intersection over Union,MIoU)3个指标来评估算法分割结果,其计算都是建立在混淆矩阵的基础上,混淆矩阵如表1所示。

表1 混淆矩阵Tab.1 Confusion matrix
Dice系数是一种集合相似度度量指标,通常用于计算两个样本的相似度,取值范围为0~1,越接近1说明分割结果越好。Dice系数公式如式(7)所示:

像素准确率表示所有预测正确的像素数占像素总数的比例,在本文中是指被正确预测为红细胞的像素数和被正确预测为非红细胞的像素数相对于像素总数的百分比,定义为:

平均像素准确率是针对每一类别的像素准确率取均值,在本文中指红细胞像素准确率和非红细胞像素准确率的平均值,定义为:

交并比是语义分割中常用的指标之一,含义是模型对某一类别的预测值和真实值之间交集和并集的比值,定义为:

平均交并比针对每一类别的交并比取均值,在本文中指红细胞和非红细胞交并比的平均值,定义为:
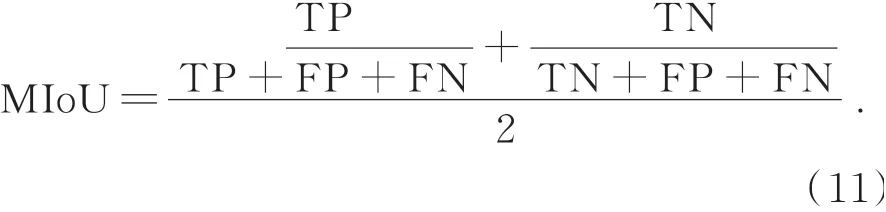
表2显示了3种算法对测试集48张图片的定量定性结果,图11为实验结果DSC、MPA、MIoU指标的箱型图。观察表2和图11,使用本文算法分割效果显著提升,Dice系数比文献[14]算法和MW-UNet++算法分别提高1.12%、0.46%;MPA比文献[14]算法和MW-UNet++算法分别提高2.82%、0.84%;MIoU比文献[14]算法和MWUNet++算法分别提高2.1%、0.79%。实验结果表明,使用本文算法分割红细胞效果更精确。

表2 3种方法的定量结果Tab.2 Quantitative results of three methods

图11 实验结果指标图Fig.11 Box-plots of three algorithms
5 结 论
针对红细胞粘连、重叠等问题,本文提出了一种基于NODE-UNet++和MW算法的单个红细胞提取方法。具体来说,NODE-UNet++网络设计了一个具有卷积块和ODE块的编码器,用来提取图像特征,使用跳跃连接来减少浅层和深层特征图的语义差距。实验结果表明,NODE-UNet++算法能够精确地提取出单个红细胞,Dice系数达96.89%,平均像素精确率达98.97%,平均交并比达96.33%,明显优于单独使用MW算法或者MW-UNet++算法提取的结果。本文算法还需要进一步改进,接下来将对NODE-UNet++网络模型进行改进,在不增加网络规模的前提下,提高网络的预测精度,改善红细胞分割算法的性能。
