拟柱孢藻N8全基因组测序及磷吸收转运通路的比较分析
2022-08-30陈志江阮紫曦肖利娟韩博平雷腊梅
陈志江 阮紫曦 程 楠 肖利娟, 彭 亮, 韩博平, 雷腊梅,
(1. 暨南大学生命科学技术学院生态学系, 广州 510632; 2. 广东省水库蓝藻水华防治中心, 广州 510632)
拉氏拟柱孢藻(Cylindrospermopsis raciborskii,简称拟柱孢藻)为念珠藻科, 柱孢藻属, 最近有研究者建议将尖头藻和拟柱孢藻合并, 称该藻为拉氏尖头藻(Raphidiopsis raciborskii)[1]。拟柱孢藻是一种具有固氮能力的丝状水华蓝藻, 对氮、磷营养盐的利用效率高, 对温度和光照波动的适应性强, 目前已广泛分布世界各地的淡水水体中, 可在热带地区的湖泊和水库中常年形成水华, 在亚热带和温带水体形成季节性水华[2,3]。拟柱孢藻还可产生拟柱孢藻毒素(Cylindrospermopsin, CYN)和石房蛤毒素(Saxitoxin, STX)等蓝藻毒素, 严重危害人类健康和淡水生态系统安全[4—6]。
全基因组测序是对未知基因组序列的物种进行个体基因组测序的一项技术, 1977年第一代测序技术诞生, 至21世纪以Roche公司的454技术、Illumina公司的Solexa技术和ABI公司的SOLiD技术为代表的第二代高通量测序的广泛应用, 极大推动了不同物种基因组研究进展[7], 随后的第三代技术向序列读长长, 精度高方向发展[8]。全基因组测序可揭示基因组的复杂性和多样性, 对了解一个物种的分子进化、基因组成和基因调控等方面具有重要的意义[9]。目前三代测序技术已在水华蓝藻中得到广泛应用, 如2016年发布的片状微囊藻(Microcystis panniformis)FACHB-1757基因组数据[10]及2018年发布的铜绿微囊藻(Microcystis aeruginosa)NIES-2481基因组数据[11], 都使用了三代测序技术。研究者们利用测序技术对蓝藻基因组的解析促进了他们对该物种的生态学、生理学和毒素合成机制等相关特性的了解, 同时基于比较基因组的分析进一步揭示了它们的全球地理分布与进化起源[12,13]。
相比较而言, 拟柱孢藻的基因组研究相对较晚。2009年, 美国国立生物技术信息中心(NCBI)公布了最早完成全基因组测序的拟柱孢藻藻株CS-505(C. raciborskiiCS-505), 该藻株的基因组相对较小, 为3.89 Mb, 在当时已完成全基因组测序的丝状蓝藻中, 拟柱孢藻是除尖头藻外基因组最小的丝状蓝藻[14]。Stucken等[14]通过基因组分析发现拟柱孢藻CS-505基因组存在水平基因转移, 并探讨了其丝体形成、异形胞发育及固氮能力的分子基础。Sinha等[15]于2014年通过比较基因组发现澳大利亚产毒与非产毒拟柱孢藻的最大区别是拟柱孢藻毒素合成酶基因簇是否存在。随着拟柱孢藻在全球的快速扩张, 人们对更多来源不同的拟柱孢藻开展了全基因组测序, 如2018年Willis等[16]和Abreu等[17]分别进行了同一地区的9株拟柱孢藻和不同地区的8株拟柱孢藻的比较基因组分析, 以期为该藻灵活的生理生态策略、个体发育及逆境应答等研究提供参考[16,17]。
磷是蓝藻生长必需的营养元素, 分无机磷、有机磷两大类, 蓝藻基因组中存在多种用于吸收不同磷源的机制[18], 其中低亲和力无机磷转运系统存在于部分蓝藻中, 以适应在高磷环境下吸收无机磷,高亲和力无机磷转运系统在蓝藻中普遍存在, 可在低磷环境下吸收无机磷。此外, 蓝藻中还存在双组分调节系统用于调节磷代谢、有机磷酸盐转运复合体介导磷酸酯的转运、C-P裂解酶裂解磷酸酯和碱性磷酸酶特异性水解磷酸单酯[18—22], 蓝藻的磷调控机制高度多样化, 以适应不同的生活环境[18]。拟柱孢藻拥有快速吸收和储存无机磷的能力, 不但能在高磷环境中生长良好, 还能适应低磷环境, 在无机磷浓度极低的环境中占优势[6,23,24], 已有研究发现在拟柱孢藻CS-505和CS-506中存在以上完整的磷调控机制[25]。目前关于拟柱孢藻磷代谢基因排列特征的描述较为匮乏, 从基因组水平上分析拟柱孢藻磷代谢途径基因将加深对拟柱孢藻生存优势形成的认识, 进一步了解磷代谢以及磷在基因型选择和进化中发挥的作用[18]。
迄今为止, 美国国立生物技术信息中心(NCBI)数据库已有27株拟柱孢藻全基因组测序信息, 其中一半以上的藻株来自澳大利亚, 目前尚未有中国地区拟柱孢藻全基因组数据的发布。近年来, 拟柱孢藻在广东省大部分水库常年存在并取代微囊藻成为危害该地区水库供水安全的首要蓝藻, 在广东省江门市镇海水库中常年优势度可达93.5%, 目前已有的研究多关注于环境因子对拟柱孢藻优势形成的影响[21,22]。本文以从镇海水库中分离的拟柱孢藻N8为材料, 进行测序及基因组分析, 并针对其亲缘关系、毒素合成基因和磷吸收转运基因进行了初步探索。本研究的开展有助于了解华南地区拟柱孢藻的基因组特性, 丰富拟柱孢藻基因组数据库,从分子水平上加深人们对拟柱孢藻生态优势形成的认识, 为有效防控拟柱孢藻水华提供理论依据。
1 材料与方法
1.1 拟柱孢藻N8的来源与培养
拟柱孢藻N8分离自广东省江门市镇海水库(22°34′7′′N、112°32′58′′E), 其藻丝体笔直不弯曲,可形成端生的长圆形或卵圆形异形胞[21]。藻株置于光照强度为35 μmol/(m2·s)、温度为(25.5±1)℃、光暗周期比为12h﹕12h的培养箱中进行培养, 使用的培养基为BG11[25]。
1.2 拟柱孢藻N8基因组DNA的提取
使用植物基因组DNA提取试剂盒(天根生化科技有限公司)按照制造商的说明从样本中提取高质量的基因组DNA。使用Nanodrop One核酸分析仪测定DNA样品260和280 nm处的光吸收值, 根据A260/A280的比值确定DNA纯度(1.8 使用PacBio RS测序系统的SMRT(Single Molecule Real-Time)[26]技术进行测序, 利用PacBio®SMRTbellTMTemplate Prep Kit(PacBio, 美国)构建文库, 在PacBio RS II平台上进行测序反应。用Pac-Bio SMRT Analysis 5.1[23]软件进行质控并组装成一条环状的基因组序列。以上工作委托北京诺禾致源生物科技有限公司完成。 使用RepeatMasker 4.1.2[27]、RNAmmer 1.2[28]、tRNA-scan-SE 2.0[29]和GeneMarkS 4.28[30]分别进行重复序列、非编码RNA、tRNA和编码基因的预测;后将编码基因与COG数据库[31]、SwissProt蛋白库[32]、GO数据库[33]和KEGG数据库[34]进行比对, 使用Circos 0.69软件[35]绘制全基因组图谱。 我们挑选了24株来自不同产地及不同产毒特性拟柱孢藻全基因组单拷贝基因进行亲缘关系分析, 手动将同一株藻的全基因组单拷贝基因序列进行排序和串联, 采用MEGA 6.0[36]对25株藻的全基因组单拷贝基因进行序列比对, 以串联序列为材料利用iqtree软件包构建系统发育树, 程序中的ModelFinder模块自动计算出最大似然法(ML)的最佳拟合进化模型为GTR+F+G4模型, 系统树各分支的置信度由重复抽样法(Bootstrap)1000次重复检测,同时使用MEGA 6.0构建最大似然(ML)进化树, 以确定进化树的可靠性, 进化树的编辑器使用iTOL的在线版本(https://itol.embl.de)。 通过antiSMASH 6.0软件(微生物次生代谢物合成基因簇数据库)[37], 结合注释信息搜索对应于次生代谢物非核糖体肽合成酶(NRPS)、聚酮化合物合酶(PKS)产生的CYN合成酶基因簇和STX合成酶基因簇。 根据KEGG代谢通路注释结果结合基因的序列相似性和位置相关性, 找到对应的核苷酸/蛋白质序列, 放到NCBI上的Nucleotice/Protein Blast上比对, 从而确定基因组中磷吸收转运基因, 包括双组分调节系统, 低亲和力、高亲和力无机磷转运系统,有机磷酸盐转运复合体, C-P裂解酶和碱性磷酸酶基因, 绘制基因簇排列图。 经过我们的分析, 挑选出8株拟柱孢藻进行磷吸收转运基因分析, 8株藻株分别代表了三大地区(欧亚地区、南美地区和澳大利亚地区)和不同产毒特性(产CYN、STX和非产毒)的拟柱孢藻。 拟柱孢藻N8的基因组覆盖率为121, 平均测序质量为0.85, Contig N50值为3865242 bp, 基因组大小为3857174 bp, 呈现一条环状的染色体, GC含量为40.13%, 注释得到的基因总数为3598, 结构RNA基因为51个, 其中tRNA基因42个, 5S-16S-23S rRNA基因各3个(图 1)。该藻株的全基因组序列已上传至NCBI数据库, 索引编号为GCA_018139025.1。 图1 拟柱孢藻N8的全基因组图谱Fig. 1 The whole genome map of C. raciborskii N8 COG注释(图 2a): 在拟柱孢藻N8功能分布的基因数目中, 细胞壁/膜/包膜合成相关基因(M)有210个, 所占比例最高, 为5.92%; 而染色质结构与动力学相关基因(B)只有1个, 所占比例最低, 为0.03%。 KEGG注释(图 2b): KEGG中PATHWAY数据库注释到拟柱孢藻N8涉及的基因通路有142条, 其中完整的或者有关键酶可以完成代谢的通路有102条, 新陈代谢系统部分存在的完整通路最多, 有76条。 GO注释(图 2c): 拟柱孢藻N8在分子功能类注释到了2814个相关基因, 其中所占比例较大的有黏合物相关基因1135个、催化活性相关基因1267个及运输活性相关基因137个, 占该组分基因的90.23%; 在细胞学组件分类部分注释到了2190个相关基因, 其中所占比例较大的有细胞器相关基因177个、高分子复合物相关基因236个、复杂大分子及细胞和细胞元件相关基因各783个, 占该组分注释基因的90.37%; 在生物学途径类注释到了4869个相关基因, 其中占比较大的有代谢过程相关基因1417个; 细胞过程相关基因1332个、定位相关基因365个、形成定位相关基因358个、生物调节相关基因329个、生物过程监管相关基因、应激反应相关基因220个和细胞成分组织或生物起源相关基因122个, 占该组分注释基因的85.01%。 图2 拟柱孢藻N8的COG (a)、KEGG (b)和GO (c)数据库注释图Fig. 2 COG (a), KEGG (b) and GO (c) database notes of C. raciborskii N8 在NCBI数据库中搜寻了27株拟柱孢藻的全基因组序列(其中CS-505和CS505为同一藻株, 采用了不同测序方法, 部分藻株数据未完善故未采纳), 表 1显示了27株拟柱孢藻的基因组基本信息。由表 1可知, 当前数据库中完成测序的藻株主要来自澳大利亚和巴西, 分别有13株和6株, 乌拉圭和美国各有2株, 新加坡、韩国、荷兰各有1株。不同来源的拟柱孢藻基因组的GC含量稳定, 维持在40.1 %左右,大小在2.74—4.16 Mb, 其中美国藻株LB2897的基因组最小, 约为2.749 Mb, 澳大利亚藻株CS505基因组最大, 约为4.159 Mb。在这些藻株中(除KL1藻株产毒未知外), 仅8株藻株不产毒, 18株藻株为产毒株, 其中来自南美的巴西和乌拉圭的拟柱孢藻产毒素STX, 来自澳大利亚(11株)和新加坡(1株)的拟柱孢藻产毒素CYN及其异构体。采用的测序技术以第二代测序技术为主, 分为Roche 454技术、Illumina Solexa技术和ABI SOLID技术, 拟柱孢藻N8、CS505和GIHE 2018采用第三代测序技术—SMRT技术。 表1 NCBI数据库中已公布的拟柱孢藻藻株基因组基本信息Tab. 1 Basic information on the genome of C. raciborskii strains downloaded from NCBI 基于拟柱孢藻的全基因组单拷贝基因构建了拟柱孢藻N8的亲缘关系树, 采用最大似然法(ML)。由图 3可知24株拟柱孢藻有3大分支, 来自南美地区的拟柱孢藻属第Ⅰ分支, 来自亚欧地区的拟柱孢藻属第Ⅱ分支, 来自澳大利亚地区的拟柱孢藻属第Ⅲ分支。 图3 基于全基因组单拷贝基因构建的系统进化树(ML)Fig. 3 Phylogenetic tree based on whole genome single copy gene construction (ML) 拟柱孢藻N8属第Ⅱ分支, 与来自韩国的拟柱孢藻GIHE 2018的亲缘关系最近。 在拟柱孢藻N8的基因组中未发现CYN合成酶基因簇和STX合成酶基因簇, 推断拟柱孢藻N8不能产生毒素CYN和STX。 对N8和其他7株来自不同地区的拟柱孢藻进行比较分析[Cr2010(荷兰), GIHE 2018(韩国), CS505、CO3和SO1(澳大利亚), CENA302和MVCC19(南美洲)], 结果表明不同地区的拟柱孢藻, 其磷代谢基因拷贝数目不同, 磷调控多样化。研究的8株拟柱孢藻基因组中都存在双组分调控系统phoB和phoR, 低亲和力无机磷转运系统与高亲和力转运系统。多数藻株有2个有机磷酸盐转运系统phnD-C-E, 来自巴西的拟柱孢藻CENA302只有1个phnD, 拟柱孢藻N8比其他藻株多1个转录调节蛋白-phnF和1个有机磷酸盐代谢蛋白(金属依赖性)-phnM。8株藻中大多有2个或2个以上phoA碱性磷酸酶基因, 来自荷兰的拟柱孢藻Cr2010仅有1个phoA基因拷贝(表 2)。 表2 八株拟柱孢藻磷吸收、调节、转运、代谢基因及拷贝数Tab. 2 Phosphorus absorption, regulation, transport, metabolism genes and copies of eight C. raciborskii strains 如图 4和图 5所示, 8株拟柱孢藻的高亲和力无机磷转运系统按照pstS-C-A-B顺序排列的,sphX在拟柱孢藻的位置不定, 可位于pstS-C-A-B的上游或下游, 拟柱孢藻的双组分调节系统phoB-phoR形成簇, 拟柱孢藻的2个有机磷酸盐转运操纵子phnC-DE, 两个转运操纵子的方向可同可不同, C-P 裂解酶基因按phnFHGIJKLM排列, 除拟柱孢藻N8和拟柱孢藻GIHE 2018外, 其余藻株的phnFH与phn-GIJKLM簇距离较远。 图4 8株拟柱孢藻的双组分调节系统和高亲和力无机磷转运系统Fig. 4 Two-component regulatory system and high affinity inorganic phosphorus transport system of eight strains of C. raciborskii 图5 8株拟柱孢藻的有机磷酸盐转运复合体和C-P裂解酶基因Fig. 5 Organic phosphonate transport complex and C-P lyase gene of eight strains of C. raciborskii 自Stucken等[14]发布第一个拟柱孢藻全基因组信息后, 近10年来陆续有不同来源的拟柱孢藻完成测序。已公布全基因组序列的拟柱孢藻主要来自澳洲和南美洲, 2019年, 2020年发表了荷兰藻株Cr2010[43]、韩国藻株GIHE2018[42]、美国藻株LB2897与KL1[45,47], 目前NCBI数据库上还没有中国地区拟柱孢藻的全基因组信息, 本研究测序的拟柱孢藻N8将成为中国地区首株具有全基因组信息的藻株。 拟柱孢藻N8与NCBI上已公布的其他国家的藻株在基因组大小、GC含量和基因数量等基本特征较为一致, NBCI上已发布的拟柱孢藻大都采用第二代测序法进行测序, 仅CS505、GIHE 2018藻株采用了三代测序技术。二代测序技术具有高效率、高通量和高精度的特点, 应用广泛, 但拼接的基因组受序列读长的限制, 其结果的准确性依赖于较高的测序覆盖度和准确的序列拼接技术[48]。拟柱孢藻N8采用第三代测序技术, 该技术改进了序列组装, 序列读长长, 精度高, 在测序的同时能够获取全基因组甲基化修饰信息, 其测序结果也相对完善[8]。N8的contig N50值为3865242 bp, 这在通过Pac-Bio平台测序的已公布的拟柱孢藻基因组中, 拟柱孢藻N8的N50值仅次于CS505的4011384 bp, 测序得到N8的基因组大小为3.857 Mb, 与澳大利亚藻株S01、S14等基因组大小相近, 其GC含量稳定, 维持在40.1 %左右。拟柱孢藻N8的编码基因有3598个,这可能与拟柱孢藻N8的重叠基因较多, 除核心基因组外, 本身也需更多特异的编码蛋白维持机体功能相关[14—17]。但也可能是本研究采用了精度更高的测序技术, 且随着数据库的不断完善, 从而有更多编码蛋白被检测到。 拟柱孢藻N8数据库注释结果与其他拟柱孢藻的注释结果有极大相似性[15,17], 具有注释功能的基因主要涉及: 蛋白质代谢、协同因子和维生素代谢、氨基酸及其衍生物、碳水化合物代谢。但不同产地、不同形状、不同产毒特征的藻株注释到的基因比例不同[15—17], 如来自南美藻株在生物学途径分类部分中涉及刺激反应、化学反应的基因占比最多, 非南美藻株在细胞学组件分类部分的膜基因占比最多, 产STX藻株在细胞学组件分类部分的包膜基因占比最多, 产CYN藻株在细胞学组件分类部分的膜基因占比最多[17], 卷曲藻株基因组中存在直藻株所没有的与卷曲形态相关的涉及碳水化合物代谢的蛋白质[16], 拟柱孢藻N8在COG注释中, 细胞壁/膜/包膜合成相关基因所占比例最多。在GO注释中, 细胞学组件分类部分所占比例较大的是高分子复合物相关基因、复杂大分子及细胞和细胞元件相关基因; 生物学途径类占比最多的是代谢过程相关基因。注释基因比例的不同反映了拟柱孢藻间不同的生态适应与生理可塑性能力[14]。 目前拟柱孢藻的全基因组数据相对匮乏, 非洲藻株尚未有全基因组数据发表。在探讨世界范围内拟柱孢藻的系统进化和起源时, 有研究认为多个遗传标记的共建树比单个遗传标记的区分能力更强大, 例如Haande等[53,54]发现pc-IGS、nifH、ITSL和rpoC1等单个基因建树不能区分澳大利亚、非洲和欧洲的藻株, 但4个遗传标记共建树能够将非洲、澳大利亚、欧洲和美洲的藻株完全区分, 基于多个基因构建的系统发育树会比单个基因构建的系统发育树的结果更加可靠, 我们通过全基因组单拷贝基因构建拟柱孢藻N8的系统发育树(ML), 发现来自广东省的拟柱孢藻N8与亚欧地区的拟柱孢藻GIHE 2018、CR12和Cr2010的亲缘关系相近, 可见同种不同株系的拟柱孢藻, 虽然地理距离隔得远,但从枝长信息看, 亲缘关系也很接近。 产毒与非产毒藻株的最大的区别是非产毒藻株中没有毒素合成酶基因簇的存在[15], 拟柱孢藻N8基因组里没有发现CYN合成酶基因簇和STX合成酶基因簇, 推断N8不产毒素CYN和STX。在NCBI公布的全基因组数据里, CS-508、CS-509、CYLP、GIHE 2018和LB2897也均为非产毒藻株[15,38,41,42,45]。 拟柱孢藻N8分离自广东省镇海水库, 该水库中TN/TP比为37—243, 显著高于浮游植物和大多数其他生物对氮、磷两种元素的相对需求-Redfield比(N/P=16), 表明该水库全年均处于磷限制性状态[24],而拟柱孢藻在镇海水库中常年的优势度可达93.5%,表明该藻能适应低磷环境。我们的前期研究也发现, N8藻株对磷的适应范围很宽, 在0.02—5.12 mg/L磷浓度下均能生长[23]。本研究进一步证实拟柱孢藻N8的基因组拥有完整的磷吸收代谢通路, 可能是该藻能适应低磷和磷波动环境的分子基础。 Bai等[55]比较了非产毒的拟柱孢藻Cylindrospermopsis raciborskiiFACHB 1096和铜绿微囊藻Microcystis aeruginosa205在不同磷源的生长情况,在K2HPO4, β-甘油磷酸酯, (2-氨基乙基)-次膦酸、草甘膦和无磷的条件下, 拟柱孢藻FACHB 1096的比生长速率和内源性碱性磷酸酶活性比铜绿微囊藻 205的更高, 这侧面反映了拟柱孢藻比其他藻类拥有更有效的磷利用机制。拟柱孢藻拥有高亲和力无机磷转运系统, 该系统在蓝藻中保守存在, 排列方式为pstS-C-A-B[18], 受低磷环境的影响, 有些蓝藻可拥有多个高亲和力无机磷转运系统[50], 在拟柱孢藻N8基因组中仅有一个完整的pstS-C-A-B基因簇, 额外存在pstS拷贝, 尽管存在多个pstS拷贝, 但一般只有一个pstS的基因表达能对磷变化做出有效性的响应[51]; 在某些藻类基因组中, 高亲和力无机磷转运复合体还包括了1个磷酸盐结合亚基sphX[18,52],sphX与pstS-C-A-B形成操纵子, 拟柱孢藻N8的sphX位于pstS-C-A-B上游, 拟柱孢藻N8具有较高的磷亲和性, 在低磷环境下能维持生长[23],sphX的存在与否及位置差异在不同蓝藻中情况不同[18], 是否对磷吸收亲和力产生影响还需进一步实验验证。低亲和力无机磷转运系统在蓝藻中不是普遍存在的[20], 而拟柱孢藻N8与其他拟柱孢藻一样拥有低亲和力无机磷转运系统[25], 推断该系统的存在有利于其在高磷环境下吸收无机磷, 更能适应环境中磷浓度的变化[6,23,25]。拟柱孢藻N8的C-P裂解酶基因按phnFHGIJKLM排列, 有2个操纵子phnCDE, 用于有机膦的吸收和同化, 拟柱孢藻N8比其他7株藻株多一个phnF与phnM, 这可能导致藻株间有机磷转化能力的差异[48,51]。拟柱孢藻N8中编码碱性磷酸酶基因为phoA, 有2个拷贝, 与其他拟柱孢藻的碱性磷酸酶拷贝数不一致[50], 蓝藻中的碱性磷酸酶活性可以随磷浓度大小而变化的, 拷贝数目不同反映了藻株间不同的有机磷利用效率[22]。由以上讨论可见,拟柱孢藻N8的磷吸收转运通路在基因拷贝数目与排列顺序等与其他藻株存在差异, 今后可从比较基因组学结合生理实验入手, 进一步探讨不同拟柱孢藻间磷吸收调节机制, 从而解析拟柱孢藻在磷限制水体中占优势的原因。1.3 全基因组测序与组装
1.4 基因组注释
1.5 亲缘关系分析
1.6 产毒潜力分析
1.7 磷吸收转运基因分析
2 结果
2.1 拟柱孢藻N8基因组的基本信息

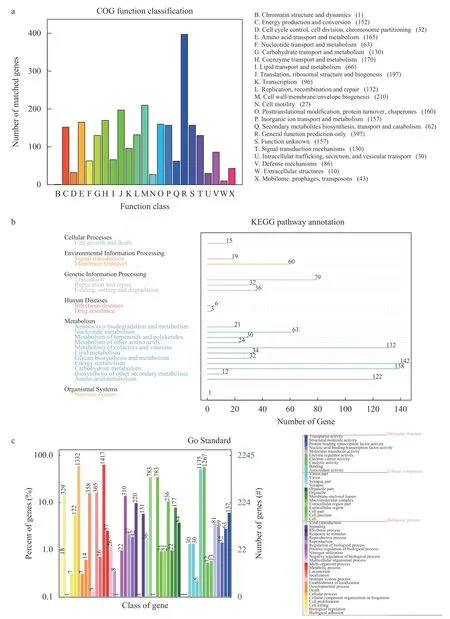
2.2 拟柱孢藻N8的数据库注释
2.3 拟柱孢藻基因组基本信息的比较

2.4 亲缘关系分析

2.5 拟柱孢藻N8的毒素合成酶基因簇分析
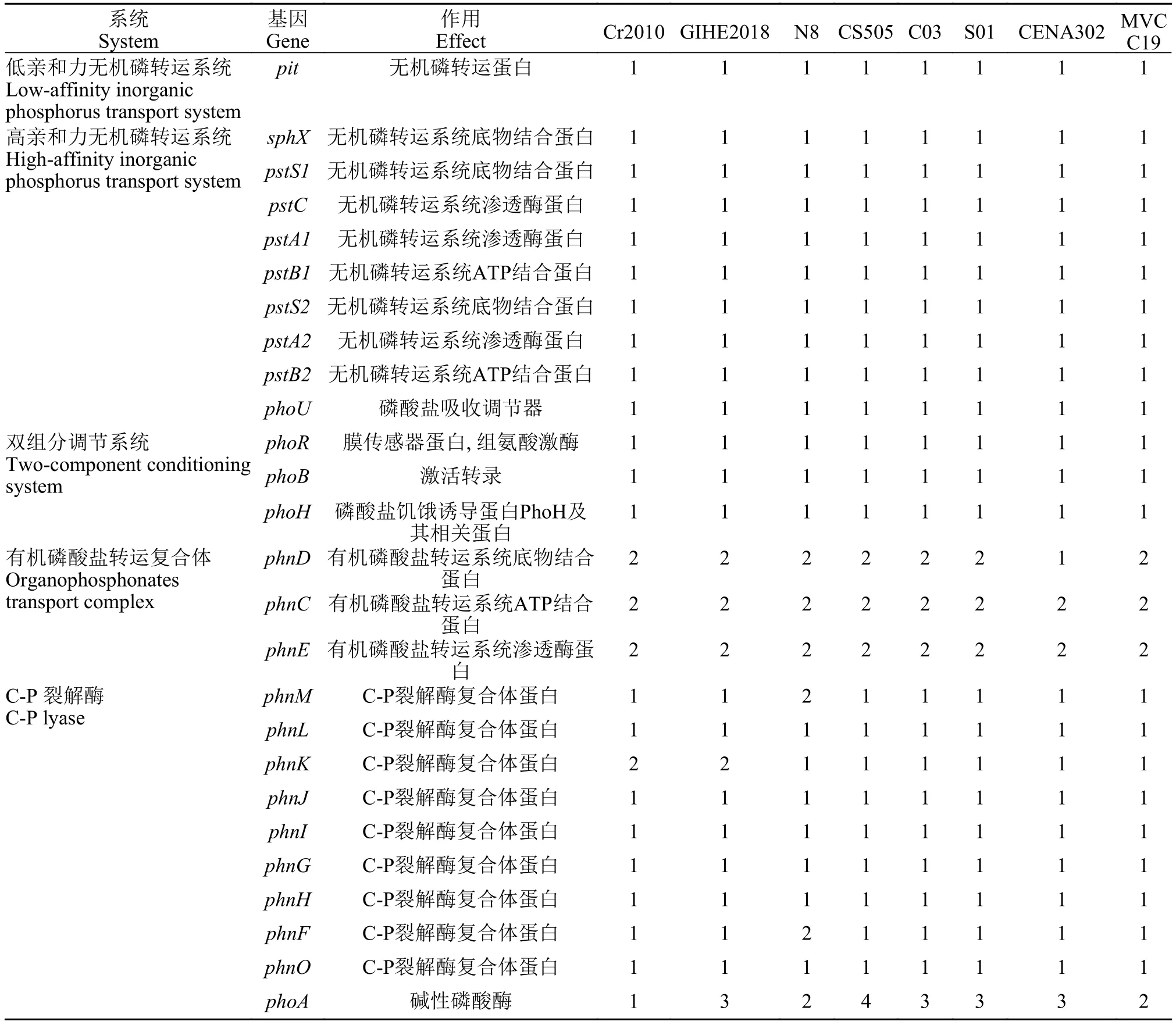
2.6 八株拟柱孢藻的磷代谢基因组成分析

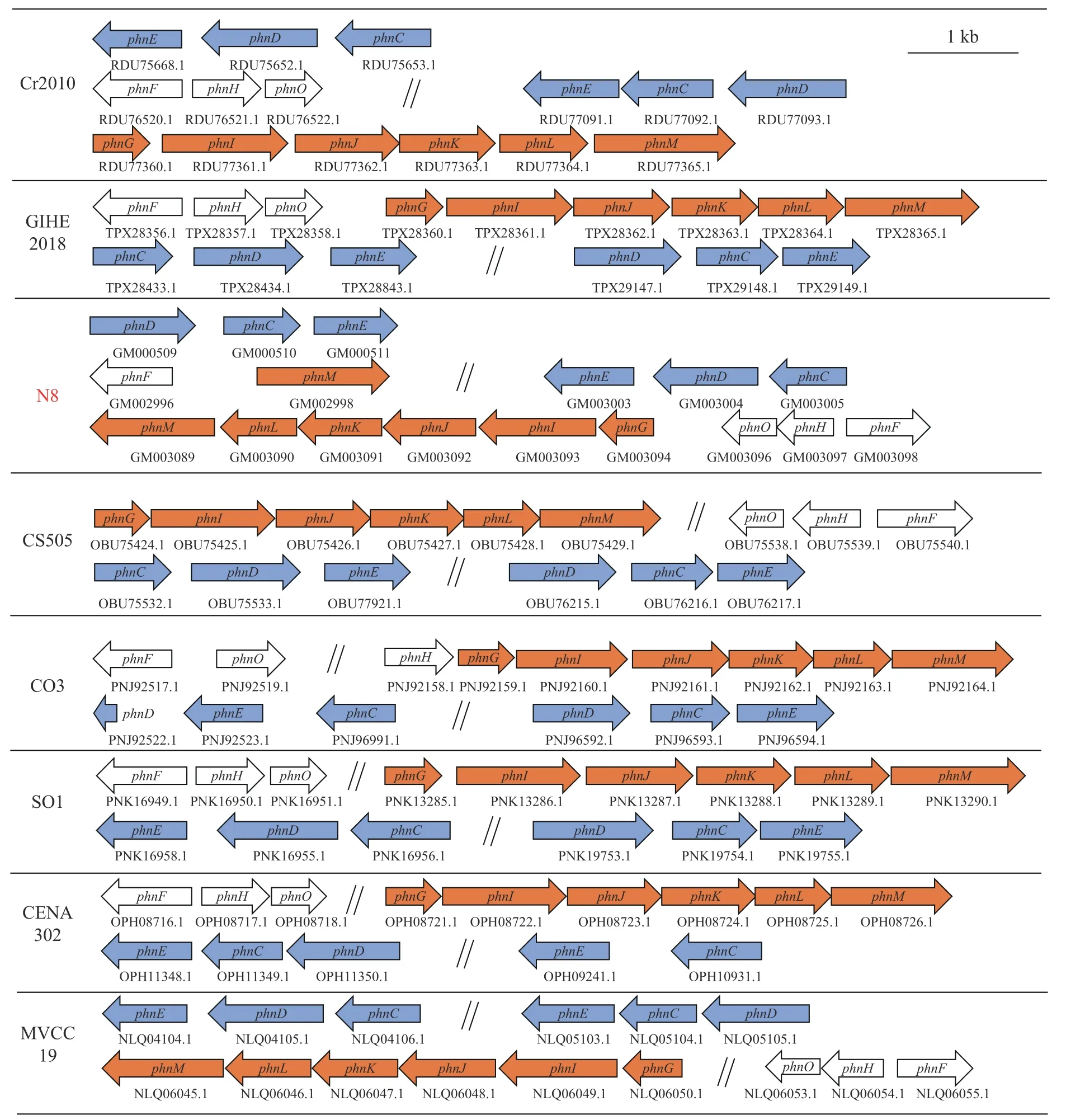
3 讨论
3.1 拟柱孢藻的基因组特征
3.2 拟柱孢藻的亲缘关系与产毒特征
3.3 拟柱孢藻的磷吸收代谢通路
