PVsiRNAPred-LSTM:基于长短时记忆神经网络预测植物病毒衍生的小干扰RNA
2022-06-26李博文贺碧芳
李博文 贺碧芳




摘 要:植物病毒衍生的小干扰RNA(Virus-derived siRNAs,vsiRNAs)能够调节多种生物学过程,在抗病毒免疫中发挥着非常重要的作用。因此,植物vsiRNAs的识别有助于了解其生物发生机制,对研究抗病毒植物具有重要意义。虽然,现在已有多种实验方法通过检测RNA来寻找vsiRNAs,但是实验测试费时费力费钱。在本文中,我们从PVsiRNAdb数据库中提取植物vsiRNAs序列,基于长短时记忆神经网络(Long Short-Term Memory neural network,LSTM)与vsiRNAs序列,开发了一种深度学习算法——PVsiRNAPred-LSTM,用于预测植物vsiRNAs。PVsiRNAPred-LSTM可以自动学习并选择与预测任务相关的重要特征。为了防止模型过拟合,我们使用了五折交叉检验来训练模型。在五折交叉检验测试中,该模型的准确率为64.38%,灵敏度(Sn)为66.44%,精确度(Pr)为60.51%,F1值为0.64,特异性(Sp)为56.63%,马修斯相关系数(MCC)为0.23,AUCROC为0.67。以上结果表明PVsiRNAPred-LSTM取得了良好的预测效果,我们希望通过PVsiRNAPred-LSTM这一生物信息学算法来预测植物vsiRNAs,帮助找到新的植物vsiRNAs。
关键词:植物病毒衍生的小干扰RNA(vsiRNAs);长短时记忆神经网络(LSTM);深度学习;五折交叉检验;生物信息学算法;vsiRNA预测
非编码RNA(Non-coding RNA,ncRNA)是转录自基因组的不编码蛋白质的RNA分子。除了在转录和转录后水平上发挥作用以外,ncRNA在基因表达的表观遗传学调控中也有着非常重要的作用。小干扰RNA(Small interfering RNA,siRNA)、microRNA(miRNA)、Piwi相互作用RNA(piRNA)是三种主要的调控型ncRNA,负责调控基因表达和宿主抗微生物免疫[1]。
siRNA是长度为20到30个核苷酸的双股RNA(dsRNA),在生物学上有多种用途。植物vsiRNAs(Virus-derived siRNAs)来源于RNA病毒的双链复制过程中的dsRNA分子。植物vsiRNAs可参与调控植物的生长、发育和抗病毒免疫等多种生物过程[2]。在受病毒感染的植物体内,vsiRNAs可以作为RNA干扰(RNA interference,RNAi)靶向入侵病毒的向导,以达到对病毒扩增的抑制效果[2-3]。除此之外,vsiRNAs还可以下调宿主基因转录,达到对宿主基因表达抑制的作用[4-5]。近年来大量研究项目表明,vsiRNAs在保护宿主植物免受病毒感染方面具有广泛应用。例如,2020年陈玲等人发表了vsiRNAs在果树病毒研究中的应用,为果树病毒研究防控提供了新思路[6]。因此,植物vsiRNAs的识别有非常重要的意义,能帮助我们进一步了解vsiRNAs的发生机制,为进一步研究抗病毒植物做出贡献。
为了存储与集中管理vsiRNAs,研究者们开发了很多的vsiRNAs数据库。例如,2019年Kumar等人开发了PVsiRNAdb数据库[7]。此外,其他课题组也构建了存储siRNAs和vsiRNAs的数据库,如siRNAdb[8]、VIRsiRNAdb[9]和HIVsirDB[10]。基于vsiRNAs数据库的数据,研究者们也开发了许多生物信息学算法来预测vsiRNAs。尽管现阶段植物vsiRNAs数据非常丰富,但是用于预测植物vsiRNAs的工具却不多。此外,虽然实验方法能够准确地识别入侵植物的RNAs序列,识别新的vsiRNAs。然而与计算方法相比,实验方法检测vsiRNAs投入的时间和人力物力及费用成本都很高,往往付出与回报不成正比,所以现在需要一种可开发性高的计算方法来预测vsiRNAs。
针对上述问题,我们提出了基于植物vsiRNAs序列组成的PVsiRNAPred-LSTM深度学习模型,用于预测植物vsiRNAs。PVsiRNAPred-LSTM模型的关键部分是LSTM模块,它可以自动学习植物vsiRNAs相关的RNA序列层次表示,降低试验成本,作为为数不多的生物信息学算法为发现新的vsiRNAs和抗病毒植物的研究提供帮助。
1 数据与预处理
1.1 数据来源
本文使用的植物vsiRNAs数据集来自2019年Kumar等人发布的PVsiRNAdb数据库[7],构造非植物vsiRNAs数据集的方法跟本课题组2019年发表的文章[11]所用方法一致,这里就不再赘述。最终训练数据集共包括12570条植物vsiRNAs序列和12570条非植物vsiRNAs序列,如图1所示。此外,我們采用双样本t检验对阳性数据集和阴性数据集的长度进行分析,两个数据集的长度分布无统计性差异(p>005)。两个数据集的长度分布为17~30个核苷酸残基。
1.2 数据预处理
本数据集内序列长度不一致,长度最短为17个核苷酸残基,最长为30个核苷酸残基。所以,首先将数据转换成相同长度的序列,不足30个核苷酸残基的序列用“X”补齐,方便后续采用Embedding进行特征编码。
2 分析方法
2.1 特征编码
目前,越来越多的研究者采用深度学习的方法预测DNA、RNA和蛋白质序列,但是这一过程中的输入必须是数值而不能是字符。构建一个实用性强且准确率高的测序模型,除了基准数据集可靠之外,还需要选取合适的特征编码方法,将序列数据不失真地转变为数值进行表征,这对于描述序列数据结构和功能属性的内在关联是非常重要的。在本研究当中,我们使用Embedding来进行特征编码,将每条序列转换为一个5×30的二维向量。
2.2 分类算法
长短时记忆神经网络(LSTM)是一种特殊的循环神经网络,它是由Hochreiter & Schmidhuber提出的。LSTM在一定程度上可以更有效地解决信息的长期依赖,从而防止梯度消失或爆炸。与传统的循环神经网络(Recurrent neural network,RNN)相比,LSTM在结构上设计了循环体结构,它主要使用两个门来控制单元状态c的内容:其中一个是遗忘门(forget gate),遗忘门决定了上一时刻的单元状态ct-1有多少保留到当前时刻ct;另外一个是输入门(input gate),输入门决定了当前时刻网络的输入xt有多少保存到单元状态ct。此外,还有一个输出门(output gate)来控制单元状态ct有多少输出到LSTM的当前输出值ht。简单地说,LSTM比普通循环神经网络有更好的表现。张永清等人在2018年申请的预测DNA-蛋白质结合的双向LSTM和CNN模型专利[12]、Ismalia Bouba等人的IncRNA基因调控关系的分析與预测等都基于LSTM进行预测建模[13]。以上研究结果表明,LSTM在预测DNA和蛋白质方面拥有广泛的应用前景。
2.3 PVsiRNAPred-LSTM的设计
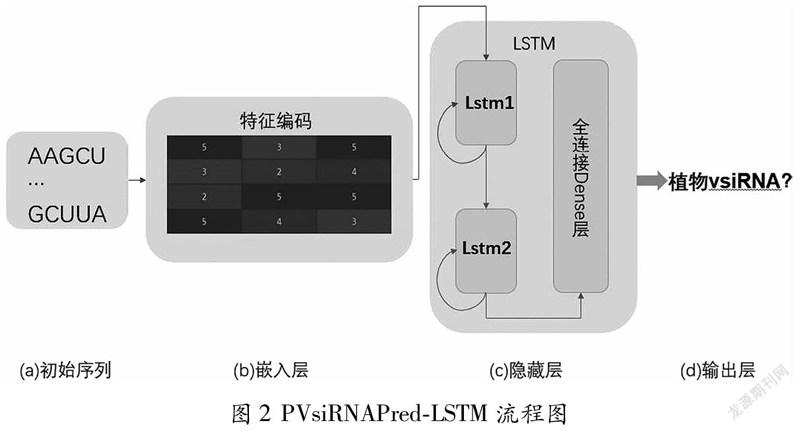
PVsiRNAPred-LSTM是一种用于预测植物病毒衍生的vsiRNAs的深度学习架构,它以RNA序列为输入,可以自动学习与植物vsiRNAs相关的RNA序列层次表示。PVsiRNAPred-LSTM模型使用TensorFlow(https://tensorflow.google.cn/)和Keras(http://keras.io)库开发。如图2所示,PVsiRNAPred-LSTM的工作流程如下:
(1)嵌入层:Embedding嵌入层将每条序列转换为一个5×30的二维向量;
(2)隐藏层:PVsiRNAPred-LSTM有两个隐藏层,分别有8和4个神经元,将第一个隐层的结果作为下一个隐层的输入,应用“tanh”非线性函数为激活函数进行激活;
(3)PVsiRNAPred-LSTM有一个全连接的Dense层,其中有两个神经元,使用“softmax”非线性函数为激活函数进行激活;
(4)输出层:最后输出层对植物vsiRNAs进行预测。
2.4 模型评价
在本文中,为了防止模型过拟合,我们采用五折交叉检验评估PVsiRNAPred-LSTM的预测性能。为了量化预测模型的性能,我们使用了六种常见的评估指标,包括灵敏度(Sensitivity,Sn)、精确度(Precision,Pr)、F1值、特异性(Specificity,Sp)、准确度(Accuracy,Acc)和马氏相关系数(MCC)。这些指标的计算公式如下:
在上述公式中,TP表示正确预测的植物vsiRNAs的数量,TN代表正确预测的非植物vsiRNAs的数量。FP表示被错误预测为植物vsiRNAs的非植物vsiRNAs数量,FN代表被错误预测为非植物vsiRNAs的植物vsiRNAs的数量。除此之外,我们也绘制了模型的受试者操作特征曲线(receiver operating characteristic curve,简称ROC曲线),同时计算了该曲线下的面积(area under the ROC,AUCROC),进一步采用AUCROC来评估模型的预测效果。
3 实验结果
3.1 PVsiRNAPred-LSTM模型预测结果
PVsiRNAPred-LSTM基于LSTM构建,可自动学习与植物vsiRNAs相关的RNA序列层次表示,预测植物vsiRNAs。在评估模型时,我们使用了五折交叉检验。基于LSTM的模型准确率为64.38%,灵敏度为66.44%,精确度为60.51%,F1值为0.64,特异性为56.63%,马氏相关系数为0.23。如图2所示,PVsiRNAPred-LSTM的AUCROC为0.67。以上结果表明PVsiRNAPred-LSTM具有良好的预测效果。
3.2 与其他传统机器学习模型的比较
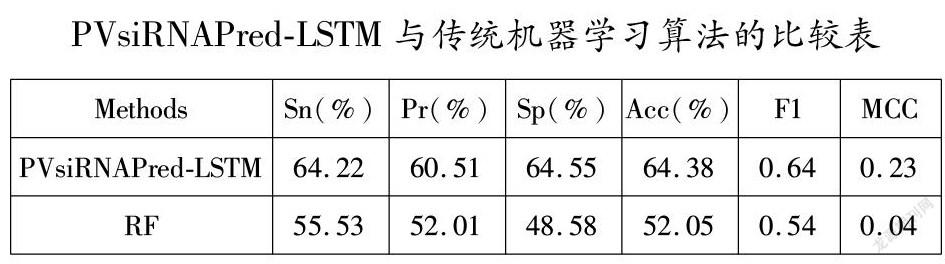
我们还采用iLearnplus软件中的几种机器学习算法来对本数据集进行预测,包括随机森林(RF)、K-最邻近(KNN)、决策树(DecisionTree)、lightGBM和支持向量机(SVM)。选取累积核苷酸频率(Accumulated Nucleotide Frequency,ANF)进行特征提取。其中,ANF特征表示核苷酸密度和RNA片段中每个核苷酸的分布。然后,使用五折交叉检验对训练的模型进行评估。如下表所示,PVsiRNAPred-LSTM分别比基于RF、KNN、DecisionTree、lightGBM和SVM的预测模型准确率高12.33%、14.3%、13.78%、11.97%和13.33%。综上所述,在基于PVsiRNAdb数据集的植物vsiRNAs的预测当中,深度学习模型要优于传统机器学习模型。
4 讨论
PVsiRNAPred-LSTM的预测性能并不令人满意。众所周知,深度学习算法的预测性能在极大程度上依赖于训练数据集的数量和质量,所以使得模型预测性能不高的因素可能有以下两个方面。第一,用于训练的数据集数量太小。第二,用于训练的数据集质量不高,这主要体现在数据不够多样化上。因此,我们认为基于LSTM的深度学习模型——PVsiRNAPred-LSTM可以在数量更多并且更多样化的植物病毒衍生的vsiRNA数据集上实现更高的性能。
文中基于LSTM的PVsiRNAPred-LSTM的模型性能明显高于传统的机器学习方法。该模型的核心是LSTM模块,它以RNA序列为输入,可以自动学习与植物vsiRNA相关的序列层次表示,最终实现对于植物vsiRNA的预测。虽然PVsiRNAPred-LSTM的预测性能不够理想。但是,对于本文提到的其他五种基于传统机器学习方法的分类器来说,该模型的预测能力还是比较优秀的。
结语
植物vsiRNAs的快速识别为了解植物vsiRNAs的生物发生和生物学功能提供重要线索。在这项研究中,我们采用包含12570条植物vsiRNAs和12570条非植物vsiRNAs的基准数据集,利用Embedding函数将序列转成特征向量,进一步建立了基于LSTM的植物vsiRNAs预测模型PvsiRNAPred-LSTM。此外,我们还发现PvsiRNAPred-LSTM的五折交叉检验准确率分别比基于RF、KNN、DecisionTree、lightGBM和SVM的预测器准确率高12.33%、143%、1378%、11.97%和13.33%。PvsiRNAPred-LSTM在五折交叉检验中取得的准确率为64.38%,灵敏度为66.44%,精确度为60.51%,F1值为0.64,特异性为5663%,马氏相关系数为0.23,AUCROC为0.67。此结果表明,深度学习方法在一定程度上要优于传统的机器学习方法,能够更好的预测植物病毒衍生的vsiRNA,此外深度学习方法在生物信息學领域有着远大的前景,可以极大地促进生物信息学领域的发展。
参考文献:
[1]Guo Z,Li Y,Ding SW.Small RNA-based antimicrobial immunity.Nat Rev Immunol,2019,19(1):31-44.
[2]Zhang C,Wu Z,Li Y,Wu J.Biogenesis,Function,and Applications of Virus-Derived Small RNAs in Plants.Front Microbiol,2015,6:1237.
[3]Szittya G,Moxon S,Pantaleo V,Toth G,Rusholme Pilcher RL,Moulton V,et al.Structural and functional analysis of viral siRNAs.PLoS Pathog,2010,6(4):e1000838.
[4]Li YF,Zheng Y,Jagadeeswaran G,Sunkar R.Characterization of small RNAs and their target genes in wheat seedlings using sequencing-based approaches.Plant Sci.2013;203-204:17-24.
[5]Miozzi L,Gambino G,Burgyan J,Pantaleo V.Genome-wide identification of viral and host transcripts targeted by viral siRNAs in Vitis vinifera.Mol Plant Pathol,2013,14(1):30-43.
[6]陈玲,段续伟,张晓明,等.植物病毒来源的小干扰RNA及其在果树病毒研究中的应用[J].果树学报,2020,37(7):1080-1088.2020.
[7]Gupta N,Zahra S,Singh A,Kumar S.PVsiRNAdb:a database for plant exclusive virus-derived small interfering RNAs.Database (Oxford),2018,2018.
[8]Chalk AM,Warfinge RE,Georgii-Hemming P,Sonnhammer EL.siRNAdb:a database of siRNA sequences.Nucleic Acids Res.2005;33(Database issue):D131-4.
[9]Thakur N,Qureshi A,Kumar M.VIRsiRNAdb:a curated database of experimentally validated viral siRNA/shRNA.Nucleic Acids Res.2012;40(Database issue):D230-6.
[10]Tyagi A,Ahmed F,Thakur N,Sharma A,Raghava GP,Kumar M.HIVsirDB:a database of HIV inhibiting siRNAs.PLoS One,2011,6(10):e25917.
[11]He B,Huang J,Chen H.PVsiRNAPred:Prediction of plant exclusive virus-derived small interfering RNAs by deep convolutional neural network.J Bioinform Comput Biol,2019,17(6):1950039.
[12]张永清,曾圆麟,卢荣钊,等.一种预测DNA-蛋白质结合的双向LSTM和CNN模型,2019.
[13]Bouba I.lncRNA基因调控关系的分析与预测[D].大连理工大学,2019.
[14]Chen Z,Zhao P,Li C,Li F,Xiang D,Chen YZ,et al.iLearnPlus:a comprehensive and automated machine-learning platform for nucleic acid and protein sequence analysis,prediction and visualization.Nucleic Acids Res,2021,49(10):e60.
作者简介:李博文(1998— ),男,汉族,山东济南人,硕士,研究方向:医学信息工程。
*通讯作者:贺碧芳。
