ZBED6基因敲除猪肾脏转录组分析
2022-05-31田文杰王丹丹王圣楠马月辉宋子仪
田文杰,王丹丹,王圣楠,马月辉,蒋 琳,宋子仪
(1.广西大学动物科学技术学院,南宁 530000;2.中国农业科学院北京畜牧兽医研究所,北京 100193)
锌指BED结构域结合蛋白6(zinc finger BED domain-containing protein 6,ZBED6)是近些年发现的胎盘类哺乳动物特有、序列高度保守的转录调控因子[1]。在家猪体内研究发现,胰岛素样生长因子2(insulin-like growth factor 2,IGF2)基因内含子3发生了G→A的突变,破坏了ZBED6对IGF2表达量的抑制,因此家猪中IGF2基因表达量增高,从而影响了内脏器官、肌肉及脂肪的生长发育[2-3]。研究显示,ZBED6基因编辑巴马香猪和两广小花猪的IGF2基因表达量上调,产肉量增多[4-6];ZBED6基因敲除小鼠IGF2基因表达量升高,肌肉增多[7];ZBED6基因可抑制黄牛IGF2基因表达及肌肉发育[4-5];ZBED6基因的遗传变异与绵羊眼肌面积、尾脂重、背部肌肉厚及背最长肌重等主要胴体性状显著相关[6];在小鼠和牛肌原细胞中沉默ZBED6基因,结果显示IGF2基因高表达,促进成肌细胞的增殖和分化[8-9],以上研究均说明了ZBED6-IGF2轴对肌肉发育的重要作用。
目前,ZBED6基因在猪上的研究主要集中于生长发育调控,对于猪肾脏代谢作用的研究鲜见报道。Markljung等[1]对ZBED6基因KO小鼠肾脏转录组研究发现,差异基因主要集中于磷酸化等代谢通路。Younis等[7]利用染色质免疫共沉淀(chromatin immunoprecipitation,ChIP)技术对小鼠C2C12细胞进行测序分析发现,在ZBED6约1 200个潜在靶基因(包含IGF2基因)中,有约22%的靶基因编码其他各种转录因子,由此推测除生长发育外,ZBED6可能具有其他复杂的生理功能。
本研究以前期通过CRISPR/Cas9技术构建成功的ZBED6 KO巴马香猪[10]与WT巴马香猪为研究对象,采集肾脏组织进行转录组分析,筛选在ZBED6 KO和WT巴马香猪肾脏组织中差异表达基因并分析相关功能,以期为进一步揭示ZBED6基因的分子调控机制提供参考依据。
1 材料与方法
1.1 样品采集
试验猪选自中国农业科学院北京畜牧兽医研究所大兴猪场实验基地,利用CRISPR/Cas9技术完成ZBED6基因敲除。选择8月龄ZBED6基因敲除雌性巴马香猪(编号为1、3、6)和野生型对照同性别巴马香猪(编号为2、4、5)各3头屠宰,采集肾脏(ZBED6 KO:肾脏1、肾脏3、肾脏6;WT:肾脏2、肾脏4、肾脏5),迅速投入液氮中保存备用。
1.2 主要试剂及仪器
RNAsimple总RNA提取试剂盒购自天根生化科技(北京)有限公司;RT-PCR试剂盒、SYBR Green试剂盒均购自TaKaRa公司;TruSeq试剂盒购自Illumina公司。Agilent 2100生物分析仪和ABI7500实时荧光定量仪均购自ABI公司。
1.3 实时荧光定量PCR检测ZBED6基因敲除对猪肾脏组织中IGF2基因表达量的影响
使用RNAsimple总RNA提取试剂盒提取肾脏组织总RNA,使用Agilent 2100生物分析仪检测RNA浓度,1.5%琼脂糖凝胶电泳检测RNA完整性,质量合格的RNA样品于-80 ℃保存备用。
使用RT-PCR试剂盒对RNA进行反转录得到cDNA。选用GAPDH作为内参基因,使用Primer Premier 5.0软件选择目的基因CDS区来设计引物,引物序列见表1。引物均由华大基因有限公司合成。
PCR反应体系20 μL:上、下游引物各0.4 μL,cDNA模板3 μL,ddH2O 7 μL,SYBR Green Mix 10 μL。PCR反应程序:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火34 s,72 ℃延伸15 s,共40个循环。每个样品设置3个生物学重复,采用2-ΔΔCt方法进行计算。

表1 引物信息
1.4 转录组测序分析
1.4.1 RNA-Seq测序 将RNA样品(浓度>100 ng/μL,总量>2.0 μg,且RIN值>8.0)送至北京贝瑞和康生物科技股份有限公司进行cDNA文库构建及高通量测序。使用Illumina Hiseq 2500测序平台分别对上述样品进行读长为150 bp的双末端测序。
1.4.2 原始数据的质控 采用FastQC对下机后数据(raw reads)质量进行基本统计;使用fastp软件截去测序数据的测序接头和低质量片段,从而获得高质量的clean reads。具体操作为:去除含有索引接头的reads;去除低质量的reads(包含N的比例>10%的reads和低质量碱基QPhred值≤10的碱基数占总碱基数量50%以上的reads);去除过滤后长度<25 bp的reads。
1.4.3 clean reads分析 参考基因组和注释文件分别采用来自Ensemble数据库的Susscrofa11.1和gtf100(https:∥asia.ensembl.org/index.html)。通过软件TopHat、Cufflink、Cuffmerge和Cuffdiff对clean data分别进行参考基因组比对、表达量的量化、转录本组装及差异表达基因分析;用FDR法对P值进行多重校正得到调整后P值,差异表达基因的筛选标准为:P<0.05和log2|FoldChange|>2。
1.5 差异表达基因的层次聚类和KEGG通路富集分析
通过R包绘制火山图及热图,显示差异表达基因的分布及在ZBED6 KO和WT猪肾脏中的表达;根据检测到的所有表达基因的FPKM值,对6个肾脏样品进行主成分分析(PCA)。检测样品相关性;对差异表达基因进行KEGG通路富集分析(https:∥biit.cs.ut.ee/gprofiler/gost),根据Fold-enrichment和调整后P值来筛选ZBED6潜在的互作基因。
1.6 实时荧光定量PCR验证差异表达基因
根据转录组测序的结果随机选取7个差异表达基因(表1),采用实时荧光定量PCR方法验证测序结果的可靠性,方法同1.3。
1.7 统计分析
组间采用t检验分析差异性,数据以平均值±标准误表示,以P<0.05为差异显著性判断标准,利用GraphPad软件进行绘图。
2 结 果
2.1 实时荧光定量PCR检测IGF2基因表达量
通过实时荧光定量PCR检测IGF2基因在ZBED6 KO和WT猪肾脏组织中的相对表达量,结果见图1。由图1可知,敲除ZBED6基因后,猪肾脏中IGF2基因表达量显著上调(P<0.05),表明ZBED6基因能够抑制猪肾脏IGF2基因的表达。

*,差异显著(P<0.05),**,差异极显著(P<0.01)。图6同*,Significant difference(P<0.05);**,Extremely significant difference (P<0.01).The same as fig.6图1 敲除ZBED6基因后猪肾脏中IGF2基因相对表达量Fig.1 Relative expression of IGF2 gene in porcine kidney after ZBED6 gene knockout
2.2 转录组测序数据的质量评估及比对
对ZBED6 KO和WT猪肾脏组织总RNA利用转录组测序技术进行分析,结果见表2。由表2可知,6个样品测序共获得78 G数据量,各样本质控率和Q30数据均在90%以上,比对率在87%以上,说明测序数据质量良好,可用于下一步生物信息学分析。
2.3 差异表达基因分析
在肾脏组织中利用RNA-Seq测序共鉴定到25 213个基因,与WT猪相比,ZBED6 KO猪中共筛选到299个差异表达基因,其中103个基因上调表达,196个基因下调表达(图2),IGF2基因同样在ZBED6 KO猪肾脏中上调表达,与实时荧光定量PCR检测结果一致。用热图展示差异表达基因在所有样品中表达量的分布情况,结果见图3。由图3可知,2组样品组内均一性较好,但ZBED6 KO和WT猪肾脏中基因表达量存在明显差异。
根据所有基因表达量FPKM值进行主成分分析,结果见图4。由图4可知,WT和ZBED6 KO猪肾脏中表达模式组内相似,但组间截然不同,证明采样和测序数据可靠。
2.4 差异表达基因KEGG通路富集分析
对获得的差异表达基因进行KEGG通路富集分析,结果显示,差异表达基因显著富集在视黄醇代谢、化学致癌、甲流、药物代谢、补体和凝聚级联6条通路中,大多数均与代谢活动相关(图5),其整体通路结果与肾脏代谢的功能相符。KEGG通路富集分析最显著的通路为视黄醇代谢通路,其中经典的生物代谢关键酶CYP1A1基因同时被富集在视黄醇和细胞色素P450对外源性药物两条代谢通路中,说明ZBED6基因敲除对巴马香猪肾脏组织代谢活动具有重要调控作用。
2.5 差异表达基因实时荧光定量PCR验证
随机选择7个差异表达基因CYP2C42、AOX1、RDH16、CYP2A19、CYP26B1、CYP1A1和RDH5,利用实时荧光定量PCR技术进行RNA-Seq测序数据准确性的验证,结果见图6。由图6可知,与WT猪相比,ZBED6 KO猪肾脏中CYP2C42、CYP2A19、RDH16、CYP1A1和RDH5基因表达量极显著或显著下调(P<0.01;P<0.05),AOX1和CYP26B1基因表达量极显著或显著上调(P<0.01;P<0.05),与RNA-Seq测序结果相一致,证实了RNA-Seq测序分析结果的准确性。

表2 测序数据质控和比对分析
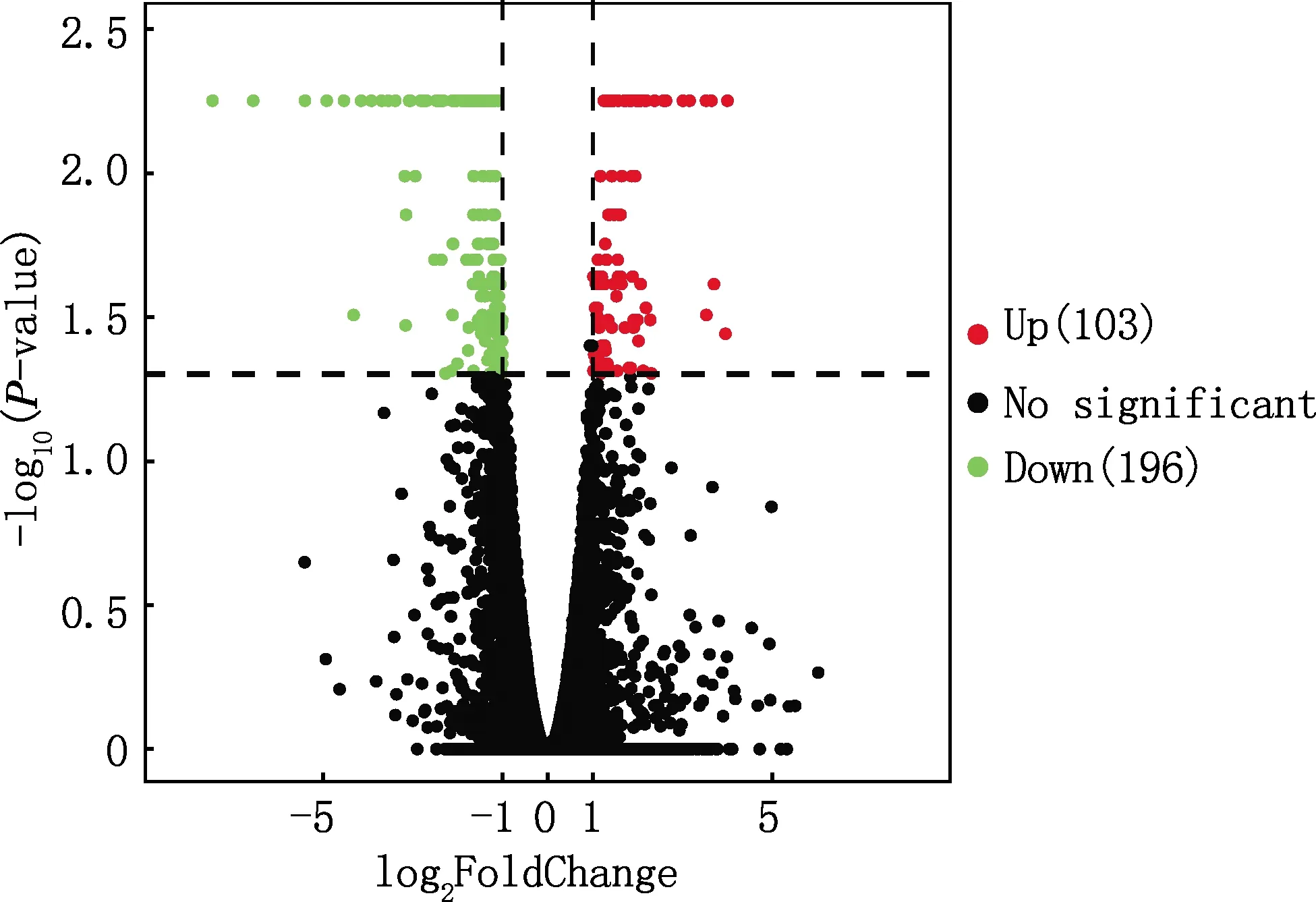
横虚线为P-value为0.05的阈值;横虚线往上右边表示上调基因(103),往上左边表示下调基因(196);往下表示非显著差异表达基因Horizontal dotted line was the threshold of P-value=0.05 in the diagram;Above the horizontaldashed line,right represents up-regulated genes(103),left represents down-regulated genes(196),below the horizontal dotted line represents non-significantly differentially expressed genes图2 差异表达基因火山图Fig.2 Volcano map of differentially expressed genes
3 讨 论
近年来,利用分子生物技术研究基因功能及调控机制已经成为主要途径。ZBED6是2009年发现并命名的转录因子,其序列在多种胎盘类哺乳动物中高度保守[1]。研究发现,ZBED6基因除影响肌肉发育外,在调控小鼠糖脂代谢[11-13]、肠癌细胞生长发育[14]等方面也发挥着重要作用。Younis等[7]利用RNA-Seq技术对ZBED6基因敲除(ZBED6 KO)小鼠肾脏组织分析发现,其差异表达基因主要富集在氧化磷酸化、运输活动等与代谢相关通路中,表明ZBED6基因敲除除能够促进肌肉生长外,对小鼠肾脏代谢反应同样具有重要作用。然而,关于ZBED6基因对猪肾脏代谢调控的研究鲜见报道。
本研究中,实时荧光定量PCR结果显示,ZBED6基因敲除后猪肾脏组织中IGF2基因表达量显著上调;RNA-Seq分析结果发现,ZBED6 KO猪肾脏组织中表达上调的103个基因中也包含IGF2基因,与Younis等[7]结果相一致,说明ZBED6同样能够抑制猪肾脏中IGF2基因的表达。近年来研究发现,小鼠ZBED6基因还可通过影响胰腺β细胞功能、胰岛素产生进而调控糖脂代谢[11-13]。本研究中,差异表达基因显著富集在6条与代谢相关的通路上,分别为视黄醇代谢、化学致癌、甲流、 药物代谢、补体和凝聚级联。

图3 差异表达基因热图Fig.3 Heat map of differentially expressed genes

图4 主成分分析图Fig.4 Principal component analysis map
提示在猪中ZBED6基因除通过抑制IGF2基因而影响肌肉生长发育外,可能同样对肾脏代谢发挥着重要作用。KEGG通路富集分析中最显著的通路为视黄醇代谢通路,视黄醇又称维生素A,是多种生物过程必需的微量营养素,包括视觉、胚胎发育、细胞分化、生长发育及免疫系统的调节[15-16]。肾脏是视黄醇一个重要的储存器官[17],通过其代谢功能维持视黄醇稳态和运转[18-19]。视黄醇利用乙醇脱氢酶(ADH)[20]、短链脱氢酶/还原酶(SDR)[21]、醛酮基还原酶(AKR)[22]和醛脱氢酶 (ALDH)[23]这4个家族成员代谢转化为视黄酸(RA)进而发挥作用。因此,视黄醇代谢是肾脏组织主要的代谢功能,为ZBED6基因调控猪肾脏代谢提供了有力支撑。

图5 差异表达基因KEGG通路富集分析Fig.5 KEGG pathway enrichment analysis of differentially expressed genes
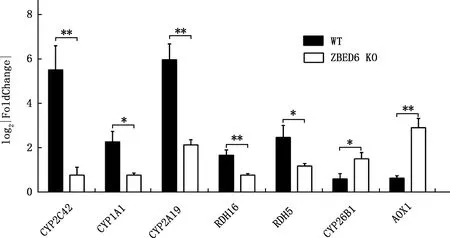
图6 7个差异表达基因实时荧光定量PCR验证Fig.6 Real-time quantitative PCR verification of 7 differentially expressed genes
聚乙二醇化姜黄素衍生物Curc-mPEG454通过上调醛氧化酶1(AOX1)、下调细胞色素P45026A1(CYP26A1)和细胞色素P45026B1 (CYP26B1),从而恢复肝脏RA水平。
CYP26B1作为视黄酸清除酶,通过氧化代谢来调节细胞中视黄酸的浓度[24],有助于哺乳动物和其他脊索动物的视黄酸代谢和体内平衡。研究表明,小鼠CYP26B1基因全身敲除会导致胚胎致死,而对出生后小鼠敲除CYP26B1基因虽不致死,但表现出寿命缩短、脂肪萎缩及视黄酸浓度增加[25]。这些结果证明,CYP26B1在视黄酸代谢中发挥着重要的作用[26]。本研究中,ZBED6 KO猪肾脏中AOX1和CYP26B1基因均显著富集到视黄醇代谢通路上,转录组分析与实时荧光定量PCR结果发现,AOX1和CYP26B1基因显著上调,推测ZBED6基因可能通过同时上调AOX1和CYP26B1基因来调控肾脏组织中视黄酸代谢的动态平衡。
视黄醇能够合成视黄醛,进而转化为视黄酸,转化后的视黄酸会与其靶蛋白进行结合,从而发挥作用。视黄醇脱氢酶5、16(RDH5、RDH16)都是一种蛋白质编码基因,与视黄醇代谢高度相关[27],其相关途径包括脂溶性维生素代谢和视黄酸生物合成。研究证明,神经生长因子(NGF)调控RDH16的从头合成并影响了视黄醇代谢途径[28]。RDH5基因作为短链脱氢酶/还原酶(SDR)家族的成员,具有催化视黄醇合成视黄醛的功能[29]。RDH5基因敲除小鼠可以存活,但在黑暗环境中的反应比较迟缓,说明RDH5基因的缺失导致小鼠体内视黄醇转化视黄醛减少,视觉能力减缓的性状一致[30-31]。此外,RDH5基因突变会导致人类患有夜盲症,证明RDH5基因具有产生视黄醛的功能[32]。本研究发现,RDH5和RDH16基因的表达量在ZBED6 KO猪肾脏组织中显著下调,提示ZBED6 KO可能通过下调RDH5和RDH16基因表达减少视黄醇转化为视黄醛。 研究发现,细胞色素P4501A1 (CYP1A1)作为生物代谢的关键酶之一,可以将小鼠和人的视黄醛催化为视黄酸[33-34],CYP1A1的诱导剂和抑制剂也会影响小鼠上皮组织中视黄酸的合成[35]。CYP1A1被富集在细胞色素P450对外源性药物的代谢作用中,早期研究发现,CYP1A1影响一些酶的活性,进而影响大鼠肝脏和肾脏对药物代谢的效率[36]。本研究中,CYP1A1基因同时被富集在视黄醇代谢和细胞色素P450对外源性药物的代谢作用2条通路中,CYP1A1基因表达量在ZBED6 KO猪肾脏组织中显著下调。 因此,ZBED6基因可能通过下调RDH5、RDH16和CYP1A1基因表达,同时上调AOX1与CYP26B1基因的表达在视黄醇-视黄醛-视黄酸的体内代谢和动态平衡调控中发挥重要功能。
4 结 论
本研究发现,ZBED6基因敲除显著上调了猪肾脏组织中IGF2基因表达量,共筛选到包括IGF2基因在内的103个上调基因和196个下调基因,CYP2C42、CYP1A1、CYP2A19、RDH16、RDH5、CYP26B1及AOX1基因是ZBED6基因调控巴马香猪肾脏视黄醇代谢的重要靶基因。
