柚多聚半乳糖醛酸酶基因家族的鉴定和分析
2022-04-27刘若南刘林婷周秋蓉张蓓高志键葛聪庄木来李延王平
刘若南 刘林婷 周秋蓉 张蓓 高志键 葛聪 庄木来 李延 王平


摘 要:草莓、桃、苹果等果实在生长后期和贮藏过程中逐渐软化,而柑橘类特别是柚在成熟后期和贮藏过程中出现粒化,严重影响其经济价值。本研究分析和鉴定了柚基因组中多聚半乳糖醛酸酶基因(CgPG)家族成员以及PG酶活的变化,旨在揭示柚果实成熟过程中CgPG表达与汁胞粒化形成的关系。本研究对柚基因组库中CgPG基因家族成员的数量、基因定位、基因结构进行生物信息学分析,并对CgPG基因家族成员进行了表达鉴定,同时测定了汁胞粒化程度以及PG活性。结果表明:从柚全基因组鉴定出26个CgPG基因,进化树分析聚类为6个亚类(Group A~Group F),除E类外,所有外显子,内含子结构和基序基本上都是保守的,Group E中的CgPG26的GDDC结构域突变为GDDA,SPNTDGI突变为APNTDGI;CgPG基因主要分布于1、2、4、5、7、9号染色体上,在2和5号染色体上都有5~6条基因的串联复制;Group A和Group B与果实成熟有关,Group A中的CgPG5和Group B中的CgPG1、CgPG24参与了果实成熟和汁胞软化,CgPG13、CgPG12、CgPG26參与柚粒化过程中的果胶降解和细胞壁稳态调节;通过表达分析,Group A中的CgPG5和Group B中的CgPG1、CgPG24可能参与了柚不易粒化果成熟过程中汁胞的软化和最后腐败,基因的显著表达表明汁胞走向衰败;Group D中的CgPG13和Group E中的CgPG12、CgPG26 3个基因可能参与了柚易粒化果成熟过程中的果胶暂时降解,基因的高表达使汁胞软化,此时正是食用的最佳时期,随后在汁胞粒化过程中基因表达显著下降。因此,CgPG基因表达呈现不同的趋势表明CgPG基因在不易粒化果和易粒化果中可能属于2个不同的调节系统,其中CgPG5、CgPG1、CgPG24属于柚成熟后期汁胞软化的果胶降解系统,CgPG13、CgPG12、CgPG26属于柚粒化的细胞壁稳态调节系统。
关键词:柚;多聚半乳糖醛酸酶;汁胞软化;汁胞粒化中图分类号:S666.3 文献标识码:A
Identification and Analysis ofpolygalacturonasegenesFamily in Pomelo
LIU Ruonan LIU Linting ZHOU Qiurong ZHANG Bei GAO Zhijian GE Cong ZHUANG Mulai LI Yan WANG Ping
1. Institute of Genetics and Breeding in Horticultural Plants, College of Horticulture, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China; 2. Pinghe Agriculture Bureau of Fujian Province, Pinghe, Fujian 363700, China; 3. College of Resources and Environment, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
Abstract: Strawberry, peach, apple and some other fruits gradually soften in the late growth period and during storage, while citrus, especially pomelo, granulated in the later ripening and storage, which seriously affects its economic value. In this study, the members of CgPG gene family in the pomelo genome library and the changes of PG enzyme activity were investigated to reveal the relationship between CgPGs expression and juice sacs granulationin the ripening process of pomelo fruit. The number, location and structure of CgPG gene family members in pomelo genome library were analyzed by bioinformatics, and the expression of CgPG gene family members was identified. The degree of granulation and the activities of PG were determined. 26 CgPG genes from pomelo genome were clustered into 6 subgroups (group A?group F) by phylogenetic tree analysis. Except for class E, all exons and introns and motifs were essentially conservative. GDDC domain ofCgPG26in Group E was mutated to GDDA, and SPNTDGI was mutated to APNTDGI respectively. The CgPG genes were mainly distributed on chromosomes 1, 2, 4, 5, 7 and 9. There were 5-6 tandem duplications of genes on both chromosomes 2 and 5. Group A and group B were related to fruit ripening.CgPG5 in group A andCgPG1, CgPG24in group B were involved in fruit ripening and juice sacs softening, andCgPG13,CgPG12, CgPG26were involved in pectin degradation and homeostatic regulation in cell wall during pomelo granulation. By expression analysis,CgPG5in Group A andCgPG1,CgPG24in Group B might be involved in softening and final decay during the ripening process of the non-granulated fruits, and the significant expression of the genes indicated the juice sacs were decomposed.CgPG5in Group A andCgPG1,CgPG24in Group B might be involved in the temporary degradation of pectin during the ripening process of easily granulated fruits. The high expression of the genes made juice sacs softened, which was the optimal time for consumption. The expressions of the genes were highly decreased during the juice sacs granulation. Therefore, CgPG genes expression showed different trends, indicating that CgPG genes might belong to two different regulatory systems in non-granular fruits and easily granular fruits. Among them,CgPG5, CgPG1,CgPG24 belonged to the pectin degradation system of juice cell softening in pomelo at late maturity, whileCgPG13, CgPG12, CgPG26 belonged to the cell wall homeostasis regulation system of pomelo granulation.
Keywords: pomelo; polygalacturonase; juice sacs softening; juice sacs granulation
DOI: 10.3969/j.issn.1000-2561.2022.04.002
多聚半乳糖醛酸酶(polygalacturonase, PG)在果胶降解的最后一步起着重要作用,是一类重要的果胶修饰酶。根据其水解活性的差异分为3种类型:内切PG、外切PG、鼠李糖PG[1-4]。根据功能分:与果实成熟相关的PG、与器官脱落相关的PG、与花粉发育相关的PG[5-6]。目前,柚CgPG基因的研究虽然较多,但缺乏对其家族的系统鉴定和分析。研究表明,在桃、猕猴桃、苹果、木瓜、番茄、梨、葡萄、草莓和香蕉等果实中,PG基因的表达影响果实的成熟和软化[7-8]。果胶结构的改变不仅影响果实的软化,也影响着次生壁的形成。柚果实粒化过程中次生壁木质化起始于胞间层,有观点认为细胞的木质化过程是老化细胞将多糖降解为单糖转移给新生细胞后木质素单体的多聚化[7-8],也有认为次生壁形成与果胶降解共调控[9]。柚基因组测序的完成和序列释放是在全基因组水平上为柚的重要功能基因的发掘和功能研究提供基础。本研究利用生物信息学方法对柚CgPG基因家族成员的保守结构域、基因结构和表达情况进行分析。以期为进一步研究与柚果实成熟软化和粒化相关的CgPG基因调控机制打下基础。
1 材料与方法
1.1 材料
以不易粒化(NGF)和易粒化(GF)特性的‘琯溪蜜柚果实汁胞为材料。不易粒化果种植于平和县小溪镇一条小溪两旁,为河流沉积河床,土壤肥沃深厚,30年健壮果树,蜜柚果实汁胞不易发生粒化、较软。易粒化果种植于平和县旧楼村农业农村局山地果场,30年健壮果树,蜜柚汁胞易发生粒化。树体正常管理。
2019年分别采取花后120 d、花后150 d、花后180 d(正常成熟采果期)、常温条件下贮藏30 d、贮藏60 d果实,每次采果18个。测定果实品质,取汁胞混匀,液氮处理后于?80℃保存。
1.2 方法
1.2.1 汁胞粒化率测定 按照汁胞粒化形态特点[10-12],蜜柚果实汁胞分为正常汁胞、凝胶汁胞和粒化汁胞。汁胞粒化率为粒化汁胞重量占汁胞总重的比值。汁胞凝胶率为凝胶汁胞重量占汁胞总重的比值。
1.2.2 PG活性测定 多聚半乳糖醛酸酶。采用比色法,取1.0 g样品,冰浴研磨匀浆,预冷的80%乙醇清洗3次,加入10 mL预冷的提取液,于4℃放置提取10 min,4℃、12 000g离心10 min,收集上清液为粗酶液。
取2支25 mL具塞刻度试管,每支试管中都分别加入1.0 mL 50 mmol/L、pH 5.5乙酸-乙酸钠缓冲液和0.5 mL 10 g/L多聚半乳糖醛酸溶液,在其中一支試管中加入0.5 mL酶提取液,另一支试管中加入0.5 mL经煮沸5 min的酶提取液作为对照,混匀后置于37℃水浴1 h。然后迅速加入1.5 mL 3.5-二硝基水杨酸试剂,在沸水浴中加热5 min,迅速冷却至室温,以蒸馏水稀释至25 mL刻度处混匀。在波长540 nm处测定吸光光度值,重复3次。
1.2.3 CgPG基因家族成员的鉴定 以拟南芥PG蛋白的氨基酸序列作为种子序列在柑橘基因组数据库(https: //www.citrusgenomedb.org/)柚分类中进行BLASTp比对搜索。为保证候选基因没有遗漏,使用柑橘(Citrus sinensis)PG蛋白和搜索到的PG蛋白的氨基酸序列在柚基因组数据库进行二次BLASTp比对搜索。利用Pfam数据库(http://pfam.Janelia.org/)、NCBI保守结构域数据库分析候选蛋白的结构域,根据候选蛋白的氨基酸序列是否具有2个及2个以上的PG基因保守结构域进行候选PG基因的筛选鉴定。此外,拟南芥中At4g20050(ORT3)虽不含有PG基因保守结构域但已被证明为PG基因,在柚基因组库中单独进行比对搜索。
1.2.4 CgPG基因结构和进化分析 CgPG基因的序列和基因组信息通过柚基因组数据库(http://citrus.hzau.edu.cn/)获得,并根据CgPG基因的序列信息使用TBtools软件对应到染色体的精确位置。通过ComputepI/Mw software(http:// www.expasy.ch/tools/ pi_tool.html)来预测CgPG蛋白的分子量和等电点。使用MEGA 6.0进行进化树构建,选择Neighbor-Joining Tree模式,Bootstrap method检测重复1000次。利用GSDS网站(http://gsds.cbi. pku.edu.cn/index.php)来分析CgPG基因的外显子-内含子结构。用DNAMAN 5软件和Weblog 3(http://weblogo. berkeley.edu/ logo.cgi)在线工具对CgPG蛋白的氨基酸序列进行比对。
1.2.5 RNA提取及表达分析 RNA提取试剂盒提取RNA,使用带有gDNA Eraser的RNA逆转录试剂盒(TaKaRa)进行逆转录。从蜜柚成熟果实汁胞转录组中筛选有表达的基因,使用Primer 5.0软件在开放阅读框两侧设计特异性引物(表1),引物合成由福州白鲸生物科技有限公司完成。使用罗氏480荧光定量分析仪进行实时定量PCR(qPCR)。使用2???CT方法分析相对表达水平,每个样品设置3个生物学重复,结果进行统计分析及绘图。
1.3数据处理
采用Excel 2010软件进行数据分析,误差分析、相关性和差异显著性分析使用PASW Statistics 18软件。
2 结果与分析
2.1CgPG基因家族的鉴定和注释
根据PG基因的保守结构域,通过对柚基因组数据库的Blast比对66个拟南芥PG基因和48个柑橘PG基因的氨基酸序列进行搜索,在柚基因组中筛选得到26个CgPG基因(表2)。分析CgPG的CDS序列在136(CgPG26)~1107(CgPG18)bp之间。CgPG蛋白的等电点在4.78(CgPG20)~9.90(CgPG7)之间,说明CgPG蛋白的酸碱性差异较大,有的显酸性,有的显碱性。除CgPG26外,25个CgPG基因具有Ⅰ、Ⅱ、Ⅲ、Ⅳ 4个结构域中的2个或2个以上结构域。
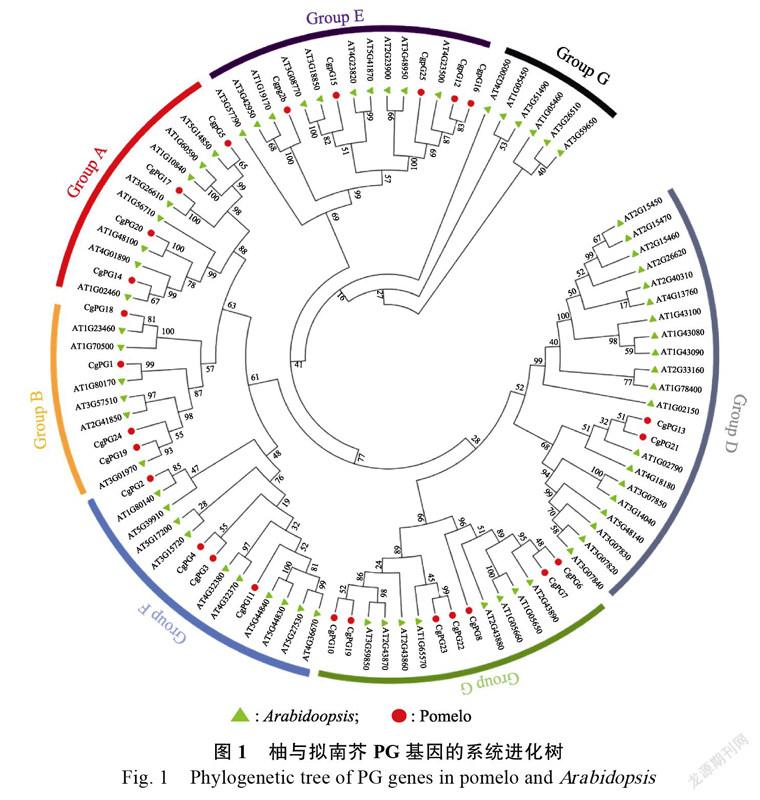
2.2 CgPG家族成员的系统发育分析
根据拟南芥PG家族基因的进化关系,将26个柚PG家族基因聚类为Group A~F 6个亚类(图1)。其中Group A含有柚PG家族基因4个,Group B 4个,Group C 7个,Group D 2个,Group E 5个,Group F 4个,没有发现Group G亚类。柚Group A和Group B中共8个,远小于柑橘的14个[12]。
2.3 CgPG蛋白多重序列比对和保守结构域分析
CgPG蛋白具有4个保守结构域,分别为SPNTDGI、GDDC、CGPGHGISI、RIK。在CgPG蛋白中,部分缺少和缺失CGPGHGISI和RIK保守结构域,也有一些保守结构域中的个别氨基酸发生了变异。在Group E中CgPG15(未知基因)和CgPG26(未知基因)的GDDC结构域分别突变为GDDL和GDDA,且CgPG26的SPNTDGI突变为APNTDGI。亚类E中PG的结构域是不保守的。除此外,其他CgPG蛋白都具有SPNTDGI和GDDC保守结构域(图2)。
2.4 CgPG家族成员的基因组分布
用TBtools中的染色体定位工具对26个CgPG基因在染色体上的位置作图,皆在染色体上准确定位(图3)。除了3号、6号、8号染色体外,其余染色体上皆有CgPG基因。1号、2号、4号、5号、7号、9号染色体上分别定位了3条、7条、2条、6条、3条、5条CgPG基因。其中,2号染色体上分布的CgPG基因最多。
2.5汁胞粒化率
柚成熟果实汁胞分为正常、凝胶、粒化3种。正常汁胞汁水饱满、透明澄澈。凝胶汁胞间有明显絮状浑浊、整体较澄澈。粒化汁胞表面硬实皱缩且发黄、絮状浑浊聚集成束[13](圖4)。不易粒化果汁胞在果实成熟和贮藏期30 d和60 d皆未出现粒化现象,逐渐软化,直至腐败。易粒化果汁胞采收期(花后180 d)蜜柚汁胞开始出现轻度粒化现象,粒化与凝胶汁胞占总汁胞的1.63%,随贮藏时间的延长,粒化程度逐渐升高,贮藏30 d,粒化与凝胶汁胞占总汁胞7.81%,贮藏60 d后处于凝胶和粒化状态汁胞达到20.2%,此时果实汁胞进入粒化的初级阶段(图5)。
2.6 PG酶活性
不易粒化果和易粒化果汁胞在各个时期PG活性趋势一致。在柚果实生长、成熟和贮藏过程中,PG活性一直上升,贮藏60 d急剧上升,达到最大值。在各个时期中,不易粒化果的汁胞PG活性都较易粒化果高,且差异显著(图6)。
2.7 CgPG基因的表达量分析
对成熟果实汁胞转录组中有表达的6个基因CgPG13、CgPG12、CgPG26、CgPG5、CgPG1和CgPG24于花后120 d、花后150 d、花后180 d(正常采收期)、贮藏30 d和贮藏60 d在不易粒化果汁胞和易粒化果汁胞进行了表达分析(图7)。
6个CgPG基因在果实成熟期表达。其中CgPG13、CgPG12、CgPG26在易粒化果中随着果实成熟基因表达量上调,贮藏30 d基因表达达到峰值,之后表达量显著下降。CgPG5、CgPG1、CgPG243个基因在不易粒化果中贮藏60 d的汁胞软化中显著上调。
3 讨论
PG基因表达于花、花粉、成熟果实和器官脱落与开裂部位[10]。植物PG基因由PG基因家族的5个共同祖先基因编码,已在拟南芥、猕猴桃、桃、番茄等部分植物中得到鉴定[4, 14]。
柚基因组中鉴定出26个CgPG基因,相对于其他植物较少,如拟南芥66个、柑橘48个、桃45个、苹果85个和梨45个等。进化树分析中,柑橘Group A~Group F中分别有5、9、2、12、3个PG基因家族成员,而柚分别有4、4、7、2、5、4个家族成员。在2和5号染色体上都有5~6条基因组成的簇状的分布,重复的PG基因序列非常相似,但可能有不同的表达模式。这些基因产生于植物进化过程中的串联复制等复制事件[3]。
在PG基因聚类分析中,一些A、B、C、D和E亚类PG基因在果实成熟过程中表达[14-15]。本研究发现A亚类CgPG5和B亚类CgPG1、CgPG24基因、D亚类的CgPG13和E亚类的CgPG12、CgPG26与果实成熟相关。
PG同工酶1(PG1)和PG同工酶2(PG2)的催化亚基在成熟过程中与果胶酯酶协同作用参与细胞壁代谢,特别是多糖降解,PG2是果实成熟期间果胶代谢中涉及的主要酶[4, 16-18]。在草莓软化过程中,下调了成熟贮藏中表达最多的2个PG基因FaPG1和FaPG2,转基因成熟果实比野生型硬度更高[19]。本研究中,不易粒化果实贮藏后期汁胞高度软化,汁胞中PG酶活性迅速上升,CgPG5、CgPG1、CgPG24 3个基因在软化汁胞型小溪果实贮藏60 d显著升高,表明3个基因可能参与了易粒化果的汁胞软化和后期腐败。其中CgPG1和CgPG24结构相似,且CgPG24与番茄PG2预测功能一致,CgPG1和CgPG24与柚果实软化相关。A亚类的CgPG5果实成熟过程中没有表达,只是在果实贮藏后期的汁胞腐败和软化时期高表达,可能是汁胞进入衰败时期的标志。
研究表明,D类和E类PG基因在多个器官中普遍表达,但主要表达在花序中[20]。本研究中,D亚类的CgPG13和同属E亚类的CgPG12、CgPG26基因在易粒化果成熟软化过程中高表达,贮藏30 d达到峰值,而当汁胞进入粒化初级阶段时,3个基因表达呈下降趋势。虽然贮藏30 d的易粒化果实并未表现出如软化果实般的显著软化,但较采后果实口感柔和,表明3个基因可能只是参与了易粒化果成熟过程中的汁胞暂时软化。这种暂时软化正好是柚汁胞食用的最佳时期。说明易粒化果汁胞粒化前的软化与番茄的软化并不一样,可能与易粒化果粒化之前的汁胞软化和调节细胞壁稳态相关。在Group E中柚CgPG26的GDDC结构域分别突变为GDDA,SPNTDGI突变为APNTDGI。亚类E中PG的结构域是不保守的。这种结构域突变或不保守可能是柚易粒化果汁胞暂时软化需要重新选择的一类基因。
本研究发现不易粒化果和易粒化果汁胞都在果实生长、成熟和贮藏过程中PG活性一直上升,贮藏60 d达到最大值,但各时期不易粒化果PG活性显著高于易粒化果。不易粒化果PG活性与CgPG5、CgPG1、CgPG24基因的后期表达基本一致,说明不易粒化果汁胞软化与番茄、桃的软化一样,是由相关PG基因驱动的。而易粒化果PG活性也在上升,这可能与整个果实的汁胞状态有关。贮藏过程中,汁胞粒化逐渐加深,60 d粒化汁胞占3.92%,进入了粒化初级阶段[13]。前人研究表明,果膠主要存在于初生壁中,次生壁仅有少量存在[21-22],还有许多汁胞初生壁果胶还在水解之中。从CgPG13、CgPG12、CgPG26基因的后期表达也可以看出,尽管与其PG活性并不一致,但仍然有一定的表达量。易粒化果PG活性表现依然呈上升趋势,但要显著低于不易粒化果PG活性,说明易粒化果暂时软化是一个复杂的过程,汁胞粒化与果胶降解存在共调控,CgPG基因表达的下降降低了PG活性,并促使汁胞向粒化方向发展,这与前人研究结果一致[19],但汁胞粒化程度的加深是否引起PG活性出现转折点还需要进一步研究。
不易粒化果汁胞向软化方向发展,CgPG5、CgPG1、CgPG24在软化汁胞型软化后期高度表达,是汁胞走向衰败的标志。而易粒化果汁胞则向粒化方向发展,CgPG13、CgPG12、CgPG26在粒化前的暂时软化过程中高度表达。CgPG基因表达呈现的不同趋势表明CgPG基因在不易粒化果和易粒化果中可能属于2个不同的系统,这是一个很有意义的现象,还需要进一步对柚粒化过程中PG对细胞壁的影响机制开展深入研究。
参考文献
- 陆胜民, 席玙芳, 金勇丰, 张耀洲. 植物多聚半乳糖醛酸酶的结构与功能—文献综述[J]. 园艺学报, 1999(6): 369-375.LU S M, XI Y F, JIN Y F, ZHANG Y Z. Structure and function of plant polygalacturonase—a literature review[J]. Acta Horticulture, 1999(6): 369-375. (in Chinese)
- OHASHI T, JINNO J, INOUE Y, ITO S, FUJIYAMA K, ISHIMIZU T. A polygalacturonase localized in the Golgi apparatus in Pisum sativum[J]. Journal of Biochemistry, 2017, 162(3): 193-201.
- MAHMOOD U, FAN Y H, WEI S Y, NIU Y, Li Y H, HUANG H L, CHEN Y L, TANG Z L, LIU L Z, QU C M, ZHANG K, LI J N, LU K. Comprehensive analysis of polygalacturonase genes offers new insights into their origin and functional evolution in land plants[J]. Genomics, 2020, 113(1): 1096-1108.
- KE X B, WANG H S, LI Y, ZHU B, ZANG Y X, HE Y, CAO J S, ZHU Z J, YU Y J. Genome-wide identification and analysis of polygalacturonase genes in Solanum lycopersicum[J]. International Journal of Molecular Sciences. 2018, 19(8): 1-18.
- PARK K C, KWON S J, KIM P H, BUREAU T, Kim N S. Gene structure dynamics and divergence of the polygalacturonase gene family of plants and fungus[J]. Genome, 2008, 51(1): 30-40.
- JIANG C Z, LU F, IMSABAI W, MEIR S, REID M S. Silencing polygalacturonase expression inhibits tomato petiole abscission[J]. Journal of Experimental Botany, 2008, 59(4): 973-979.
- KHAN N, FATIMA F, HAIDER M S, SHAZADEE H, LIU Z, ZHENG T, FANG J. Genome-wide identification and expression profiling of the polygalacturonase (PG) and pectin methylesterase (PME) genes in grapevine (Vitis vinifera L.)[J]. International Journal of Molecular Sciences, 2019, 20(13): 1-17.
- FIGUEROA C R, PIMENTEL P, GAETE-EASTMAN C, MOYA M, HERRARA R, CALIGARI P D S, MOYA-LE?N M A. Softening rate of the Chilean strawberry (Fragaria chiloensis) fruit reflects the expression of polygalacturonase and pectate lyase genes[J]. Postharvest Biology and Technology, 2008, 49(2): 210-220.
- LIN Y C, L I W, SUN Y H, KUMARI S, WEI H R, LI Q, TUNLAYAA S, SEDEROFF R R, CHIANG V L. SND1 transcription factor–directed quantitative functional hierarchical genetic regulatory network in wood formation in Populus trichocarpa[J]. The Plant Cell, 2013, 25(11): 4324-4341.
- YANG Y, YU Y, LIANG Y, ANDERSON C T, CAO J A. Profusion of molecular scissors for pectins: classification, expression, and functions of plant polygalacturonases[J]. Frontiers in Plant Science, 2018, 9: 1208.
- GARRIGA M, MU?OZ C A, CALIGARI P D S, RETAMALES J B. Effect of salt stress on genotypes of commercial (Fragaria ? ananassa) and Chilean strawberry (F. chiloensis)[J]. Scientia Horticulturae, 2015, 195: 37-47.
- SITRIT Y, HADFIELD K A, BENNETT A B, BTADFORD K J, DOWINE A B. Expression of a polygalacturonase associated with tomato seed germination[J]. Plant Physiol 1999, 121(2): 419-428.
- 张振珏, 谢志南, 许文宝. 琯溪蜜柚汁囊分化和粒化过程的解剖学观察[J]. 植物学报, 1999(1): 16-19. ZHANG Z J, XIE Z N, XU W B. Anatomical observation of the sac differentiation and granulation of Guanxi honey pomelo[J]. Acta Botany, 1999(1): 16-19. (in Chinese)
- 葛 廷, 黄 雪, 谢让金. 多聚半乳糖醛酸基因在果树中的研究进展[J]. 植物生理学报, 2019, 55(8): 1075-1088. GE T, HUANG X, XIE R J. Research progress of polygalacturonic acid gene in fruit trees[J]. Acta Plant Physiology, 2019, 55(8): 1075-1088. (in Chinese)
- 李 慧, 丛 郁, 常有宏, 蔺 经, 盛宝龙. 翠冠梨PG基因家族两成员的克隆及其表达与货架期果实软化的关系[J]. 果树学报, 2012, 29(1): 17-23. LI H, CONG Y, CHANG Y H, LIN J, SHENG B L. The relationship between the cloning and expression of two members of the PG gene family of Cuiguan pear and fruit softening during shelf life[J]. Fruit Tree Journal, 2012, 29(1): 17-23. (in Chinese)
- DEAN D P, DANNY C ALEXANDER, ALAN B BENNETT. Molecular cloning of tomato fruit polygalacturonase: analysis of polygalacturonase mRNA levels during ripening[J]. Proceedings of the National Academy of Sciences, 1986, 83(17): 6420-6424.
- PANIAGUA C, RIC-V P, GARCIAG J A, LOPEZC G, BLANCOP R, MUNOZB J, SCHUCKEL J, KNOX J P, MATAS A J, QUESADA M A, POSE S, MERCADO J A. Elucidating the role of polygalacturonase genes in strawberry fruit softening[J]. Journal of Experimental Botany, 2020, 71(22): 7103-7117.
- BIRD C R, SMITH C J S, RAY J A, MOUREAU P, BEVAN M W, BIRD A S, HUGHES S, MORRIS P C, GRIERSON D, SCHUCH W. The tomato polygalacturonase gene and ripening-specific expression in transgenic plants[J]. Plant Molecular Biology1988, 11(5): 651-662.
- RAKESH B, JOE A, GALLAGHER, LEONARDO D, GOMEZ, MAURICE B. Genetic engineering of grass cell wall polysaccharides for biorefining[J]. Plant Biotechnology Journal, 2017, 15(9): 1071-1092.
- QIAN M, ZHANG Y, YAN X Y, HAN M Y, LI J J, LI F, Li FURUI, ZHANG D, ZHAO C P. Identification and expression analysis of polygalacturonase family members during peach fruit softening[J]. International Journal of Molecular Sciences, 2016, 17(11): 1933.
- KERRY H C, DEBRA M. The structure, function, and biosynthesis of plant cell wall pectic polysaccharides[J]. Carbohydrate Research, 2009, 344(14): 1879-1900.
- SELVENDRAN R R. Developments in the chemistry and biochemistry of pectic and hemicellulosic polymers[J]. Journal of Cell Science, 1985, 2: 51-88.
