基于TMT蛋白组学及生物信息学分析牦牛抗冻差异蛋白
2022-04-21贾银海张成福张强姬秋梅黄光云姜辉文信旺黄明光彭夏云吴柱月
贾银海 张成福 张强 姬秋梅 黄光云 姜辉 文信旺 黄明光 彭夏云 吴柱月


摘要:【目的】從蛋白水平阐明牦牛抗寒性能机理,并进一步从营养学角度提高其代谢性能,为牦牛有效抵御外界恶劣气候条件提供科学依据。【方法】应用TMT蛋白组学技术对寒冷季节(1月)和温暖季节(8月)的牦牛抗冻蛋白进行挖掘,并对鉴定到的牦牛抗冻蛋白进行亚细胞定位、结构域、GO功能富集、KEGG信号通路注释、蛋白相互作用等生物信息学分析。【结果】从牦牛耳组织中共鉴定获得21856个肽段(Peptide),其中特有肽段(Unique peptide)序列为18452个,定量获得4519个蛋白,最终筛选出144个差异蛋白,其中上调蛋白89个、下调蛋白55个。144个牦牛抗冻差异蛋白亚细胞定位到7个条目上,分别是细胞核蛋白56个、细胞质蛋白51个、质膜蛋白24个、细胞外蛋白23个、线粒体蛋白18个、细胞骨架蛋白1个和溶酶体蛋白1个;共鉴定到194个结构域。GO功能富集分析结果显示,生物过程主要富集到细胞过程蛋白79个、代谢过程蛋白70个和生物调控蛋白42个等,分子功能主要富集到结合功能蛋白75个和催化活性蛋白64个等,细胞组分主要富集到细胞部分蛋白89个和细胞蛋白89个等。144个牦牛抗冻差异蛋白在KEGG数据库中注释到205条KEGG信号通路,主要涉及核糖体、氮代谢、胞质DNA感受、动物体内生热作用、氧化磷酸化、白细胞介素-17及钙离子信号等通路。牦牛抗冻差异蛋白相互作用网络分析发现L8IHE5的关联度最高,且冷诱导RNA结合蛋白(CIRP)和HSP70结合蛋白在蛋白相互作用网络中具有更多的相互作用关系。【结论】基于TMT蛋白组学对牦牛抗冻差异蛋白进行挖掘,结果鉴定获得144个抗冻差异蛋白(上调蛋白89个,下调蛋白55个),其中CIRP和HSP70在冷应激条件下呈上调趋势,能促使牦牛肌体适应低温环境,可作为牦牛抗冻性育种的候选分子标记。
关键词: 牦牛;TMT蛋白组学;生物信息学;抗冻性;差异蛋白
中图分类号: S823.85 文献标志码: A 文章编号:2095-1191(2022)01-0001-11
TMT proteomics and bioinformatics to analyze differential proteins in cold resistance of yaks
JIA Yin-hai1, ZHANG Cheng-fu2, ZHANG Qiang2, JI Qiu-mei2, HUANG Guang-yun1,JIANG Hui2, WEN Xin-wang1, HUANG Ming-guang1, PENG Xia-yun1, WU Zhu-yue1
(1Animal Husbandry Research Institute of Guangxi/Guangxi Key Laboratory of Livestock Genetic Improvement, Nanning 530001, China; 2 Institute of Animal Science and Veterinary, Tibet Academy of Agricultural and Animal Husbandry Sciences/State Key Laboratory of Hulless Barley and Yak Germplasm Resources and
Genetic Improvement, Lhasa 850000, China)
Abstract:【Objective】To elucidate the mechanism of cold resistance of yak from protein level, and to further improve its metabolic performance from the perspective of nutrition, so as to provide scientific basis for yak to effectively resist harsh climate conditions. 【Method】The yak antifreeze proteins were extracted by TMT proteomics in cold season (January) and warm season(August), and the subcellular localization analysis, domain analysis, GO functional analysis, KEGG signaling pathway annotation analysis , protein interaction were analyzed. 【Result】A total of 21856 peptides were identified, of which 18452 were specific peptides and 4519 proteins were identified. There were 144 subcellular proteins, including 89 up-regulated proteins and 55 down-regulated proteins. 144 yak antifreeze differential proteins were subcellular mapped to 7 items, including 56 nuclear proteins, 51 cytoplasmic proteins, 24 plasma membrane proteins, 23 extracellular proteins, 18 mitochondrial proteins, 1 cytoskeleton protein and 1 lysosomal protein. A total of 194 domains were identified. GO enrichment of function analysis indicated that, biological process mainly enriched to cell protein 79, 70 metabolic process protein and 42 regulation protein, molecular function mainly enriched to combine functional protein 75 and 64 catalytic activity of the protein, cell component part mainly enriched to the 89 cell protein and 89 cell proteins. The 144 yak antifreeze differential proteins were annotated into 205 KEGG signaling pathways in KEGG database, which mainly involved in ribosomes, nitrogen metabolism, cytoplasmic DNA sensing, in vivo thermogenesis, oxidative phosphorylation, interleukin-17 and calcium ion signaling. It was found that L8IHE5 had the highest correlation, and cold-induced RNA-binding proteins(CIRP) and HSP70 binding protein had more interaction in the network. 【Conclusion】According to TMT proteomics, 144 differential antifreeze proteins(89 up-regulated proteins and 55 down-regulated proteins) are identified, among which CIRP and HSP70 are up-regulated under cold stress, which can promote yak body to adapt to low temperature environment, and can be used as candidate molecular markers for yak antifreeze breeding.3000D976-7878-45DE-B639-788EACCAAD63
Key words: yak; TMT proteomics; bioinformatics; cold resistance; differential proteins
Foundation items: Guangxi Science and Technology Major Special Project(Guike AA17204028); Tibet Academy of Agricultural and Animal Husbandry Sciences/State Key Laboratory of Hulless Barley and Yak Germplasm Resources and Genetic Improvement Open-project(XZNKY-2020-C-007K05)
0 引言
【研究意義】牦牛具有很强的抗逆能力,属于世界第三大极地动物,是当今世界未被污染的三大物种(北极熊、企鹅和牦牛)之一。青藏高原的牦牛数量占全球牦牛总量的90%以上。据统计,2018年青藏高原1.2亿ha的高寒草场上约有1600万头牦牛,是世界上重要的牦牛资源库和主要生产基地(郝力仕,2019)。在藏区牧民经济中,牦牛的乳、肉、皮、毛、绒及其加工产品均是其他家畜无法替代;但高原气候条件恶劣,冬季缺乏饲草料,导致牦牛营养不良及个体生产性能下降,严重制约着牦牛产业的可持续发展。低温环境所引起的冷应激,会导致牦牛抵抗能力降低及采食量下降,甚至致使怀孕牦牛流产等(贾银海等,2021)。因此,揭示牦牛抗冷适应能力的作用机理,对保护牦牛品种资源及促进牦牛产业的可持续发展具有重要意义。【前人研究进展】动物在受到冷应激时,其机体通过增加产热以维持体温恒定。在冷应激条件下,肌体会产生一系列变化,通过震颤或非震颤产热(郝力仕,2019)。冷应激不仅影响动物的生产性能,还会对动物的免疫能力及抗氧化能力等造成不同程度的影响(郭爽等,2021;贾银海等,2021)。决定动物抗寒性的因素诸多,包括皮肤表面积、产热性能、蒸发损耗、代谢损耗、脂肪厚度、皮肤厚度、体毛长度和细度及环境湿度等,许多动物在低温条件下可合成重结晶抑制效应的抗冻蛋白,降低体内的体液冰点,从而最大限度地保持体液的液体状态(de Maayer et al.,2014)。至今,有关抗冻蛋白(Antifreeze protein,AFP)的研究主要集中在鱼类(Inglis et al.,2006;Gamham et al.,2008)、昆虫(Meister et al.,2015)、植物(Ji et al.,2017;Sperotto et al.,2018)及菌类(Mu?oz et al.,2017)等物种上,其中又以鱼类的研究最深入(钟其旺和樊廷俊,2002;Martínez-Páramo et al.,2009;Duman,2015)。Kim等(2019)对南极鱼亚目物种基因组中的高度保守核苷酸片段(Conserved nucleotide elements,CNEs)进化模式进行分析,结果发现南极鱼类对血红细胞发生过程的调控是适应低温的一个重要机制;Daane等(2020)研究表明,在长期的低温环境下CNEs存在快速突变功能丢失的趋势;Ren等(2021)从基因层面开展研究,发现通路MAPK在鱼类低温应激中占据重要位置,且可能与其他重要的通路如TGF-Bata等存在复杂联系。在高寒地区,植物在面对低温环境也会产生抗寒机制的产物,但其含量远低于鱼类的抗冻蛋白(Ji et al.,2017)。在大部分昆虫体内都存在抗冻蛋白,其含量较鱼类抗冻蛋白高10~100倍,且其蛋白结构存在明显差异(Meister et al.,2015)。此外,有研究已将抗冻蛋白添加至化妆品中,通过皮肤吸收能起到保护皮肤、防止皮肤冻伤的作用,即可用于高级防冻化妆品(Evans and Fletcher,2004;巩子路,2015)。可见,抗冻蛋白对于不同物种发挥抗冷性能均有重要意义。【本研究切入点】不同物种为适应外界环境条件的变化,通常会产生应对环境条件变化的相关产物,目前国内外针对抗冻蛋白已有深入研究(钟其旺和樊廷俊,2002;Martínez-Páramo et al.,2009;Duman,2015),但有关我国牦牛抗逆性及其抗冻蛋白方面的研究鲜见报道。【拟解决的关键问题】基于TMT(Tandem mass tag)蛋白组学及生物信息学分析对牦牛抗冻蛋白进行筛选研究,旨在阐明牦牛抗寒性能机理,并进一步从营养学角度提高其代谢性能,为牦牛有效抵御外界恶劣气候条件提供科学依据。
1 材料与方法
1. 1 试验材料
供试牦牛由西藏农牧科学院畜牧兽医研究所改则县古姆乡和斯布牦牛养殖基地提供,分别于气候寒冷的冬春季(1月)和气候温和的夏季(8月)采集6~8岁经产牦牛耳组织样品,各8头,-80 ℃低温保存备用。TMT分子标记试剂盒购自美国ThermoFisher公司,BCA试剂盒购自南京诺威赞生物科技股份有限公司,乙腈和超纯水购自Fisher Chemical公司,SDS、三氟乙酸、碘代乙酰胺、二硫苏糖醇(DTT)、尿素及四乙基溴化铵等试剂购自Sigma公司。
1. 2 蛋白提取和肽段酶解
分别设寒冷季节组和温暖季节组,其中,寒冷季节组3个重复(CON-1、CON-2和CON-3),温暖季节组3个重复(T-1、T-2和T-3)。采用SDT裂解法(4% SDS,100 mmol/L Tris/HCl,0.1 mol/L DTT,pH 7.6)提取蛋白,取适量蛋白使用FASP(Filter aided proteome preparation)进行胰蛋白酶酶解(Wi?niewski et al.,2009),并以C18 Cartridge对肽段进行脱盐;冻干后的肽段加入40 μL 0.1%甲酸溶液复溶,应用TMT标记不同样品中的同一蛋白,再以BCA法进行定量分析(贾银海,2018)。3000D976-7878-45DE-B639-788EACCAAD63
1. 3 TMT分子标记
取100 μg肽段按TMT分子標记试剂盒说明分别对各样品进行标记(葛泽勇,2018)。RP(反相)分级:采用High pH Reversed-Phase Peptide Fractiona-tion Kit对TMT标记的每组肽段等量混合后进行分级(葛泽勇,2018)。以乙腈和0.1%三氟乙酸(TFA)进行色谱柱平衡,将TMT标记肽段混合样品上样,低速离心脱盐,然后依次用不同梯度浓度的高pH乙腈溶液对结合肽段进行梯度洗脱。真空干燥后,以12 μL 0.1%脂肪酸(FA)进行复溶,于280 nm处测定肽段浓度。阳离子交换柱(SCX)分级:采用AKTA Purifier 100对TMT标记的每组肽段混合后进行分级(葛泽勇,2018)。缓冲液A液[10 mmol/L KH2PO4,25%乙腈,pH 3.0],B液(10 mmol/L KH2PO4,500 mmol/L KCl,25%乙腈,pH 3.0)。A液平衡色谱柱,以1 mL/min的流速经色谱柱进行分离。B液梯度如下:0%,25 min;0%~10%,25~32 min;10%~20%,32~42 min;20%~45%,42~47 min;45%~100%,47~52 min;100%,52~60 min;60 min后B液重设为0%(蒋少秋,2020)。洗脱过程监测214 nm处的吸光值(OD214),每隔1 min收集1次洗脱组分,冻干后采用C18 Cartridge进行脱盐(蒋少秋,2020)。
1. 4 LC-MS/MS数据采集
采用纳升流速的HPLC液相系统Easy nLC对每份样品进行分离(张弛,2020)。缓冲液A液为0.1%甲酸水溶液,B液为0.1%甲酸乙腈水溶液(乙腈为84%)。95%的A液平衡色谱柱,样品通过进样器上样至C18反相分析柱(Thermo Scientific Acclaim PepMap100,100 μm×2 cm,nanoViper C18),采用B液线性梯度分离,以300 nL/min的流速流经分析柱(Thermo Scientific EASY column,10 cm,ID75 μm,3 μm,C18-A2),再用Q-Exactive质谱仪进行质谱分析,并采集数据(杨重晖,2020)。
1. 5 蛋白鉴定及定量分析
采用Mascot 2.2和Proteome Discoverer 1.4对质谱分析原始数据进行鉴定及定量分析(贾银海,2018)。
1. 6 生物信息学分析
1. 6. 1 亚细胞定位分析 采用CELLO(http://cello.life.nctu.edu.tw/)进行亚细胞定位预测,即以多重支持向量机(multi-class SVM)的机器学习方法对公共数据库中已知亚细胞定位信息的蛋白序列数据进行建模,预测待检索蛋白亚细胞定位情况。
1. 6. 2 蛋白结构域分析 使用Pfam数据库的InterProScan数据包,采用运行扫描算法对蛋白序列进行功能表征预测,以获得目标蛋白序列在Pfam数据库中的结构域注释信息。
1. 6. 3 GO功能富集分析 利用Blast2GO对目标蛋白集合进行GO功能富集分析,具体分析过程可归纳为序列比对(BLAST)、GO条目提取(Mapping)、GO注释(Annotation)及InterProScan补充注释(Annotation augmentation)等(贾银海,2018)。
1. 6. 4 KEGG信号通路注释分析 利用KAAS(KEGG automatic annotation server)对目标蛋白集合进行KEGG信号通路注释分析(贾银海,2018)。
1. 6. 5 蛋白相互作用网络分析 依据IntAct(http://www.ebi.ac.uk/intact/main.xhtml)或STRING(http://string-db.org/)数据库分析蛋白间的连接数,预测蛋白与蛋白间的相互作用关系,同时以CytoScape 3.2.1绘制蛋白相互作用网络结构图并进行分析(贾银海,2018)。
1. 6. 6 蛋白层次聚类分析 首先对目标蛋白集合的定量信息进行归一化处理,即归一化至(-1,1)区间;使用ComplexHeatmap Version 3.4从抗冻差异蛋白样本及其表达量2个维度进行分类,并生成层次聚类热图(杨重晖,2020)。
2 结果与分析
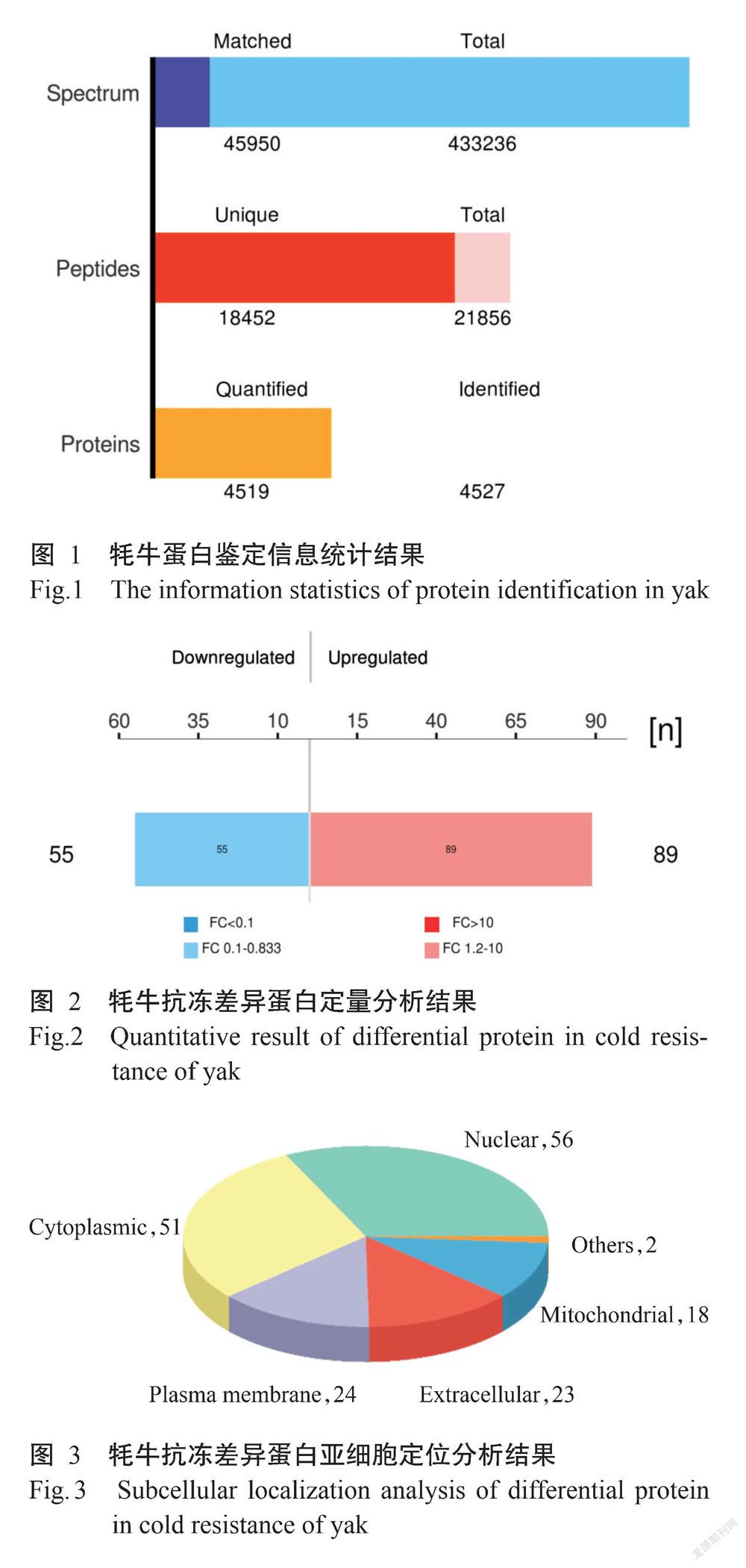
2. 1 牦牛抗冻蛋白鉴定结果
由图1和图2可知,经Mascot 2.2鉴定和Proteome Discoverer 1.4定量分析共获得433236张总谱图(Total spectra)。其中蛋白谱图(Spectra)45950张;肽段(Peptide)数量为21856个,其中特有肽段(Unique peptide)数量为18452个;蛋白(Protein)数量为4527个,定量到的蛋白数量为4519个。对牦牛抗冻差异蛋白进行筛选,结果筛选获得144个差异蛋白,其中上调蛋白89个、下调蛋白55个。
2. 2 亚细胞定位预测结果
CELLO亚细胞定位预测结果表明,144个牦牛抗冻差异蛋白定位到7个条目上(图3),分别是细胞核(Nuclear)蛋白56个、细胞质(Cytoplasmic)蛋白51个、质膜(Plasma membrane)蛋白24个、细胞外(Extracellular)蛋白23个、线粒体(Mitochondrial)蛋白18个及其他蛋白2个[细胞骨架(Cytoskeleton)蛋白1个和溶酶体(Lysosome)蛋白1个]。
2. 3 蛋白结构域分析结果
通过评价鉴定获得蛋白在某个结构域条目下的富集水平及对应的差异蛋白,对牦牛抗冻差异蛋白进行结构域分析,共鉴定到194个结构域,排名前20位的结构域分析结果如图4所示。其中,Uteroglobin family(子宫珠蛋白家族)、Copper/zinc supero-xide dismutase(SODC)(铜/锌超氧化物歧化酶)、Zn-finger in Ran binding protein and others(含锌指 Ran 结合结构域蛋白)及Acetyltransferase(GANT)family(C-酰胺化酶家族)是机体参与抗冻应答的相关蛋白。3000D976-7878-45DE-B639-788EACCAAD63
2. 4 GO功能富集分析结果
对牦牛抗冻差异蛋白进行GO功能富集分析,结果(图5)显示,生物过程(Biological process)主要富集到细胞过程(Cellular process)蛋白79个、代谢过程(Metabolic process)蛋白70个和生物调控(Biological regulation)蛋白42个等,分子功能(Molecular function)主要富集到结合功能(Binding)蛋白75个和催化活性(Catalytic activity)蛋白64个等,细胞组分(Cellular component)主要富集到细胞部分(Cell part)蛋白89个和细胞(cell)蛋白89个等。在生物过程中,Cellular carbohydrate metabolic process(细胞碳水化合物代谢过程)上调,而Energy reserve metabolic process(能量储备代谢过程)、Oxidation-reduction process(氧化还原反应过程)、Cellular glucan metabolic process(细胞糖代谢过程)、Regulation of glucose metabolic process(细胞糖代谢调控过程)及Cellular carbohydrate catabolic process(细胞碳水化合物分解代谢过程)等条目下调;在分子功能中,Oxidoreductase activity(氧化还原酶活性)上调,而Antioxidant activity(抗氧化活性)、Transaminase activity(转氨酶活性)及Catalytic activity(催化活性)等条目下调;在细胞组分中,Integral component of membrane(膜的整体组分)及Intrinsic component of mem-brane(膜的内在成分)等条目上调,而Mitochondrial outer membrane(线粒体外膜)、Postsynapse(突触后膜)、Nuclear outer membrane-endoplasmic reticulum membrane network(核外膜—内质网膜网络)等条目下调。
2. 5 KEGG信号通路注释分析结果
根据KEGG数据库对牦牛抗冻差异蛋白进行分类注释,结果显示共注释到205条KEGG信号通路。表1为注释获得的前10条KEGG信号通路,主要涉及核糖体、氮代谢、胞质DNA感受、动物体内生热作用、氧化磷酸化、白细胞介素-17及钙离子信号等通路。
2. 6 牦牛抗冻差异蛋白相互作用网络分析结果
由CytoScape 3.2.1绘制的牦牛抗冻差异蛋白相互作用网络结构图(图6)可看出,关联度排名前10位的抗冻差异蛋白为L8IHE5、L8I961、A0A6B0RV13、A0A6B0RUX6、A0A6B0QW44、A0A6B0QZV0、L8I1Z6、A0A6B0RR94、A0A6B0RJ65和L8IPU4,关联度最高的是L8IHE5。其中,A0A6B0RV13为冷诱导RNA结合蛋白(Cold inducible RNA binding protein,CIRP),L8I1Z6为HSP70结合蛋白。CIRP和HSP70蛋白在冷应激条件下均呈上调趋势,可作为牦牛抗冻性育种的候选蛋白基因。
2. 7 牦牛抗冻差异蛋白的表达量聚类分析结果
由图7可知,对6个样品组的抗冻差异蛋白定量数据(样本维度和表达量维度)进行分层聚类分析,每列代表1组样品(横坐标为样品信息),每行代表1个蛋白(纵坐标为显著差异表达蛋白),其中,红色代表显著上调蛋白,蓝色代表显著下调蛋白,灰色部分代表无蛋白信息。分层聚类分析结果显示:寒冷季节组(CON-1、CON-2和CON-3)牦牛抗冻差异蛋白表达量的上调和下调趋势较一致,CON-2和CON-3先聚为一类,再与CON-1聚为一类;温暖季节组(T-1、T-2和T-3)牦牛抗冻差异蛋白表达量的上调或下调趋势与寒冷季节的基本一致,T-2和T-3先聚为一类,再与T-1聚为一类,最终形成与寒冷季节组(CON-1、CON-2和CON-3)明显不同的簇。
3 讨论
TMT是Thermo Scientific公司推出的一种应用广泛、基于體外同位素标记的相对与绝对定量蛋白组学技术,利用同位素试剂标记蛋白酶解后产生的多肽,可同时比对分析多达16种样品间的蛋白表达量(Ross et al.,2004)。TMT标记的不同样本中同一蛋白均表现出相同的质荷比,等量混匀后,经高质量、高精度和高分辨率的质谱分析,可保持良好的质量偏差,其鉴定结果准确可靠;经数据库分析即可获得不同样本中各蛋白的相对定量比值,进而筛选出差异表达蛋白。本研究采用TMT结合液相色谱和串联质谱联用的方法成功对牦牛耳组织蛋白进行分离和鉴定,结果鉴定获得特有肽段(Unique peptide)序列18452个,鉴定获得4527个蛋白(Protein),其中定量到的蛋白数量为4519个。可见,TMT可标记所有肽段,适用于任何类型的样本鉴定,具有高通量的特点,且可信度高。
本研究应用TMT蛋白组学技术对寒冷季节和温暖季节的牦牛抗冻蛋白进行挖掘,并对鉴定到的牦牛抗冻蛋白进行亚细胞定位、结构域、GO功能富集及KEGG信号通路注释等生物信息学分析。在亚细胞定位分析中,144个牦牛抗冻差异蛋白共鉴定到7个条目上,分别是细胞核蛋白56个、细胞质蛋白51个、质膜蛋白24个、细胞外蛋白23个、线粒体蛋白18个、细胞骨架蛋白1个及溶酶体蛋白1个。在结构域分析中,共鉴定到194个结构域,其中,子宫珠蛋白家族上调,可能与动物肌体防御外界不同环境条件变化,促进肺组织发育及提高自身免疫力有关(厉双慧等,2020);铜/锌超氧化物歧化酶和C-酰胺化酶家族下调,则与高寒缺氧条件下机体内摄入的抗氧化营养素(铜和锌)不足,不能维持自由基低浓度的动态平衡,新陈代谢功能及抵抗力降低有关,与贾银海等(2021)研究牦牛毛微量元素与其抗寒性能的关联分析结果一致。GO功能富集分析结果显示,在生物过程中,细胞碳水化合物代谢过程上调,而能量储备代谢过程、氧化还原反应过程、细胞糖代谢过程、细胞糖代谢调控过程及细胞碳水化合物分解代谢过程等下调;在分子功能中,氧化还原酶活性上调,而抗氧化活性、转氨酶活性及催化活性等下调;在细胞组分中,膜的整体组分和膜的内在成分等上调,线粒体外膜、突触后膜及核外膜—内质网膜网络等则下调。根据KEGG数据库对牦牛抗冻差异蛋白进行分类注释,结果共注释到205条KEGG信号通路,主要涉及核糖体、氮代谢、胞质DNA感受、动物体内生热作用、氧化磷酸化、白细胞介素-17及钙离子信号等通路。3000D976-7878-45DE-B639-788EACCAAD63
本研究还鉴定出冷诱导RNA结合蛋白(CIRP),该蛋白是在哺乳动物细胞内首个发现的冷休克蛋白,由172个氨基酸残基组成,含有2个明显的结构域(Lee et al.,2015)。已有研究表明,缺氧、紫外线辐射、缺糖、热应激和过氧化氢均可调节大鼠血清中CIRP的表达,提示CIRP是一种应激反应蛋白(Lleonart,2010)。在应激条件下,肠系膜细胞可从细胞核迁移至细胞质,通过其靶基因3'端的结合位点调节mRNA稳定性(Sakurai et al.,2006)。CIRP参与多种细胞过程,包括细胞增殖、细胞存活、昼夜节律调节、端粒维持及肿瘤的形成和发展(Lleonart,2010)。CIRP在25 ℃下被诱导产生(Sakurai et al.,2006),特别是在低温、缺氧和强紫外线的胁迫下,其表达量显著增加,表明冷诱导产生的CIRP能促使牦牛肌体适应低温环境(Al-Fageeh and Smales,2009)。
热休克蛋白(HSPs)是由细胞核内高度保守的热应激基因所编码(Lanneau et al.,2008)。HSPs作为蛋白分子伴侣存在,参与细胞内蛋白肽链空间结构的形成,促进蛋白向内质网移动,对调节细胞稳态具有重要作用(黄建芳等,2015;伍钢等,2020)。此外,HSPs能被冷应激过后的复温过程所诱导(Lanneau et al.,2008)。在冷应激条件下HSPs能迅速表达,因此可作为冷应激反应的分子生物标记。本研究从牦牛耳组织蛋白中鉴定获得HSP70、HSP90、HSP110和HSP27,尤其是HSP70当肌体受到冷应激时会显著升高,与其能与细胞内其他分子以不稳定的结构结合有关。
4 结论
基于TMT蛋白组学对牦牛抗冻差异蛋白进行挖掘,结果鉴定获得144个抗冻差异蛋白(上调蛋白89个,下调蛋白55个),其中CIRP和HSP70在冷应激条件下呈上调趋势,能促使牦牛肌体适应低温环境,可作为牦牛抗冻性育种的候选分子标记。
参考文献:
葛泽勇. 2018. 基于TMT技术对湖羊高低活力精子的差异蛋白组分析[D]. 合肥:安徽农业大学. [Ge Z Y. 2018. Proteome analysis of sperm between high- and low-motility in Hu sheep using TMT approach[D]. Hefei:Anhui Agriculture University.] doi:10.26919/d.cnki.gannu.2018.000031.
巩子路. 2015. 新疆冷水鱼中抗冻蛋白的分离纯化与结构鉴定[D]. 石河子:石河子大学. [Gong Z L. 2015. Separation,purification and primary structure analysis of antifreeze protein of Xinjiang cold water fish[D]. Shihezi:Shihezi University.] doi:10.7666/d.D717855.
郭爽,孟繁宇,徐熙亮,杨健,李亚东,王煦,王洪宇. 2021. 冷应激对奶牛生产性能的影响及其防控措施概述[J]. 天津农业科学,27(3):61-64. [Guo S,Meng F Y,Xu X L,Yang J,Li Y D,Wang X,Wang H Y. 2021. Effects of the cold stress on production in dairy cow and its prevention and control measures[J]. Tianjin Agricultural Sciences,27(3):61-64.] doi:10.3969/j.issn.1006-6500.2021.03.013.
郝力仕. 2019. 牦牛暖季补饲对改善肉品质的作用及机理研究[D]. 兰州:兰州大学. [Hao L S. 2019. Effect of supplementary feeding in warm season on meat quality of yaks and underlying mechanisms[D]. Lanzhou:Lanzhou University].
黄建芳,鄢胜飞,吴敏,陈秋明,周亭亭,蒋钦杨,郭亚芬,兰干球. 2015. 陆川猪热休克蛋白90基因的克隆与序列分析[J]. 南方农业学报,46(8):1505-1510. [Huang J F,Yan S F,Wu M,Chen Q M,Zhou T T,Jiang Q Y,Guo Y F,Lan G Q. 2015. Gene cloning and sequence analysis of heat shock protein 90 in Luchuan pig[J]. Journal of Sou-thern Agriculture,46(8):1505-1510.] doi:10.3969/j:issn. 2095-1191.2015.08.1505.
賈银海,张成福,姬秋梅,黄光云,张强,姜辉,黄明光,滕少花,朱文,吴柱月. 2021. 牦牛毛微量元素含量与其抗寒性能得关联性分析[J]. 中国牛业科学,47(1):7-9. [Jia Y H,Zhang C F,Ji Q M,Huang G Y,Zhang Q,Jiang H,Huang M G,Teng S H,Zhu W,Wu Z Y. 2021. Study on relationship between the content of trace elements and cold resistance of yak[J]. China Cattle Science,47(1):7-9.]3000D976-7878-45DE-B639-788EACCAAD63
贾银海. 2018. 基于唾液蛋白组学开发鉴定水牛发情方法的研究[D]. 南宁:广西大学. [Jia Y H. 2018. Research on the estrus identification methods of the development based on the salivary proteomics in the buffalo[D]. Nanning:Guangxi University]. doi:10.7666/d.Y3537256.
蒋少秋. 2020. 基于iTRAQ技术对CNV的蛋白组学研究及TAB1对CNV的保护作用[D]. 重庆:重庆医科大学. [Jiang S Q. 2020. Research based on iTRAQ technology in CNV rat and protective role of TAB1 in CNV[D]. Chong-qing:Chongqing Medical University.] doi:10.27674/d.cnki. gcyku.2020.000131.
厉双慧,陈媚媚,柳广南,李文涛. 2020. 子宫珠蛋白相关蛋白1在肺癌中的研究进展[J]. 广西医科大学学报,37(1):141-144. [Li S H,Chen M M,Liu G N,Li W T. 2020. Research progress of uteroglobin-related protein 1 in lung cancer[J]. Journal of Guangxi Medical University,37(1):141-144.] doi:10.16190/j.cnki.45-1211/r.2020.01.025.
伍钢,肖鹏,彭侃霖,吴青青,王安平,郑文亚. 2020. HSP90、HSP70、HSP27在不同发育时期猫卵巢的表达规律研究[J]. 黑龙江畜牧兽医,(17):150-152. [Wu G,Xiao P,Peng K L,Wu Q Q,Wang A P,Zheng W Y. 2020. The expression of HSP90,HSP70 and HSP27 in cat ovary at different developmental stages[J]. Heilongjiang Animal Science and Veterinary Medicine,(17):150-152.] doi:10.13881/j.cnki. hljxmsy.2020.03.0928.
杨重晖. 2020. 中药鹿茸分子鉴别研究及鹿茸蛋白差異性研究[D]. 长春:长春中医药大学. [Yang C H. 2020. Study on the molecular identification of antler and the difference of antler protein[D]. Changchun:Changchun University of Chinese Medicine.] doi:10.26980/d.cnki.gcczc.2020.00 0122.
张弛. 2020. 猪细小病毒荧光定量PCR检测方法的建立及其感染PK-15细胞后的蛋白质组学研究[D]. 郑州:河南农业大学. [Zhang C. 2020. Establishment of a real-time fluorescent quantitative PCR detection method for Porcine parvovirus and the proteomics study of infected PK-15 cells[D]. Zhengzhou:Henan Agriculture University.] doi:10.27117/d.cnki.ghenu.2020.000196.
钟其旺,樊廷俊. 2002. 鱼类抗冻蛋白的研究进展[J]. 生物化学与生物物理学报,34(2):124-130. [Zhong Q W,Fan T J. 2002. Advances in fish antifreeze protein research[J]. Acta Biochimica et Biophysica Sinica,34(2):124-130.] doi:10.1007/BF02943277.
Al-Fageeh M B,Smales C M. 2009. Cold-inducible RNA bin-ding protein (CIRP) expression is modulated by alternative mRNAs[J]. RNA,15(6):1164-1176. doi:10.1261/rna.1179109.
Daane J M,Auvinet J,Stoebenau A,Yergeau D,Harris M P,Detrich H W. 2020. Developmental constraint shaped genome evolution and erythrocyte loss in Antarctic fishes following paleoclimate change[J]. PLoS Genetics,16(10):e1009173. doi:10.1371/journal.pgen.1009173.
de Maayer P,Anderson D,Cary C,Cowan D A. 2014. Some like it cold:Understanding the survival strategies of psychrophiles[J]. EMBO Reports,15(5):508-517. doi:10. 1002/embr.201338170.3000D976-7878-45DE-B639-788EACCAAD63
Duman J G. 2015. Animal ice-binding(antifreeze) proteins and glycolipids:An overview with emphasis on physiological function[J]. The Journal of Experimental Biology,218(12):1846-1855. doi:10.1242/jeb.116905.
Evans R P,Fletcher G L. 2004. Isolation and purification of antifreeze proteins from skin tissues of snailfish,cunner and sea raven[J]. Biochimica et Biophysica Acta,1700(2):209-217. doi:10.1016/j.bbapap.2004.05.006.
Gamham C P,Gilbert J A,Hartman C P,Campbell R L,Laybourn-Parry J,Davies P L. 2008. A Ca2+-dependent bacterial antifreeze protein domain has a novel beta helical ice-binding fold[J]. Biochem Journal,411(1):171-180. doi:10.1042/BJ20071372.
Inglis S R,McGann M J,Price W S,Harding M M. 2006. Diffusion NMR studies on fish antifreeze proteins and synthetic analogues[J]. FEBS Letters,580(16):3911-3915. doi:10.1016/j.febslet.2006.06.022.
Ji L,Zhou P,Zhu Y,Liu F,Li R B,Qiu Y F. 2017. Proteomic analysis of rice seedlings under cold stress[J]. The Protein Journal,36(4):299-307. doi:10.1007/s10930-017-9721-2.
Kim B M,Amore S A,Kang S,Ahn D H,Kim J H,KimI C,Lee J H,Lee S G,Lee H,Lee J,Kim H W,Desvigens T,Batzal P,Sydes J,Titus T,Wilson C A,Catchen J M,Warren W C,Schartl M,Detrich H W,Postlethwait J H,Park H. 2019. Antarctic blackfin icefish genome reveals adaptations to extreme environments[J]. Nature Ecology & Evolution,3(3):469-478.doi:10.1038/s41559-019-0812-7.
Lanneau D,Brunet M,Frisan E,Solary E,Fontenay M,Garrido C. 2008. Heat shock proteins:Essential proteins for apoptosis regulation[J]. Journal of Cellular and Molecular Medicine,12(3):743-761. doi:10.1111/j.1582-4934. 2008.00273.x.
Lee H N,Ahn S M,Jang H H. 2015. Cold-inducible RNA-binding protein,CIRP,inhibits DNA damage-induced apo-ptosis by regulating p53[J]. Biochemical and Biophysical Research Communications,464(3):916-921. doi:10.1016/ j.bbrc.2015.07.066.
Lleonart M. 2010. A new generation of proto-oncogenes:Cold-inducible RNA binding proteins[J]. Biochimica et Biophysica Acta,1805(1):43-52. doi:10.1016/j.bbcan. 2009.11.001.
Martínez-Páramo S,Barbosa V,Pérez-Cerezales S,Robles V,Herráez M P. 2009. Cryoprotective effects of antifreeze proteins delivered into zebrafish embryos[J]. Cryobiology,58 (2):128-133. doi:10.1016/j.cryobiol.2008.11.013.
Meister K,Lotze S,Olijve L L C,Devries A L,Duman J G,Voets I K,Bakker H J. 2015. Investigation of the ice-binding site of an insect antifreeze protein using sum-frequency generation spectroscopy[J]. The Journal of Physical Chemistry Letters,6(7):1162-1167. doi:10.1021/acs.jpclett.5b00281.3000D976-7878-45DE-B639-788EACCAAD63
Mu?oz P A,Márquez S L,González-Nilo F D,Márquez-Miranda V,Blamey J M. 2017. Structure and application of antifreeze proteins from Antarctic bacteria[J]. Microbial Cell Factories,16:210-216. doi:10.1186/s12934-017-0737-2.
Ren J,Long Y,Liu R,Song G L,Li Q,Cui Z B. 2021. Cha-racterization of biological pathways regulating acute cold resistance of zebrafish[J]. International Journal of Mole-cular Sciences,22(6):3028. doi:10.3390/ijms22063028.
Ross P L,Huang Y N,Marchese J N,Williamson B,Parker K,Hattan S,Khainovski N,Pillai S,Daniels S,Purkayastha S,Juhasz P,Martin S,Bartlet-Jones M,He F,Jacobson A,Pappin D J. 2004. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents[J]. Molecular & Cellular Proteomics,3(12):1154-1169. doi:10.1074/mcp.M400129-MCP200.
Sakurai K,Itoh H,Higashitsuji H,Nonoguchi K,Liu Y,Watanabe H,Nakano T,Fukumoto M,Chiba T,Fujita J. 2006. CIRP protects against tumor necrosis factor-alphainduced apoptosis via activation of extracellular signal-regulated kinase[J]. Biochimica et Biophysica Acta,1763(3):290-295. doi:10.1016/j.bbamcr.2006.02.007.
Sperotto R A,de Araujo Junior A T,Adamski J M,Cargnelutti D,Ricachenevsky F K,de Oliveira B H N,da Cruz R P,dos Santos R P,da Silva L P,Fett J P. 2018. Deep RNAseq indicates protective mechanisms of cold-tolerant indica rice plants during early vegetative stage[J]. Plant Cell Reports,37(2):347-375. doi:10.1007/s00299-017-2234-9.
Wi?niewski J R,Zougman A,Nagaraj N,Mann M. 2009. Universal sample preparation method for proteome analysis[J]. Nature Methods,6(5):359-362. doi:10.1038/nmeth. 1322.
(責任编辑 兰宗宝)3000D976-7878-45DE-B639-788EACCAAD63
