基于生物信息学方法构建重度抑郁症发病的miRNA-mRNA调控网络
2022-04-20李果张青萍刘永辉吴鹏吴成挺周娇娇
李果,张青萍,刘永辉,吴鹏,吴成挺,周娇娇
1 广西中医药大学研究生学院,南宁 530023;2 广西中医药大学第一附属医院脑病科
重度抑郁症(MDD)是一种常见的精神类疾病,日益威胁着人类的健康,但其确切的发病机制仍未阐明,亦无极其有效的干预方案[1]。因此,探讨MDD的潜在发病机制、研发MDD 的有效治疗方法是全世界亟需解决的公共卫生问题和社会问题。微小RNA(miRNA)是具有调控功能的非编码短小RNA,通过调控下游靶基因参与细胞的生长发育过程。研究表明,MDD的发生发展与miRNA异常表达密切相关[2],但尚未发现参与MDD 发生发展的特异性miRNA。生物信息学方法是联合多学科领域信息的一种新兴技术,通过整合分析疾病相关数据,可更高效、系统地揭示疾病的分子机制,并从宏观层面揭示疾病发生发展的复杂过程。2021年1月—6月,本研究通过生物信息学方法挖掘MDD 患者外周血中差异表达的miRNA 和基因,建立潜在的miRNAmRNA 调控网络,并筛选出关键miRNA 和基因,为阐明MDD的发病机制提供理论依据。
1 材料与方法
1.1 miRNA 微阵列数据集选择 通过国家生物技术信息中心(NCBI)基因表达综合数据库(GEO 数据库),以“major depression”“blood”“miRNA”作为关键词,搜索出关于MDD 患者外周血的miRNA 表达数据集,最终筛选出符合条件的数据集GSE81152。该数据集由GPL21814 平台合成,包含19 例正常人和30 例MDD 患者的外周血(分别作为对照组和观察组),正常人均无个人和家族精神病病史,MDD 患者来源于爱尔兰科克市的精神卫生服务机构和圣帕特里克大学医院,均未接受治疗。
1.2 差异表达miRNA(DE-miRNA)筛选 下载GSE81152 数据集,并分出对照组和观察组数据,采用R软件的“limma”软件包,对两组表达矩阵数据进行常规背景校正和分位数归一化;通过高级的t检验方法比较两组基因表达水平,以P<0.05 和|logFC|>0.15作为基本条件筛选DE-miRNA,最后使用R 软件的“Gplot”软件包对比较后的结果进行可视化,即构建差异分析的火山图。
1.3 DE-miRNA 上游转录因子和下游靶基因预测 使用FunRich 软件预测DE-miRNA 上游的潜在转录因子,并筛选出影响DE-miRNA 表达的主要上游转录因子,以P<0.05 作为鉴定转录因子的阙值。使用miRWalk 和TargetScan 数据库分别预测上、下调DE-miRNA 的下游靶基因,再筛选出两个数据库共同的上调DE-miRNA 的下游靶基因和下调DEmiRNA的下游靶基因。
1.4 DE-miRNA 下游目标基因筛选 通过GEO 数据库检索并下载MDD 患者外周血的基因表达芯片GSE98793 和GSE76826,其中GSE98793 数据集包含64 例份健康对照人群和128 例份MDD 患者的全血样本,GSE76826 数据集包含12 例份健康对照人群和10 例份MDD 患者的外周血样本。采用R 软件的“limma”软件包分别对下载整理好的两个数据集进行校正和归一化,分别比较两组外周血相关基因的表达水平,并筛选出同时满足P<0.05 和|logFC|>0.15 的差异表达基因(DEG),并构建火山图。将上调DE-miRNA 的下游靶基因和下调的DEG 进行交集,将下调DE-miRNA 的下游靶基因和上调的DEG进行交集,最终获得DE-miRNA下游目标基因。
1.5 京都基因与基因组百科全书(KEGG)富集分析 通过4个不同的数据库(Enrichr数据库、DAVID数据库、Metascape 数据库和GlueGO 数据库),对DE-miRNA 下游目标基因分别进行KEGG 富集分析,筛选出主要的信号通路,设置条件为P<0.05。
1.6 miRNA-mRNA 网络构建 利用String 数据库构建目标基因的蛋白质—蛋白质互作(PPI)网络,设置网络最低要求互作得分为0.15。通过miRNA 预测的靶基因结果找到目标基因对应的miRNA,使用cytoscape3.6.1 软件构建miRNA-mRNA 调控网络图;最后用插件“cytohubba”进行网络拓扑分析,根据MCC、MNC 和Degree 三种不同的计算方法筛选出核心miRNA和核心基因。
2 结果
2.1 DE-miRNA 筛选结果 在获得的DE-miRNA中,上调的DE-miRNA 有8 个(hsa-miR-3129-3p、hsamiR-4428、hsa-miR-3122、hsa-miR-4783-5p、hsa-miR-186-5p、hsa-miR-24-3p、hsa-miR-185-5p、hsa-miR-3156-5p),下调的DE-miRNA 有10 个(hsa-miR-668、hsa-miR-496、hsa-miR-655、hsa-miR-3664-3p、hsamiR-1208、hsa-miR-129-5p、hsa-miR-214-5p、hsamiR-1275、hsa-miR-539-5p、hsa-miR-1185-2-3p)。见OSID码图1、2。
2.2 DE-miRNA 上游转录因子和下游靶基因预测结果 在FunRich 软件筛选出的DE-miRNA 主要上游转录因子有5个,分别是特异性蛋白1(Sp1)、特异性蛋白4(Sp4)、早期生长反应1(EGR1)、锌指蛋白143(ZNF143)和锌指蛋白161(ZFP161),见OSID 码图3。miRWalk 和TargetScan 数据库共同的上调DE-miRNA 和下调DE-miRNA 的下游靶基因分别有13 351和15 549个。
2.3 DE-miRNA 下游目标基因筛选结果 在GSE98793 数据集中上、下调的DEG 分别有231、241个(OSID 码图4A),在GSE76826 数据集中上、下调的DEG分别有3 290、3 377个(OSID码图4B)。上调DE-miRNA 的下游靶基因与下调DEG 的交集得到47 个基因、下调DE-miRNA 的下游靶基因与上调DEG 的交集得到48 个基因,最终共得到95 个下游目标基因(OSID码图5)。
2.4 下游目标基因的KEGG 富集分析结果 下游目标基因在造血细胞系、Th17细胞分化、人类T细胞白血病病毒1感染、mTOR信号通路中显著富集。见OSID码图6。
2.5 miRNA-mRNA 网络构建结果 PPI 网络的网络节点数为95、边数为205、平均节点度为4.32、平均局部聚类系数为0.325。miRNA-mRNA 调控网络图,见OSID 码图7。网络拓扑分析后筛选出3 个核心miRNA(hsa-miR-4428、hsa-miR-3664-3p 和hsamiR-539-5p)和5 个核心基因(IL-7R、IKZF3、CD3D、CD3G和MS4A1)。见表1。
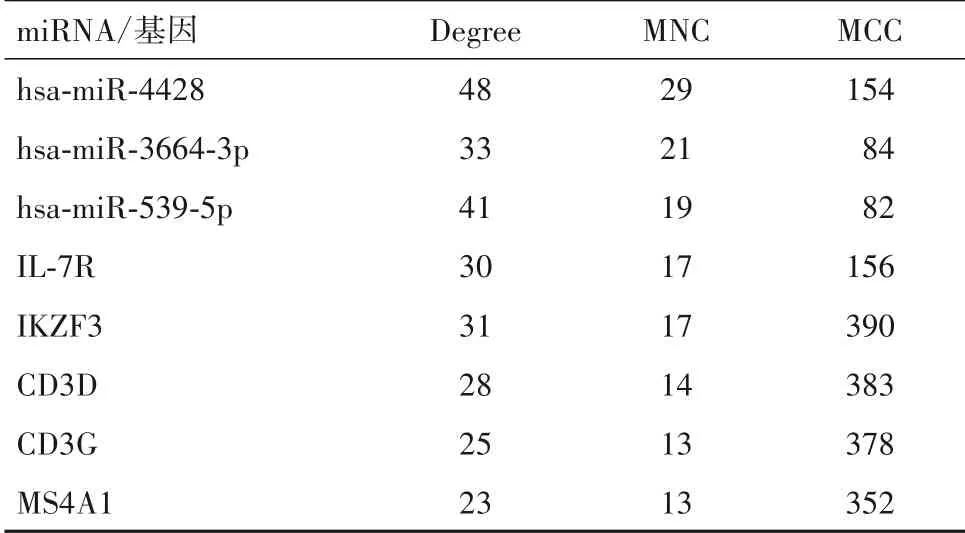
表1 核心miRNA和基因在3种计算方法中的得分(分)
3 讨论
研究显示,miRNA 通过调节基因的表达在神经精神疾病中起着重要作用,其失调导致下游基因表达水平的改变也是MDD 的发病原因之一[2-3]。但是目前关于MDD 患者比较全面的miRNA-mRNA 调控网络相关研究报道较少。因此本研究先对GEO 数据库中MDD 患者的miRNA 数据芯片进行差异分析,最终得到8 个上调的DE-miRNA 和10 个下调的DE-miRNA,其部分分析结果也有相关的研究报道。miR-24-3p在MDD 患者的脑组织(腹侧前额叶皮层)中表达上调,予抗抑郁治疗后血液中miR-24-3p 表达则明显下降[4];MDD 患者血清中miR-185 表达明显高于健康对照人群[5];另外,miR-185-5p 在双相情感障碍患者(含有MDD 患者)中的表达也明显上调[6]。有研究表明,miR-185可通过抑制额叶皮质中TrkB-T1 的表达,导致精神类疾病发生[7]。miR-129-5p在MDD小鼠脑内合成显著降低,其表达改变可能与TRPC3/6 和TREK1 活性变化导致神经元离子失衡有关[8]。虽然目前暂未找到miR-214-5p 在MDD中的相关研究,但DENG 等[9]通过动物实验发现在抑郁小鼠内侧前额叶皮层中miR-214-3p 表达明显增高,且认为该miRNA 是通过抑制了β-catenin 表达而导致了小鼠的抑郁行为,有效降低miR-214-3p 表达可达到治疗MDD 的效果。这与本研究筛选的结果有所不同,这种差异可能与所取的样本、种类或实验室方法不同有关,后期仍需更多实验研究来探讨。
先前有研究表明,miRNA 的表达可受转录因子调控,并进一步共同调节下游mRNA的转录和翻译,是细胞代谢的主要调节剂[3]。因此本研究利用Fun-Rich 软件预测了调节DE-miRNA 并可能在MDD 中发挥作用的转录因子。Sp1 和Sp4 属于转录因子家族的知名成员,均参与多种基本生物学过程,如细胞生长、分化、凋亡等,在多种精神疾病中的调节作用也已得到充分证明[10],但其在MDD 中的影响尚不明确。EGR1 被定义为锌指转录因子,可通过参与大脑中神经元活动的关键过程而影响神经精神疾病的发展,包括MDD。COVINGTON 等[11]发现,EGR1 在MDD 患者大脑前额皮质中呈低表达,因此上调EGR1 表达可能是抗抑郁治疗的一种有效手段。但ZHANG 等[12]却发现,EGR1 在大脑海马中的表达是上调的,且通过抑制EGR1 表达可快速发挥抗抑郁效果。这可能是实验方法及所取样本不同而导致的,有关这些的转录因子在MDD 中的作用尚有争议,有待于将来进一步研究。
本研究通过对DE-miRNA 靶基因与DEG 的整合,共筛选出95 个目标基因并进行了KEGG 富集分析,其结果显示其在造血细胞系、Th17 细胞分化、人类T 细胞白血病病毒1 感染和mTOR 信号通路中显著富集。造血细胞系、Th17 细胞分化和人类T 细胞白血病病毒1 感染途径均与体内免疫应答过程相关,体内免疫炎症反应与MDD 有密切联系,而神经炎症是导致情绪障碍(包括MDD)的原因之一,且抑制炎症反应在抗抑郁治疗中也有不错的效果,但免疫反应与MDD 的复杂网络关系还有待更深入研究[13-14]。mTOR 信号通路是由含有mTOR 的复合物引起下游反应的一个途径,因mTOR 可与多种蛋白质结合形成两种不同的复合体(mTORC1 和mTORC2),可将mTOR 信号通路分为mTORC1 途径和mTORC2 途径。mTORC1 途径通过控制蛋白质的磷酸化调节多种生物过程,且mTORC1 通路失调可涉及到多种神经系统疾病,如MDD、精神分裂症[15]。因此,激活mTORC1 信号引起下游反应能够抑制抑郁样行为[16]。而mTORC2 途径则主要调节细胞存活、细胞代谢和细胞骨架组织,但其可通过影响下游AKT/GSK3β 途径导致MDD 的发生[17]。研究发现,抗抑郁药可先通过促进大脑海马及前额叶皮层内AKT 和ERK1/2 因子的磷酸化,激活MAPK/ERK 和PI3K/AKT途径而诱导下游的mTOR信号通路,最后介导脑组织中的突触途径发挥抗抑郁的作用[18]。
本研究最后通过构建miRNA-mRNA 调控网络,筛选出核心miRNA(hsa-miR-4428、hsa-miR-3664-3p和hsa-miR-539-5p)和 核 心 基 因(IL-7R、IKZF3、CD3D、CD3G 和MS4A1)。目前关于miRNA 的研究越来越多,但miR-4428 和miR-3664-3p 在MDD 中的研究相对甚少。CIUCULETE 等[19]认为,miR-3664-5p 表达改变是导致MDD 发生的重要因素,而miR-3664-3p是否有相同的作用尚无定论。miRNA(包括miR-539)可通过靶向调节HTR2A 基因多态性导致MDD 的发生[20]。研究表明,大脑组织中miR-539-3p在精神类疾病的发生中发挥重要作用[21]。IL-7R 基因编码的蛋白质是白细胞介素7(IL-7)的受体,参与体内免疫炎症反应,而免疫炎症反应与MDD 的发生密切相关[13]。另外,IKZF3 表达对CD4+T 淋巴细胞中抗炎细胞因子IL-10 的表达有影响,CD3D 和CD3G 基因在T 淋巴细胞的增殖发育过程中发挥作用,而MDD 又与IL-10、T 淋巴细胞比例改变有关[22-24]。此外,MS4A1(即CD20)在B 淋巴细胞增殖和分化中具有重要的调节作用,可通过影响B 淋巴细胞数量导致MDD 的发生[25]。这些重要的基因主要在免疫系统中发挥作用,而免疫反应又是导致MDD 的重要途径,因此针对这些基因通过免疫系统来参与MDD 的发生可能是研究其机制的相关切入点。
综上所述,本研究成功构建了MDD 患者的
miRNA-mRNA 调 控 网 络,其 中hsa-miR-4428、hsamiR-3664-3p 和hsa-miR-539-5p 等 核 心miRNA 通 过影响IL-7R、IKZF3和CD3D等核心基因表达,并进一步调节免疫系统,在MDD 的发生发展中发挥重要作用。本研究通过生物信息学方法构建了涉及MDD发病机理的miRNA-mRNA 调控网络,有利于更全面地阐明其潜在机制,也为MDD 的诊断及开发新的治疗药物提供了一个新思路。但是本文是根据大数据预测出其可能存在的分子机制,部分预测结果仍需进一步实验验证。
