基于SRAP分子标记构建何首乌核心种质库
2021-12-18李嘉惠欧晓华邓文静罗可可张宏意何梦玲严寒静
李嘉惠 欧晓华 邓文静 罗可可 张宏意 何梦玲 严寒静








摘 要: 為保护何首乌遗传种质多样性,该文运用SRAP分子标记方法探究了44个产地327份何首乌种质的遗传多样性和居群结构,采用3种取样策略及6种比例抽样模式构建核心种质库,经t检验比较后筛选较优的核心种质构建方法和具有代表性的核心种质。结果表明:(1)何首乌种质遗传多样性较丰富,其观察等位基因数(Na)、有效等位基因数(Ne)、Shannon信息指数(I)和Nei’s遗传多样性指数(H)分别为1.984 3、1.454 9、0.271 7和0.417 9,另外何首乌种质居群间遗传分化程度大,基因交流较少,基因差异分化系数(Gst)为0.753 1,基因交流值(Nm)仅有0.163 9。(2)根据样品间遗传距离,采用邻接法对样品进行聚类,聚类结果显示327份样品主要分成四大类,样品分组结果与地理分布相符,相同采集点样品可聚在同一类中。(3)居群结构分析结果显示,当K=16时,其ΔK值最大,说明样品分成16个类群时最佳,同时大部分何首乌样品的血统组成较单一(Q>0.600),相同采集点样品血统组成相似,可大致归在同一类群中。(4)t检验结果显示,采用居群结构分类-比例取样和10%抽样比例构建的种质库,4个遗传参数的保留率高,Na、Ne、I和H的保留值分别为99.0%、101.9%、106.4%和105.9%,样品量较少,且多样性与原种质库无明显差异(P>0.05)。(5)核心种质库由34份样品组成,包括9份栽培样品和25份野生样品,主要来源于四川、重庆和贵州。该研究构建的核心种质库能代表原种质库的遗传多样性,可为种质资源的收集和新品种的选育提供参考,所采用的研究方法对其他植物核心种质库的构建具有一定参考意义。
关键词: 何首乌, 核心种质构建, SRAP, 遗传多样性, 居群结构
中图分类号: Q948
文献标识码: A
文章编号: 1000-3142(2021)11-1920-11
Construction of core germplasm bank of Fallopia multiflora using SRAP molecular markers
LI Jiahui1, OU Xiaohua1, DENG Wenjing1, LUO Keke1, ZHANG Hongyi1,2, HE Mengling1,2, YAN Hanjing1,2*
( 1. School of Traditional Chinese Medicine Guangdong Pharmaceutical University, Guangzhou 510006, China; 2. Key Laboratory of State
Administration of Traditional Chinese Medicine for Production & Development of Cantons Medicinal Materials, Guangzhou 510006, China)
Abstract: In order to protect the genetic diversity of Fallopia multiflora, in this study, SRAP molecular marker method was used to explore the genetic diversity and population structure of 327 F. multiflora samples from 44 habitats. Three sampling strategies and six proportional sampling modes were used to construct the core germplasm bank. After t-Test comparison, the better core germplasm construction method and the representative core germplasm samples were selected. The results were as follows: (1) The genetic diversity of F. multiflora germplasm was abundant, and number of observed alleles (Na), number of effective alleles (Ne), Shannon information index (I) and Nei’s genetic diversity index (H) were 1.984 3, 1.454 9, 0.271 7 and 0.417 9, respectively. In addition, the population of F. multiflora germplasm showed a high degree of genetic differentiation, but less gene exchange, with a gene differentiation coefficient (Gst) of 0.753 1 and a gene flow (Nm) of 0.163 9. (2)According to the genetic distance between samples, the Neighbor-Joining method was used to cluster the samples. The results showed that the 327 samples were mainly divided into four categories. The grouping results were consistent with the geographical distribution, and the samples at the same collection point could be clustered into the same category. (3) Population structure analysis showed that the biggest ΔK value was obtained when K=16, indicating that the sample should be divided into 16 groups, at the same time, the lineages composition of most F. multiflora samples were relatively simple, and the samples from the same collection point had similar lineages and could be roughly grouped in the same group. (4) The t-test showed that when population structure classification-proportional sampling and 10% sampling proportion were used to construct the germplasm bank, the retention rate of the four genetic parameters was high. The retention rate of Na, Ne, I and H values were 99.0%, 101.9%, 106.4% and 105.9%, respectively, the sample size was small, and the diversity was not significantly different from the original germplasm bank (P>0.05). (5) The core germplasm bank consisted of 34 samples, including 9 cultivated samples and 25 wild samples, mainly from Sichuan, Chongqing and Guizhou provinces. The core germplasm bank constructed in this experiment can represent the genetic diversity of the original germplasm bank, and the results can provide reference for the collection of germplasm resources and breeding of new varieties. The method used in the experiment has certain reference significance for the construction of other plant core germplasm banks.
Key words: Fallopia multiflora, construction of core germplasm, SRAP
何首乌(Fallopia multiflora)为蓼科多年生草本植物,适应力强,在我国204个县(市)均有分布(陈亚等,2011)。其喜光耐贫瘠,地下膨大的块根和地上生长的茎藤(首乌藤)均可入药,以块根入药为主,生用可以截疟解毒、润肠通便,炮制后可补益精血、固肾乌须。随着人们生活水平的提高,具有补益作用的何首乌市场需求量日渐攀升。药材市场中,野生何首乌是何首乌商品的重要来源。然而,研究人员调查结果显示,野生何首乌的资源供应量正以每年约15%的速度递减(刘红昌等,2013)。何首乌的繁殖方式包括种子繁殖、扦插繁殖、组织培养快速繁殖及压条繁殖等,其中扦插繁殖操作简单、成活率高,是目前农户规模化种植的常用方式。但是,长期的扦插繁殖会导致品种退化,从而影响何首乌药材的质量(曾文丹等,2016)。野生资源的枯竭和栽培种质的退化会造成物种遗传多样性的减少,降低生存和适应环境变化能力,同时也会导致优良基因丢失,严重影响何首乌的可持续发展和利用。因此,加强种质研究,进而构建何首乌的核心种质库,对何首乌资源实施有针对性和高效保护是亟待进行的任务。然而,至今尚无何首乌核心种质构建的相关报道。
SRAP(sequence related amplified polymophism)标记方法由Li & Quiros(2001)于2001年创立,相对其他标记方法,其具备分布普遍、可靠性和重复性高、技术难度低和多态性高等特点,在植物遗传基因图谱、植物遗传多样性和亲缘关系的分析以及基因转录图谱的构建上均有普遍应用(杨金华,2019)。程远辉(2007)在研究重庆地区何首乌遗传多样性时,较长时间使用RAPD标记方法进行研究,由于结果显示扩增条带少、重复性差,因此最终选择SRAP标记方法进行研究。可见,SRAP标记方法在何首乌遗传多样性研究上的优势。本研究利用前期筛选的13对SRAP引物对44个产地327份何首乌样品的遗传多样性进行研究,采用邻接法聚类-比例取样、居群结构分类-比例取样和最小距离逐步聚类取样3种取样策略,以及5%、10%、15%、20%、25%和30%共6種比例抽样模式,通过t检验对上述方法构建的初始种质进行评价,筛选出较好的取样策略和抽样比例,从而构建何首乌核心种质库。
1 材料与方法
1.1 材料
2017—2019年,采集327份何首乌叶片样品,经广东药科大学中药学院刘基柱副教授鉴定为蓼科何首乌属植物何首乌(Fallopia multiflora)。样品覆盖我国12个省44个地点,其中218份为栽培居群样品,109份为野生居群样品。栽培居群样品中包含1份棱枝何首乌(Fallopia multiflora var. angulatum),原种来自广西,栽培于广东药科大学药用植物园,具体信息见表1。
1.2 PCR扩增、产物检测及数据统计方法
选用植物DNA提取试剂盒(宝生物工程有限公司,产品型号9768)对何首乌叶片的基因组DNA进行提取。利用前期筛选得到的13对引物对327份何首乌基因组DNA进行扩增,引物信息见表2。反应体系的总体积为20 μL,包括10 μL premix(宝生物工程有限公司,产品型号RR902A),正反向引物(引物母液浓度为10 ng·μL-1)各1 μL,DNA模板40 ng,并用ddH20补充体积至20 μL;PCR反应程序为SRAP标记方法的一般程序,即94 ℃预变性4 min;94 ℃ 1 min,35 ℃ 1 min,72 ℃ 1 min,共5个循环;94 ℃ 1 min,50 ℃ 1 min,72 ℃ 1 min,共35个循环;72 ℃延伸5 min。
使用条带识别软件Quantity-One加人工识别的方式统计非变性聚丙烯酰胺凝胶电泳图上各泳道的条带迁移距离,相同位置有条带则记为1,无则为0,并建立二维数据矩阵。利用NTsyspc、Popgen 32、MEGA 7和Structure 2.3.4软件对数据进行统计分析,获得何首乌种质的遗传多样性参数、遗传距离、样品聚类和居群结构分析结果。在居群结构分析时,假设何首乌居群可以分成K=1~23个,并采用Evanno et al.(2005)的方法计算ΔK值,从而获得最佳的类群数。
1.3 取样策略和抽样方法
设置3种取样策略,即邻接法聚类-比例取样、居群结构分组-比例取样和最小距离逐步聚类取样;6个抽样比例,分别为 5%、10%、15%、20%、25%、30%,尝试构建何首乌的核心种质,取样方法的具体步骤如下。
(1)邻接法聚类-比例取样:根据原居群样品间遗传距离,通过邻接法聚类对样品进行分组,以各组的Nei’s基因多样性指数(H)比值决定各组样品的取样数量,当该组抽样数目大于组内样品数时,则按H比值把取样数量分配到其他组。
(2)居群结构分组-比例取样:通过Structure 2.3.4软件的居群分析,获得原居群的最佳分类组数,根据Q值对样品进行分类,并以各组的Nei’s基因多样性指数(H)比值决定各组样品的取样数量,当组中抽样数目大于组内样品数时,则按H比值把取样数量分配到其他组。
(3)最小距离逐步聚类取样:根据原群体各样品的遗传距离进行聚类分析,在聚类结果中找出遗传距离最小的一组,在该组中剔除其中一份样品,另一份样品保留下来,和其余样品重新计算遗传距离并进行聚类分析,循环上述步骤,直至样品数量符合抽样比例。
1.4 核心种质库检验和评价
采用t检验比较初始核心种质库和原种质库的各项遗传多样性参数。
2 结果与分析
2.1 样品扩增结果
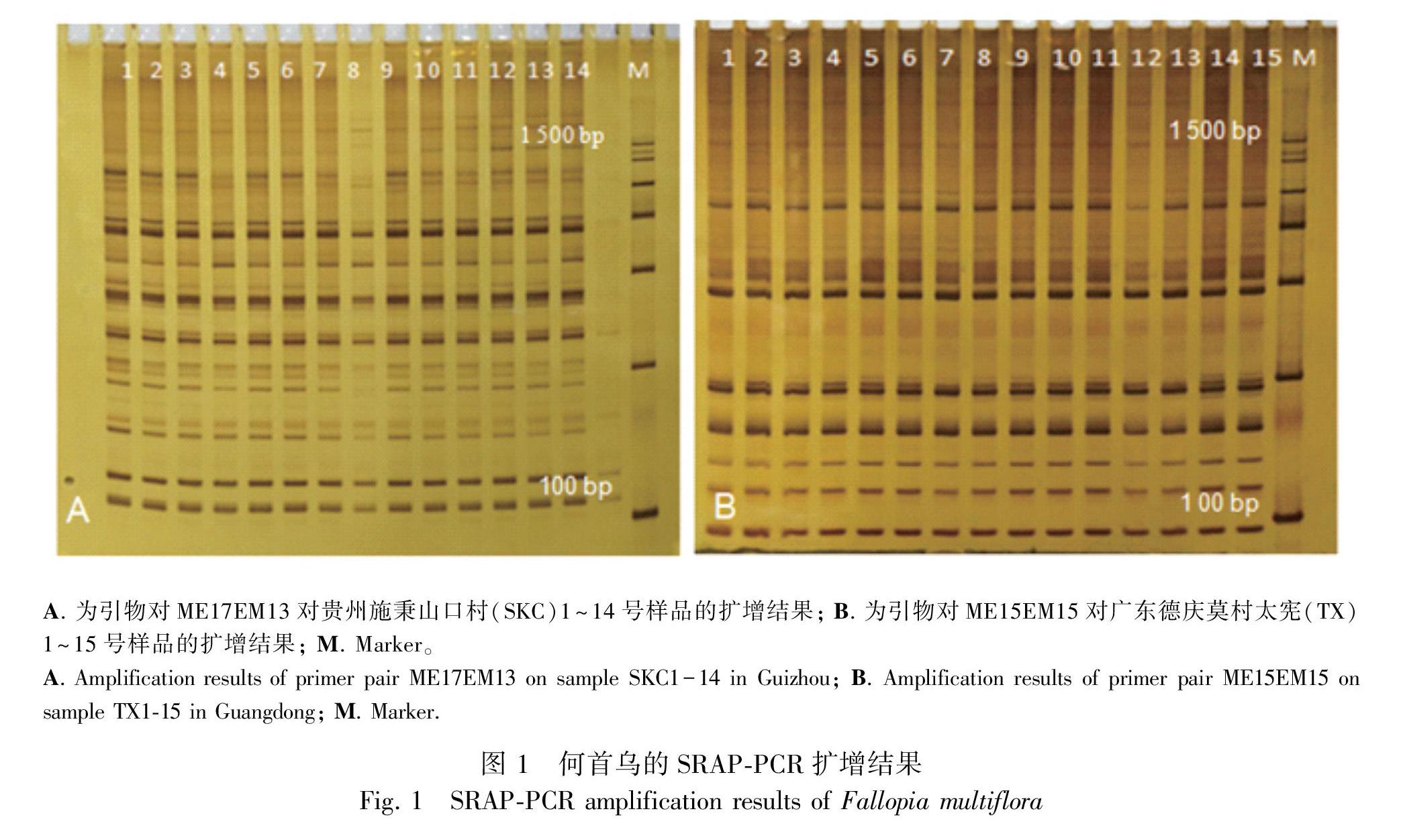
图1为何首乌样品基因组DNA经SRAP-PCR扩增、电泳和银染后得到的聚丙烯酰胺凝胶图,结果显示样品扩增条带中等丰富,条带能充分分离且清晰平整,即实验筛选的SRAP引物和采用的扩增、电泳、银染体系均适合何首乌的种质研究。
2.2 种质遗传多样性、样品聚类和居群结构分类
何首乌种质遗传多样性结果如表3所示,13对引物在栽培和野生居群样品中的扩增条带数及多态位点数丰富,野生居群样品的4个遗传多样性参数均大于栽培居群样品,说明野生何首乌的遗传多样性比栽培居群的大,两种类型何首乌的居群间基因多样性均大于居群内多样性,表明种质多样性主要来自居群间。整个何首乌种质资源的基因差异分化系数(Gst)为0.753 1(>0.5),表明居群间遗传分化程度大,但居群间的基因交流较少,基因交流值(Nm)仅有0.163 9(<1.0)。另外,栽培居群间分化程度极高,但基因交流极小,推测是生产过程主要采用营养繁殖,居群间缺少基因交流所致。
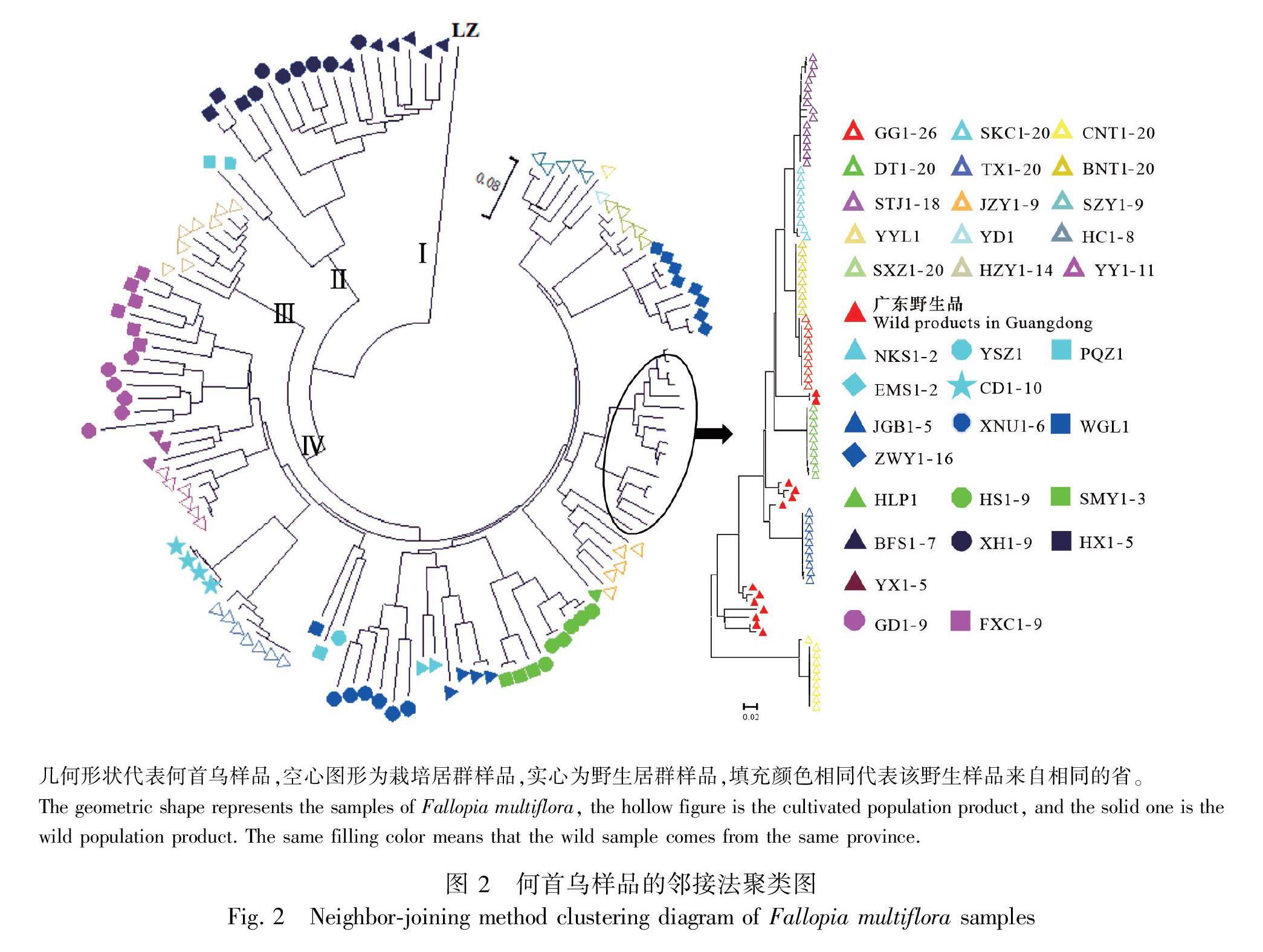
通过邻接法获得何首乌样品聚类分组结果(图2),何首乌样品主要分成4大分支:第1分支为棱枝何首乌,由于它与其余何首乌样品均有较大的遗传距离,因此独立成支;第2分支包括四川的EMS1~2和贵州的所有野生居群样品;第3分支为河南郑州的样品;第4分支由其余样品组成。
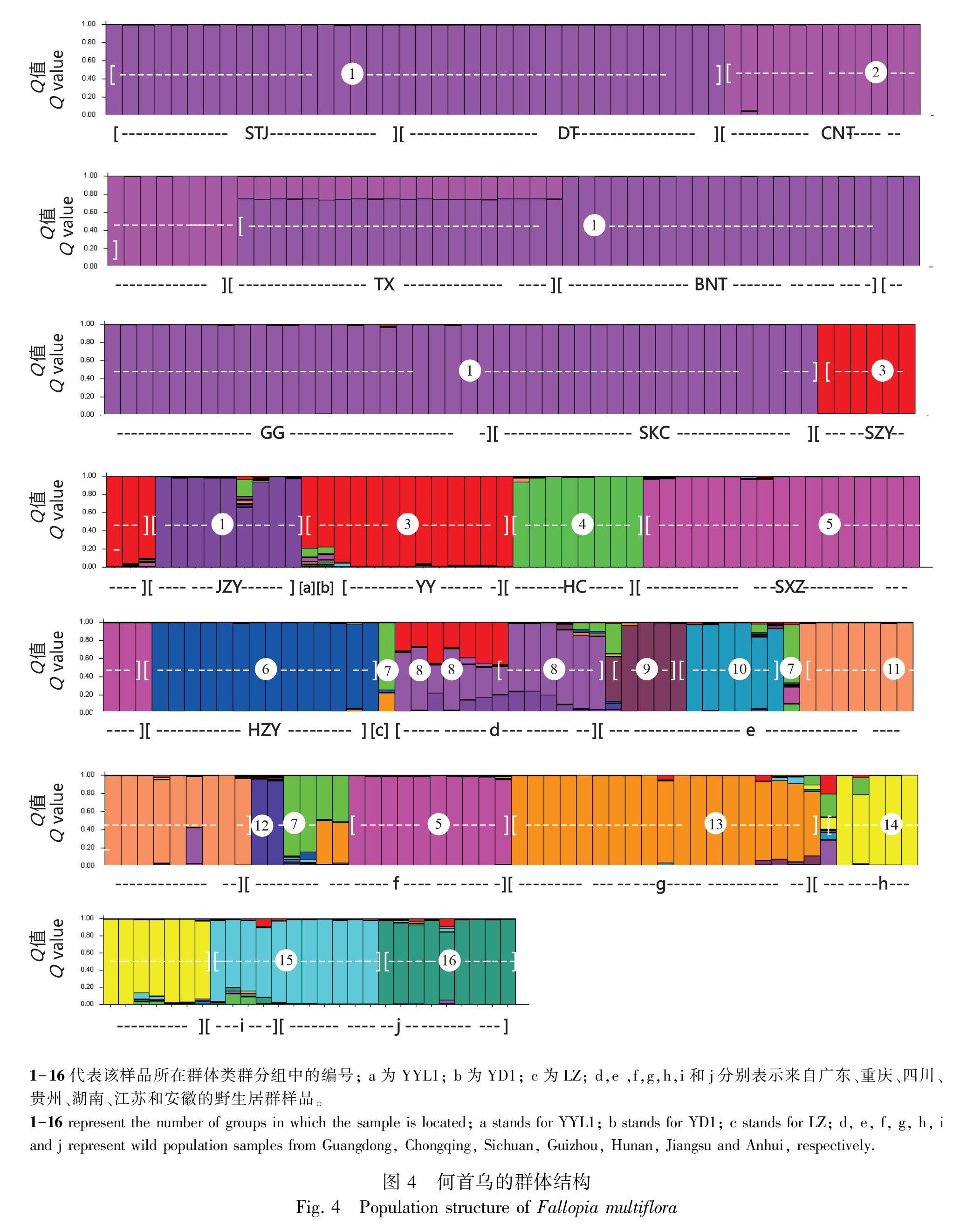
经Structure 2.3.4统计分析,当K=16时,其ΔK值最大,说明样品分成16个类群时最佳(图3),并由此获得居群结构图(图4),根据刘丽华等(2009)的方法,把样品归纳如表4所示,其中319份样品的Q≥0.600,说明大部分何首乌种质的遗传结构简单,而EMS(1~2)、HLP1、JGB1、DZ1、JGC1及HT(1~2)样品则血统相对复杂,实验将这些样品归在同一类群中,用于后续核心种质的构建。
2.3 不同取样策略和抽样比例的比较
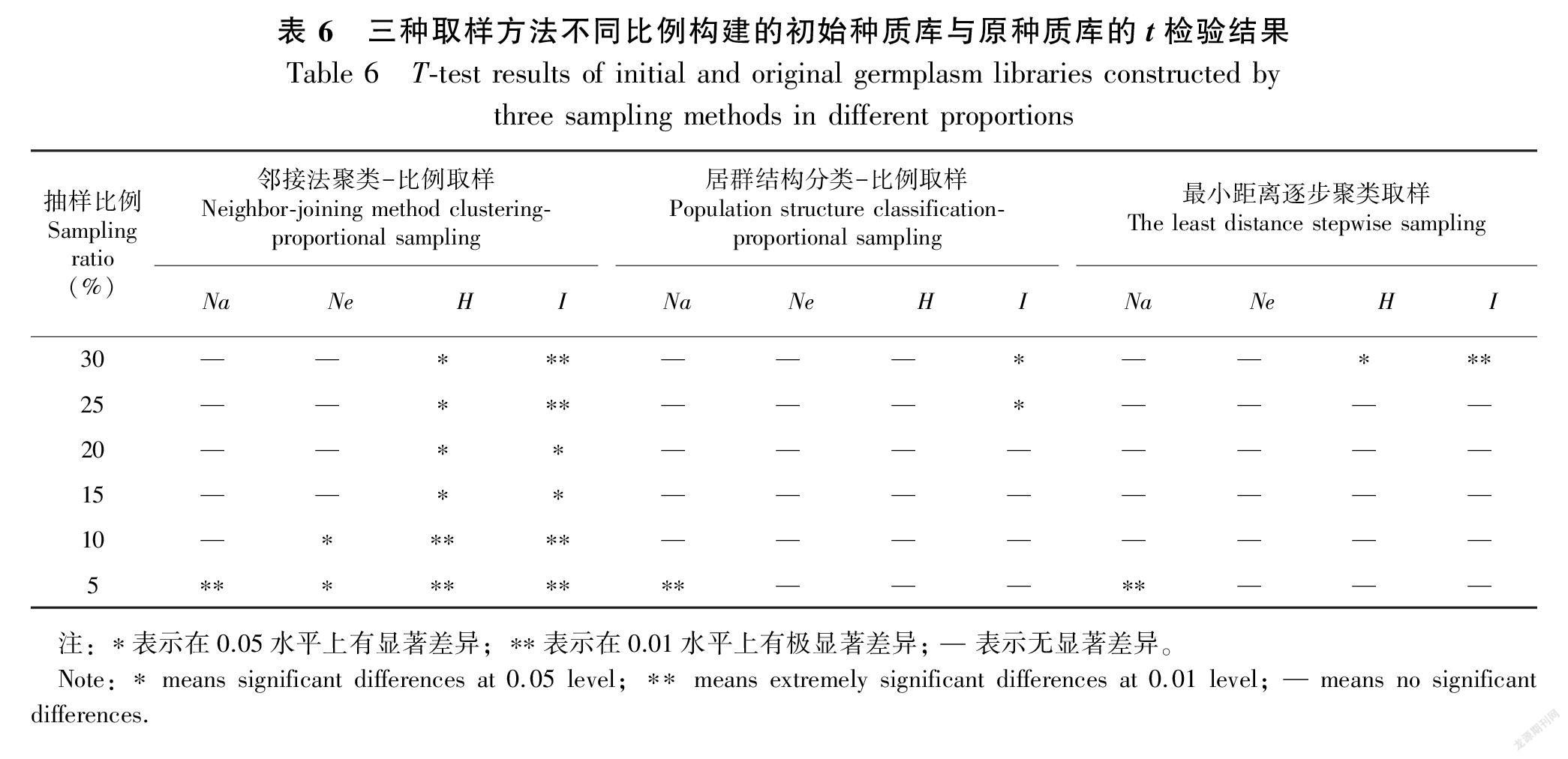
何首乌样品在不同取样策略和抽样比例下所组成的初始种质库,其遗传多样性参数如表5所示, 随抽样比例增大, 不同取样策略其Na值和多态位点百分数逐渐增大,而Ne值、H值和I值则先下降后上升,且3个参数的保留率均大于100%,即6个抽样比例构建的初始种质遗传多样性均大于原种质。
经t检验比较5%~30%抽样比例下初始种质库和原种质库的4个遗传参数,结果见表6。邻接法聚类-比例取样策略在不同抽样比例下初始种质库的H值和I值与原种质库均存在显著或极显著的差异,说明采用该方法构建核心种质,种质库样品遗传多样性不能反映原种质的真实情况,而居群结构分类-比例取样和最小距离逐步聚类取样方法在10%~20%抽样比例下,4个遗传参数与原种质库均无显著差异,当比例为10%时,两种方法的4个遗传参数值均最大,且居群结构分类-比例取样对原种质遗传多样性的保留上更占优势,根据核心种质库构建的原则:去除冗余,并以最小的资源量代表整个种质资源的遗传多样性。因此,选择居群结构分类-比例取样在10%比例下构建核心种质库。
2.4 何首乌核心种质库构建
采用居群结构分类-比例取样抽取原种质库10%的样品所组成的核心种质库如表7所示,样品数量共34份,由9份栽培居群样品和25份野生居群样品组成。栽培居群样品包括与其他样品存在明显差异的棱枝何首乌,而野生居群样品主要分布在四川、重庆和贵州,该地区样品数量约占核心种质库总数的47%。
3 讨论与结论
焦贤贤(2018)的研究表明,核心种质的构建可以为植物的保护和利用提供理论指导,并有效地提高种质资源的管理、研究和利用效率。目前,世界各国已创立1 300余个种质资源库,保存了600多万份各类种质资源,而我国现存的约有40万份(李荣荣,2020)。虽然药用植物种质资源是中药的源头,但由于药用植物的核心种质库的建立起步较晚,因此在种质库的构建上一般是沿用农作物的方法(林丹,2018)。
核心种质的构建,实质上是利用收集到的数据,根据一定原则把类似的种质资源进行分组,选取合适的总体取样规模,在每组中用合理的取样比例和取样方法进行抽样, 使建立的核心种质不仅能尽量地代表原种质的遗传多样性,而且能最大程度地反映原始群体的遗传结构(王心迪,2016)。核心种质构建一般包括4个步骤,即数据采集及分析、材料分组、样品抽样和核心种质检验及评价。其中,样品抽样包括抽样方法和抽样比例的确立,是核心种质构建中的重要环节。因此,抽样策略的选择对核心种质库的优劣起到关键性作用。
在材料分组上,将搜集到的材料先以相似特征为依据将种质分类,再以一定取样方法选择不同类群中的种质所形成的样本,其遗传差异比选取同种群体的样本高。分组的标准和方法较多样,目前使用最多的是聚类分组方法(姜丽媛,2018)。本研究采用SRAR分子标记方法获得何首乌的聚类分组结果,同时通过居群结构分析獲得样品的类群分组结果,作为后续样品选择的基础。
在取样策略上,主要包括分层取样法、逐步聚类随机取样法、最小距离逐步聚类法、主成分分析法和位点优先逐步聚类法。其中,分层取样法包括4种, 分别为比例法、 对数法、 平方根法和遗传几何形状代表何首乌样品,空心图形为栽培居群样品,实心为野生居群样品,填充颜色相同代表该野生样品来自相同的省。
多样性法(杨孟莉,2016)。本研究获得的是离散型指标数据,主要通过遗传参数的统计来分析种质的多样性,由于物种多样性在群体中分布是不均匀的,因此在前期的聚类分组和居群结构分组的基础上,根据每组遗传多样性指数的比例来确定组内抽样的数目, 最终形成了邻接法聚类-比例取样和居群结构分类-比例取样两种抽样方法。另外,由于最小逐步聚类方法为核心种质构建的常用方法,因此将其纳入何首乌核心种质构建的抽样策略中。
本研究中,何首乌核心种质库的构建结果表明,虽然邻接法聚类分组-比例取样能使初始种质库的遗传参数获得较高的值,但与原种质库存在显著差异(P<0.05), 笔者认为可能是邻接法聚类对样品的分组不够精细,取样时较为随机所致。虽然实验采用的最小距离逐步聚类方法能较好地构建何首乌的核心种质,但步骤较为繁琐,而居群结构分类-比例取样方法目前虽然较为少用,但实验结果显示,该方法构建的核心种质库代表性强且多样性保留较高,它可以对样品进行精细的类群划分,利用多样性比值确定取样数量避免了样品抽取数量分配不均匀的情况,减少了冗余,且操作比最小距离逐步聚类取样简便,上述比较提示该方法可以推广应用于其他物种核心种质库的构建中。
本研究采用居群结构分类-比例取样,在10%的抽样比例下获得了最佳的核心种质库,关于抽样比例,大多数植物资源核心种质的整体取样比例在5%~30%之间, 目前还没有研究可以确定统一的比例(吴茵,2017)。因此,需要根据不同植物种质资源的收集程度、遗传多样性状况和遗传结构等方面的具体特点,设定适合的取样比例(焦贤贤,2018)。
核心种质的构建并非完美无缺, 尽管在构建中选取了最佳的比例、最佳的抽样策略、最大限度地对原始种质进行筛选,但仍旧会丢失某些变异类型,并且随着资源不断地收集,会出现新的变异类型。因此,为了使物种的多样性得到长久的保存和有效利用,应该实时对种质库进行调整(姜丽媛,2018)。本研究中,重庆和四川的野生居群样品占核心种质的多数, 在聚类和居群结构分析时发现, 上述两个产地的野生样品与其他产地样品相比,在聚类分组中呈分散分布,说明两地样品遗传多样性高,提示该地区何首乌种质遗传多样性丰富,今后可以加强两地何首乌样本的收集和研究,进而不断完善核心种质库。
参考文献:
CHENG YH, 2007. Study on natural resources of Polygonum multiflorum Thunb in Chongqing base on SRAP analysis [D]. Chongqing: Southwest University: 23. [程远辉, 2007. 基于SRAP分析的重庆何首乌种质资源整理研究 [D]. 重庆: 西南大学: 23.]
CHEN Y, JIANG B, ZENG YE, 2011. Study on the geographic distribution and ecological environmental characteristics of Polygonum multiflorum based on GIS [J]. J Chin Pharm, 22(39): 3726-3728. [陈亚, 江滨, 曾元儿, 2011. 基于地理信息系统的何首乌地理分布及分布区的生境特征研究 [J]. 中国药房, 22(39): 3726-3728.]
EVANNO G, REGNAUT S, GOUDET J, 2005. Detecting the number of clusters individuals using the software structure: a simulation study [J]. Mol Ecol, 14(8): 2611-2620.
JIANG LY, 2018. The wild germplasm resources evaluation and core collection establishment of Rosa rugosa [D]. Tai’ an: Shandong Agricultural University: 11-12. [姜丽媛, 2018. 濒危植物野生玫瑰种质资源评价与核心种质构建 [D]. 泰安: 山东农业大学: 11-12.]
JIAO XX, 2018. Construction of core collection of wax gourd [D]. Nanning: Guangxi University: 6-11. [焦贤贤, 2018. 冬瓜核心种质的构建 [D]. 南宁: 广西大学: 6-11.]
LI G, QUIROS CF, 2001. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica [J]. Theor Appl Genet, 103(2/3): 455-461.
LI RR, 2020. Diversity analysis and construction of primary core collection in Tibetan turnip [D]. Hangzhou: Zhejiang University: 11. [李榮荣, 2020. 西藏芜菁遗传多样性分析与初级核心种质的构建 [D]. 杭州: 浙江大学: 11.]
LIN D, 2018. Analysis of genetic diversity and preliminary construction of core construction of core collection of Aquilariasinensis (Lour.) Spreng [D]. Guangzhou: Guangdong Pharmaceutical University: 4-41. [林丹, 2018. 白木香遗传多样性分析及核心种质的初步构建 [D]. 广州: 广东药科大学: 4-41.]
LIU HC, LUO CL, LI JL, et al., 2013. Habitat survey and biological characteristics study of Polygonum multiflorum germplasms in Guizhou region [J]. J Chin Med Mat, 36(6): 864-870. [刘红昌, 罗春丽, 李金玲, 等, 2013. 何首乌不同种质生境调查及在贵州地域的生物学特性研究 [J]. 中药材, 36(6): 864-870.]
LIU LH, WANG LX, ZHAO CP, et al., 2009. Genetic diversity and alterations of population structure in restorers of dual cross-line hybrid wheat with thermo-photoperiod sensitive male sterile [J]. Chin J Biochem Mol Biol, 25(9): 867-875. [劉丽华, 王立新, 赵昌平, 等, 2009. 光温敏二系杂交小麦恢复系遗传多样性和群体结构分析 [J]. 中国生物化学与分子生物学报, 25(9): 867-875.]
LIU SY, 1991.A new variety of Polygonum multiflorum [J]. Plant Divers, 13(4): 390. [刘寿养, 1991. 何首乌一新变种 [J]. 云南植物研究, 13(4): 390.]
WANG XD, 2016. Construction and evaluation of core collectios of Momordica charantia [D]. Nanning: Guangxi University: 3. [王心迪, 2016. 苦瓜核心种质的构建与评价 [D]. 南宁: 广西大学: 3.]
WU Y, 2017. Genetic diversity analysis and establishment of the core collection of pepper germplasm resources based on SRAP, SSR markers [D]. Nanchang: Jiangxi Agricultural University: 6. [吴茵, 2017. 基于SRAP、SSR标记的辣椒种质遗传多样性分析与核心种质构建 [D]. 南昌: 江西农业大学: 6.]
YAN HJ, FANG ZJ, ZHANG HY, et al., 2011. Fallopia multiflora var. angulata, a new combination in the Polygonaceae from China [J]. Novon, 21(3): 388-391.
YANG JH, 2019. Application of SRAP marker in plant research [J]. Seed Sci Technol, 37(7): 55. [杨金华, 2019. SRAP标记在植物研究中的应用 [J]. 种子科技, 37(7): 55.]
YANG ML, 2016. The germplasm evaluation and core collection construction of Cornus officinalis based on ISSR markers [D]. Zhengzhou : Henan Agricultural University: 9. [杨孟莉, 2016. 基于ISSR分子标记的山茱萸种质资源评价与核心种质构建 [D]. 郑州: 河南农业大学: 9.]
ZENG WD, LU LY, XIE XY, et al., 2016. Advances in reproductive technology of Polygonum multiflorum [J]. Chin Trop Agric, 70(3): 66-68. [曾文丹, 陆柳英, 谢向誉, 等, 2016. 何首乌繁殖技术研究进展 [J]. 中国热带农业, 70(3): 66-68.]
(责任编辑 蒋巧媛)
