小管福寿螺SNP标记开发及其在物种鉴定中的应用
2021-10-20林友福李雪霞殷文莉
林 英,肖 麒,林友福,李雪霞,殷文莉,李 宏,陈 炼
(1.南京师范大学生命科学学院,江苏 南京 210023) (2.南京林业大学生物与环境学院,江苏 南京 210037)
福寿螺属(Pomacea)隶属于腹足纲(Gastropoda)新进腹足目(Caeogastropoda)瓶螺科(Ampullariidae),是原产于南美洲亚马逊河流域的一类大型淡水螺[1]. 因人类有意引种或无意传播,福寿螺属多个物种现已入侵到亚洲、北美、欧洲及大洋洲的许多国家和地区,成为严重的入侵性有害生物[2]. 小管福寿螺(Pomaceacanaliculata)和斑点福寿螺(P.maculata)是福寿螺属中常见且入侵性最强的两种物种[3]. 其中,小管福寿螺被国际自然保护联盟(IUCN)列为全球100种恶性外来物种之一[4]. 小管福寿螺自20世纪80年代初作为食物引入到我国以来,迅速定殖并扩展到很多地区,成为我国长江以南大部分省区的严重农业有害生物,并且呈现由南向北扩散的趋势[5].
福寿螺属传统的分类方法主要依靠形态学特征[6]. 然而,福寿螺属物种形态相似,且具有较高的形态可塑性,难以实现物种的准确鉴定和鉴别[7]. 过去认为,引入中国的福寿螺属生物仅有小管福寿螺,Lv等[8]利用线粒体COI基因序列对我国54个福寿螺地理种群进行系统发育关系分析,中国大陆地区的福寿螺种群结构复杂,可能有多种来源和扩散模式,并且发现入侵中国的福寿螺除了有小管福寿螺和斑点福寿螺这两种之外,还存在一个中国特有的隐存种团(Cryptic groups). 近期研究基于线粒体COI基因序列的系统发生分析和形态学特征确定其为中国分布的第三种入侵性福寿螺(P.occultanov. sp.)[9].
虽然福寿螺种间形态相似,但其生物学、生态学特征以及原产地的地理分布不同,入侵能力和扩散趋势等方面存在差异[3]. 利用有效的鉴定方式对福寿螺进行准确的种类鉴别,对于控制其快速扩散、制定有效的防治措施具有重要意义. 现有研究大多利用基于线粒体COI基因和核基因EF1α的序列分析[10]或多重PCR扩增技术[11-12]来鉴别福寿螺种类. 然而,长片段扩增对于样本的质量要求较高,并且鉴定成本高. 此外,遗传多样性和种群结构是影响入侵种定殖的重要因素,利用分子标记进行种群遗传学研究有利于开展入侵物种的控制与管理[13]. SNP标记作为第三代分子遗传标记,具有数量丰富、分布广泛、遗传稳定性高、二等位性、分析成本较低且DNA降解耐受性较高等特点[14-15],近年来在群体遗传分析及动物个体、品种鉴定方面的研究应用较多. 本研究基于小管福寿螺和斑点福寿螺全基因组重测序数据,借助Sanger测序法验证开发多态性SNP位点,为后续群体遗传学研究的开展提供了工具. 同时,针对多态性位点根据其在小管福寿螺和斑点福寿螺间的遗传分化指数(Fst)及基因频率信息,进一步筛选在两物种中存在明显区别的SNP位点,为两种福寿螺提供高效、便捷、准确的鉴定方法.

A-B:小管福寿螺;C-D:斑点福寿螺图1 福寿螺的螺壳形态Fig.1 Shell morphology of Pomacea spp.
1 材料与方法
1.1 样品采集及物种鉴定
2019年3月至7月共采集来自重庆(N=10)、四川成都(N=5)、浙江丽水(N=30)、江苏苏州(N=3)的48只野生福寿螺,螺长范围为30 mm~45 mm.
剪取福寿螺腹足肌提取基因组总DNA,DNA提取使用百奥莱博科技(北京)有限公司柱式软体动物DNA提取试剂盒(BTN101111),方法参照试剂盒的使用说明. 利用1.5%琼脂糖凝胶电泳检测基因组DNA的完整性.
由于福寿螺属物种如小管福寿螺和斑点福寿螺外部形态相似,难以区分,在螺壳形态鉴定基础上(图1)结合线粒体COI基因序列进行物种鉴定[3,9]. 结果表明,本研究样本中包括30只小管福寿螺,均来自浙江丽水和18只斑点福寿螺,来自重庆、四川成都和江苏苏州.
1.2 SNP位点筛选
本研究基于课题组前期已获得的75只小管福寿螺和23只斑点福寿螺全基因组重测序数据(数据未发表),将原始数据(raw data)去除接头序列和数据质控后,利用BWA软件[16],将有效测序数据(clean data)比对小管福寿螺参考基因组(ASM307304v1)[17],比对结果经Picard(http://broadinstitute.github.io/picard/)去除重复. 使用GATK 4.0软件[18]进行SNP检测分析,筛选测序深度不低于7X、覆盖所有群体95%以上个体、次等位基因频率(MAF)不低于0.05的SNP位点用于SNP标记开发. 使用ANNOVAR软件[19]对SNP进行注释.
基于上述成功开发的多态性SNP位点,利用VCFtools(v0.1.16版本)[20]软件进行Fst值的计算,同时基于重测序基因型数据,利用GENEPOP 4.0.10软件[21]分别统计这些SNP位点在小管福寿螺和斑点福寿螺中的等位基因频率信息,挑选Fst值和等位基因频率在两种福寿螺间存在较大差异的SNP位点用于物种鉴定.
1.3 基于Sanger测序SNP验证
基于小管福寿螺基因组测序信息,将筛选的位点定位在基因组上,利用TBtools软件[22]提取该SNP位点上下游各200 bp碱基序列,利用Primer Premier 5.0软件[23]进行引物设计. 随机选取4个小管福寿螺样本,进行PCR扩增,利用Sanger测序验证SNP位点是否存在双峰,若扩增产物出现不同峰值,即认为该位点在群体内存在多态性. PCR反应体系为20 μL:10 μL Premix Taq,引物各1 μL,1 μL DNA和7 μL ddH2O;反应条件:94 ℃预变性5 min,94 ℃变性30 s,合适的退火温度下30 s,72 ℃延伸1 min,28个循环,最后72 ℃延伸10 min. 针对存在双峰的位点,用相同的PCR条件,对剩下的福寿螺样本进行扩增测序. 引物合成和测序均由上海迈浦生物科技有限公司完成.
1.4 数据分析
将测序得到的序列用MEGA X软件[24]分析基因序列的SNP位点,对比测序后的数据峰图,找到峰值图中对应MEGA X序列里观察到的SNP位点,二倍体纯合位点记为两个相同的碱基,显示为单峰图;而杂合位点则记为两个不同的碱基,显示为套峰,进行基因型统计. 使用Cervus 3.0软件[25]计算SNP位点的观测杂合度(observed heterozygosities,Ho),期望杂合度(expected heterozygosities,He)和多态信息含量(polymorphism information content,PIC). 使用GENEPOP 4.0.10软件进行位点间的连锁不平衡(linkage disequilibrium)检验和哈迪温伯格平衡偏离检验(Hardy-Weinberg equilibrium,HWE).
针对筛选得到的用于小管福寿螺和斑点福寿螺物种鉴定的SNP位点,依据这些SNP位点在重测序数据中得到的基因频率信息(表1),以及48个福寿螺样本进行Sanger测序得到的基因型信息,对这48个个体进行物种鉴定,验证这些SNP位点用于物种鉴定的准确性. 具体分析方法参考程方圆等[26]和Jiang等[27]的研究:如果使用一个SNP位点,以SNP4为例(表1),某样本测序结果为TT,计算该SNP位点小管福寿螺T基因频率(0.133)的乘积,以及斑点福寿螺该SNP位点T基因频率(0.935)的乘积,如果小管福寿螺的乘积高于斑点福寿螺,则支持此样本为小管福寿螺,反之,则为斑点福寿螺. 如果使用2个及以上SNP位点,先按上述单个SNP标记方法分别独立计算,再将每个SNP位点得到的数值依次相乘获得综合判定数值,如果小管福寿螺的综合判定数值高,则支持此样本为小管福寿螺,反之,则为斑点福寿螺.

表1 5个用于物种鉴定的SNP分子标记基因频率Table 1 Gene frequency of 5 diagnostic SNP markers
利用单个(或多个)SNP位点得到的某个物种鉴定结果与实际物种鉴定结果一致时,记录判断正确次数1次. 基于单个SNP位点以及多个位点对48个个体进行判断,分别统计判断正确次数,计算验证准确率. 计算公式如下:R=Ncorrect/Nall×100%,式中:R为验证准确率即判断正确次数占总次数的比值;Ncorrect为判断正确的次数;Nall为判断总次数[26].
2 结果与讨论
2.1 SNP标记的鉴定和筛选
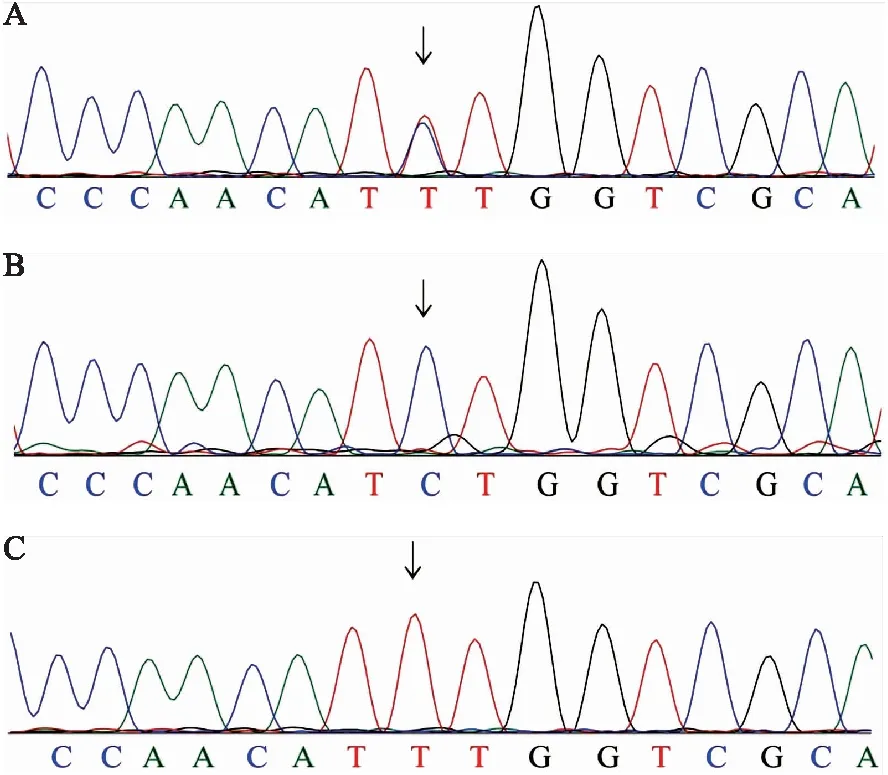
A:CT 基因型;B:CC 基因型;C:TT 基因型图2 位点SNP11分型结果Fig.2 The results of genotyping in SNP11
通过与小管福寿螺参考基因组序列比较,在小管福寿螺和斑点福寿螺全基因组范围内共检测到的SNP数量为27 950 000个,通过进一步过滤得到46 693个高质量SNP位点,其中碱基转换(transition)占55.56%,高于碱基颠换(transversion)44.44%. 位于3′-非编码区、5′-非编码区、基因上游、基因下游、基因间区、剪切位点和内含子的SNP数量分别为2 593、1 088、3 392、3 007、13 454、6和24 146个,位于外显子的SNP数量为2423个(5%),随机选取34个SNP位点设计引物.
2.2 SNP分型
在34对引物中,有30对引物成功扩增出DNA片段. 以位点SNP11为例,测序结果如图2所示,峰图清晰规则,无杂峰,目标位点在不同样本中单峰(纯合)或套峰(杂合)明显,即SNP11为潜在的SNP位点,在4个福寿螺DNA样品的扩增测序中,共筛选到22个潜在的SNP位点(表2),准确率为64.70%. Yang等[28]基于广东鲂(Megalobramaterminalis)简化基因组测序数据开发并验证了30个SNP位点,准确率为83%;有研究对大西洋鳕(Gadusmorhua)的全基因组进行测序并开发和验证 SNP,其准确率为74.60%[29]. SNP标记开发的准确率可能与不同的测序方法、质量及深度有关[30].

表2 小管福寿螺22个SNP位点信息Table 2 Genetic information for 22 SNPs in Pomacea canaliculata

续表2 Table 2 continued
通过计算上述22个SNP位点在小管福寿螺和斑点福寿螺间的Fst值,发现有5个SNP位点Fst值位于前1%,且等位基因频率信息在两种福寿螺间有明显差异(表1). 小管福寿螺各位点等位基因频率差值在0.706~0.786,平均值为0.744,而该差值在斑点福寿螺数据中较大,为0.740~0.956,平均值为0.870. 两种福寿螺各位点等位基因频率差值在0.763~0.832,平均值为0.807,说明这些标记具有较高的鉴别能力(表1). Jiang等[27]基于前人研究的二代测序数据利用Snapshot分型方法开发了12个SNP位点用于鉴别蒙古狼(Canislupus)和家犬(C.l.familiaris),各位点在两物种中等位基因频率差值在0.640~0.889,平均值为0.797,具有良好的鉴别能力.
2.3 SNP位点多态性分析
对上述22个SNP位点在30个小管福寿螺样本中进行多态性分析,结果如表2所示,22个SNP位点的平均等位基因数为2,观测杂合度(Ho)和期望杂合度(He)范围分别为0~0.933和0.033~0.549,多态信息含量(PIC)范围为0.032~0.466.PIC值是衡量DNA座位变异程度高低的重要指标[31]. 根据Botstein等[32]划分的原则,PIC<0.25为低度多态性,0.25
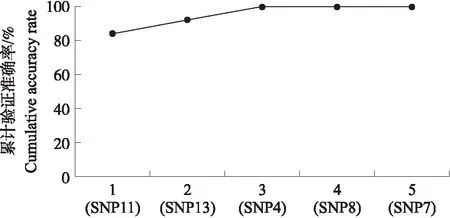
横坐标:分子标记个数,括号内为依次增加的SNP标记;纵坐标:分子标记累计验证准确率.图3 分子标记和累计验证准确率Fig.3 SNP markers and cumulative accuracy rate
2.4 物种鉴定SNP位点验证
尽管基因组中拥有数以百万计的SNP位点,但使用更多的SNP位点并不代表会达到更高的分辨率. SNP是基于等位基因频率的标记,它们在物种水平上的分辨率取决于物种之间等位基因频率的差异[27]. 基于5个物种鉴定SNP位点在小管福寿螺和斑点福寿螺中的等位基因频率信息,对48个福寿螺样本进行物种鉴别验证,结果表明当5个SNP位点分别单独使用时,判别准确率为94.74%(SNP4)、97.37%(SNP7)、94.74%(SNP8)、84.21%(SNP11)和92.10%(SNP13). 当同时使用多个SNP位点时,判别准确率结果如图3所示,同时使用前3个位点SNP11、SNP13和SNP4时准确率即可达到100%. Lapègue等[33]利用高通量测序数据,在入侵物种长牡蛎(Crassostreagigas)和葡萄牙牡蛎(C.angulata)中成功筛选出具有较大Fst值和等位基因频率差异的15个SNP位点用于两个物种的准确鉴定. 本研究中使用较少的SNP位点可以达到100%的分辨率.
3 结论
本研究首次基于福寿螺重测序数据,成功开发22个多态性SNP标记,可用于小管福寿螺群体遗传多样性、遗传结构等相关研究,且进一步筛选5个SNP位点用于入侵中国的小管福寿螺和斑点福寿螺物种鉴定,通过Sanger测序法在48个福寿螺样本中进行分型验证,结果表明,物种鉴定准确率较高(>84.21%),当同时使用3~5个SNP位点时准确率可达到100%. 为小管福寿螺和斑点福寿螺准确的种类鉴别提供了有效的工具,对于控制其快速扩散、制定有效的防控措施具有重要意义.
