两种养殖模式下黑鲷肠道及养殖水体菌群结构特征的研究
2021-10-12聂志娟邵乃磷张志伟胡佳雯徐钢春
聂志娟,邵乃磷,张志伟,胡佳雯,徐 跑,徐钢春
(1中国水产科学研究院淡水渔业研究中心农业农村部淡水渔业和种质资源利用重点实验室,江苏无锡214081;2江苏省海洋水产研究所,江苏省海水鱼类遗传育种重点实验室,江苏南通226007;3南京农业大学渔业学院,江苏无锡214081)
0 引言
黑鲷(Acanthopagrus schlegelii),隶属鲈形目(Perciformes),鲷科(Sparidae)棘鲷属(Acanthopagrus),为暖温性底层鱼类,广泛分布于中国东南、朝鲜半岛南部、日本及东南亚等地附近海域,因其具有生长快、抗病力强、广盐性、广温性、营养丰富等多种生物学优良特性,已成为了中国海水集约化养殖的重要经济鱼类品种[1-2],并形成了网箱养殖、传统池塘养殖、池塘工程化循环水养殖等多种黑鲷养殖模式[3]。其中池塘流水槽循环水养殖,是中国渔业转型升级、创新发展的一种健康生态养殖新模式,具有节水节地、高产量、低渔药使用量、污物资源化利用、养殖全程可控等优点[4]。
不同养殖水体中,都栖息生存着大量的微生物群体,其种类繁多、功能复杂、遗传代谢方式多样,在营养物质的循环和外源物质的生物转化中起着非常重要的作用,调节物质元素循环过程的平衡并维持生态系统的健康[5-6]。生活在水体中养殖动物,其肠道作为食物消化吸收重要器官,同样吸附寄生丰富的微生物,形成特有的肠道微生态系统,参与宿主鱼生长免疫等代谢调控[7-8]。肠道微生物菌群组成受到许多内源性和外源性因素的影响,如物种、生活方式、喂养习惯、饮食、营养状况、生活条件等[9-10]。在外源性环境因素中,生长水体与微生物紧密相关,在很大程度上影响鱼类肠道细菌群落的结构和丰度[11]。不同养殖模式造就不同的生长环境,其群落组成结构存在明显差异[12-13],对养殖动物具有很大影响。众多研究表明,养殖模式能影响养殖水质[14]、鱼类的生长性能[15]、肌肉品质[16]、免疫性能[17]等,然而水产养殖中水质调节,鱼类生长、免疫、消化、吸收等都与环境中的微生物息息相关[18-19],因此,深入了解水产养殖环境微生物群落结构信息对整个水产养殖业的生态绿色健康发展有非常重要的意义。
传统的微生物种群结构研究建立在菌群分离和培养技术基础上,但因整个生态系统中99%的微生物是不可培养,此方法因费时费力,培养菌群有限而发展受限[20]。随着测序技术的发展和生物信息学的进步,变性梯度凝胶电泳[21]、扩增子[22]和宏基因组[23]测序被相聚用于表征和分析各种环境下的微生物群落,获取微生物的分类图谱,进行菌群多样性和菌群功能预测分析。本研究利用16S rRNA基因高通量测序技术,将首次在黑鲷传统池塘养殖和池塘流水槽循环水养殖两种养殖模式下,从养殖水体、黑鲷肠道细菌群落结构和多样性的角度,探究2种养殖模式对黑鲷肠道及养殖水体微生物群落结构以及组成的影响。
1 材料与方法
1.1 试验鱼来源
养殖实验在江苏省南通市通州湾渔业发展有限公司进行。实验所用黑鲷苗种由江苏省海洋水产研究所海水增养殖技术及种苗中心自主培育,分别在池塘循环水流水槽(砖混结构)和传统养殖池塘2种养殖模式条件下饲喂60天。每种养殖模式3个平行组。流水槽由气提推水增氧区、养殖区(长20 m×宽5 m×水深2 m)、集排污区(30 m×5 m)和水质净化区(5.33 hm2);传统池塘面积为0.13 hm2,水深2.5 m。海水水质清新、无污染,符合《渔业水质标准》。
1.2 样品采集与处理
养殖60天后,进行实验采样,采样前试验鱼饥饿24 h。每个试验平行组随机取4尾鱼,保证每个模式取12尾鱼。采用MS-222(200 mg/L)将其麻醉,先用75%的乙醇擦拭体表,在无菌环境下立即解剖采集肠道,无菌解剖刀刮取肠道内容物,每个平行组2尾鱼的肠道内容物合并为1个样品,冷冻保存,后续用于总DNA提取。在养殖水体均匀分散10处点各采集3 L水样,混匀,用0.22 μm滤膜抽滤收集,分装6份,冷冻保存。
1.3 样本总DNA提取、PCR扩增及测序
根据E.Z.N.A.® soil试剂盒(Omega Bio-tek,Norcross,GA,U.S.)说明书进行总DNA抽提,DNA浓度和纯度利用NanoDrop2000进行检测,利用1%琼脂糖凝胶电泳检测DNA提取质量;用338F(5’-ACTCCTACGGGAGGCAGCAG-3’) 和 806R(5’-GGACTACHVGGGTWTCTAAT-3’)引物对 V3-V4 可变区进行PCR扩增,扩增程序为:95℃预变性3 min,27个循环(95℃变性30 s,55℃退火30 s,72℃延伸30 s),最后72℃延伸10 min(PCR仪:ABI GeneAmp® 9700型)。扩增体系为20 μL,4 μL 5*FastPfu缓冲液,2 μL 2.5 mmol/L dNTPs,0.8 μL 引 物 (5 μmol/L),0.4 μL FastPfu聚合酶;10 ng DNA模板。
使用2%琼脂糖凝胶回收PCR产物,利用AxyPrep DNA Gel Extraction Kit(Axygen Biosciences,Union City,CA,USA)进行纯化,Tris-HCl洗脱,2%琼脂糖电泳检测。利用QuantiFluor™-ST(Promega,USA)进行检测定量。根据Illumina MiSeq平台(Illumina,SanDiego,USA)标准操作规程将纯化后的扩增片段构建PE 2*300的文库。构建文库步骤:(1)连接“Y”字形接头;(2)使用磁珠筛选去除接头自连片段;(3)利用PCR扩增进行文库模板的富集;(4)氢氧化钠变性,产生单链DNA片段。利用Illumina公司的Miseq PE300平台进行测序(上海美吉生物医药科技有限公司)。
1.4 数据处理与分析
原始测序序列使用Trimmomatic软件质控,使用FLASH软件进行拼接:(1)设置50 bp的窗口,如果窗口内的平均质量值低于20,从窗口前端位置截去该碱基后端所有序列,之后再去除质控后长度低于50 bp的序列;(2)根据重叠碱基overlap将两端序列进行拼接,拼接时overlap之间的最大错配率为0.2,长度需大于10 bp。(3)根据序列首尾两端的barcode和引物将序列拆分至每个样本,barcode需精确匹配,去除存在模糊碱基的序列。使用的UPARSE软件,根据97%的相似度对序列进行OTU聚类,并在聚类的过程中去除单序列和嵌合体。利用RDP对每条序列进行物种分类注释,比对Silva数据库(SSU123),设置比对阈值为70%。
2 结果与分析
2.1 基于16S rRNA测序的菌群多样性特征
4组24个样品所测得的原始数据共1315602条,通过对测序所得的序列进行预处理,过滤低质量、长度短和模糊序列后,获得用于后续分析的有效合格16Sr DNA序列1086274条,每个样本产生的有效序列数目在33013~ 70087之间,有效序列的平均长度为432 bp,序列覆盖度>99.7%,表明测序真实反映各样品的微生物组成。对所有序列进行97%相似水平OTU划分并物种注释,可划分为1730个OTU,分类为34个门、72个纲、168个目、324个科、719个属、1091个种。
黑鲷肠道和养殖水体中细菌群落的物种丰富度和多样性结果如图1所示。Sobs和Ace指数均表明,2种养殖模式条件下,黑鲷肠道菌群丰富度极显著高于其对应的养殖水体(P≤0.01);IP模式水体菌群丰富度显著高于EP模式水体(P≤0.01);IP模式肠道菌群丰富度与EP模式肠道差异不显著(P>0.05)。Simpson指数显示,EP和IP两种模式养殖黑鲷肠道菌群多样性无显著性差异(P>0.05);EP模式水体菌群多样性高于IP模式水体(P≤0.05),而Pd指数显示相反的结果(P≤0.05)。Pd指数为谱系多样性指数,它同时考虑物种丰度以及进化距离。Pd指数显示,EP和IP两种模式黑鲷肠道菌群多样性均极显著高于其对应的养殖水体(0.001
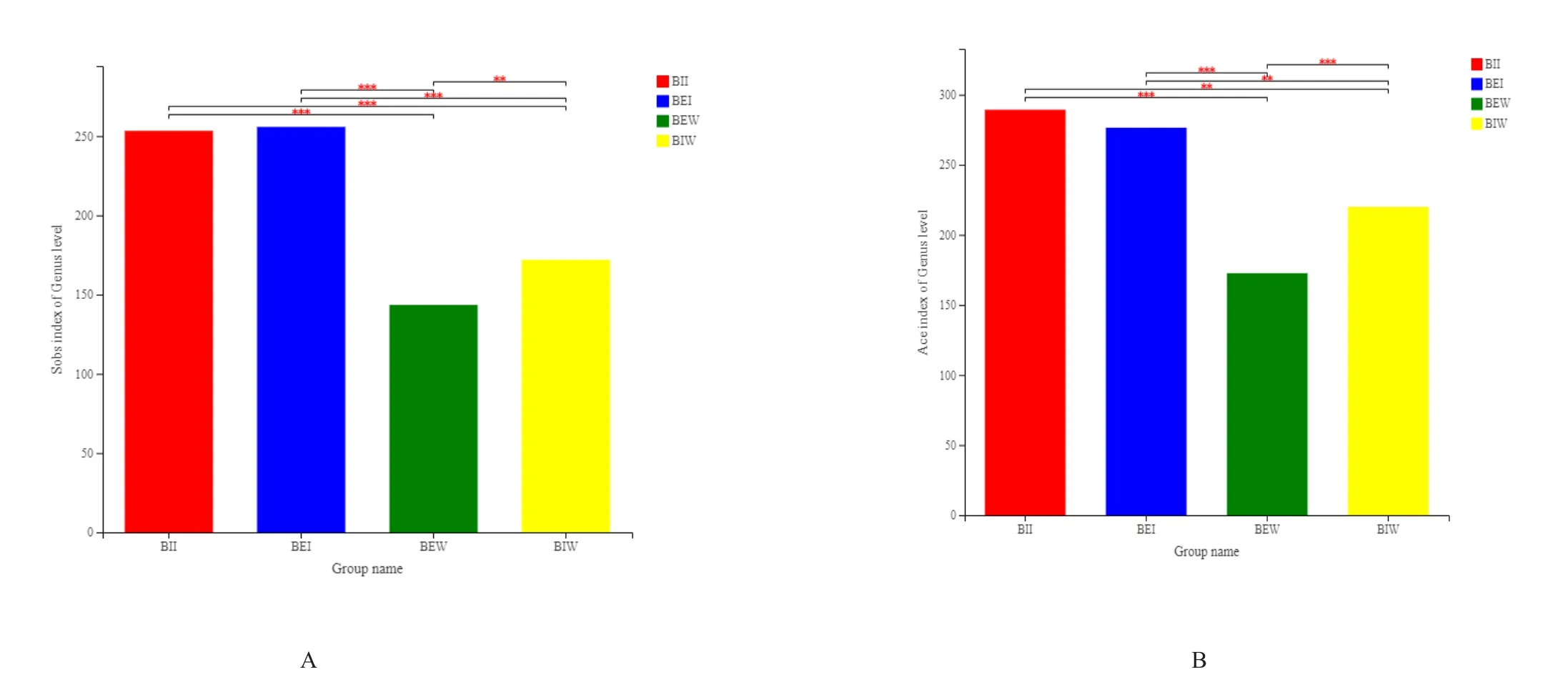

图1 两种模式下4组样品中细菌丰富性和多样性
利用主坐标分析(PCoA)反映两种模式下菌群群落的β多样性,主成分1(PC1)贡献率36.68%,主成分2(PC2)的贡献率为18.86%(图2A),不同组的肠道、养殖水体菌群明显分开,且组内重复样品菌群群落聚集在一起,这表明样品具有很好的重复性,组间具有明显的微生物群落差异,种间差异明显大于种类差异(P≤0.01)。样品菌群层次聚类分析结果与PCoA类似,4组样品分别聚为一簇,而其中两种模式养殖水体样品菌群结构距离更近(图2B)。

图2 两种模式下所有样品微生物群落PCoA排序图(A)和层级聚类分析(B)
2.2 两种模式下菌群结构组成
由图3可以看出,在门水平上,两种养殖模式下菌群结构组成种类比较相似,优势菌群主要由变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、蓝藻细菌(Cyanobacteria)、厚壁菌门 (Firmicutes)、拟 杆 菌 门(Bacteroidetes)和绿弯菌门(Chloroflexi)组成,其丰度之和为98.32%~ 99.22%。为了减少群落的复杂性和冗余性,相对丰度低于1%的群落都归为稀有种,并为“others”[24]。
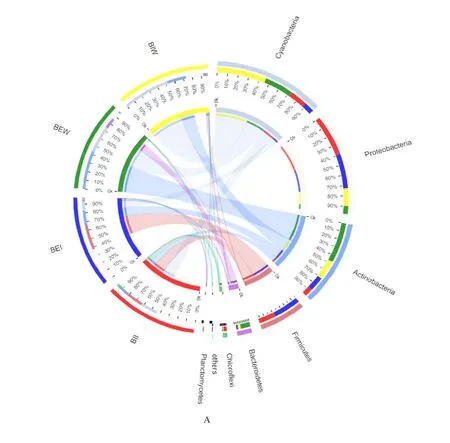
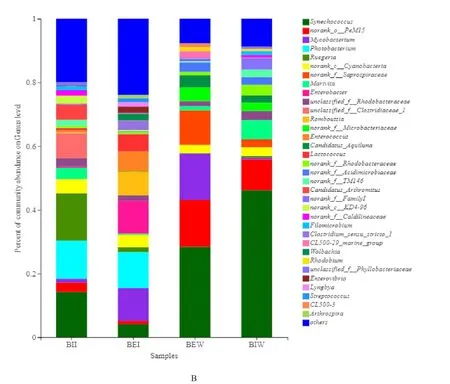
图3 两种模式下4组样品菌群组成(A:门水平,B:属水平)
由图4可以看出,在属水平上,EP模式肠道菌群丰度大于1%的菌属有17个,优势菌属主要包括发光杆菌属 Photobacterium(11.30%),肠杆菌属Enterobacter(10.31%),分枝杆菌属Mycobacterium(10.30%),Romboutsia(7.37%),肠球菌属 Enterococcus(6.29%),乳球菌属Lactococcus(5.38%);IP模式肠道菌群丰度大于1%的菌属有15个,优势菌属主要包括鲁杰氏菌属Ruegeria(14.84%),聚球藻属Synechococcus(14.24%),发光杆菌属 Photobacterium(11.97%),未分类梭状芽孢杆菌属unclassified_f_clostridiaceae(7.96%);EP模式水样菌群丰度大于1%的菌属有14个,优势菌属主要为:聚球藻属(28.41%),norank_o_PeM15(14.71%),分 枝 杆 菌 属 (14.60%),norank_f_Saprospiraceae(10.86%);IP模式水样菌群丰度大于1%的菌属有13个,优势菌属主要为:聚球藻属(46.19%),norank_o_PeM15(9.71%),Marivita(5.99%)。
2.3 两种模式下差异菌属分析
运用Wilcoxon秩和检验检测不同组中菌群群落中表现出的丰度差异的物种。对所有4组样品进行属水平差异分析,并显示丰度在前20的菌属,图4中可看出,18个具有统计学显著差异(0.01≤P≤0.05),占90%;3个具有极显著差异(0.001≤P≤0.01),占15%;14个具有极显著差异(P≤0.001),占70%。具有显著差异的主要菌属为聚球藻属、norank_o__PeM15、分枝杆菌属、鲁杰氏菌属、norank_f__Saprospiraceae、Marivita、肠杆菌属、unclassified_f__Clostridiaceae_1、Romboutsia、norank_f__Microbacteriaceae、肠球菌属、Candidatus_Aquiluna、乳 球 菌 属 、norank_f__Rho dobacteraceae、norank_f__Acidimicrobi aceae、norank_f__TM146、Candidatus_Arthromitus。分别对相似生境的组样品进行两两比较分析,在不同模式下的黑鲷肠道样品中,排名前15的丰富菌属中有10个菌属有显著性差异(P≤0.05),占66.67%,其中聚球藻属、Ruegeria、norank_o__PeM15、Marivita、norank_C__K D4-96(IP>EP),分枝杆菌属、肠杆菌属、Romboutsia、肠球菌、乳球菌属IP<EP)。在不同模式下的养殖水体样品中,排名前15的丰富菌属中有13个菌属有显著性差异(P≤0.05),占86.67%,其中聚球藻属、Marivita、unclassified_f__Rhodobacteraceae、norank_f__Rhodob acteraceae、norank_f__FamilyI、norank_f__TM146(IP>EP),norank_o__PeM15、分支杆菌属 、norank_f__Saprospiraceae、norank_f__Microbacteriac eae、Candidatus_Aquiluna、CL500-29_marine_group、红菌属Rhodobium(IP<EP)。

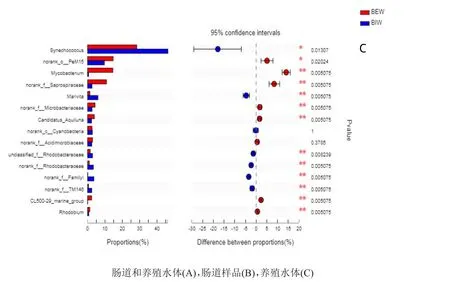
图4 两种模式下菌属差异分析
3 讨论与结论
本研究中,高通量测序获得1086274条有效序列,序列覆盖度超过99.7%,结合Sobs、Ace、Simpson、Pd指数显示黑鲷肠道和养殖水体中细菌群落非常丰富和多样,这与众多研究结果显示一致[25-27],验证了高通量测序在微生物群落结构研究中的绝对优势[28]。不同养殖模式黑鲷肠道和其相应的养殖水体之间,与不同的养殖模式水体之间的微生物丰富度和多样性存在显著差异,说明不同养殖模式形成的不同养殖环境具有不同的微生物菌群结构;而不同养殖模式黑鲷肠道之间无显著差异,因为试验黑鲷来自同一批繁育鱼苗,肠道对于菌群是特殊的寄生环境,相同的宿主肠道对菌群具有一定的宿主选择性。分析微生物多样性研究中,较多的研究者选择Simpson和Shannon指数[29-30],而采用Pd指数相对较少,在本研究中,Simpson指数显示EP模式水体菌群多样性高于IP模式水体,而Pd指数显示结果恰好相反。张志敏[31]对南方鲇孵化后8dpf、35dpf和125dpf的肠道、摄食食物和生长水样微生物多样性比较分析,得出3个不同阶段,除了Pd指数外,水体中的多样性指数普遍高于摄食食物。因此,在进行多样性分析时尽量选择两种以上的分析方法,多角度评价阐述。
和很多动物一样,生存环境对水生动物的具有特定的选择性压力,最终逐渐促使其对环境的适应。不同环境具有不同的环境因子胁迫,所形成的微生态也是有差异的,同时环境微生态对鱼类具有免疫压力[31],大量的研究从水质调控、生态进化及宿主的环境影响角度探究鱼类微生物群落组成特征[32-34]。而对于不同养殖模式菌群结构研究较少。李存玉等[35]对工厂化和池塘养殖牙鲆肠道菌群结构差异性和系统进化分析表明,两种养殖条件下牙鲆肠道菌群结构与饵料中菌群关系密切,此外受养殖水环境中菌群影响较大。池塘养殖和海上吊笼养殖仿刺参肠道菌群的结构存在较大差异,且在池塘养殖仿刺参肠道内茅孢杆菌等益生菌的占比较高,海上吊笼养殖仿刺参分离得到较多的弧菌属细菌[36]。本文在池塘循环水流水和传统池塘黑鲷养殖两种模式下开展菌群结构研究,其组成在门水平种类比较相似,优势菌群主要由变形菌门、放线菌门、蓝藻细菌、厚壁菌门、拟杆菌门和绿弯菌门组成,这与许多报道一致[37-39]。此外,在所有样品菌属差异分析得出丰度前20的菌属中90%具有显著差异,结合层次聚类、主成分分析表明四组样品明显分开聚类,说明养殖模式可以影响微生物群落组成结构[40]。
两种模式黑鲷肠道样品中,前15的丰富菌属中有10类的菌属有显著性差异,占66.67%,其中,聚球藻、鲁杰氏菌和Marivita菌属是循环水池塘养殖模式下黑鲷肠道中丰富显著优势可鉴定菌属群。聚球藻是海洋光合自养原核生物的优势代表类群,是全球碳循环的主要参与者和初级生产力的主要贡献者,在海洋微食物网中生物量循环迅速,能量转换效率高[41]。陈竺等[42]研究得出聚球蓝细菌可作为饲料添加剂,能够提高免疫相关酶的活性,增强幼虾对白斑综合征病毒感染的防御能力。Miura等[43]从水族馆保存的一个健康鱼群中成功分离到3株Ruegeria sp.菌株,抑制了珊瑚弧菌P1的生长,得出Ruegeria sp.在保护珊瑚免受致病性弧菌感染方面的潜在作用。林国荣等[44]在对有无浒苔水华的海水养殖池塘中菌群组成分析发现,Marivita是无浒苔水华池塘中的优势菌群。传统池塘养殖黑鲷肠道样品中分支杆菌属、肠杆菌属、Romboutsia、肠球菌属、乳球菌属丰度具有显著优势,其中丰度差异最高的分支杆菌属,能导致分枝杆菌病,是一种海洋硬骨鱼病原菌[45],其中主要病原菌海洋分歧杆菌是一种产生慢性病的细菌,在某些情况下可能导致暴发性死亡[46]。Wilson等[47]研究尼罗罗非鱼感染海洋分枝杆菌时脾脏黑素巨噬细胞中心反应,得出感染海洋分枝杆菌的罗非鱼黑色素瘤噬细胞MMCs中黑色素含量升高,其MMCs作为工具来确定鱼的健康状况[48-49],分枝杆菌感染被描述为形成一种非常难以治疗的慢性病。肠杆菌是肠道正常菌,但是部分也是条件致病菌,具有抗生素的固有和获得性耐药[50],Seker等[51]报道了阴沟肠杆菌对健康鲻鱼具有较强的致病性,可造成大量死亡。陈雪峰[52]等从浙江湖州罗氏沼虾育苗场暴发的幼体大规模发病死亡新病害中分离确定的病原菌为阴沟肠杆菌,阴沟肠杆菌在幼体体内进行了大量增殖,成为绝对优势种,进而导致幼体感染死亡。
本研究表明,不同黑鲷养殖模式形成不同的菌群结构,池塘循环水养殖模式下黑鲷肠道中具有明显差异的优势菌属大多为有益菌,而传统池塘养殖黑鲷肠道多为慢性致病菌或条件致病菌属。鱼类通过肠道微生物结构改变来增强免疫能力是其适应各自生境的一种手段,为此,获得相关的研究结果,丰富对不同养殖模式微生物群落特征信息的了解,同时为循环水养殖新模式的推广提供有力的科学依据。
