基于深度学习和组织病理图像的癌症分类研究进展
2021-10-12陈丽萌李锦涛
颜 锐,陈丽萌,李锦涛,任 菲
1中国科学院计算技术研究所, 北京 1001902中国科学院大学,北京 1000493中国医学科学院 北京协和医学院 北京协和医院肾内科,北京 100730
受人口老龄化、环境污染、不良生活习惯和膳食结构变化等因素的影响,癌症已成为全世界共同面临的重大公共健康问题[1]。作为癌症诊断的 “金标准”,病理诊断的准确性和及时性直接影响患者治疗方案的选择和预后。在传统病理诊断流程中,病理医生在显微镜下直接对病理切片进行镜检,然后根据经验独立作出病理诊断。随着全切片扫描图像 (whole slide images,WSI)技术的快速发展,病理切片的数字化给病理医生带来了诸多好处,如无须再通过显微镜观察病理切片,而是可以通过屏幕更加方便快捷地反复观看和定量分析病理图像。WSI的推广应用,促使病理学进入了新的发展阶段。
当然,病理学的发展目前仍然面临巨大挑战。首先,世界上普遍缺乏病理学家,而这一问题在我国尤为突出,2018年,我国有执照的病理科医生仅10 000余人,缺口达90%[2];其次,病理诊断是否正确完全取决于病理医生的专业知识和诊断经验,受个人主观因素的影响,诊断结果往往并不一致。面对上述挑战,迫切需要开发客观且准确的病理图像分析方法。在所有病理图像分析任务中,最重要的是分类任务,其是细胞核定位[3]、有丝分裂检测[4]、腺体分割[5]、病理图像检索[6]等深入研究的基石。
在自然图像和医学影像领域,基于深度学习的图像分类方法已取得显著成效[7],但由于病理WSI存在分辨率极高、病理特征差异细微、细胞重叠、颜色分布不均匀等特点,WSI自动分类一直缺乏突破性进展。近年来,一些创新性的方法不断涌现,使得基于病理图像的自动癌症分类成为可能。本文从病理图像分类常用的4种深度学习方法入手,对基于深度学习和组织病理图像的癌症分类(包括分级)研究进行回顾和总结,并对该领域未来发展进行展望。
1 常用的病理图像分类深度学习方法
对于病理图像,一般较易获得WSI对应的分类标签(诊断结果)。一张几万×几万像素的WSI对应一个分类标签,切分WSI可得到几万张几百×几百像素的图像小块(Patch),即几万张Patch对应一个分类标签,此为典型的弱监督学习场景。而某些医学问题需根据先验知识对病理图像的特定区域(比如癌症区域)先进行标注,这种标注可以是对Patch的逐个标注,也可以是描绘感兴趣区域的轮廓。实际处理时,对感兴趣区域轮廓的标注亦转换为对Patch的逐个标注,即一张Patch对应一个分类标签,此为典型的监督学习场景。病理图像分类常用的监督学习和弱监督学习方法包括:卷积神经网络(convolutional neural network, CNN)、循环神经网络(recurrent neural network, RNN)、图卷积神经网络(graph convolutional network, GCN)和多示例学习(multiple instance learning, MIL)。
1.1 CNN
CNN是计算机视觉领域使用最广泛的深度学习方法。传统的全连接神经网络直接应用于图像任务会导致一些问题,比如图像展开成向量后空间信息的丢失以及全连接参数量巨大导致的过拟合现象。CNN针对此类问题在局部感受野、权值共享、卷积和池化操作等方面采取了新的改进机制,在分类、检测、分割、检索、生成和去噪等任务上均取得了显著效果[8]。常见的用于图像分类的CNN包括VGG[9]、ResNet[10]、GoogLeNet[11]、DenseNet[12]、Xception[13]、MobileNet[14]等。在现有的硬件和算法条件下,直接采用CNN对WSI进行处理并不可行,但可将对WSI的处理问题分解为对Patch的处理子问题,从而适用常规的CNN方法。
1.2 RNN
RNN主要用于处理和预测序列数据,其最重要的变体之一是长短时记忆网络(long short term memory,LSTM)。LSTM 单元之间相互连接形成有向循环,从而创建了网络的内部状态,使网络能够记住远处输入,解决了数据之间的长期依赖问题[15]。由于WSI分辨率极高,故往往将WSI切分成Patch后再进行处理,这导致切分后Patch之间的近邻关系或拓扑结构部分丢失,而RNN及其变体的优势正好弥补了这一缺点。RNN在进行WSI分析前,一般先采用CNN提取Patch的特征表示向量,然后基于特征表示向量再使用RNN进行融合,二者可共同进行端到端训练,也可分为两阶段进行训练。
1.3 GCN
GCN在具有空间拓扑结构的图数据方面存在巨大优势,如交通网络、蛋白质相互作用网络、社交网络、文献引用网络等[16]。GCN可以处理节点级别的任务和图级别的任务,其中节点级别的任务包括节点分类、链路预测等;图级别的任务包括图分类、图生成等。现有的GCN主要分为谱方法和空间方法两类,谱方法利用图上傅里叶变换和卷积定理从谱域定义图卷积,空间方法则通过在节点域定义节点相关性实现图卷积。对于WSI,切分出的Patch之间具有各种空间拓扑结构,这和组织结构的复杂性及癌症的异质性有关。将Patch作为图的顶点,然后根据Patch之间的距离构建图的边,即可将WSI转换为图结构的数据,从而利用GCN对WSI进行图级别和节点级别的分析。
1.4 MIL
MIL是一种弱监督学习方法[17-18],通过对包(包有标签,示例无标签)的学习建立MIL模型,并将该模型应用于未知包的预测。既往MIL在图像上的应用仅是把图像小块向量化作为示例,并未与CNN结合起来。自2018年起,Ilse等[19]提出的基于注意力的深度MIL将MIL与CNN有效结合,为MIL在图像领域的应用揭开了新的序幕。对于WSI分类问题,虽然获取像素级别的人工标注非常困难且价格昂贵,但WSI级别的分类标签较易获取。一张几万×几万像素的WSI,即几万张Patch,对应一个分类标签,此场景正好与MIL的适用场景相吻合。
上述4种常用的深度学习方法已应用于乳腺癌、结/直肠癌、肺癌、前列腺癌等多个领域的病理图像分类(表1)。
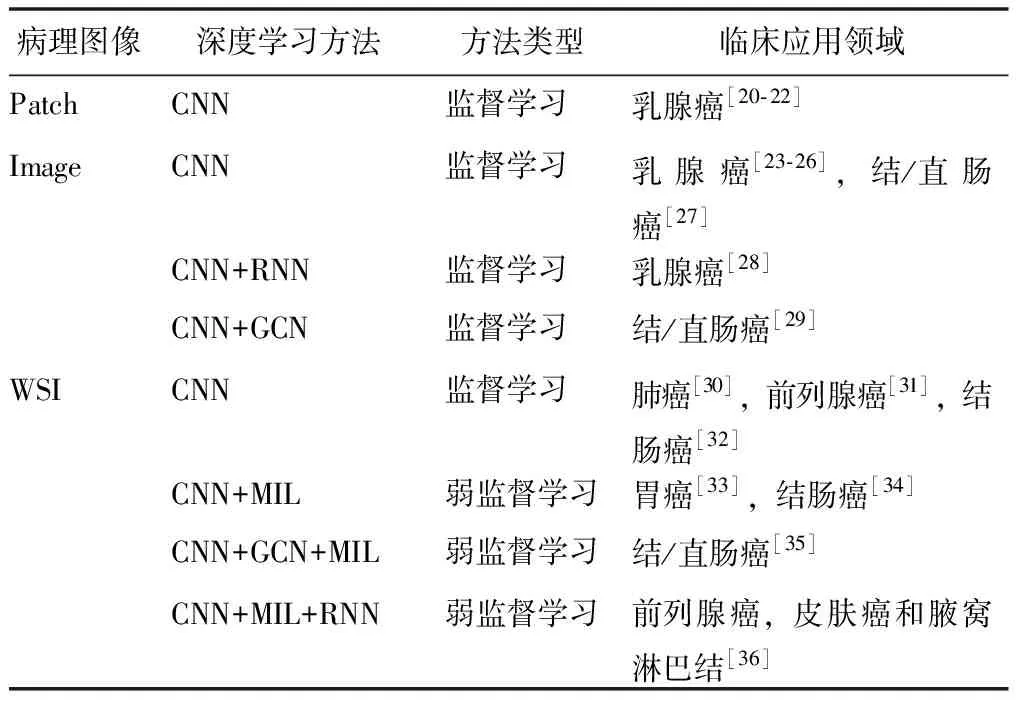
表1 4种常用的深度学习方法的临床应用
2 癌症分类
准确的癌症分类可以帮助医生对患者的治疗作出整体规划,同时评估患者预后。根据研究成果出现的时间顺序,下文逐一介绍基于Patch(几百×几百像素)、Image(几千×几千像素)和WSI(几万×几万像素)的癌症分类相关研究进展。3种不同尺度病理图像示例见图1。
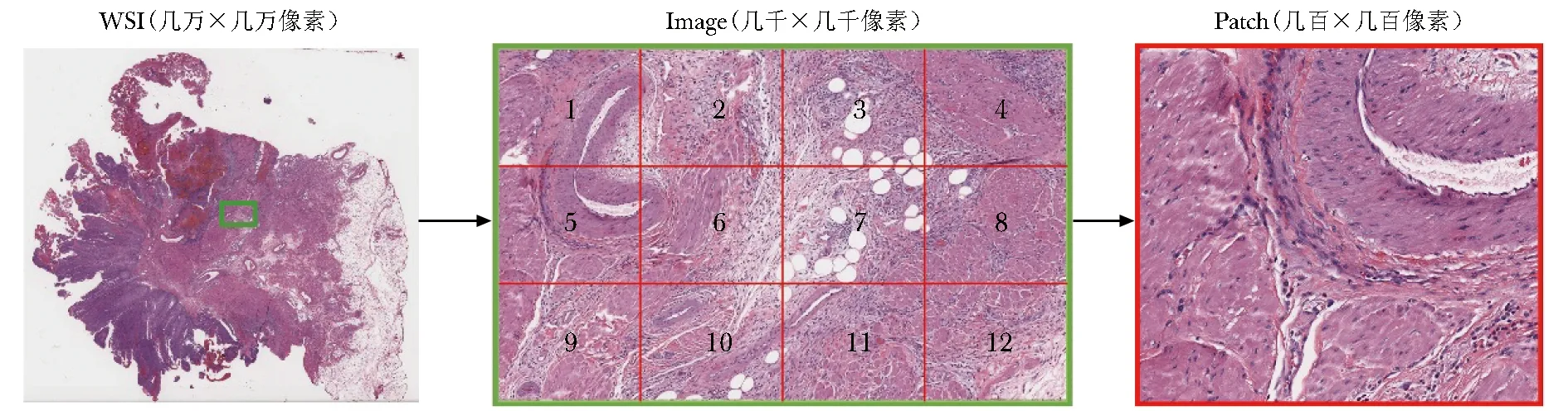
图1 不同尺度病理图像示意图
2.1 基于Patch的癌症分类
基于深度学习和病理图像的癌症分类始于Patch分类,其一般方法框架如图2所示。Spanhol等[37]于2016年公开了乳腺癌良/恶性二分类的病理图像数据集BreaKHis。在此数据集的基础上,Spanhol等[20]利用AlexNet网络和多种融合策略进行Patch分类,分类准确度比传统机器学习算法提高了6%。Bayramoglu等[21]在BreaKHis数据集基础上使用了不依赖于图像放大倍数的深度学习方法,Patch分类准确度约为83%。Araújo等[22]进一步考虑了乳腺癌病理图像的四分类(正常组织、良性组织、原位癌和浸润性癌)问题,先利用CNN提取Patch的特征,然后利用支持向量机(support vector machine,SVM)算法对特征进行分类,该团队在构建的乳腺癌分类数据集基础上,Patch平均分类准确度为77.8%。
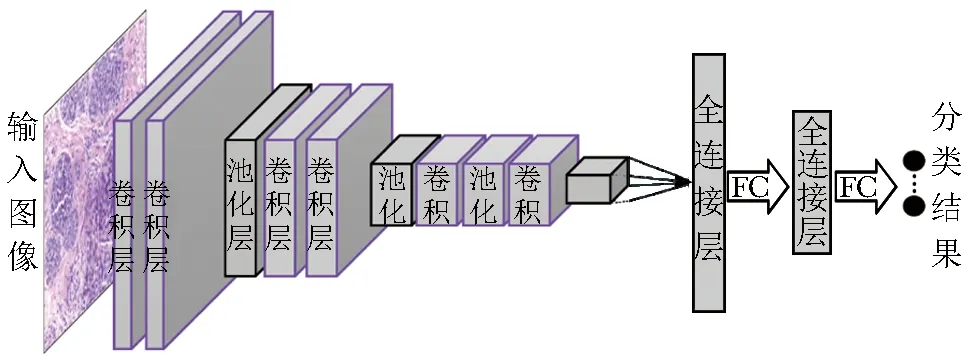
图2 基于深度学习的病理图像(Patch)分类方法的典型框架
2.2 基于Image的癌症分类
基于Image的癌症分类研究是从Patch到WSI的重要过渡。如果简单地将高分辨率的图像压缩为低分辨率的图像,则不可避免地会丢失大量有用的图像信息,导致较差的分类效果。目前,基于Image的癌症分类算法采用了大致相同的研究思路:首先将Image切分成大小相等的Patch,然后采用CNN对每张Patch进行分类,最后将一整张Image的所有Patch分类结果整合在一起以确定最后的Image分类结果。总的来说,基于深度学习的Image病理图像分类方法按时间顺序的演变路径总结如下:(1)CNN + 多数投票法; (2)CNN + SVM; (3)CNN + 迁移学习 + 多数投票法或SVM; (4)CNN + 迁移学习 + Patch捆绑 + 多数投票法或SVM。其中,SVM也可以是其他传统的有监督的机器学习分类算法。
基于Image的癌症分类研究是从ICIAR2018国际挑战赛[38]中的乳腺癌分类任务开始流行的。ICIAR2018公开数据集的病理图像分辨率为2048×1536像素,围绕此数据集相继开展了多项Image分类算法研究。Vesal等[23]使用基于迁移学习的GoogLeNet和ResNet预训练模型,首先对Image的每个Patch进行分类,再使用多数投票法得到Image的分类结果。Vang等[24]提出先使用GoogLeNet进行Patch分类,然后通过包含多数投票、梯度增强机和逻辑回归的集成融合框架进行Image分类。Rakhlin等[25]使用了一种称为深度卷积特征表示的方法,首先对病理图像进行通用网络编码,得到低维的稀疏描述符,然后用梯度增强树进行分类。Yan等[26]先将一张Image切分为大小相等的12张Patch,采用ResNet提取12个特征向量代表这12张Patch,然后将其中位置相邻的Patch绑定在一起进行分类,最后通过对绑定的Patch分类结果进行多数投票决定癌症分类类别。上述方法均是基于Patch得到最终的Image分类结果,要么直接采用多数投票法和传统的机器学习分类器,要么仅集成Patch之间的短距离空间关系,却忽略了Patch之间的长距离空间关系的重要作用。针对这一问题,本研究团队提出了一种混合神经网络算法以同时保留Patch之间长、短距离空间关系,该方法首先将高分辨率的Image切分成Patch,然后利用一种通过 CNN 提取图像的多级特征的方法得到 Patch 的特征表示,最后利用双向LSTM融合Patch的特征表示进行Image分类,整体框架如图3所示[28]。研究结果表明,对于乳腺癌四分类任务,该方法获得了91.3%的平均准确度。

图3 基于深度学习的病理图像(Image)分类方法的典型框架[28]
2.3 基于WSI的癌症分类
相较于Patch和Image分类,WSI分类面临的挑战更加艰巨,如分辨率极高、标注不足等。这些难点导致WSI虽然包含更多的信息量,但分类效果并不如Patch和Image好,为应对此类挑战,研究者进行了大量探索。例如,为将肺的WSI分为正常、肺腺癌或鳞状细胞癌3类,Coudray等[39]先对WSI进行简单的预处理,然后开发了基于迁移学习的InceptionV3分类模型对Patch进行分类,最后对Patch的分类结果进行多数投票,或对Patch的分类概率进行平均,从而得到WSI的分类结果;整个模型的受试者工作特征曲线下面积(area under the curve,AUC)为0.97,显著高于既往模型,且高于病理学家的分类准确度。Adnan等[40]利用GCN进行WSI分类,并将WSI的特征表示学习过程分为2个阶段进行:首先从WSI中采样重要的Patch并将其建模为全连接的图,然后通过GCN将此图转换为向量表示即WSI的特征表示;研究者从The Cancer Genome Atlas(TCGA)数据集中收集了1026张肺癌WSI,结果表明该方法能够很好地区分肺腺癌和肺鳞状细胞癌2种肺癌亚型,准确度为88.8%,AUC为0.89。
像素级的WSI标注代价昂贵,越来越多的研究者尝试采用弱监督学习进行WSI分类。Chikontwe等[34]提出了一种可以同时学习示例级嵌入和包级嵌入的MIL框架用于WSI分类,且其提出的中心损失函数可在嵌入空间中最小化类内距离,该方法在2个结/直肠癌病理图像数据集上分别取得了92.31%和98.72%的分类准确度。Campanella等[36]采用仅需病理报告中的诊断结果作为标注的MIL方法进行WSI分类,具体流程:首先在Patch层面采用CNN和MIL进行训练,从而得到WSI中的Patch分类概率,然后基于概率大小筛选Patch,利用RNN融合选出的Patch从而得到WSI分类结果。这项研究收集了来自15 187例患者的44 732张WSI。基于前列腺癌、基底细胞癌和乳腺癌转移至腋窝淋巴结的WSI测试数据集得出的AUC均高于0.98。该方法的临床应用将使病理科医生在保证100%灵敏度的前提下筛除65%~75%的WSI,从而将精力集中于重要病例的诊断上。Chen等[32]也使用弱监督学习的方法进行了WSI分类,与常用的弱监督学习方法MIL不同的是,其仅在CNN的基础上通过提出修正的交叉熵损失函数和上界转换损失函数进行WSI的弱监督分类,使得在仅有WSI标签进行模型训练的情况下,Patch的分类结果可以达到病理学家的水平。Wang等[30]同样使用弱监督学习方法进行了肺癌四分类(腺癌、鳞状细胞癌、小细胞肺癌和正常组织),同时指出少量的粗注释有助于进一步提高WSI分类的准确度。该方法首先利用基于Patch的全卷积神经网络来选择有区分性的块,然后采用不同的Block特征选择方法和特征聚合策略,得到WSI的全局描述子,最后将全局描述子输入随机森林算法从而得到WSI分类结果,整体框架如图4所示。Wang等[33]提出了胃部WSI三分类(正常、异型增生和癌症)的重校准MIL框架,该框架首先利用CNN网络选择出每张WSI中有区分性的示例,然后仅根据这些示例训练重校准MIL框架,使其能够捕获示例间的依存关系,并根据从融合特征中学习的重要性系数重新校准示例特征。

图4 基于深度学习的病理图像(WSI)分类方法的典型框架[30]
3 癌症分级
癌症分级属于细粒度分类任务,不同分级的病理图像之间差别非常细微,仅采用一般的深度学习方法无法胜任此类任务。因此,针对不同的癌症分级任务,研究者设计了不同网络结构和方法框架。本研究团队提出了可在端到端学习过程中集中注意力于细胞核相关特征的网络(NANet)进行乳腺癌分级[27]。NANet包含两个分支:主干分支用来提取病理图像的特征表示,细胞核分支用来提取细胞核相关的特征表示,通过NANet中的“指导”模块,在不同卷积层中重点关注细胞核相关的特征。“指导”模块在端到端的学习中起到了一种类似于注意力机制的作用,从而使得网络整体上学习到更加细粒度且更具代表性的特征表示以进行乳腺癌分级。当前的结/直肠癌分级方法仅利用了Patch信息,而通过评估细胞水平的信息以及腺体的形态考量整个组织的微环境非常重要,为克服目前这一分级方法的不足,Zhou等[29]提出了一种新的细胞图卷积神经网络(CGC-Net)。CGC-Net将Image转换为图,其中每个节点用原始病理图像中的一个细胞核表示,节点之间的边(细胞相互作用)用节点相似性表示,结果表明,相较于传统基于Patch的方法,将Image建模为图可有效处理更大像素的Image(约为Patch的16倍),并可对复杂的组织微环境进行建模。Nagpal等[31]提出了用于前列腺活检标本全扫描图像的格里森分级深度学习系统,该系统包括两个阶段:第一阶段采用通过神经网络搜索技术得到的CNN对WSI中每个Patch进行分类,第二阶段采用支持向量机和从结果热图中提取的特征进行整体等级组的分类,其分级效果与亚病理专家的诊断能力一致。Raju等[35]综合利用了GCN和基于注意力机制的MIL,构建了结/直肠癌TNM分级系统。具体方法:首先从WSI中随机选取一些Patch,并采用基于纹理的方法提取这些Patch的特征表示;然后基于Patch构建出一系列的图以保留Patch之间的空间关系,并将这些图作为后续的基于注意力机制的MIL的示例。该方法分类准确度为81.1%,显著高于目前最好的结/直肠癌TNM分级方法。
4 展望
基于深度学习和组织病理图像的癌症分类研究为计算机辅助诊断及预后预测提供了新的模式,但目前仍无法在临床上广泛应用。笔者通过分析其中原因,提出如下亟需解决的问题和未来可能的发展方向。
首先,WSI最大的特点是分辨率极高,简单地把深度学习领域对自然图像分析的方法迁移过来应用是不够的。需研究专门针对WSI的深度学习研究框架,如果对WSI的分类也可以实现端到端的学习和推断,未来则可能得到更好的WSI分类效果。
其次,与自然图像动辄百万张的标注相比,病理图像的标注(特别是像素级标注)困难且价格高昂。针对这一问题,一方面可从弱监督算法方面缓解标注缺乏的问题,另一方面需要投入更大的力量去构建足够大的基准数据集。
再次,目前关于WSI的大多数研究集中于设计更加高效的网络模型,这样单方面的研究存在很大的局限性。针对超高分辨率图像的深度学习方法研究,需从网络模型优化和并行计算等不同角度同时展开。
最后,目前对病理图像的研究和医院中其他模态数据的研究是割裂的。应融合多组学、影像、检查检验、电子病历等一切可用的不同模态的数据进行癌症分类研究,从而更好地辅助诊断和预后预测。
作者贡献:颜锐、陈丽萌、李锦涛、任菲负责论文选题构思和校对;颜锐、任菲负责文献收集,论文撰写、修订。
利益冲突:无
