施肥方式对干旱半干旱地区土壤氨氧化微生物数量和群落的影响
2021-07-09郭岩彬孟凡乔邵小明刘宝驹吴文良
陈 竹,郭岩彬,孟凡乔,邵小明,刘宝驹,吴文良,*
1 中国农业大学资源与环境学院, 北京 100000 2 贵州大学农学院, 贵阳 550000
硝化作用介导铵态氮向硝态氮转化的过程, 它不仅影响氮对植物的有效性, 还与面源污染和 温室气体排放有关[1]。氨氧化是硝化作用的限速步骤, 也是评估土壤氮循环的重要指标[2]。氨氧化古菌 (ammonia-oxidizing archaea, AOA) 、氨氧化细菌 (ammonia-oxidizing bacteria, AOB)[2- 4]和新近发现的全程氨氧化微生物 (complete ammonia oxidizer, Comammox)[5]是氨氧化的主要参与者。AOA是强酸性土壤中氨氧化作用的主要驱动者[6], 但AOA最高仅能耐受20 mM的铵, 这远低于多数AOB适宜的铵浓度 (50—1000 mmol/L[7]), 因此在高氮或碱性土壤中, 氨氧化过程多由AOB主导[8-9]。
长期定位施肥会对氨氧化微生物的活性、群落丰度和结构产生深刻影响[10]。大量研究表明,中性和碱性土壤上施用氮肥只改变AOB的群落组成和丰度,酸性土壤上则影响AOA[11- 14]。但杨亚东等[15]和罗培宇等[16]对水稻土和东北棕壤研究结果表明,施用氮肥后AOA和AOB的群落丰度和多样性都显著升高。这表明淹水、低温和干旱等极端环境下可能产生独特的氨氧化微生物种群,它们对施肥的响应有待进一步研究。
干旱半干旱生态系统占全球陆地面积的 41%, 全球约有 38% 的人口居住于此[17], 发展和可持续的矛盾在这一地区非常突出。目前, 对干旱半干旱地区农田土壤氨氧化微生物影响的报道较少。本研究以内蒙古农牧业科学院旱作实验站的长期定位实验为基础, 利用实时荧光定量PCR、末端限制性片段长度多态性(Terminal Restriction Fragment Length Polymorphism, T-RFLP) 和克隆文库分析:1) 长期施用化肥、有机肥对干旱半干旱地区农田土壤AOB 和 AOA 群落数量、结构以及活性的影响;2) 影响 AOB、AOA 群落变化的主要环境因子。
1 材料与方法
1.1 实验设计与样品采集
本研究土壤样品采自中国农业大学武川长期定位试验地 (40°59′N, 110°33′E), 土壤为栗钙土[18]。该地属于温带大陆性季风气候, 年平均气温2.5℃, 年降雨量343.6 mm。试验始于2004 年, 采用马铃薯—油菜—莜麦一年一熟轮作制。共设置5 个处理, 肥料用量及配比根据测土配方施肥确定:对照 (CK, 不使用肥料)、氮 (N, 单施尿素, 60 N kg/hm2)、有机肥 (O, 单施羊粪, 37.5 N kg/hm2, 15 P2O5kg/hm2, 55.5 K2O kg/hm2)、氮磷钾 (NPK, 尿素+磷酸二铵+氯化钾, 60 N kg/hm2, 45 P2O5kg/hm2, 30 K2O kg/hm2)、氮磷钾+有机肥 (NPK+O, 尿素+磷酸二铵+氯化钾 (60 N kg/hm2, 45 P2O5kg/hm2, 30 K2O kg/hm2) +羊粪 (37.5 N kg/hm2, 15 P2O5kg/hm2, 55.5 K2O kg/hm2))。化肥混匀后在播种时开沟侧施, 有机肥于每年作物播种前均匀撒在地表, 随翻土施入。每个处理三个重复, 随机区组排列, 小区面积3 m2, 试验区面积50 m2。样品于2012年7月采集,地上部作物为莜麦。每小区用土钻随机采集5个土壤样品后均匀混合。土壤样品过 2 mm 筛后, 用于DNA提取和酶活测定的样品保存于 -80℃, 其余样品风干后保存待测。
1.2 土壤理化性质测定
土壤 pH 以水土比 1∶2.5 测定。土壤铵态氮及硝态氮用流动分析仪 (Bran-Luebbe, Germany) 测定。土壤有机质和全氮用碳氮分析仪 (Thermo Fischer, USA) 测定。土壤磷酸盐的测定依据 Olsen等[19]的方法。土壤PNR采用氯酸钾抑制法测定[20]。
1.3 DNA的提取及氨氧化微生物 amoA基因实时荧光定量PCR测定
土壤DNA由 0.25 g 土壤用 PowerSoil DNA Isolation Kit (Mobio, USA) 依据说明书提取。AOB 的amoA基因实时荧光定量 PCR 引物为amoA- 1F/amoA- 2R[21]。反应程序为:94℃预变性 120 s;94℃ 变性 45 s, 57℃ 退火 1 min, 72℃ 延伸 45 s, 共35个循环。AOA 的amoA基因实时荧光定量 PCR 引物为 CrenamoA23f/CrenamoA616r[22]。反应程序为:94℃ 预变性 2 min;94℃ 变性 45 s, 53℃ 退火 1 min, 72℃ 延伸 45 s, 共35 个循环。反应体系均为20 μL:10 μL SYBR Premix Ex Taq (Takara, 大连), 上下游引物各1 μL, 2 μL BSA, 1 μL 的模板或标线DNA, 6 μL 灭菌超纯水。标准曲线以AOA和AOB的amoA基因克隆制备质粒。AOA 在83℃ 收集反应荧光信号, 氨氧化细菌在 81℃ 收集反应荧光信号以防止引物二聚体所引起的误差。反应完成后设置溶解曲线用以检验产物的特异性, 其程序为 55℃ 至 99℃ 之间, 每 1℃读数一次。qPCR扩增反应在 ABI 7500 Real-time PCR (ABI, USA) 上进行。
1.4 amoA基因的T-RFLP分析
AOBamoA基因的T-RFLP 分析用amoA- 2R和6-carboxyfluorescein (FAM) 标记的amoA- 1F 作为引物。PCR程序为 94℃ 变性 5 min;然后 94℃ 变性 30 s、57℃ 退火 30 s、72℃ 延伸 60 s, 共 35个循环;最后72℃ 延伸 10 min。电泳检测 PCR 产物特异性, 用 PCR 产物纯化试剂盒 (Axygen, USA) 纯化。纯化产物用限制性内切酶MspI (NEB, UK) 酶切。AOAamoA基因的T-RFLP 分析用 CrenamoA616r 和 FAM 标记的CrenamoA23f 作为引物。PCR程序为 94℃ 变性 5 min;然后 94℃ 变性30 s、60℃ 退火 30 s、72℃ 延伸 60 s, 共 35个循环;最后 72℃ 延伸 10 min。电泳检测 PCR 产物特异性, 然后用 PCR产物纯化试剂盒 (Axygen) 纯化PCR产物。纯化产物用限制性内切酶MboI (NEB) 酶切。酶切产物与甲酰胺和 GeneScanTM- 500 Liz (ABI) 混合。酶切片段长度用 ABI-PRISM 3030XL Genetic Analyzer (ABI, UK) 软件检测。每个峰的相对丰度用 GeneMarker version 2.2 (http://www.softgenetics.com) 软件进行分析。
1.5 克隆文库的构建与分析
将每个处理3个重复的DNA单独进行 PCR 扩增,扩增体系同1.4。每个处理的PCR产物等体积混合后用pEASY-T1 载体 (TransGen Biotech, Beijing) 进行克隆并测序。序列由BLAST-N在线比对验证。AOB 和 AOA 分别构建5个文库,每个文库包含12 条序列。用 MEGA v.4.0以 neighbor-joining 法构建进化树。
1.6 统计分析
使用 one-way ANOVA 的 Duncan′s 检验在P< 0.05 水平计算各指标在处理间的差异显著性。用 Spearman 相关性指数计算各指标之间的相关性。用R语言 (http://www.R-project.org) 的“Vegan”程序包进行ANOSIM 分析以比较处理间AOB和AOA 的群落结构的相似性, 冗余分析 (redundancy analysis, RDA) 结合BIOENV来分析环境因子对群落的影响, 控制方差膨胀因子使其小于10, 并用蒙特卡洛置换检验分析环境因子和群落之间的相关性。
2 结果
2.1 土壤理化性质
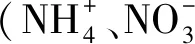
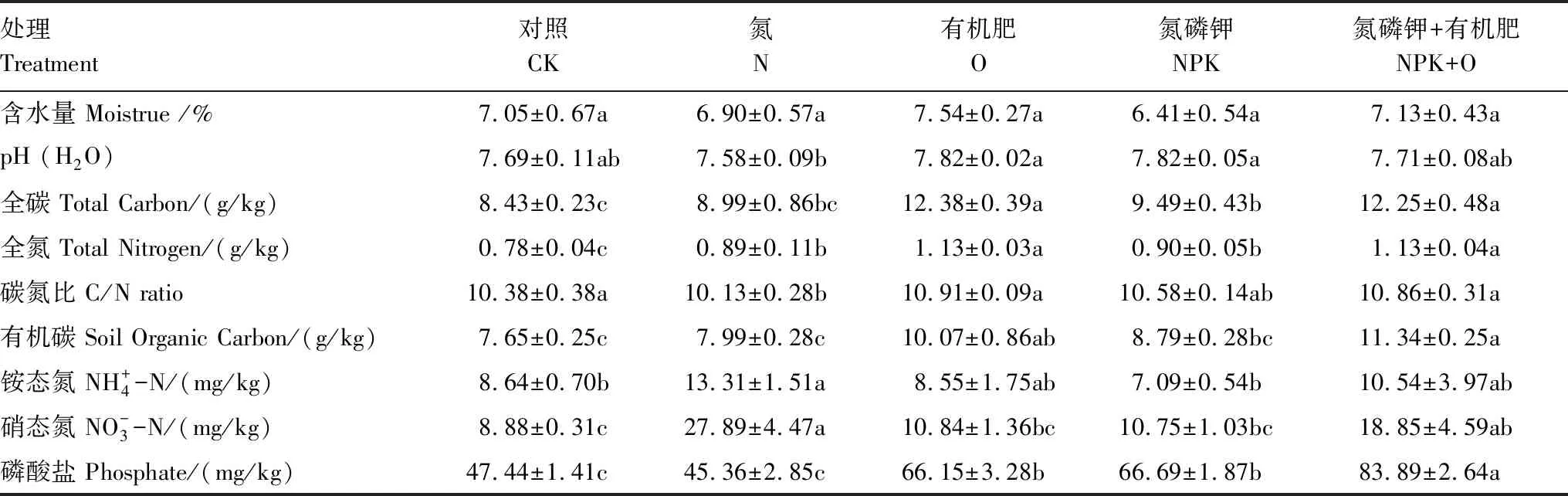
表1 土壤理化性质
2.2 硝化潜式和氨氧化微生物群落丰度

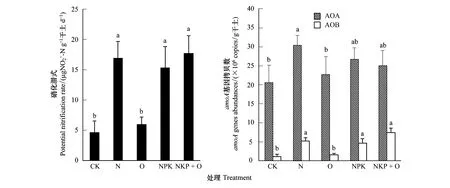
图1 不同处理土壤的硝化潜式和氨氧化细菌和氨氧化古菌的数量Fig.1 Potential nitrification rate, bacterial and archaea amoA genes abundances in different treatment *处理:不施肥 (CK);氮 (N);有机肥 (O);氮磷钾 (NPK);氮磷钾+有机肥 (NPK+O);平均值+标准误 (n=3);不同字母表示不同处理间有显著差异性 (P < 0.05)

表2 AOA、AOB丰度与PNR、土壤化学性质的相关性

2.3 氨氧化微生物群落结构
基于AOAamoA基因的T-RFLP分析得到5个末端限制性片段(图2)。ANOSIM 分析表明各处理间土壤 AOA 群落间没有显著性差异 (R=0.08,P=0.202; 表3)。基于AOBamoA基因的T-RFLP分析得到4个末端限制性片段, 包括tRF 60 bp、tRF 156 bp、tRF 235 bp和tRF 256 bp (图2)。tRF 156 bp 在CK处理土壤中的相对丰度显著高于其他处理, N处理土壤中tRF 256 bp 的相对丰度最高。O处理土壤中tRF 60 bp 的相对丰度显著高于其他处理。ANOSIM分析表明各处理间土壤AOB 群落都有显著性差异 (P< 0.05; 表3)。
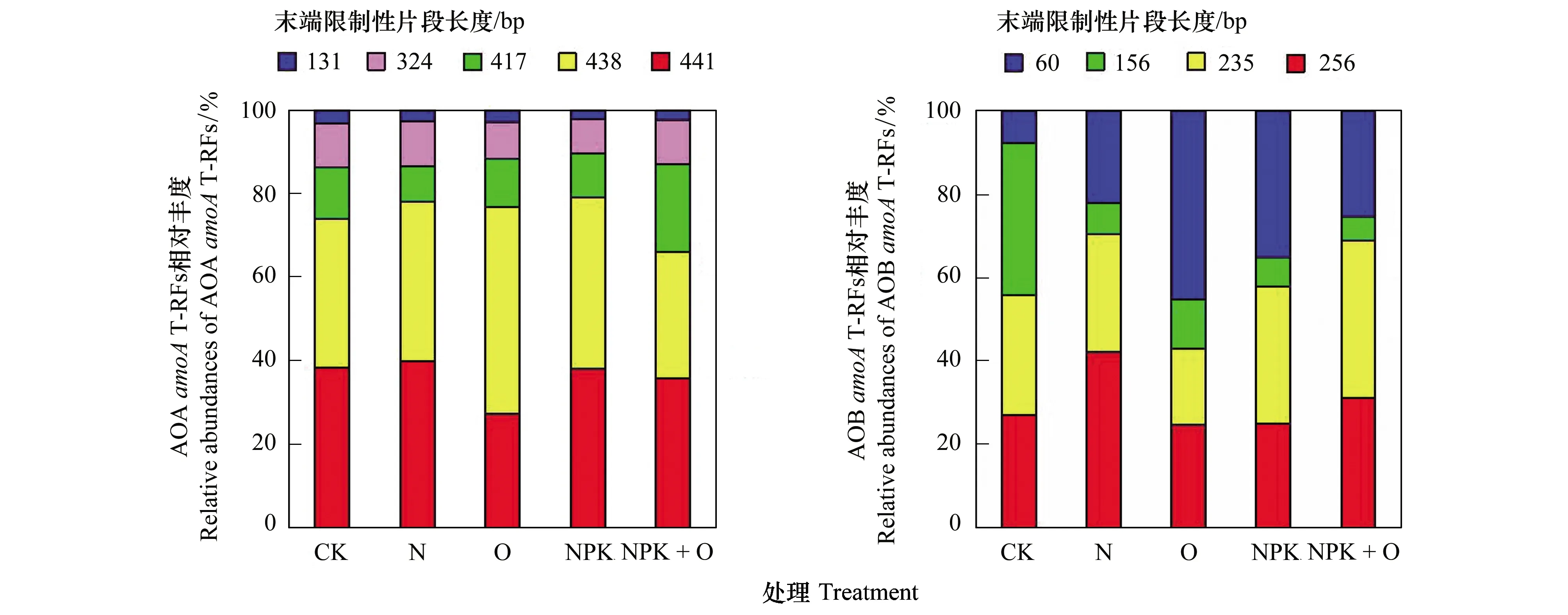
图2 不同处理中土壤氨氧化古菌和氨氧化细菌的群落结构Fig.2 T-RFLP fingerprints of the archaea and bacterial amoA gene fragments in different treatment *平均值+标准误 (n=3)
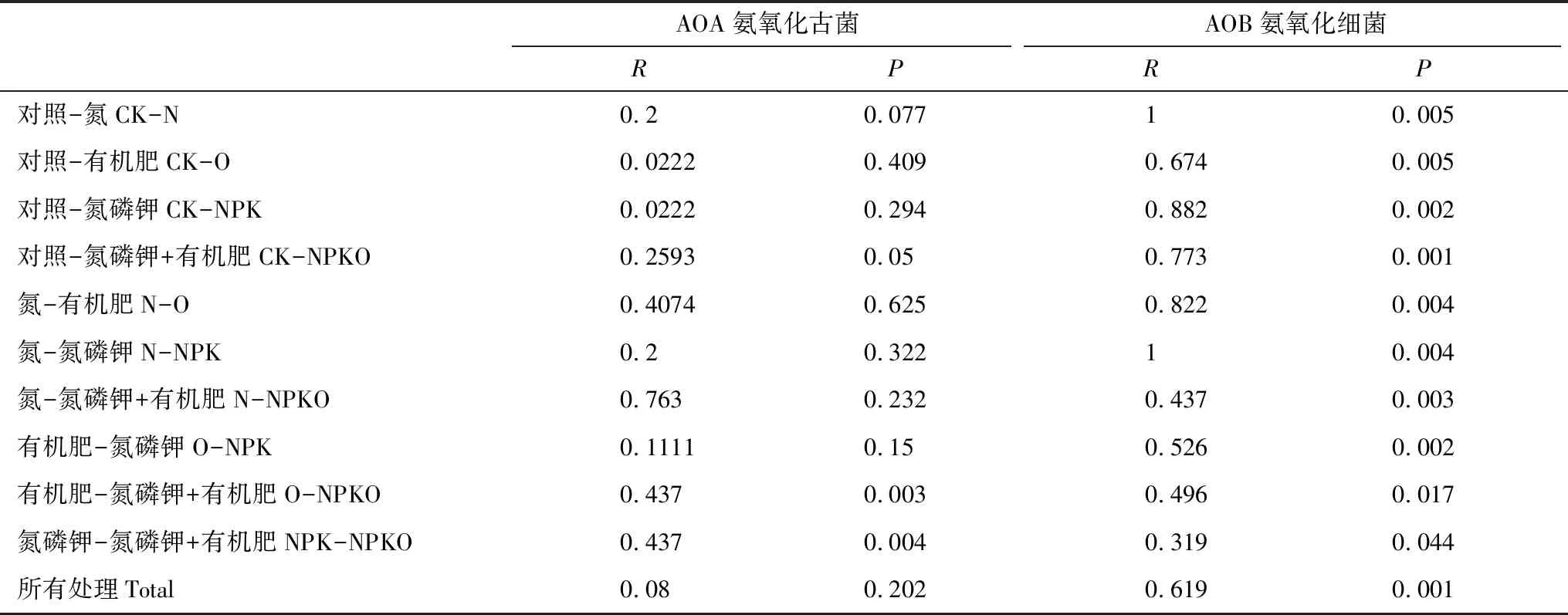
表3 氨氧化细菌和古菌群落的ANOSIM分析
通过构建 AOB 文库, 检测到 4 个 tRF 片段, 包括 tRF 256 bp、tRF 235 bp、tRF 156 bp 和tRF 62 bp (图3)。系统发育树表明所有序列都属于亚硝化螺菌 (Nitrosospira), 主要分为NitrosospiraCluster 3a.1、Cluster 3a.2、Cluster 3b、Cluster 9和Cluster 2-related。CK土壤中主要种群为NitrosospiraCluster 3a.1 (75%),而在N、O、NPK 和NPK+O处理的土壤中NitrosospiraCluster 3a.2 的比例更高(>50%)。属于NitrosospiraCluster 3b的序列仅在O处理的土壤中检测到,其模拟酶切tRF片段大小为 60 bp。仅在N处理的土壤中发现模拟酶切tRF片段大小为 256 bp 的序列,它与Nitrosospirasp. Nsp58 (Cluster 2-related) 有很高的相似性 (97% sequence identity)。RDA 和BIOENV分析表明pH和土壤含水量可以完全解释AOB 群落的变化 (100%),是影响 AOB 群落结构变化的关键因子 (rho=0.179,Pr< 0.05) (图4)。通过构建 AOA 文库, 检测到 7个 tRF 片段, 包括 tRF75 bp、 tRF 131 bp、 tRF 324 bp、 tRF 417 bp、tRF 438 bp、tRF 441 bp 和tRF 550 bp, 系统发育树表明所有序列都属于Cluster I.1b (Nitrososphaere, 图5)。
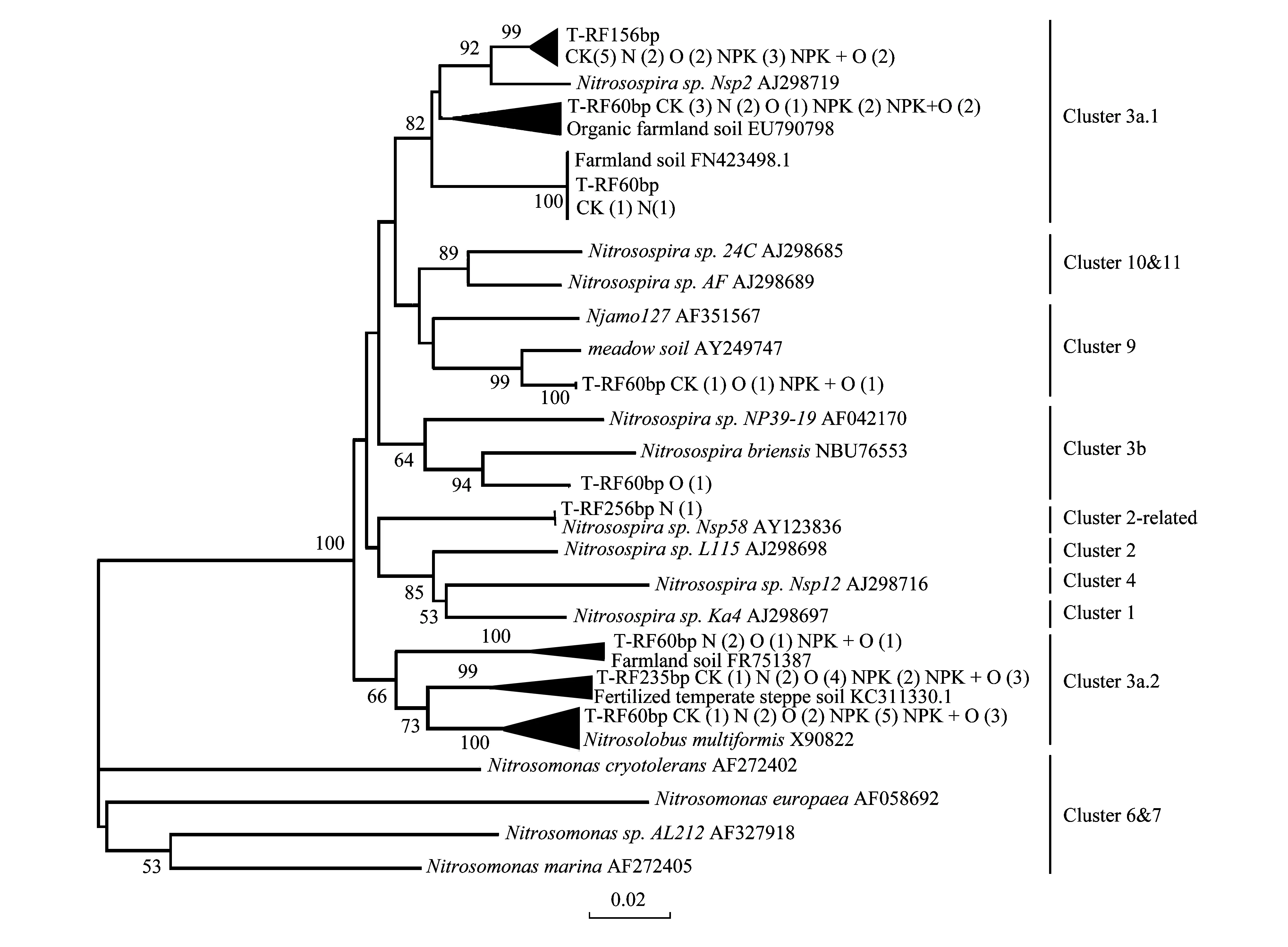
图3 不同处理中氨氧化细菌 amoA 基因的遗传进化树Fig.3 Neighbor-joining phylogenetic tree of the bacterial amoA sequences in different fertilization regimess
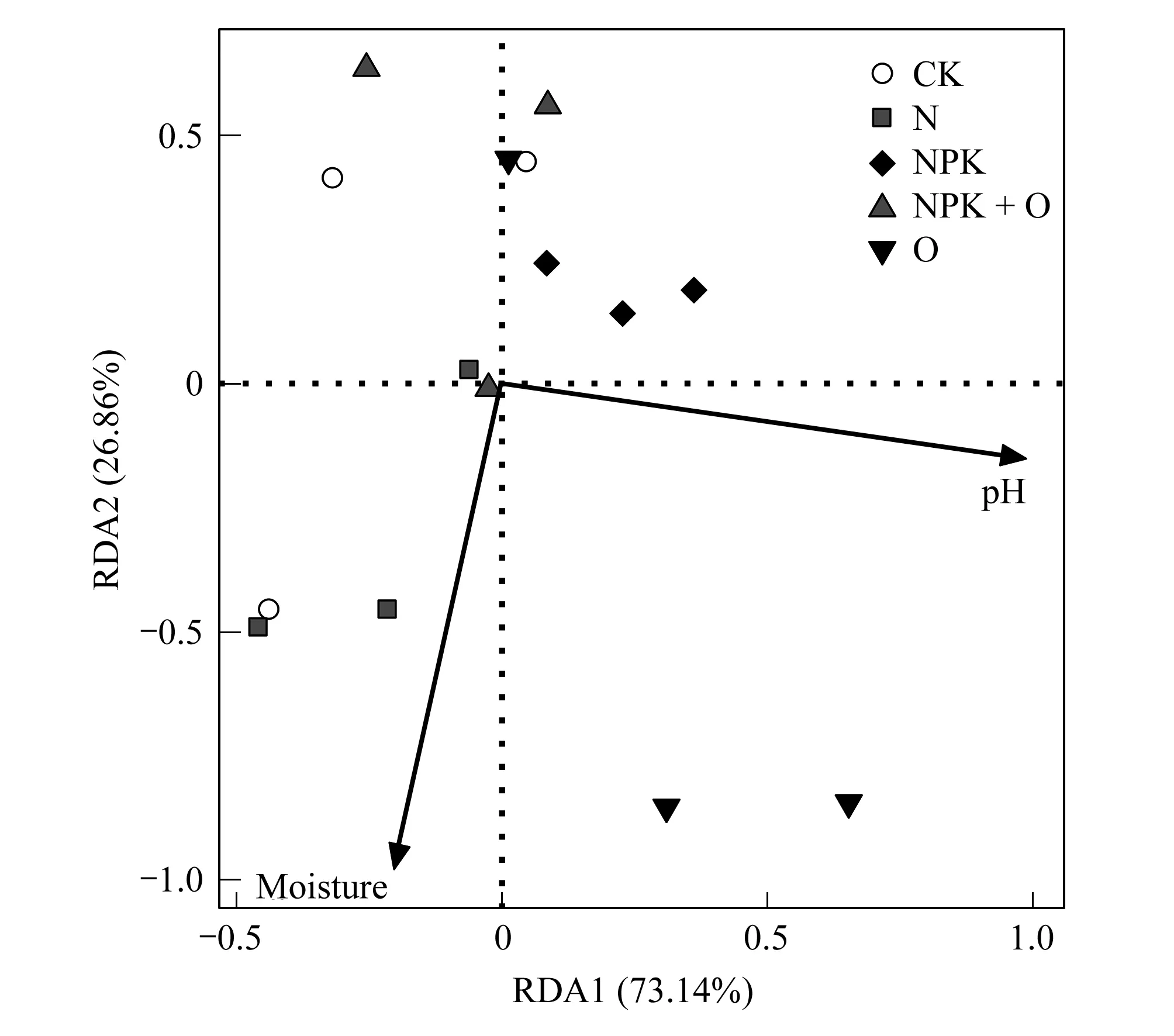
图4 不同土地利用方式下土壤氨氧化细菌群落结构RDA分析Fig.4 RDA of soil parameters effect on ammonia-oxidizing bacterial communities using the T-RFLP data *不施肥 (CK);氮 (N);有机肥 (O);氮磷钾 (NPK);氮磷钾+有机肥 (NPK+O)
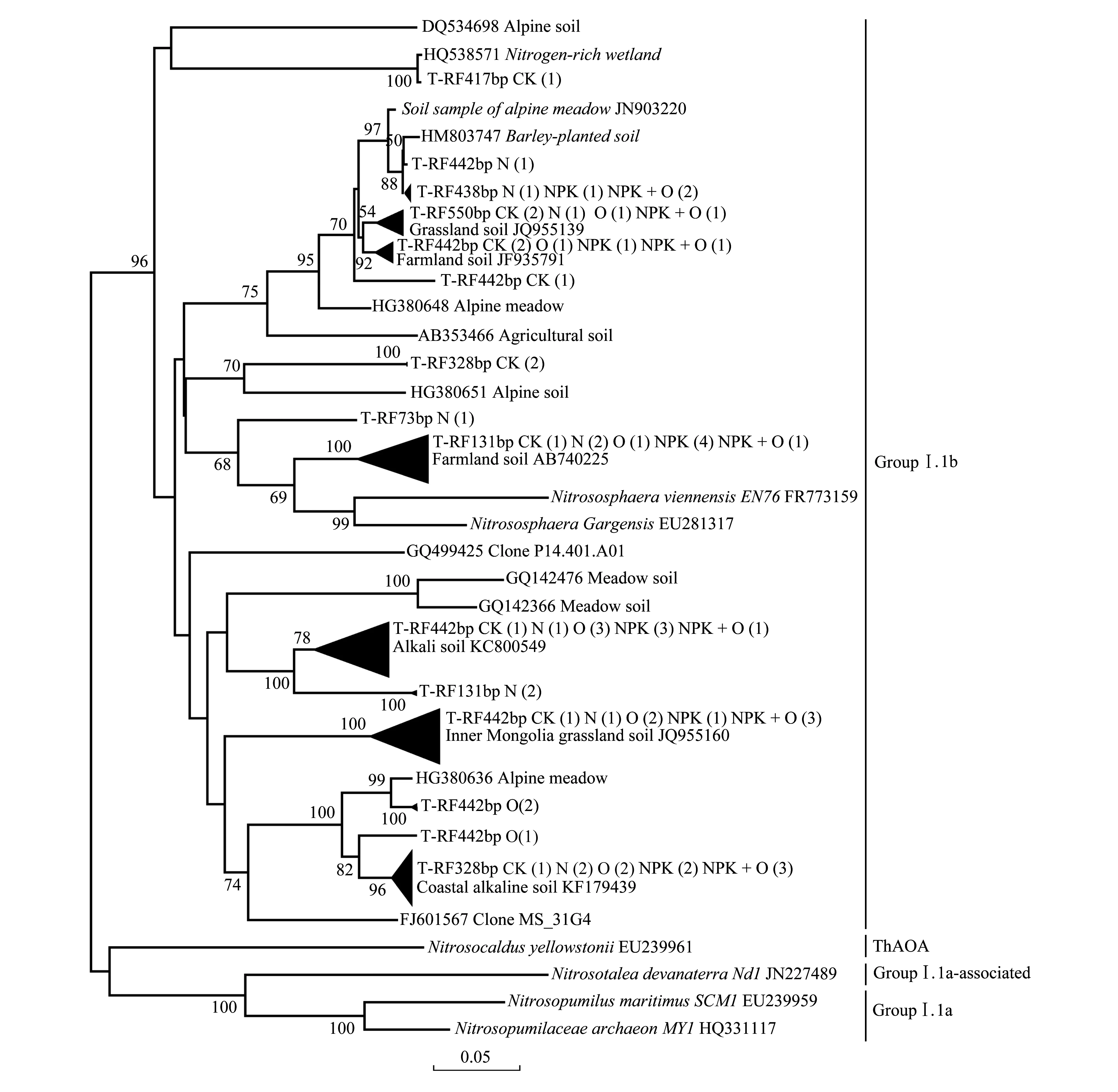
图5 不同处理中氨氧化古菌 amoA 基因的遗传进化树Fig.5 Neighbor-joining phylogenetic tree of the archaea amoA sequences in different fertilization regimess
3 讨论
不同施肥处理土壤AOAamoA基因拷贝数在 (1.65×107—3.34×107)拷贝数/g干土之间, 高于AOBamoA的基因拷贝数 (5.99×105—1.21×107) 拷贝数/g干土, 这与在潮土[13]、碱性壤土[11]、红壤[12]上的研究结果一致。多数研究表明, 碱性土壤中施用氮肥不影响AOA群落丰度[11, 23], 但在本研究中, 无论施用化肥还是有机肥, AOA数量都有升高的趋势,其中,单施尿素处理土壤AOA数量显著高于CK (图1), 这与Hu等[24]在干旱半干旱草原上的研究结果一致。在干旱环境中, 极低的土壤含水量会阻断物质传递, 受根系和微生物吸收铵时分泌氢离子影响,土壤中可能形成大量利于AOA繁殖的酸性微环境。
N、NPK和NPK+O处理土壤的AOBamoA基因拷贝数和PNR显著高于CK (图1), 这与Shen[11]、Cui[23]等在碱性土壤上的结果一致, 表明施用化肥后会促进AOB的增长, 增大硝态氮淋洗的风险。AOBamoA基因克隆文库的结果表明, 93%以上的序列都属于Nitrosospiracluster 3 (图 4)。Nitrosospiriacluster 3 在农田土壤 AOB 群落中的主导地位已经被广泛验证[1,10]。T-RFLP分析和文库分析均表明,Nitrosospiriacluster 3a.1是CK处理中的优势种群, 而施用氮肥后,优势种群转变为Nitrosospiriacluster 3a.2 (图 2, 图 3)。这与Guo[25]和Chen[26]等的研究结论一致。与CK相比, 施用有机肥没有增加AOBamoA基因拷贝数 (图1)。有机肥中的铵态氮矿化缓慢, 而且有机肥的施用可能促使其他土壤微生物生长并与AOB竞争无机氮[27-28]。值得注意的是,Nitrosospiriacluster 3b的比例在O处理土壤中显著升高 (图 2, 图 3), 表明与Nitrosospiriacluster 3a相比,Nitrosospiriacluster 3b更适应有机氮源[12,29]。