克氏原螯虾养殖群体的SLAF测序及遗传多样性分析
2021-04-15刘亚楠刘洁魏上李亚琳李明肖俊邱高峰陆颖
刘亚楠 刘洁 魏上 李亚琳 李明 肖俊 邱高峰 陆颖

摘要:【目的】探究克氏原螯蝦养殖群体内和群体间的遗传多样性,为引种和群体间杂交以改善养殖群体种质提供参考依据。【方法】以湖北、江西、安徽、浙江和江苏5省克氏原螯虾主产地14个养殖群体的120个个体为研究对象,采用简化基因组测序技术——特定区点扩增片段测序技术(SLAF-seq)进行基因组测序,获得基因组SNP基因型数据,构建群体系统发育进化树,并进行群体结构、主成分和遗传多样性分析。【结果】共鉴定出741147个单核苷酸多态性(SNP)位点,群体系统进化分析表明,14个群体的种源主要来自浙江金华和江苏宿迁,然后再向各地引种迁徙。群体结构分析结果与系统进化分析结果相吻合。主成分分析结果揭示了安徽长丰和滁州群体、湖北荆州龙口及和平2个群体,以及浙江东阳群体等5个群体与其他群体间存在相对较远的亲缘关系,是杂交引种的潜在种源。群体遗传多样性分析显示,14个群体的Ho介于0.2171~0.2801,平均值为 0.2476,He介于0.3424~0.3598,平均值为0.3534,PIC介于0.2750~0.2878,群体间相差不大,接近0.25,各群体均接近遗传多样性中等水平的下限。【结论】14个克氏原螯虾养殖群体内的遗传多样性较低,群体间的亲缘关系较接近,品种单一、长期内交迹象明显。通过群体的基因组重测序,能以较低的成本实现对养殖群体遗传多样性的定期监测。
关键词: 克氏原螯虾;SLAF-seq;SNP;遗传多样性;群体遗传学
中图分类号: S932 文献标志码: A 文章编号:2095-1191(2021)12-3265-09
Analysis of genetic diversity among the farmed Procambarus clarkii populations using the SLAF-seq technology
LIU Ya-nan1,2, LIU Jie1,2, WEI Shang1,2, LI Ya-lin1,2, LI Ming3,
XIAO Jun4, QIU Gao-feng1,2, LU Ying1,2*
(1Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education,Shanghai Ocean University, Shanghai 201306, China; 2International Research Center for Marine Bioscience,Ministry of Science and Technology, Shanghai Ocean University, Shanghai 201306, China; 3Aquatic Products Technology Extension
Station of Jinhua City, Jinhua, Zhejiang 321017, China; 4Guangxi Academy of Fishery Sciences/Guangxi
Key Laboratory of Aquatic Genetic Breeding and Healthy Aquaculture, Nanning 530021, China)
Abstract:【Objective】This study was designed to investigate the intra-group and inter-group genetic diversity of farmed Procambarus clarkii populations,and to provide fundamental data bases for improving the germplasm of breeding groups through required introduction and crossbreeding. 【Method】A total of 120 individuals of 14 farmed populations collected in Hubei, Jiangxi, Anhui, Zhejiang and Jiangsu were sequenced with a Specific-Locus Amplified Fragment Sequencing (SLAF-seq) technology, one of the restriction site-associated DNA sequencing technologies. The single nucleotide polymorphisms (SNPs) were identified to conduct the analyzation of phylogeny, population structure, principal component and genetic diversity. 【Result】According to the identified 741147 SNPs in this study, the phylogenic analysis suggested that the 14 populations originated from two main origins, Jinhua and Suqian. Then they were introduced and migrated to different places. The results of the population structure analysis agreed with the phylogenetic analysis. The principal component analysis revealed that the five populations, including Changfeng (Anhui), Chuzhou (Anhui), Longkou, Jingzhou (Hubei), Heping, Jingzhou (Hubei) and Dongyang (Zhejiang) had relatively distant genetic relationships to the others, which might be the potential provenances for the future hybridization. The estimated genetic diversity of the 14 populations exhibited that Ho ranged from 0.2171 to 0.2801 with a mean of 0.2476, He from 0.3424 to 0.3598 with a mean of 0.3534 and PIC from 0.2750 to 0.2878, the difference among populations was small, especially for the PIC all close to 0.25. Each population approached the lower limit of intermediate levels of genetic diversity. 【Conclusion】Comparison of sequence variations reveals a considerably low genetic diversity within and among the 14 farmed P. clarkii populations. The kinship between groups is close, with obvious signs of single breed and long-term crossing. As a low-cost and effective approach,the SLAF-seq sequencing should be suited to regularly monitor the genetic diversity of the farmed populations.
Key words: Procambarus clarkii; SLAF-seq; SNP; genetic diversity; population genetics
Foundation item: National Key Research and Development Program of “Blue Granary Science and Technology Innovation”(2018YFD0900101);Zhejiang One Million Project of Rice-fishery Integrated Planting and Raising(zjjhdy20200701);Guangxi Natural Science Foundation(2016GXNSFFA380002)
0 引言
【研究意义】克氏原螯虾(Procambarus clarkii)俗称小龙虾,属甲壳纲(Crustacea)十足目(Decapoda)鳌虾科(Cabaridae)(刘其根等,2008),原产于北美洲墨西哥。克氏原螯虾具有较高的营养价值及较强的环境适应能力,不仅对生长水质要求较低,甚至可以存活于高污染性的水体中。此外,其广谱的杂食性使之还能有效清除鱼类排泄物,对水体清洁有重要意义。近年来螯虾种群已迅速蔓延到东亚、欧洲和北非尼罗河流域(宋光同等,2018;Yi et al.,2018;徐滨等,2019)。克氏原螯虾约在20世纪30年代末由日本传入我国(张爱军和沈继红,2005),现已在我国广泛分布。鳌虾被作为水产资源物种以来,其养殖业发展迅速,养殖面积也越来越大,已成为一种重要的水产养殖物种(严维辉等,2019)。但近年来,克氏原螯虾的养殖面临个体变小、疫病多发、种质退化等问题,已对其养殖产业的可持续发展造成明显影响。因此,了解不同养殖群体的遗传多样性信息和亲缘关系,为种间交流、品种优化提供参考依据,对克氏原螯虾养殖业的健康、可持续发展具有重要指导意义。【前人研究进展】为明确克氏原螯虾养殖群体的遗传信息,国内外学者主要采用微卫星序列的测序和分析。Barbaresi等(2003,2007)对欧洲5个种群的遗传变异分析表明引种种群来自葡萄牙、法国和意大利等地区,通过分析西欧12个克氏原螯虾群体中5个微卫星位点的遗传变异,发现其传播可能涉及远距离跳跃扩散和微观地理范围内的自然扩散。王长忠等(2009)运用微卫星序列分析长江下游4个种群的遗传多样性,结果表明随着克氏原螯虾群体在长江流域的自然扩散和人为迁移,其遗传多样性出现不同程度的降低。曹玲亮等(2010)开展了安徽水系中克氏原螯虾种群的遗传学研究,发现该地区种群的遗传多样性水平和杂合度相对较高,水系间的交流是种群扩散的重要途径。彭刚等(2010)根据7对微卫星引物的扩增产物,比较了3个克氏原螯虾野生和养殖群体间的遗传多样性,得出长江流域野生群体之间具有较高遗传多樣性的结论。邢智珺等(2014)利用8个微卫星标记对江苏省8个地区克氏原螯虾的遗传多样性进行分析,发现群体存在中等程度的分化。黄小芳等(2020)利用8对微卫星引物对克氏原螯虾群体基因组DNA进行PCR检测,分析其遗传多样性,数据显示各群体间存在广泛的基因交流,仅存在低度至中度的遗传分化。Zhong等(2020)利用线粒体DNA和微卫星技术对广西5个克氏原螯虾群体的遗传多样性进行研究,结果表明遗传变异是由种群内因素决定的,广西南部的群体遗传多样性较低,应对其种质资源进行改良。从现有研究来看,普遍存在着不足之处,如调查取样的样品种群数量较少,样品地理分布代表性不足;微卫星方法研究所能获得的有效分子标记数量太少,结果的可靠性低;很少使用第二代高通量基因组测序技术和方法等。【本研究切入点】目前,国内研究极少涉及克氏原螯虾养殖群体的遗传多样性分析。此外,克氏原螯虾基因组具有高度复杂性,有94对染色体(张莎等,2018),基因组大小近3.5 Gb,参考基因组序列尚未发表。因此,采用简化基因组测序技术——特定区点扩增片段测序技术(Specific-locus amplified fragment sequencing,SLAF-seq)(Sun et al.,2013)对其开展遗传多样性分析十分必要。该技术通过在基因组酶切测序后获得的大量序列标签上获得的单核苷酸多态性(SNP)位点分析,能在较低测序成本下对群体遗传多样性实现无参分析(不依赖参考基因组的序列分析)。由于SNP的数量大,分辩率高,能适用于遗传差异较小的养殖群体的遗传多样性分析。【拟解决的关键问题】采用基因组测序的方法对分布于湖北、江西、安徽、浙江和江苏5省的14个克氏原螯虾养殖群体进行测序,获得比以往研究更丰富的基因组多态性序列标签,以分析养殖群体间的遗传多样性,为判断和指导克氏原螯虾养殖过程中是否需要进行及时引种或群体间杂交提供重要依据。
1 材料与方法
1. 1 样品收集和基因组DNA提取
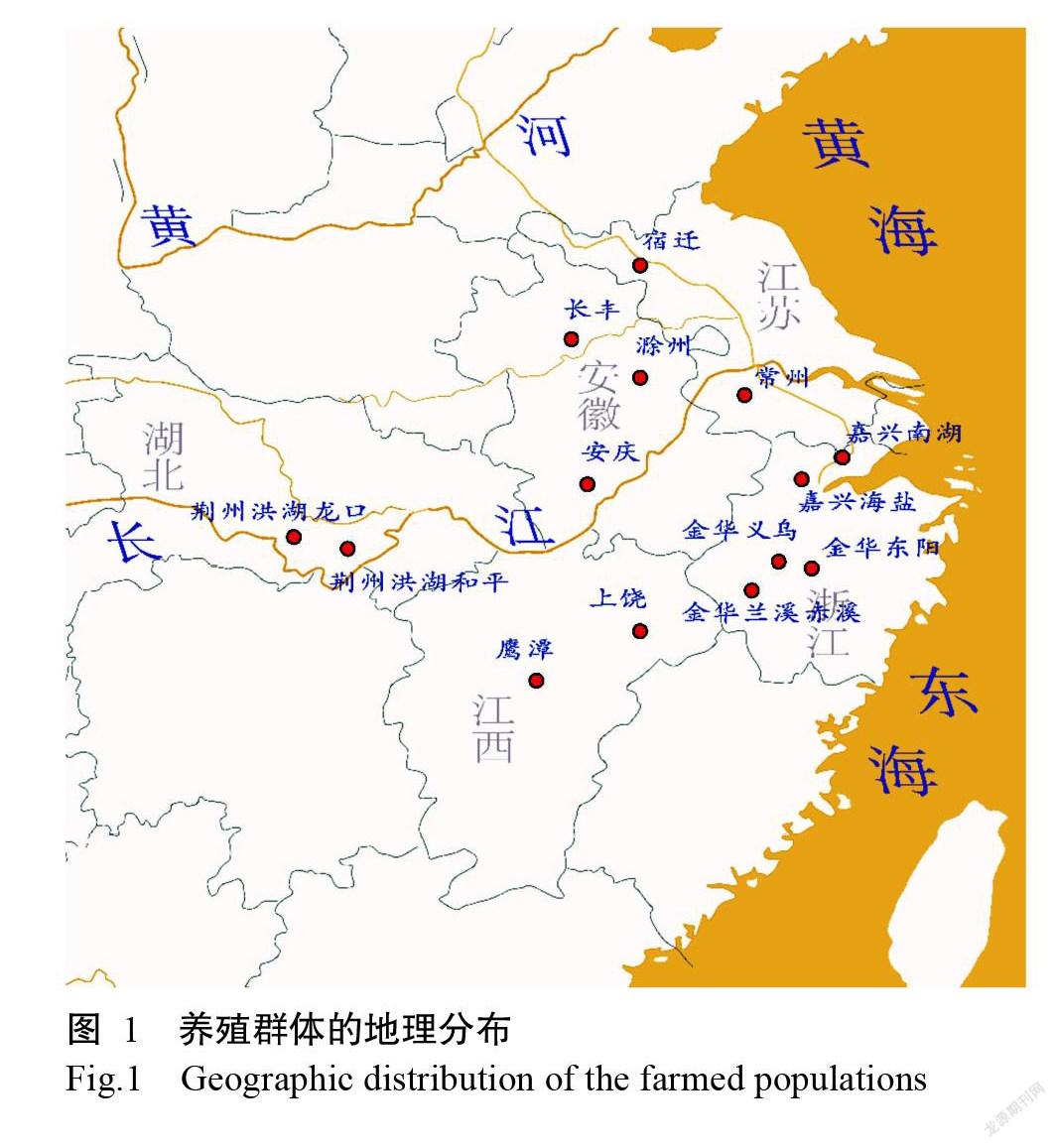
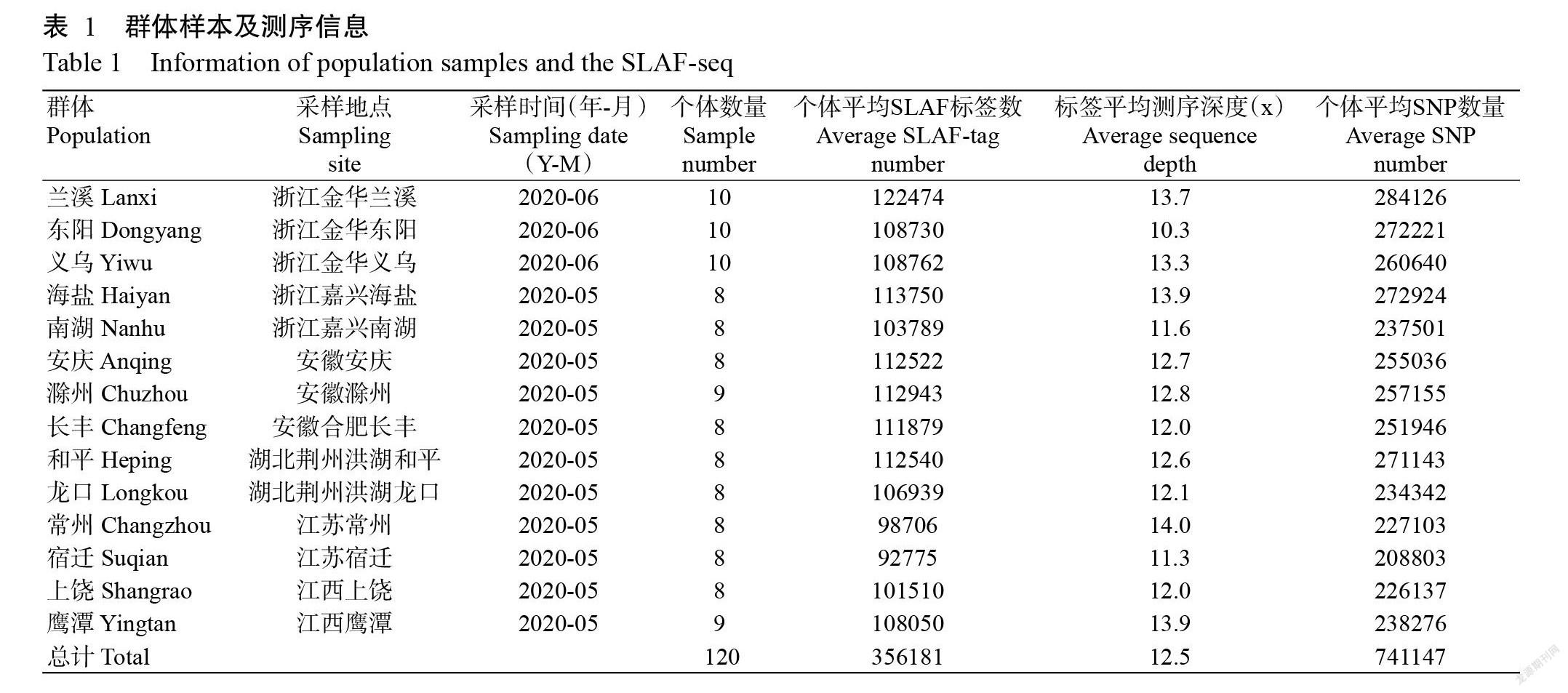
2020年5—6月采集来自浙江、安徽、湖北、江西和江苏5省的14个养殖群体(图1和表1),每个养殖群体选取8~10个个体的肌肉组织,共120份样品,利用液氮速冻后,采用CTAB法提取基因组DNA(闫苗苗等,2008),用Agilent 5400检测DNA浓度。
1. 2 简化基因组测序和标签SNP鉴定
预酶切实验后,选择限制性内切酶切HaeIII对基因组DNA进行酶切,将酶切得到的片段,即长度约400 bp的SLAF标签,按照测序的流程构建Paire-dend测序文库,使用HiSeq XTen测序平台进行测序,获得双向长度各为150 bp的原始序列。原始序列经过去除接头和质量评估后的干净序列(Clean reads),按序列相似性进行聚类,来源于同一个SLAF标签被聚类到一起。鉴定SNP时,每个SLAF标签中测序深度最高的序列类型作为参考序列,使用BWA(Li and Durbin,2009)将Clean reads比对到对应参考序列上,使用GATK(Mckenna et al.,2010)和Samtools(Li et al.,2009)工具,默认参数下,鉴定出SNP。
1. 3 群体遗传结构和亲缘关系分析
鉴定后的SNP,使用MEGA X(Kumar et al.,2018),基于邻接法(Neighbor-joining),采用Kimura 2-parameter模型(Davidson and Campo,2020),Bootstrap重复1000次,构建样品的系统发育进化树。基于SNP,使用EIGENSOFT(Price et al.,2006)进行主成分分析(PCA)。根据系统发育进化树分析结果,计算多态信息含量(PIC),并使用Stacks v1.42计算群体观测杂合度(Ho)和期望杂合度(He)等遗传多样性指数。使用ADMIXTURE(Alexander et al.,2009)分析群体结构,并进行聚类,最后根据最低交叉验证错误率(CV error)值来判断最优分群数。PIC按Botstein等(1980)的公式计算:
PIC=1-([i=1nPi2])-[i=1n-1j=i+1n2Pi2]Pj?
式中,Pi和Pj分别为第i和第j个等位基因在群体中的频率,n为等位基因数。
2 结果与分析
2. 1 群体系统进化分析
由表1可知,用于测序的14个养殖群体共120个个体涵盖了长江中游、下游和钱塘江等水系。对120个个体样品进行SLAF测序后,共得到191.2 M初始Reads。序列经过聚类后,获得超过35万个SLAF标签,每个标签的平均测序深度约为12.5 x。对群体中存在多态性的SLAF标签进行分析后,鉴定出741147个SNP位点,个体平均SNP数量在208803~284126,即使是拥有SNP数量最少的个体也携带16万以上的SNP。如此数量巨大的SNP为后续准确的群体遗传学分析打下了数据基础。
转换和颠换是SNP的主要类型,对这些SNP的转换与颠换信息进行详细统计,结果(表2)显示,在克氏原鳌虾各养殖群体中,转换与颠换的比例在1.4∶1左右,高于完全随机情形下的1∶2。同大多数后生动物相似,存在转换——颠换的偏差(Seplyarskiy et al.,2012)。通常,在密码子中发生的颠换更易导致氨基酸的改变,且转换比起颠换更容易,因此这在进化上是合理的。
利用这些SNP,构建120个克氏原螯虾个体的系统发育进化树(图2)。系统发育进化树显示样本在遗传上主要聚成2个簇(clade):一支由浙江金华的义乌和兰溪群体与浙江嘉兴的海盐和南湖2个群体构成;另一支由浙江金华东阳的群体和其余4省的其他群体构成。由个体在系统发育进化树上的分布可看出,浙江义乌、江西鹰潭、湖北洪湖和平及龙口、安徽滁州、安徽合肥长丰和江苏常州等养殖点的个体几乎均单独聚集在一起,说明这些养殖点品种较单一,几乎没有与其他养殖点间进行过杂交。而如浙江金华兰溪、浙江嘉兴海盐和南湖、安徽安庆、江西上饶等群体,个体的分布与其他养殖点的个体有较明显的混杂,表明该养殖点品种与其他养殖点品种间交流较多。系统进化分析揭示某些在地理分布上有较远距离的群体却显示出较近的亲缘关系,如浙江金华东阳与湖北荆州的2个群体,江苏常州与江西上饶、安徽安庆的3个群体间在进化关系上较接近。
根据所有样品的系统进化关系,可大致预测出克氏原螯虾养殖种群的迁徙过程(图3)。由图3可看出,取样点的克氏原螯虾最初可能来自两个祖先群体,在这些养殖点中,目前能回溯到与这两个祖先亲缘关系最近的养殖点,一个是浙江义乌或兰溪,另一个是江苏宿迁。然后再由这两个地方,分别向各地引种迁徙。第一条线路,从浙江义乌/兰溪到嘉兴海盐再到嘉兴南湖,但随后这几个地方的养殖群体不断交流。第二条线路较复杂,从江苏宿迁开始即分成两支:一支先到江西鹰潭,再到浙江东阳,最后到湖北荆州的龙口和和平;另一支先到安徽安庆,随后又分成三支,分别迁移至滁州、长丰至上饶,其中上饶的那支再到达常州。此外,浙江金华东阳的群体源自江苏宿迁,与金华的另两个群体不同。
2. 2 群体结构分析
基于群体的SNP位点,通过ADMIXTURE(Ale-xander et al.,2009)对克氏原螯虾群体的群体结构进行分析(图4)。从图4可看出,当K=2时,对应的CV error值最低,表明14個群体可能来源于两个祖先,该结果与系统进化分析结果吻合。另外,K=3时CV error值也较低,且与K=2时的CV error值很接近。而在系统进化分析中,浙江金华义乌和浙江金华兰溪的群体亲缘关系很近,很难区分哪个更接近祖先种,因此在一定程度上,较难区分所有群体是双起源的还是三起源的,与群体结构的分析结果相对应。由于金华义乌与兰溪的地理距离很近,可能本身就来自同一个群体。所以,更倾向于这些种群最初来自于两个种源群体。
2. 3 群体主成分分析
为进一步明确群体间亲缘关系,利用741147个SNP位点通过EIGENSOFT进行PCA分析(图5)。从三维聚类后的情况看出(第一、二、三主成分分别为PC1、PC2和PC3),安徽长丰和滁州群体亲缘关系较近,聚集成1个簇(Cluster);湖北荆州洪湖的2个群体及浙江东阳的群体,分别聚集成3个独立的簇。这4个簇都游离于由剩下的9个群体形成的主簇(Main cluster)之外。在主簇中,江苏常州和浙江金华兰溪群体的亲缘关系最远,与系统发育的结果相似。PCA分析结果表明:湖北的2个群体,安徽的长丰和滁州及浙江东阳与其他群体间存在相对较远的亲缘关系。
2. 4 群体遗传多样性分析
为评估群体的遗传多样性,通过计算Ho、He和PIC等参数来考察各个群体的遗传变异程度,是遗传资源保护的基础研究内容(Talebi et al.,2008),常用来表示群体遗传多样性情况(Pan et al.,2016)。从基于SNP位点计算得到14个群体的Ho、He和PIC数值统计结果(图6)来看,Ho介于0.2171~0.2801,平均值为0.2476,He介于0.3424~0.3598,平均值为0.3534。其中,浙江嘉兴南湖群体的He最高,但Ho最低,表明这个群体经过较长时间的内交,遗传多样性较低。14个群体的PIC均处于0.2750~0.2878,群体间相差不大,接近0.25,表明这14个养殖群体的遗传变异,虽然还居于中等水平(刘楚吾等,2010),但已非常接近中等水平的下限。从14个群体的He和PIC来看,浙江群体的遗传多样性大多较低,可能会面临更多的种质退化风险。根据PCA分析结果和遗传多样性分析结果,当浙江群体需要进行引种时,可选择遗传多样性相对较高及单独成簇的安徽长丰和滁州的群体。
3 讨论
与传统的微卫星等分子标记相比,通过鉴定SNP检测遗传多样性,不再以DNA长度差异作为检测手段,而是直接通过检测序列变化,是更加高效的遗传标记,在大多数基因组中,这是最丰富和稳定的遗传变异形式(Liu et al.,2012)。对于缺少参考基因组的物种,特别是基因组复杂的物种来说,简化基因组测序技术不仅节省费用,测序分析周期短,而且获得的大量多态性位点分析结果较接近深度全基因组测序结果(胡亚亚等,2018)。以往的克氏原螯虾群体遗传多样性研究中,多采用SSR标记和ISSR标记,利用的微卫星位点数很少,而且这些方法适用于个体差异较显著的野生群体,不适用于个体差异较小的养殖群体。克氏原螯虾作为一种重要的水产物种,人工养殖规模大,养殖群体的遗传多样性检测直接与生长实践相关联,定期监测养殖群体对水产养殖的可持续发展极为重要。
定期检测养殖群体的生物多样性,有助于及时采取措施,降低种质退化风险。与国内其他重要经济养殖品种如中华绒螯蟹、中国对虾等相比(刘萍等,2004;马海涛等,2007),克氏原螯虾的遗传多样性相对偏低。过去对克氏原螯虾的研究主要关注生态和引种种源方面,对克氏原鳌虾遗传多样性的检测方法研究较少。定期检测养殖群体中遗传多样性水平,并适时引种是解决克氏原螯虾产业面临的种质退化问题的有效措施。本研究以湖北、江西、安徽、浙江和江苏5省的克氏原螯虾养殖群体为研究对象,通过SLAF测序,利用获得的741147个SNP位点进行遗传多样性分析,结果发现14个群体的PIC在0.2750~0.2878,与其他利用微卫星标记的研究结果相比相对较低。相比于以往利用微卫星标记评估的报告结果(谭云飞等,2020),本研究计算所得的PIC值较低,推测养殖群体的遗传多样性已明显低于野生群体,与此前报道关于克氏原螯虾养殖群体的数据出入较大的原因可能是所选养殖场封闭式养殖时间较长,与其他养殖点间的交流过少,近亲繁殖严重。当然,杂交亲本的选择还需要更多更细致的遗传差异数据进行分析。
此外,群体的聚类分析结果表明,其系统发育关系并没有按照长江中游、中下游和钱塘江等水系的地理位置划分,说明养殖群体的迁移可能是人为引种或养殖的结果。与预测的群体迁移结果相似的是,从PCA分析中发现的游离在外的5个群体,均位于迁移过程的末梢。国内这些养殖点的种源相对较单一,不同养殖群体间的交流较少,许多养殖点可能已经或将要面临因过度近亲繁殖而带来的负面影响。如成年体型越来越小,生长缓慢,抗病力降低、病害频发、种质退化(舒新亚,2010)。近年来广泛发生的“五月瘟”(魏文燕等,2020),也是种群遗传多样性降低的结果。纤毛虫病(钟亮等,2021)也是养殖过程中的常见病害之一,患病后,虫体寄生在龙虾外壳上,影响正常蜕壳生长,进而失去商业价值。因此,及时引种、提高养殖群体的遗传多样性,是目前解决这些问题的最便捷方式。
基于生物大数据分析,对不同地区克氏原螯虾养殖群体的差异、群体内个体的差异进行及时调查,适时引种及加强不同地区群体间的交流,才能加强克氏原螯虾优良种质资源的保护,且定期监测还能为引种提供重要参考依据。本研究中首次发现浙江金华东阳的群体与另外2个金华的养殖群体有较大的遗传差异,种质来源不同,将来可作为一个就近引种的选择。同时,也发现浙江群体的遗传多样性低于其他省的养殖群体,可能与其内交及与其他地区交流过少有关。为保证各地区克氏原螯虾的遗传多样性,建议可适当从遗传多样性较高的安徽和湖北等地引种进行杂交,选育出多样性更加丰富的群体进行繁殖,加强不同地区、不同水系间群体的交流。通过大数据监测,一方面有利于保护好现有的克氏原螯虾物种基因库;另一方面有利于充分利用其遗传多样性,挖掘育种潜力,用科学方法及时准确掌握克氏原螯虾的种质状况,以促进其养殖业的健康稳定发展。
4 结论
14个克氏原螯虾养殖群体内的遗传多样性较低,群体间的亲缘关系较接近,品种单一、长期内交迹象明显,面临较高的种质退化风险。通过群体的基因组重测序获得的多态性信息远多于传统的微卫星方法,能以较低的成本实现对养殖群体遗传多样性的全面定期监测。
参考文献:
曹玲亮,周立志,张保卫. 2010. 安徽三大水系入侵物種克氏原螯虾的种群遗传格局[J]. 生物多样性,18(4):398-407. [Cao L L,Zhou L Z,Zhang B W. 2010. Genetic patterns of an invasive Procambarus clarkii population in the three river basins of Anhui Province[J]. Biodiversity Science,18(4):398-407.] doi:10.3724/SP.J.1003.2010.398.
胡亚亚,刘兰服,冀红柳,韩美坤,焦伟静,高志远,马志民. 2018. 简化基因组测序技术研究进展[J]. 江苏师范大学学报(自然科学版),36(4):63-68. [ Hu Y Y,Liu L F,Ji H L,Han M K,Jiao W J,Gao Z Y,Ma Z M. 2018. Research progress on the reduced-representation genome sequencing technique[J]. Journal of Jiangsu Normal University(Natural Science Edition),36(4):63-68.] doi:10.3969/ j.issn.2095-4298.2018.04.012.
黄小芳,唐章生,刘俊丹,张宏燕,钟一治,卢智发,侯树鉴,王大鹏,陆专灵. 2020. 广西不同地区克氏原螯虾群体遗传多样性微卫星分析[J]. 南方农业学报,51(2):437-444.[Huang X F,Tang Z S,Liu J D,Zhang H Y,Zhong Y Z,Lu Z F,Hou S J,Wang D P,Lu Z L. 2020. Genetic diversity microsatellite analysis of Procambarus clarkii populations in different regions of Guangxi[J]. Journal of Sou-thern Agriculture,51(2):437-444.] doi:10.3969/j.issn.2095- 1191.2020.02.025.
刘楚吾,黎锦明,刘丽,郭昱嵩. 2010. 中国龙虾微卫星标记的筛选及遗传多样性分析[J]. 遗传,32(7):737-743. [Liu C W,Li J M,Liu L,Guo Y S. 2010. Screening and gene-tic diversity analysis of microsatellite markers in Chinese lobster(Panulirus stimpsoni)[J]. Hereditas,32(7):737-743.] doi:10.3724/SP.J.1005.2010.00737.
刘萍,孟宪红,何玉英,孔杰,李健,王清印. 2004. 中国对虾(Fenneropenaeus chinensis)黄、渤海3个野生地理群遗传多样性的微卫星DNA分析[J]. 海洋与湖沼,35(3):252-257. [Liu P,Meng X H,He Y Y,Kong J,Li J,Wang Q Y. 2004. Genetic diversity in three wild populations of Fenneropenaeus chinensis in Yellow and Bohai Seas as revealed by microsatellite DNA[J]. Oceanologia et Limnologia Sinica,35(3):252-257.] doi:10.3321/j.issn:0029-814X.2004.03.009.
刘其根,李应森,陈蓝荪. 2008. 克氏原螯虾的生物学[J]. 水產科技情报,35(1):21-23. [Liu Q G,Li Y S,Chen L S. 2008. Ecological culture of red swamp crawfish Procambarus clarkii(I)[J]. Fisheries Science & Technology Information,35(1):21-23.] doi:10.3969/j.issn.1001-1994. 2008.01.011.
马海涛,常玉梅,于冬梅,孙效文. 2007. 利用微卫星分子标记分析四个中华绒螯蟹群体的遗传多样性(英文)[J]. 动物学研究, 28(2):126-133. [Ma H T,Chang Y M,Yu D M,Sun X W. 2007. Microsatellite variations among four populations of Eriocheir sinensis[J]. Zoological Research,28(2):126-133.] doi:10.3321/j.issn:0254-5853.2007.02. 003.
彭刚,刘伟杰,李佳佳,严维辉,唐建清. 2010. 长江流域3个克氏原螯虾野生群体遗传结构的微卫星分析[J]. 江苏农业学报,26(5):1115-1117. [Peng G,Liu W J,Li J J,Yan W H,Tang J Q. 2010. Microsatellite DNA analysis of genetic structure of three wild populations of Procambarus clarkii in the Yangtze River basin[J]. Jiangsu Journal of Agricultural Sciences,26(5):1115-1117.] doi:10.3969/j.issn.1000-4440.2010.05.040.
舒新亚. 2010. 克氏原螯虾产业发展及存在的问题[J]. 中国水产,(8):22-25. [Shu X Y. 2010. The development and existing problems of clarkiis original crayfish industry[J]. China Fisheries,(8):22-25.] doi:10.3969/j.issn.1002- 6681.2010.08.011.
宋光同,何吉祥,吴本丽,陈静,黄龙,汪翔,武松. 2018. 克氏原螯虾雌性生殖系统发育及组织结构观察[J]. 江西农业学报,30(2):68-75. [Song G T,He J X,Wu B L,Chen J,Huang L,Wang X,Wu S. 2018. Study on development of female reproductive system and histological structure of Procambarus clarkii[J]. Acta Agriculturae Jiangxi,30(2):68-75.] doi:10.19386/j.cnki.jxnyxb.2018.02.16.
谭云飞,蓬国辉,熊礼静,彭波,吴毅博,宋朝伟,白旭峰. 2020. 长江中下游流域13个克氏原螯虾群体遗传多样性和遗传结构分析[J]. 华中农业大学学报,39(2):33-39. [Tan Y F,Peng G H,Xiong L J,Peng B,Wu Y B,Song C W,Bai X F. 2020. Genetic diversity and structure ana-lysis of 13 red swamp crayfish(Procambarus clarkii)popu-lations in Yangtze River basin[J]. Journal of Huazhong Agricultural University,39(2):33-39.] doi:10.13300/j.cnki.hnlkxb.2020.02.005.
王長忠,李忠,梁宏伟,呼光富,吴勤超,邹桂伟,罗相忠. 2009. 长江下游地区4个克氏原螯虾群体的遗传多样性分析[J]. 生物多样性,17(5):518-523.[Wang C Z,Li Z,Liang H W,Hu G F,Wu Q C,Zou G W,Luo X Z. 2009. Genetic diversity in four Procambarus clarkii populations in the lower reaches of the Yangtze River[J]. Biodiversity Science,17(5):518-523.] doi:10.3724/SP.J.1003. 2009.09017.
魏文燕,杨马,刘家星,李良玉,吴艳蓉. 2020. 小龙虾“五月瘟”成因及防控措施[J]. 科学养鱼,(6):50-51. [Wei W Y,Yang M,Liu J X,Li L Y,Wu Y R. 2020. Causes and control measures of crayfish “May plague”[J]. Scientific Fish Farming,(6):50-51.] doi:10.3969/j. issn1004-843X. 2020.06.026.
邢智珺,姜虎成,陆伟,钱照君,于宏伟,李家乐. 2014. 江苏8个克氏原螯虾群体遗传多样性微卫星分析[J]. 上海海洋大学学报, 23(5):656-662. [Xing Z J,Jiang H C,Lu W,Qian Z J,Yu H W,Li J L. 2014. Genetic diversity analysis of eight procambarus clarkii stocks in Jiangsu Province based on microsatellites[J]. Journal of Shanghai Ocean University,23(5):656-662.]
徐滨,李忠,魏开金,马宝珊,朱祥云,徐进. 2019. 6个克氏原螯虾群体形态学分析[J]. 淡水渔业,49(6):27-32. [Xu B,Li Z,Wei K J,Ma B S,Zhu X Y,Xu J. 2019. Morphological variations among six populations of Procambarus clarkia[J]. Freshwater Fisheries,49(6):27-32.] doi:10.13721/j.cnki.dsyy.2019.06.005.
闫苗苗,魏光成,潘效红,马怀雷,李伟振. 2008. 一种适用于动物与植物总DNA提取的方法——改良CTAB法(英文)[J]. Agricultural Science & Technology,9(2):39-41. [Yan M M,Wei G C,Pan X H,Ma H L,Li W Z. 2008. A method suitable for extracting genomic DNA from animal and plant—Modified CTAB method[J]. Agricultural Science & Technology,9(2):39-41.] doi:10.16175/j.cnki. 1009-4229.2008.02.001.
严维辉,唐建清,许志强,李佳佳. 2019. 克氏原螯虾大棚育苗试验总结[J]. 水产养殖,40(12):8-9. [Yan W H,Tang J Q,Xu Z Q,Li J J. 2019. Summary of greenhouse seedling experiment of Procambarus clarkii[J]. Journal of Aquaculture,40(12):8-9.] doi:10.3969/j.issn.1004-2091. 2019.12.005.
张爱军,沈继红. 2005. 龙虾的综合加工利用[J]. 中国资源综合利用,(9):35-36. [Zhang A J,Shen J H. 2005. The comprehensive machining utilization of Chinese lobster[J]. China Resources Comprehensive Utilization,(9):35-36.] doi:10.3969/j.issn.1008-9500.2005.09.011.
张莎,俞树惠,邱高峰. 2018. 克氏原螯虾染色体及其核型[J]. 水产学报,42(10):1513-1519. [Zhang S,Yu S H,Qiu G F. 2018. Chromosome and karyotype of the crayfish(Procambarus clarkii)[J]. Journal of Fisheries of China,42(10):1513-1519.] doi:10.11964/jfc.20171211084.
钟亮,黄小丽,徐铭,李良玉. 2021. 小龙虾纤毛虫病的诊断与防治[J]. 科学养鱼,(1):50-51. [Zhong L,Huang X L,Xu M,Li L Y. 2021. Diagnosis and control of ciliosis in crayfish[J]. Scientific Fish Farming,(1):50-51.] doi:10.3969/j.issn1004-843X.2021.01.027.
Alexander D H,Novembre J,Lange K. 2009. Fast model-based estimation of ancestry in unrelated individuals[J]. Genome Research,19(9):1655-1664. doi:10.1101/gr.094 052.109.
Barbaresi S,Fani R,Gherardi F,Mengoni A,Souty-Grosset C. 2003. Genetic variability in European populations of an invasive American crayfish:Preliminary results[J]. Biolo-gical Invasions,5(3):269-274. doi:10.1023/A:10261335 19707.
Barbaresi S,Gherardi F,Mengoni A,Souty-Grosset C. 2007. Genetics and invasion biology in fresh waters:A pilot study of Procambarus clarkii in Europe[M]//Gherardi F. Biological invaders in inland waters:Profiles, distribution, and threats. Dordrecht:Springer:381-400.doi:10.1007/978-1-4020-6029-8_20.
Botstein D,White R L,Skolnick M,Davis R W. 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics,3(3):314-331.
Davidson R,Campo A M D. 2020. Combinatorial and computational investigations of neighbor-joining bias[J]. Frontiers in Genetics,(11):584785. doi:10.3389/fgene.2020. 584785.
Kumar S,Stecher G,Li M,Knyaz C,Tamura K. 2018. MEGA X:Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution,35(6):1547-1549. doi:10.1093/molbev/msy096.
Li H,Durbin R. 2009. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics,25(14):1754-1760. doi:10.1093/bioinformatics/btp324.
Li H,Handsaker B,Wysoker A,Fennell T,Ruan J,Homer N,Marth G,Abecasis G,Durbin R. 2009. The sequence alignment/map format and SAMtools[J]. Bioinformatics,25(16):2078-2079. doi:10.1093/bioinformatics/btp352.
Liu J,Huang S M,Sun M Y,Liu S Y,Liu Y M,Wang W X,Zhang X R,Wang H Z,Hua W. 2012. An improved allele-specific PCR primer design method for SNP marker analysis and its application[J]. Plant Methods,8(1):34. doi:10.1186/1746-4811-8-34.
Mckenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A,Garimella K,Altshuler D,Gabriel S,Daly M,DePristo M A. 2010. The Genome Analysis Toolkit:A MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research,20:1297-1303. doi:10.1101/gr.107524.110.
Pan Y Z,Wang X Q,Sun G L,Li F S,Gong X. 2016. Application of RAD sequencing for evaluating the genetic diversity of domesticated Panax notoginseng(Araliaceae)[J]. PLoS One,11(11):e0166419. doi:10.1371/journal.pone.0166419.
Price A L,Patterson N J,Plenge R M,Weinblatt M E,Shadick N A,Reich D. 2006. Principal components analysis corrects for stratification in genome-wide association studies[J]. Nature Genetics,38(8):904-909. doi:10.1038/ ng1847.
Seplyarskiy V B,Kharchenko P,Kondrashov A S,Bazykin G A. 2012. Heterogeneity of the transition/transversion ratio in Drosophila and hominidae genomes[J]. Molecular Biology and Evolution,29(8):1943-1955. doi:10.1093/molbev/mss071.
Sun X W,Liu D Y,Zhang X F,Li W B,Liu H,Hong W G,Jiang C B,Guan N,Ma C X,Zeng H P,Xu C H,Song J,Huang L,Wang C M,Shi J J,Wang R,Zheng X H,Lu C Y,Wang X W,Zheng X K. 2013. SLAF-seq:An efficient method of large-scale De Novo SNP discovery and genotyping using high-throughput sequencing[J]. PLoS One,8(3):e58700. doi:10.1371/journal.pone.0058700.
Talebi R,Naji A M,Fayaz F. 2008. Geographical patterns of genetic diversity in cultivated chickpea(Cicer arietinum L.)characterized by amplified fragment length polymorphism[J]. Plant,Soil and Environment,54(10):447-452. doi:10.17221/399-PSE.
Yi S K,Li Y H,Shi L L,Zhang L,Li Q B,Chen J. 2018. Characterization of population genetic structure of red swamp crayfish,Procambarus clarkii,in China[J]. Scientific Reports,8(1):5586. doi:10.1038/s41598-018-23986-z.
Zhong Y Z,Tang Z S,Huang L M,Wang D P,Lu Z L. 2020. Genetic diversity of Procambarus clarkii populations based on mitochondrial DNA and microsatellite markers in different areas of Guangxi,China[J]. Mitochondrial DNA Part A,31(2):48-56. doi:10.1080/24701394.2020. 1721484.
(責任编辑 王 晖)
