基于2b-RAD简化基因组测序的甜瓜遗传多样性分析
2021-03-10曹燕燕刁倩楠陈幼源张永平
曹燕燕,刁倩楠,陈幼源,张永平
(上海市农业科学院 园艺研究所,上海市设施园艺技术重点实验室,上海 201403)
甜瓜(CucumismeloL.)为葫芦科甜瓜属一年生蔓性草本植物,是国内外重要的园艺和经济作物。甜瓜果实不仅味美香甜,多汁爽口,而且具有很高的营养和药用价值,因此深受广大消费者的喜爱,特别是在夏季水果市场中占有十分重要的地位。为了满足市民多元化高质量的消费需求,迫切需要育种工作者培育出更多新的高品质甜瓜品种。甜瓜种质资源是进行新品种选育以及开展科研工作的重要基础,因此加强甜瓜特异种质资源的开发和利用对促进甜瓜产业的发展和科研水平的提升具有重要意义[1]。
作为葫芦科植物中表型变异最为丰富的作物之一,甜瓜在株型、叶片和果实等形态特征方面具有广泛的多样性,尤其是果皮颜色、果面覆纹、果肉颜色、果肉硬度、可溶性固形物含量以及风味等的果实表型的变异最为丰富[2-4]。目前,关于甜瓜遗传多样性的研究国内外已有报道。例如,Escribano S.等[5]研究了不同皮色希腊甜瓜的遗传多样性;王炜勇等[6]利用表型性状研究了浙江省沿海地区27份薄皮甜瓜地方品种的遗传多样性;胡建斌等[7]对中国各地257份甜瓜地方种质资源果实与种子的形态性状进行了遗传多样性分析;王吉明等[8]基于果实性状对从国外引进的100份野生甜瓜种质资源的遗传多样性进行了研究;闫洪朗等[9]对江浙沪地区的65个甜瓜品种果实性状进行了遗传多样性分析。以上这些报道都是基于表型性状进行甜瓜遗传多样性研究。
分子标记技术是评价种质资源遗传多样性的重要手段。目前已有利用分子标记研究甜瓜遗传多样性的报道,所用标记主要涉及AFLP、SRAP、RAPD、SSR以及ISSR等[10-14]。SNP标记作为目前遗传学研究中最为流行的分子标记,其与传统分子标记相比具有在基因组中分布广泛、密度高、遗传稳定以及易于分析等优势。2b-RAD是一种基于IIB型限制性核酸内切酶的简化基因组测序技术,也是近年来在非模式生物中进行SNP开发最经济有效的测序方法之一,具有酶切DNA片段大小一致、文库构建简单快速、标签密度易于调节、重复性和准确性高以及成本低廉等优点[15]。该技术已在果蝇、扇贝、线虫、对虾、拟南芥、水稻、玉米和茶树等物种的基因型分型、高精度连锁图谱构建、群体遗传结构和遗传多样性分析、QTL(quantitative trait locus)定位以及系统演化分析等研究中得到广泛应用[16-22]。迄今尚未有利用2b-RAD技术对甜瓜种质开展遗传多样性研究的相关报道。
厚皮甜瓜是区别于薄皮甜瓜的一个类型,包括厚薄皮杂交类型甜瓜、网纹厚皮甜瓜和哈密瓜品种,具有耐低温弱光、耐湿以及抗病的特点[13]。因其经济效益比较可观,在上海具有较广的种植面积。本研究以从国内外搜集和自主选育的在果实性状方面(包括果皮颜色、果面网纹和果肉颜色)差异比较大的28份高代稳定自交系厚皮甜瓜为材料,利用2b-RAD技术对其在分子水平进行遗传多样性及亲缘关系分析,以期为今后甜瓜SNP分子标记的开发和分子标记辅助选择育种提供科学依据。
1 材料和方法
1.1 供试材料
28份供试甜瓜种质材料均由上海市农业科学院园艺研究所提供,其编号信息和果实主要性状详见表1。

表1 28份甜瓜种质及其果实主要性状
1.2 方 法
1.2.1 基因组DNA提取和2b-RAD文库构建采集苗期甜瓜叶片,10株混合取样。液氮速冻后,置于-80 ℃冰箱保存备用。基因组DNA采用CTAB法进行提取[23],通过琼脂糖检测和紫外可见分光光度计对抽提样本基因组DNA的质量和浓度进行检测。样本DNA抽提质检合格后,利用2b-RAD五标签串联技术进行测序文库构建,采用标准型5′-NNN-3′接头与BsaXI酶切标签连接。文库质控合格后在Illumina nova平台进行Paired-end测序。2b-RAD测序由上海欧易生物医学科技有限公司完成。
1.2.2 测序质量分析利用Illumina nova测序平台得到原始测序序列后,使用Pear软件(Version 0.9.6)对其进行拼接。为保证后续数据分析结果的准确性,将拼接好的序列按照以下条件进行过滤:过滤删除含有N碱基比例大于8%的序列;过滤删除低质量序列 (质量值低于Q30的碱基数超过整条序列碱基数15%)。依据建库时各个样本所在的位置,提取出各样本对应的序列,过滤删除不含酶切识别位点的序列后得到各样本的高质量测序序列,即含酶切位点的序列。
1.2.3 全基因组范围SNP筛查和分型分析对于有参照基因组的标记分型,依据 RAD-typing的分型策略[24],标记分型如下:(1)从甜瓜参考基因组序列(ftp://cucurbitgenomics.org/pub/cucurbit/ genome/ melon/v3.6.1/CM3.6.1_pseudomol.fa.gz)中提取包含BsaX I 酶切位点的标签作为参考序列;(2)利用SOAP软件[25]将各样本的高质量序列比对到参考序列上(参数为-r 0-M 4 -v 2);(3)利用最大似然法(maximum likelihood, ML)进行SNP标记分型。为保证后续分析的准确性,分型完成后通过以下指标对分型结果进一步过滤:剔除只含有1种等位基因的位点;剔除基因组碱基为N的位点;剔除一个标签内多于2个SNP的标签;剔除同一位置2种分型的位点;剔除所有样本中低于80%个体可以分型的位点;剔除最小等位基因频率低于0.01的位点;剔除等位基因大于2的位点。
1.3 数据处理
分别利用SnpEff软件(V4.1g)、Genepop软件(V1.0.5)、Vcftools软件(V0.1.14)、Admixture软件(V1.3.0)和Treebest软件(Version: 1.9.2)进行SNP注释,遗传分化系数(Fst)统计分析,SNP位点的观测杂合度(Ho)、期望杂合度(He)、观测等位基因数(Na)、有效等位基因数(Ne)、多态信息含量(PIC)和核苷酸多样性(Pi)分析,群体结构分析以及进化树分析。
2 结果与分析
2.1 2b-RAD测序结果分析
利用2b-RAD测序技术构建28份甜瓜种质的标签测序文库(1份种质即为1个样本),结果(表2)显示,测序共获得391 918 500条原始序列,平均每个样本的测序序列数为13 997 089。参照Wang等[15]的方法对原始序列进行质量控制,在28个样本的原始序列中筛选出含有BsaX Ⅰ酶切位点的高质量序列比例均在82%以上(表2),说明利用此方法构建的甜瓜样本测序文库质量较好。

表2 2b-RAD测序数据基本分析
2.2 SNP分子标记在甜瓜中的分布
参照RADtyping分型策略[24],对比甜瓜参考基因组,根据比对结果统计样本获得的标签数和测序深度信息。结果显示,28个甜瓜样本中比对到参考基因组上的标签数在67 732~70 919之间,平均标签数为69 684,测序深度值从21.8~128.36不等,平均测序深度为49×(表3)。从28个样本的共同测序标签中共筛选出10 318个高质量的SNP位点,包括9 988个定位于染色体上的SNP位点和330个非染色体SNP位点。从SNP位点在每条染色体上的数量可以看出,6号染色体上存在的SNP位点最多,为1 233个,其次是1号染色体,为1 162个(图1,A)。这些SNP位点主要分布于基因间区以及基因的上下游序列(图1,B)。

表3 28个甜瓜样本的标签数和测序深度
2.3 SNP 突变类型分析
SNP分型结果中各位点的突变类型具体如下:G/A转换类型占所有碱基突变类型的33.94%,T/C转换类型占33.35%;T/A和T/G颠换类型分别占所有碱基突变类型的 9.17%和8.80%,G/C和C/A颠换类型分别占5.96%和 8.78%。SNPs中发生转换与发生颠换的比值(Ts/Tv)为 2.15。
2.4 28个甜瓜样本的SNP遗传多样性分析
本次分型只统计了二等位基因型,因此每个SNP位点观测等位基因数Na值均为2。SNP遗传多样性分析结果显示,每个二等位SNP位点观测杂合度Ho数值在0~1之间,平均值为0.068;期望杂合度He数值在0.035~0.500之间,平均值为0.291;有效等位基因数Ne数值在1.036~2.000之间,平均值为1.484;多态信息含量PIC数值在0.035~0.375之间,平均值为0.237;核苷酸多样性Pi数值在0.036~0.511之间,平均值为0.297;最小等位基因频率数值在0.018~0.500之间,平均值为0.214;位点的分型率数值在82.14%~100%之间,平均值为96.54%。

A. 各染色体SNPs的数量分布;B. SNPs在基因组中的分布图1 SNPs 在甜瓜基因组中的分布情况A. Distribution of the number of SNPs in each chromosome; B. Distribution of SNPs in genomeFig.1 Distribution of SNPs in melon genome
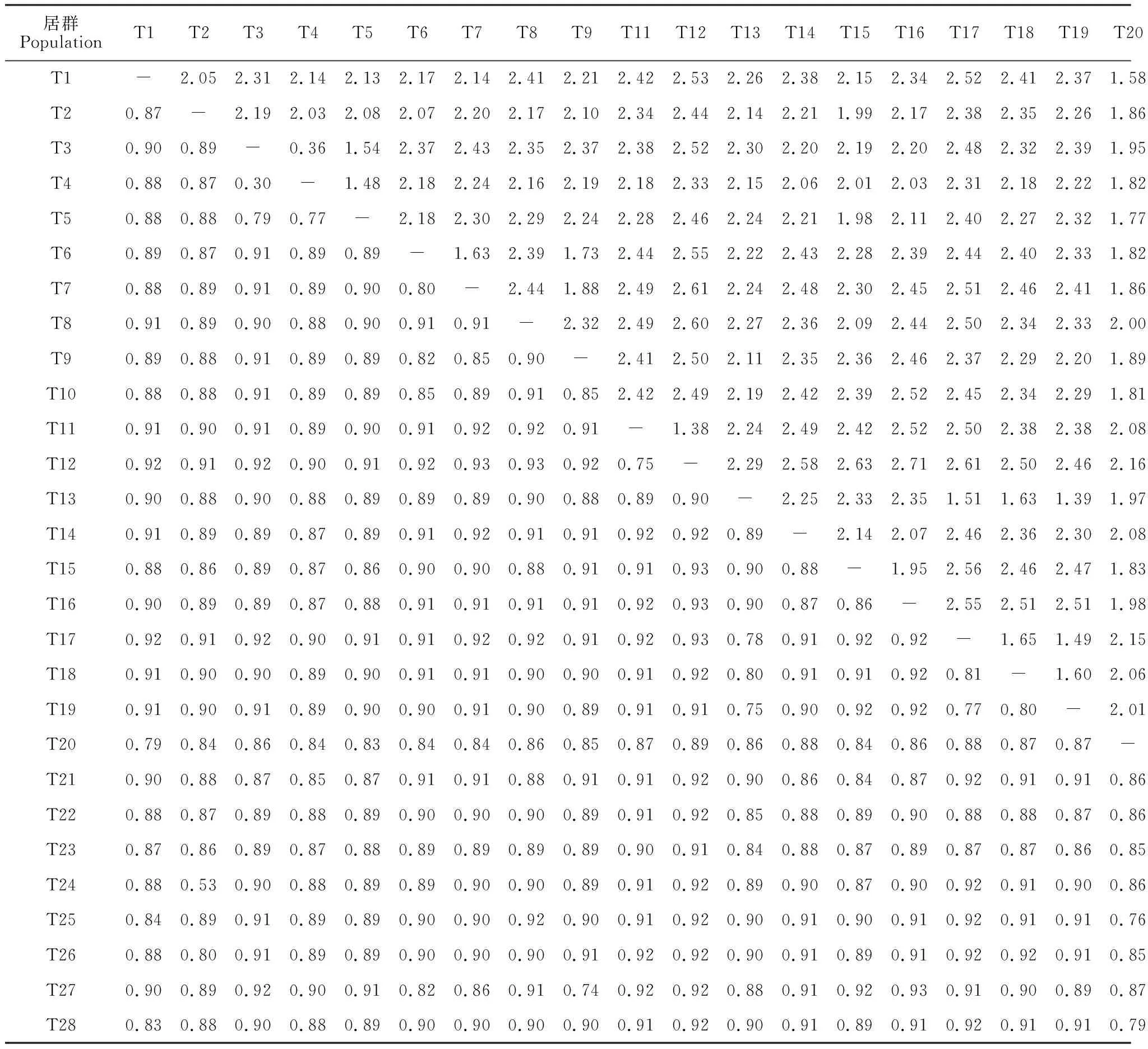
表4 28个甜瓜样本遗传分化系数(Fst, 下三角)和遗传距离(DR, 上三角)
2.5 甜瓜样本间遗传变异及分化分析
遗传分析结果(表4)显示,28个甜瓜样本两两之间的遗传分化系数(Fst)在 0.188~0.933之间,平均值为0.885,遗传距离(DR)范围在0.208~2.706之间,平均值为2.218,表明各样本之间均存在高度的遗传分化(一般认为,若群体Fst值在0~0.05之间,表明其各亚群间不存在分化;若Fst值在0.05~0.15之间,为中度分化;若Fst值在0.15~0.25之间,则为高度分化);其中,T22和T23两个样本之间的遗传分化系数和遗传距离最小,T12和T16两个样本之间的遗传分化系数和遗传距离最大(表4)。
依据果皮颜色分类,28份甜瓜种质可分为4个群体,分别为白皮群体(包括T1、T6、T14、T22、T23和T28)、黄皮群体(包括T3、T4、T5、T11、T12、T16和T20)、青皮群体(包括T2、T7、T8、T9、T15、T21、T24和T26)和绿皮群体(包括T10、T13、T17、T18、T19、T25和T27)。遗传分析结果(表5)显示,这4个群体两两之间的Fst值从0.052~0.188不等,群体间遗传距离DR值从0.054~0.208不等,其中白皮群体和青皮群体间的Fst值最小,黄皮群体和绿皮群体间的Fst值最大(表5)。各群体之间的Fst值均大于0.05,表明其均存在中度及以上程度的遗传分化。
依据果面网纹性状分类,28份甜瓜种质可分为3个群体,分别为光皮群体(包括T12、T14、T16、T21、T22和T23)、稀网群体(包括T1、T3、T4、T8、T11、T15和T26)和密网群体(包括T2、T5、T6、T7、T9、T10、T13、T17、T18、T19、T20、T24、T25、T27和T28)。遗传分析结果(表6)显示,这3个群体两两之间的Fst值分别为0.004、0.096和0.029,DR值分别为0.004、0.101和0.029,表明光皮群体和密网群体之间存在中度遗传分化,光皮群体和稀网群体之间以及稀网群体和密网群体之间无显著分化。
依据果肉颜色分类,28份甜瓜种质可分为3个群体,分别为白肉群体(包括T4、T14、T16、T22和T23)、桔肉群体(包括T1、T2、T5、T6、T7、T9、T10、T11、T12、T17、T18、T20、T27和T28)以及绿肉群体(包括T3、T8、T13、T15、T19、T21、T24、T25和T26)。遗传分析结果显示,这3个群体两两之间的Fst值分别为0.126、0.033和0.027,DR值分别为0.134、0.033和0.027,表明白肉群体和桔肉群体之间存在中度遗传分化,白肉群体和绿肉群体之间以及桔肉群体和绿肉群体之间无明显分化(表7)。
综合以上结果表明,28份甜瓜种质依据果皮颜色分类的各群体之间的遗传分化系数总体上是最大的,即遗传分化程度最高,并且各群体之间均存在中度及以上程度的遗传分化。
2.6 群体遗传结构分析
利用10 318个SNPs标记28份甜瓜种质,根据其果实性状,再结合K值最大似然值分类,将这些种质划分为3个类群(图2)。3个类群分别用红色、绿色和蓝色代表,即类群1、类群2和类群3。由图3可以看出,大部分条带(每个条带代表1份种质)都由2~3种颜色组成,表明3个类群大部分种质之间存在不同程度的基因交流。种质所属类群,可根据颜色占条带的比例推断。

表5 基于果皮颜色分类的28个甜瓜样本群体间的遗传分化系数(Fst)和遗传距离(DR)

表6 基于果面网纹性状分类的28个甜瓜样本群体间的遗传分化系数(Fst)和遗传距离(DR)

红色代表类群1;绿色代表类群2;蓝色代表类群3图2 28份甜瓜种质SNPs分析群体结构(K=3)Red color blocks represent group 1; Green color blocks represent group 2; Blue color blocks represent group 3Fig.2 Analysis of population structure using identified SNPs for 28 melon germplasms (K=3)

表7 基于果肉颜色分类的28个甜瓜样本群体间的遗传分化系数(Fst)和遗传距离(DR)
2.7 系统进化树构建及聚类分析
从聚类树形图(图3)可以看出,28份甜瓜种质被分为 3类,第一类包括T2、T3、T4、T5、T8、T14、T15、T16、T21、T24和T26共11份种质,第二类包括T1、T6、T7、T9、T10、T20、T25、T27和T28共9份种质,第三类包括T11、T12、T13、T17、T18、T19、T22和T23共8份种质。其中,第一类主要为自主选育出的高代自交系甜瓜种质,第二类主要为从新疆引进或从新疆品种中选育出来的高代自交系种质,第三类主要为从日本引进或从日本品种中选育出来的高代自交系种质。
3 讨 论
丰富多样的种质资源是选育高产、优质和高抗新品种以及研究物种起源、进化和分类的基础。甜瓜作为葫芦科植物中表型变异最为丰富的作物之一,育种工作者利用不同方法针对不同方向对其进行了广泛研究[6-9, 26-30]。胡建斌等[7]研究发现甜瓜果实性状的变异是甜瓜表型变异的主要来源。虽然已有研究人员利用不同的甜瓜种质资源对甜瓜果实表型性状进行了遗传多样性分析[8-9, 31-32],但目前仍未见利用2b-RAD技术对其在分子水平上进行研究的相关报道。

居群编号同表1图3 28份甜瓜种质基于SNP标记的聚类Population numbers are the same as those in Table 1Fig.3 Phylogenetic tree of 28 melon germplasms revealed by SNP markers
2b-RAD 技术利用IIB型限制性内切酶对基因组进行酶切后产生等长的33~36 bp的酶切标签,经富集后用于下游的高通量测序反应,结合生物信息学分析在全基因组范围内对SNP位点进行高通量筛选和基因分型分析,获得的批量的 SNP 位点可为群体遗传多样性分析奠定良好的基础[24, 33]。研究表明,该技术生物学重复的准确性高达99% 以上[34]。本研究经2b-RAD测序后,每个样本得到的原始序列数从912 682~30 740 808不等,质量过滤后,平均每个样本中获得含有BsaXI 酶切位点的高质量序列数占测序原始序列数的82%以上,每个个体的测序深度值在21.8~128.36之间,平均测序深度为49×,达到了高准确度下的分型标准[35],表明本次构建的文库质量较高,测序结果准确可靠。
SNP碱基替换类型分为转换(Ts)与颠换(Tv)两类,转换发生在A与G或C与T之间,颠换发生在A 与C、A与T、G与C以及G与T之间。研究表明,不同物种间转换与颠换的比值存在很大差异。砂梨(Pyruspyrifolia)转录组测序中转换与颠换的比值为1.71[36];桑葚不同时期转录组测序中转换与颠换的比值约为1.6[37];澳洲坚果(Macadamiaintegrifolia)重测序结果显示SNPs转换和颠换的比率为0.77[38];中国明对虾(Fenneropenaeuschinensis)2b-RAD测序结果中转换与颠换的比值为1.402[19];马氏珠母贝(Pinctadafucata)血细胞转录组测序中转换与颠换的比值为0.5[39]。本研究2b-RAD测序结果显示SNPs转换与颠换的比值(Ts/Tv)为2.15。产生这种差异的原因可能与不同物种在进化过程中承受的选择压力有关[40]。
多态信息含量(PIC)和杂合度(Ho)是2个较好的衡量基因多态性的指标[19]。当PIC< 0.25时,表示该位点为低度多态位点;PIC介于0.25~0.50之间时,表示该位点为中度多态性位点;PIC> 0.50时,表示该位点为高度多态性位点[41]。本研究中28个甜瓜样本SNPs位点的PIC值在0.035~0.375之间,表明各SNP位点呈低度多态性或中度多态性。28个样本SNPs位点的Ho值从0~1不等,表明各位点的杂合度存在较大差异。
种群的遗传分化指数(Fst)是衡量群体间遗传分化程度的重要参数。本研究中依据果皮颜色、果面网纹和果肉颜色3个性状进行分类的各群体之间的Fst值分别从0.052~0.188、0.004~0.096以及0.027~0.126不等,表明依据果皮颜色分类的各群体之间遗传分化程度最高,群体间的遗传结构差异最为显著,而依据果面网纹形成情况进行分类的各群体之间整体遗传分化程度最低。
本研究以果皮颜色、果面网纹和果肉颜色3个性状差异比较大的28份甜瓜种质为材料进行遗传多样性分析研究,共鉴定到10 318个SNP位点。后续可从以上位点中筛选可重复、易分辩、强专一的 SNP位点集合,用于构建甜瓜种质指纹图谱、果实性状相关的QTL定位分析以及种子纯度鉴定研究,同时也可将其应用于甜瓜分子标记辅助选择育种工作中以加快新品种的选育进程。
目前,Fergany M.等[42]通过形态学和SSR标记研究认为甜瓜亲缘关系与其生态地理分布具有高度相关性;程振家等[10]通过AFLP标记研究认为厚皮甜瓜聚类结果除了与果面网纹性状有关外,与生态地理来源也具有一定的相关性;徐志红等[43]通过形态学分析认为厚皮甜瓜的聚类结果与成熟期有一定的相关性。本研究通过系统进化树分析将28份厚皮甜瓜种质划分为3类,其中第二类大部分为从新疆引进或从新疆品种中选育出来的纯合种质,第三类大部分为从日本引进或从日本品种中选育出来的纯合种质,说明本研究中甜瓜聚类结果与地理来源具有一定关系,这与程振家等[10]和Fergany M.等[42]的研究结果十分相似。
本研究中依据分子水平的聚类分析结果与育种者对育种材料的分类不完全一致。育种者主要是根据甜瓜果实的果皮颜色、果肉颜色和有无网纹等性状对其进行分类,而这只是一种表型分类。由于参试的28份材料有些是由各种类型的甜瓜杂交后选育出的纯合种质,这些种质可能含有多个品种的血缘。因此基于表型的分类不能真实地反映各材料间亲缘关系的远近。程振家等[10]的研究结果也表明基于分子水平的分类方法能够更加准确地揭示材料之间的内在遗传联系。本研究结果也提示我们,在今后的甜瓜育种工作中要综合材料的表型性状和分子水平的分析结果,选择不同地理来源、表型差异显著、亲缘关系远的材料进行杂交,这样才更容易培育出优质、高产和抗病的甜瓜新品种。
作者贡献:张永平、曹燕燕、刁倩楠和陈幼源进行试验设计;曹燕燕进行数据分析和论文写作;张永平进行最后的论文修改。全体作者都阅读并同意最终的文本。
