鼻咽癌STGC3基因转录调控元件分析与鉴定*
2020-12-31李素云吴冬梅刘慧晴袁靖哲尹菊梅贺修胜
李素云 吴冬梅 刘慧晴 唐 磊 袁靖哲 尹菊梅 贺修胜△
(南华大学衡阳医学院,1 应用解剖与生殖医学研究所,2 肿瘤研究所, 3 2018级临床医学专业,4 2017级临床医学专业,衡阳 421001)
鼻咽癌(nasopharyngeal carcinoma, NPC)是常见的头颈部恶性肿瘤之一,起源于鼻咽部黏膜上皮。STGC3 基因是本课题组前期通过ESTs(expressed sequence tags,ESTs),采用定位候选克隆方法,对鼻咽癌LOH 3p21 区域比对分析与鉴定,所得到的鼻咽癌抑瘤基因[1]。前期研究表明,STGC3 基因在正常鼻咽和鼻咽癌组织中的表达差异具有统计学意义,是鼻咽癌表达下调的基因,但其表达调控机制不清楚[2-7]。本研究运用生物信息学,预测STGC3 基因核心启动子区转录因子结合位点,采用电泳迁移率变动分析(EMSA)和染色质免疫共沉淀(ChIP)实验,体内外证实与STGC3 基因启动子特异性结合位点,从而鉴定出STGC3 基因表达调控的元件,以期为研究STGC3基因表达调控机制提供前期基础。
1 材料和方法
1.1 生物信息学分析软件
AliBaba2.1:http://www.gene-regμLation.com/pub/programs/alibaba2/index.html。TESS:http://www.cbil.upenn.edu/tess。TFSEARCH:http://mbs.cbrc.jp/research/db/TFSEARCH.html。
1.2 细胞
人鼻咽癌细胞系(CNE2)由本研究所保存,用10 %新生小牛血清的RPMI1640 培养基,置于恒温37℃、含5% CO2饱和湿度细胞培养箱内培养。
1.3 实验试剂
RPMI1640培养液、胎牛血清、青霉素、链霉素(双抗)购自Gibico公司;DMSO购自杭州四季青公司;高保真DNA聚合酶购自Fermentas公司;DNA Marker DL2000购自广州美津生物公司;胰酶购自HyClone公 司;BCA Protein Assay Kit、核蛋白抽提试剂盒、LightShift Chemiluminescent EMSA Kit、Halt Protease Inhibition Cocktail Kit购自Pierce公司;Sp1抗体、磁场、ChIP试剂盒购自Millipore公司;正电荷尼龙膜购自Amersham公司;RNA提取试剂盒、逆转录试剂盒购自Omega公司;2×Taq PCR Master Mix、EB购自Sigma公司;琼脂糖、无水乙醇、氯仿、甲醛分析纯购自国产分析纯公司。
1.4 引物设计
运用生物信息学,预测STGC3基因核心启动子区转录因子Sp1结合位点所在序列为-832 ~ -807 bp,采用Primer 5.0设计ChIP实验、PCR实验中转录因子Sp1(Sp1)所需的引物,由上海生工公司合成(表1)。
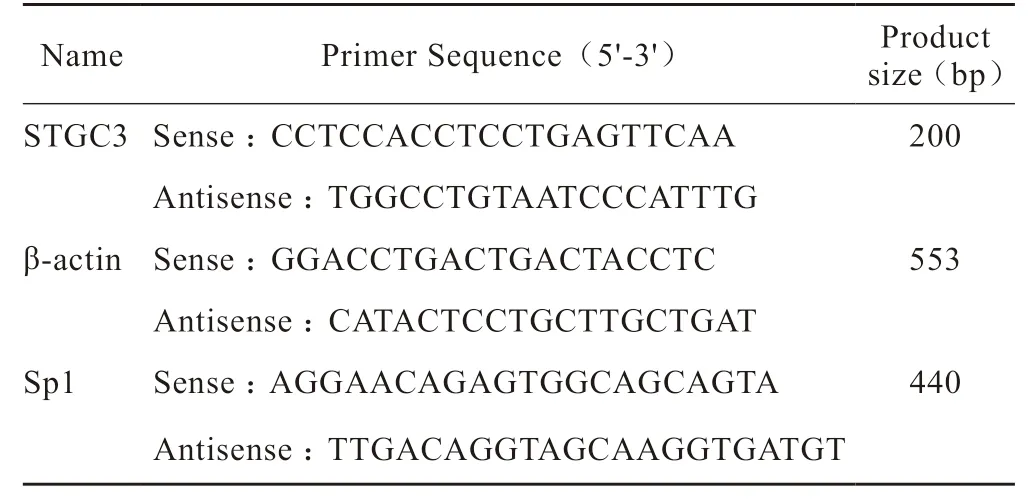
表1 PCR 引物序列与产物长度Tab 1 PCR primer sequence and product size
1.5 CNE2 细胞核蛋白抽提步骤
取呈对数生长期的细胞;弃培养液,胰酶消化,将悬液转移至1.5 mL无酶EP管;离心:4℃、1 500 r/min、10 min,弃上清;用预冷的PBS洗细胞,同条件离心,弃上清液;重复离心1次;加冰上预 冷 的CERⅠ200 μL(含Halt Protease Inhibitor, 2 μL),轻轻振摇15 s,重悬细胞,冰上置10 min;加冰上预冷的CERⅡ11 μL,轻轻振摇5 s,冰上置1 min;振摇5s后,离心:4 ℃、13 000 r/min、 5 min;迅速转上清,即胞质蛋白提取物,至一洁净的冰上预冷的EP管中,标记后,置于-80℃冰箱备用;向沉淀中加入冰上预冷的NER100 μL(含Halt Protease Inhibitor, 1 μL),轻轻振摇15 s,重悬细胞,冰上置10 min;离心:4℃、13 000 r/min、5 min;迅速转上清,即胞核蛋白提取物,至另一冰上预冷的无酶EP管中;BCA法测定核蛋白浓度,存于-80℃冰箱备用。
1.6 EMSA 检测
制备6%非变性聚丙烯酰胺凝胶;预电泳:用0.25×TBE 缓冲液,120 V 电压、冰浴中预电泳 1 h;DNA-蛋白结合反应:以AliBaba 2.1 预测结果为基准,结果显示STGC3 基因核心启动子区 -825 bp ~-816 bp 区域只有Sp1 转录因子的结合位点,截取该区域26 bp(-932 ~-807 bp)寡核苷酸序列制作特异性探针,探针序列为:
Wt-Sp1, 5'-AAAAAGGCCGGGCGCGGTGG CTCACG-3', Mt-Sp1, 5'- AAAAAGGCCAAGCA TGGTGGCTCACG-3', 并对Wt-Sp1 和Mt-Sp1 结合的DNA 双链探针的5'-端进行生物素标记,生物素标记野生型和突变型探针,以及未标记的野生型探针均由北京微奥基因科技有限公司合成;上样;电泳及转膜;紫外交联:转膜完成后,将膜取出,置于一干燥的滤纸上,溴酚蓝面向上,将膜放置于紫外灯(波长254 nm)下约10 cm 处,紫外交联10 min;化学发光检测生物素标记的DNA。
1.7 ChIP 分析
体内交联与裂解;超声波断裂DNA;免疫沉淀已交联的蛋白/DNA 复合物;洗脱蛋白/DNA 复合物与解交联蛋白/DNA 复合物使DNA 游离;Spin Columns 洗脱DNA;PCR 扩增,其中PCR 扩增反应:根据Primer 5.0 设计的引物,进行PCR 扩增,反应条件为95℃预变性5 min 后循环,94℃变性 30 s,54℃退火50 s,72℃延伸30 s,35 个循环后于72 ℃终延伸5 min,扩增反应结束后,取扩增产物5 μL 与1 μL 6×上样缓冲液混匀后,于1%琼脂糖进行凝胶电泳,检测扩增片段。
2 结果
2.1 STGC3 基因转录调控元件预测
AliBaba 2.1、TESS 和TFSEARCH 3 种在线软件,预测并分析STGC3 基因核心启动子-934 ~ -653 bp 区域序列内存在的转录调控元件。结果显示,该基因核心启动区域内存在转录调控元件较多,综合分析预测结果,确定以AliBaba 2.1 软件预测结果作为本实验结果。如图1 所示,在软件默认参数条件下,即:Pairsim to known sites:50;Mat width in bp:10;Min num of sites:4;Min mat. Conservation:75%;Sim of seq to mat:1%;Factor class level:4,结果显示,STGC3 基因核心启动区域存在AP-1、Sp1、GAL4、HNF-3B、USF、C/EBPalp 等21 个相关调控元件,其中9 个为Sp1 转录因子结合位点。严格限定其参数后(Pairsim to known sites :64 ;Min mat. Conservation :80%),结果显示,STGC3 基因核心启动区域有5个转录相关调控元件,其中2 个为Sp1 转录因子结合位点(图2)。

图1 参数默认时,STGC3 基因核心启动区转录调控元件预测结果Fig 1 The predicted result of transcription regulatory elements in the core promoter region of STGC3 gene by default
2.2 EMSA 鉴定STGC3 基因转录调控元件
根据生物信息预测结果,STGC3 基因存在转录因子Sp1 结合位点,进行EMSA 实验,选择STGC3 基因核心启动子区中转录因子Sp1 结合位点的26 bp(-832 ~-807 bp)寡核苷酸序列,人工合成5'末端生物素辣根过氧化物酶标记的野生型(Biotin-Wt Sp1)和突变型(Biotin-Mt Sp1)双链探针,同时合成未生物素辣根过氧化物酶标记的野生型(Wt Sp1)探针,未标记的野生型探针(Wt Sp1)作为特异性竞争结合反应(图3)。将所合成的探针与实验所提取的CNE2 细胞核蛋白质进行EMSA 实验,以确定转录因子Sp1 与其特异性结合位点是否特异性结合。结果显示,Sp1 探针与CNE2 细胞提取的核蛋白结合,形成特异性DNA-蛋白质复合物条带 (图4,第2 泳道);当加入100倍和50 倍的末端未标记野生探针(Wt Sp1)竞争性探针后,特异性DNA-蛋白质复合物条带消失(图4,第3 ~4 泳道);加入突变探针,未形成特异性DNA-蛋白质复合物条带 (图4,第5 泳道)。实验结果表明STGC3 核心启动子区存在的转录因子结合位点。
下划线为Sp1 核心序列;方格中为突变碱基;Wt Sp1 为未标记的野生型探针;Biotin-Mt Sp1 为生物素标记的突变型探针;Biotin-Wt Sp1 为生物素标记的野生型探针(图3)。

图2 严格限定参数后,STGC3 基因核心启动区的转录调控元件预测结果Fig 2 The prediction result of transcription regulatory elements in the core promoter region of STGC3 gene after strictly defining parameters

图3 人工合成的Sp1 寡核苷酸双链探针序列Fig 3 The synthetic SP1 oligonucleotide double strand probe sequence
2.3 ChIP 鉴定STGC3 基因转录调控元件
采用ChIP 进一步鉴定STGC3 基因核心启动区存在转录调控元件。实验中为排除可能出现假阳性或假阴性结果,实验分5 组,分别是阳性对照组,即没有用抗体处理的含有总基因DNA 的imput,阴性对照组是用IgG 处理的DNA,同时设Anti-Polymerase Ⅱ和No antibody 组排除假阳性及假阴性,结果显示,转录因子Sp1 抗体实验组与阳性对照Input 组、假阳性Anti-Polymerase Ⅱ组均检测到与目的片段大小一致的200 bp 扩增片段,而阴性IgG 组和假阴性No antibody 组均未产生目的条带(图5)。结果表明,STGC3 基因核心启动子区含有转录因子Sp1 结合位点,STGC3 基因转录调控元件预测正确。

图4 EMSA 鉴定STGC3 基因转录调控元件结果Fig 4 Transcription regulatory element of idenffied by EMSA
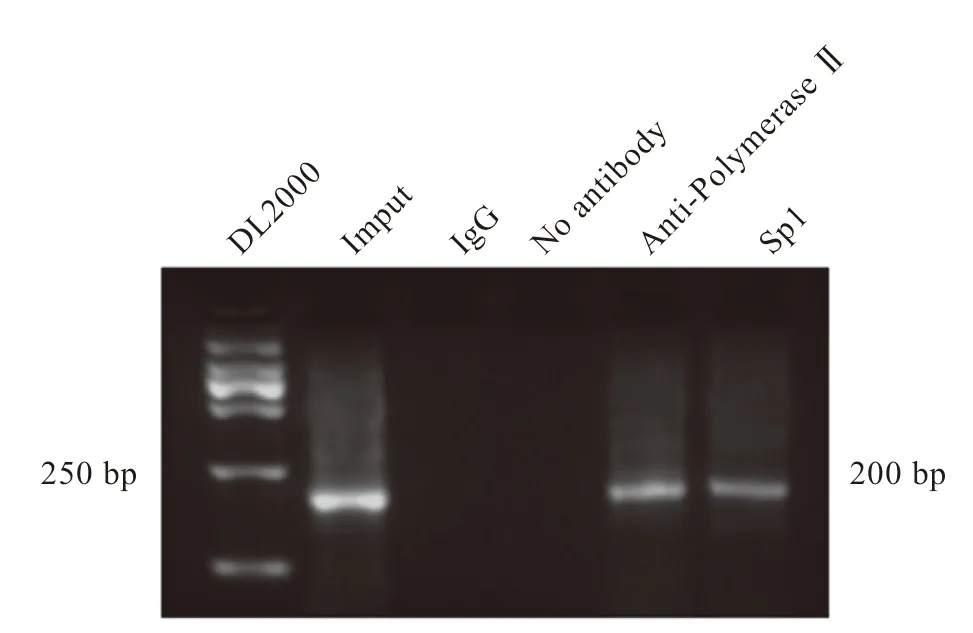
图5 ChIP 鉴定STGC3 基因转录调控元件Fig 5 Transcription regulatory element of STGC3 gene identified by ChIP
3 讨论
随着生命科学技术的迅速发展,海量信息数据库不断完善,生物信息学已成为当今基因结构及功能研究中不可缺少的重要工具之一。生物信息学是一个庞大的系统,可以对基因组中海量调控序列数据进行研究,如启动子及转录因子结合位点预测 分析[8]。
激发转录的不可缺少的部分是核心启动子,核心启动子区域内通常有一些重要的调控元件[9]。前期研究采用生物信息学,预测STGC3 基因的启动子,将启动子构建报告基因载体,对报告基因表达产物活性进行双荧光素酶活性检测,确定了STGC3 基因核心启动子区[10]。为揭示STGC3 基因的转录调控分子机制,运用3 种不同的在线软件AliBaba 2.1、TESS和TFSEARCH, 对STGC3基因核心启动子可能存在的转录调控元件预测,结果表明,当AliBaba 2.1 软件默认参数时预测,显示该核心启动子区存在多个重要调控元件如AP-1、Sp1、GAL4、HNF-3B、USF、C/EBPalp等,且Sp1转录因子结合位点最多;严格调整其参数,结果显示,STGC3基因核心启动子区存在5个转录调控元件,其中Sp1转录因子结合位点同样最多。Sp1普遍存在于生物各种细胞与组织中,是DNA 结合蛋白,能特异性与DNA 结合位点结合,调控富含GC序列的细胞和病毒基因启动子的转录[11-12]。
EMSA 和ChIP 技术,是当前研究基因转录调控元件的经典方法,从体外、体内验证转录调控元件中转录因子和转录因子结合位点及两者的相互作用关系。EMSA 是体外检测蛋白质和DNA 序列相互结合的技术,最初用于DNA 结合蛋白质与相关DNA 结合序列相互作用,可定性和定量,现已用于RNA 结合蛋白和特异性RNA 序列结合研究[13]。由于有些转录因子不具有结合的特异性,即便转录因子和寡核苷酸探针结合,不能说明在体情况下转录因子不能和其他寡核苷酸结合,有一定的局限性。ChIP 技术是体内检测与特异性蛋白结合的DNA 序列,此项技术最大的优点是在活体状态下,检测蛋白质和目的DNA 结合情况,减少了体外实验的误差,在活体细胞中,首先对与调节蛋白质结合的DNA 分离,通过一定的方法随机剪切染色质,用调节蛋白抗体沉淀目的染色质,再用PCR 检测目的DNA 片段。
EMSA结果证实,STGC3核心启动区内有转录因子Sp1结合位点,其结合具有特异性。ChIP实验结果表明STGC3基因转录调控元件预测与实验吻合。上述实验说明STGC3基因核心启动子区有转录因子Sp1结合位点,STGC3基因核心启动子区转录结合位点为STGC3基因的转录调控元件。本实验对STGC3基因转录调控元件的鉴定,将为STGC3基因转录调控的相关分子机制提供实验依据。
参与文献
[1]贺修胜, 肖志强, 陈主初, 等.STGC3 新基因的克隆及功能初步分析[J]. 癌症,2004, 23(10): 1110-1115.
[2]邓敏, 贺修胜, 罗桥,等.Tet 调控STGC3 基因表达CNE2 细胞系的建立及其功能初步研究[J].生物化学与生物物理进展, 2006, 33(1):39-44.
[3]胡波, 邱青朝,贺修胜,等.雌激素对转STGC3 基因CNE2 细胞系生长增殖的影响[J].生物化学与生物物理进展, 2007, 34(5):538-545.
[4]邱青朝, 胡波, 贺修胜, 等.Tet 调控STGC3 基因表达的CNE2 细胞系裸鼠成瘤实验性研究[J].生物化学与生物物理进展, 2007, 34(4):359-365.
[5]He X S, Deng M, Yang S, et al. The tumor supressor function of STGC3 and its reduced expression in nasopharyngeal carcinoma[J]. Cell Mol Biol Lett,2008,13(3):339-352.
[6]李丽, 贺修胜, 罗桥, 等.STGC3 基因缺失对CNE2 细胞生长增殖能力的影响[J].生物化学与生物物理进展, 2011,38(3):248-253.
[7]Qiu Q C, Hu B, He X P, et al. STGC3 inhibits xenograft tumor growth of nasopharyngeal carcinoma cells by altering the expression of proteins associated with apoptosis [J]. Genet Mol Biol, 2012,35(1):18-26.
[8]Xiong Q,Jiang X,Liu X et al. Prediction of IER5 structure and function using a bioinformatics approach[J]. Mol Med Rep,2019,19(6):4631-4636.
[9]Levy S, Hannenhalli S. Identification of transcription factor binding sites in the human genome sequence[J]. Mamm Genome, 2002,13(9):510-514.
[10]Li SY, Wang L L, Sheng H X,et al. Identification and analysis of the promotor region of STGC3 gene[J]. Arch Med Sci,2015,11(5):1095-1100.
[11]Vizcaíno C, Mansilla S, Portugal J, et al. Sp1 transcription factor : a long-standing target in cancer chemotherapy[J]. Pharmacol Ther, 2015,152(4):111-124.
[12]Noda K,Yamazaki M, Iwai Y, et al. IL-1β and TNF-α regulate mouse amelotin gene transcription in gingival epithelial cells[J]. J Oral Sci,2018, 60(3):388-398.
[13]Pan Y,Comiskey D F,Kelly L E, et al. Regulation of photoreceptor gene transcription via a highly conserved transcriptional regulatory element by vsx gene products[J]. Mol Vis,2016, 14 (22):1421-1428.