微卫星DNA标记评估湛江光裸星虫养殖群体的遗传多样性
2020-12-28姚泽彬姚维彭锦胜王庆恒郭昱嵩
姚泽彬 姚维 彭锦胜 王庆恒 郭昱嵩

摘 要 为评估湛江地区光裸星虫养殖群体的种质现状,利用11对荧光标记微卫星引物对光裸星虫的1个野生群体和2个养殖群体,每个群体取30~31个样本进行遗传多样性分析。结果表明,11 个微卫星位点在不同群体呈现出不同程度的多态性,野生群体和2个养殖群体的平均等位基因数(Na)在4.182~4.636,平均有效等位基因数(Ne)在2.447~2.632,平均观测杂合度(Ho)、期望杂合度(He)分别在0.442~0.471、0.525~0.536,平均多态信息含量(PIC)为0.461~0.475。Hardy-Weinber 平衡分析显示,3 个群体的大部分位点未偏离平衡。在每个群体中进行连锁不平衡分析,发现有4对位点间显著(P<0.05)或者极显著(P<0.01)偏离连锁平衡。分子方差分析(AMOVA)显示,光裸星虫大部分的变异来自于群体内而非群体间。综合分析认为,本研究采集的2个光裸星虫养殖群体遗传多样性丰富,暂未出现近交衰退现象。
关键词 光裸星虫;遗传多样性;微卫星标记
中图分类号:S932 文献标志码:A DOI:10.19415/j.cnki.1673-890x.2020.31.001
光裸星虫(Sipunculus nudus L.,1766),隶属于方格星虫纲(Sipunculidea)方格星虫目(Sipunculiformes)方格星虫科(Sipunculidae)方格星虫属(Sipunculus),俗称“沙虫”,生长于温带及热带水域的潮间带,以沙粒间的底栖藻类、有机碎屑等为食,在中国沿海均有分布。光裸星虫营养成分丰富,味道鲜美,高蛋白、低脂肪,具有较高的食用价值[1],但自然采捕难度大,且过度采捕、海区污染、滩涂破坏等威胁光裸星虫的种质资源[2],自然采捕已经难以满足市场的长期需求。近年,光裸星虫的人工养殖日益成熟[3-7],然而,养殖业的快速发展常会因无序引种、苗种异地交易等导致种质资源混杂,进而影响遗传多样性的长期稳定。因此,有必要对人工养殖光裸星虫的遗传多样性进行监测和评估。
光裸星虫的遗传多样性研究目前主要集中在对分子标记和线粒体DNA的应用上,王庆恒等运用RAPD技术对4个光裸星虫地理群体的遗传多样性进行研究,发现不同地理群体存在较大的遗传分化[8]。郭昱嵩等利用微卫星DNA分子标记分析了北部湾3个光裸星虫地理群体的遗传多样性,得出“群体间存在较大的基因流、已产生较低水平的遗传分化”的结论[9]。Du等利用线粒体COⅠ序列分析了3个地理野生群体的遗传多样性差异,认为光裸星虫目前具有较高的遗传多样性和独特的种群结构[10]。彭银辉等基于线粒体控制区序列开展了6个地理野生群体光裸方格星虫的遗传多样性研究,发现遗传分化在群体间并不显著,光裸方格星虫具有较高的遗传多样性,且不同地理群体存在频繁的基因交流[11]。周于娜等利用线粒体控制区序列分析了光裸星虫不同地理野生群体和不同地理养殖群体的遗传差异情况,认为养殖群体光裸星虫的遗传多样性略低于野生群体,养殖群体正在逐渐积累遗传变异[12]。
微卫星DNA(microsatellite DNA),又称简单序列重复(SSR,simple sequence repeat),是1~6个碱基串联重复序列。SSR标记具有稳定性好、多态性丰富、引物通用性较好和共显性遗传等特点,是研究群体遗传学的常用手段。目前,运用微卫星标记研究光裸星虫遗传多样性的报道较少。
本研究运用科研团队自主开发的SSR标记,筛选出多态性高的标记对光裸星虫3个群体的遗传多样性进行检测,并以野生群体作为参考群体来评估养殖光裸星虫的遗传多样性,从而为光裸星虫种质资源保护和合理利用提供参考依据,还可为养殖光裸星虫的种苗优劣评估提供基础数据支持。
1 材料与方法
1.1 实验材料及DNA提取
实验用野生光裸星虫采集自湛江遂溪县下六镇(遂溪野生群体,SX)的自然滩涂。2个湛江养殖群体分别采集自湛江碧海湾水产有限公司乐民繁育基地(乐民养殖群体,LM)和广东海洋大学覃斗(覃斗养殖群体,TD)试验基地。参照文献[13]的苯酚/氯仿抽提法,稍作修改后用于提取光裸星虫基因组DNA,经1%的琼脂糖凝胶电泳检测后,用凝胶成像仪进行观察,估算DNA的浓度和纯度。
1.2 微卫星序列的引物筛选
本实验中项目组自主开发20对光裸星虫微卫星引物[14],委托上海生工生物工程有限公司合成。将合成好的微卫星引物进行群体初筛选,筛选实验用20个随机个体,提取DNA后对所有引物进行预扩增,根据预扩增的结果筛选出11对多态性引物(见表1)。预扩增体系与程序如下:PCR预扩增总体系为10 μL,包含DNA模板0.5 μL,上下游引物(5 μmol·L-1)各0.5 μL,PCR Mix 5 μL,剩余體积用ddH2O 补齐至10 μL。PCR反应程序为:94 ℃预变性80 s;94 ℃
30 s,退火(温度依据引物而定)30 s,72 ℃ 45 s,30个循环;72 ℃延伸5 min。
PCR产物在8%非变性聚丙烯酰胺凝胶分离,银染显色,以出现清晰主带为标准,筛选出多态性引物和每对引物最适退火温度,等位基因大小以 20 bp DNA Ladder Marker为参照标准进行判读。
1.3 PCR扩增及微卫星检测
委托上海生工生物工程有限公司合成荧光引物,用于光裸星虫3个群体样本的扩增,每个群体30~32个个体。
PCR扩增体系为25 μL,包含DNA模板2 μL,上下游引物(5 μmol·L-1)各1 μL,Taq酶0.125 μL,10×PCR Buffer(含Mg2+) 2.5 μL,dNTPs (2.2 mmol·L-1) 2 μL,剩余体积用ddH2O 补齐至25 μL。每个微卫星标记随机抽取4个PCR产物进行检测,用1%琼脂糖凝胶进行电泳,然后放入凝胶成像仪中观察,检测PCR产物是否正常。将检测到无异常的PCR产物送往上海生工生物工程有限公司进行毛细管电泳检测,检测仪器为3730XL序列分析仪(ABI公司,美国)。
1.4 数据分析
使用GenAlEx 6.503软件对生物公司反馈的基因片段长度等数据进行遗传分析,计算11个位点的有效等位基因数(Ne)、等位基因数(Na) 、期望杂合度(He)、观测杂合度(Ho)、Hardy-Weinberg平衡检验、固定指数(Fis)、群体间遗传距离(DA)、遗传相似系数(I)和分子方差分析(AMOVA)。利用PIC-CAL0.6软件计算每个微卫星位点的多态信息含量(PIC)。利用PopGen32软件对每个群体的连锁不平衡进行检测。无效等位基因用Micro-checker软件进行分析。群体间基于Nei's遗传距离的聚类树使用MEGA-X软件进行绘制。
2 结果与分析
2.1 微卫星引物的筛选
筛选实验结果显示,微卫星多态位点有11个,其余9个为单态位点。所筛选出来的11对多态性引物的特征见表1。
2.2 等位基因数和有效等位基因数
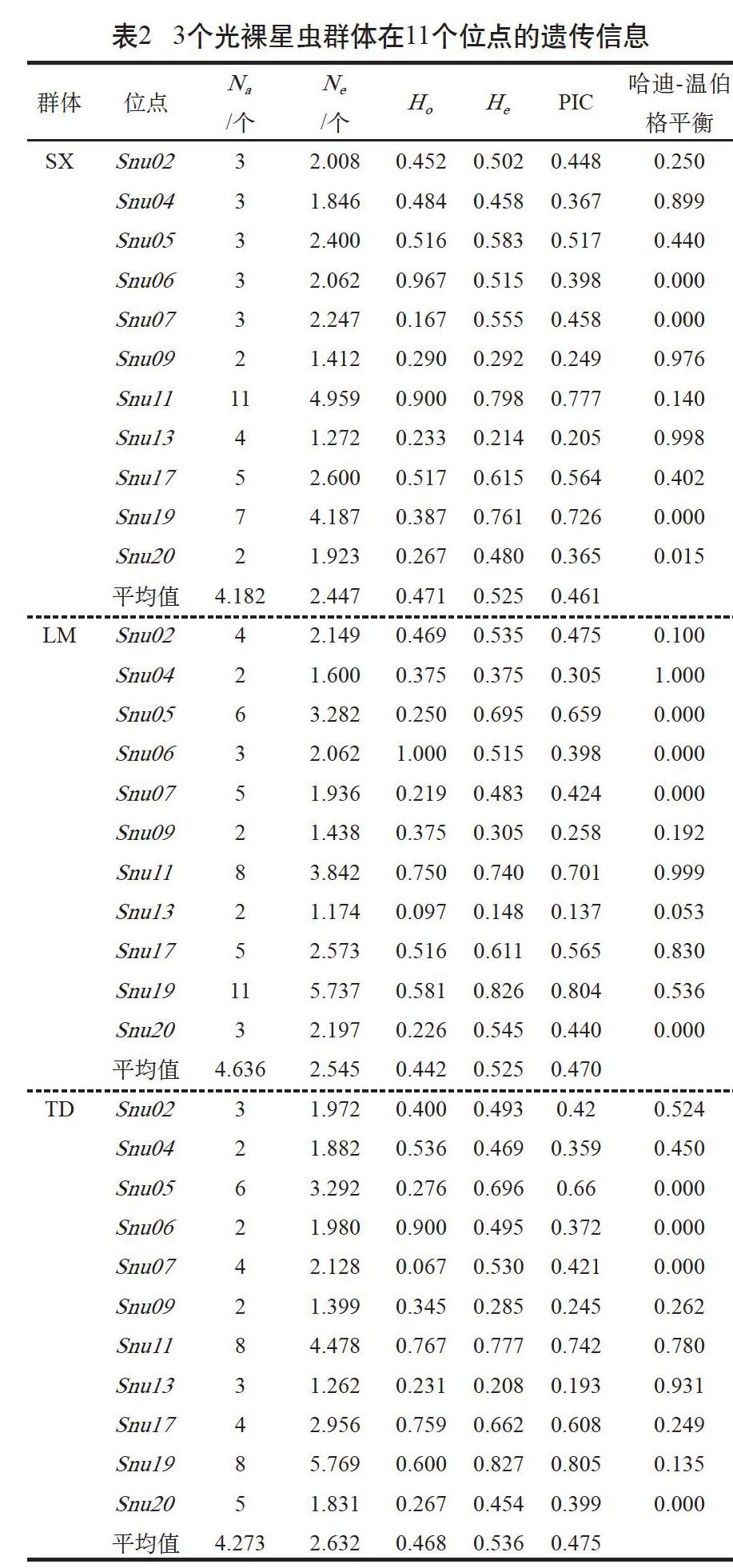
3个光裸星虫群体的11个微卫星标记均表现出多态性,由表2可知,3个群体中,遂溪野生群体的平均Na和平均Ne均最低,为4.182个和2.447个;乐民养殖群体的平均Na最高,为4.636个;覃斗养殖群体的平均Ne最高,为2.632个。
2.3 遗传杂合度、Hardy-Weinberg平衡与连锁不平衡
表2数据显示,3个群体的平均观测杂合度Ho和期望杂合度He的范围分别在0.442~0.471和0.525~0.536,其中,平均Ho遂溪野生群体最大,为0.471,乐民养殖群体最小,为0.442;覃斗养殖群体的平均He在3个群体中最大,为0.536,另外2个群体的平均He均为0.525。经Hardy-Weinberg平衡的卡方检验,在3个群体11个多态微卫星位点中大部分未偏离平衡状态(P>0.05),每个群体均有4个位点偏离平衡状态(P<0.05),其中Snu06、Snu07和Snu20位点在3个群体中均偏离平衡,Snu05位点在2个养殖群体偏离平衡状态,Snu19位点在野生群体偏离平衡。在每个群体中进行连锁不平衡分析,结果表明4对位点间显著(P<0.05)或者极显著(P<0.01)偏离连锁平衡;在乐民养殖群体中有2对连锁不平衡(Snu05/Snu19、Snu07/Snu13),野生群体和覃斗养殖群体均只出现1对相同的连锁不平衡(Snu7/Snu19)。
2.4 多态信息含量(PIC)与无效等位基因
3个群体11个微卫星位点的平均PIC在0.461~0.475,遂溪野生群体最小,2个养殖群体相对较大。Micro-checker检测发现,Snu07、Snu19和Snu20位点在3个群体中均存在无效等位基因,Snu05位点在2个养殖群体中存在无效等位基因,其余位点在各个群体中均不存在无效等位基因。
2.5 群体间遗传分化分析
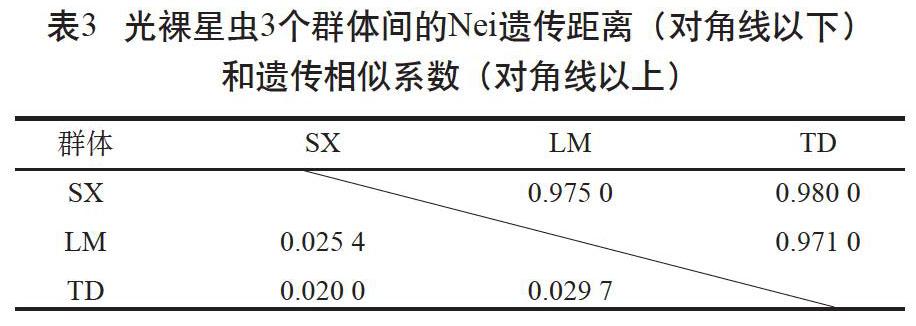
利用GenAlEx 6.503软件计算得到了3个群体间的Nei遗传距离(DA)和遗传相似系数(I)(见表3)。乐民养殖群体和覃斗养殖群体间的DA最大,I最小;遂溪野生群体和覃斗养殖群体间的DA最小,I最大。3个群体间的DA为0.020~0.034。根据Nei遗传距离采用邻接法和UPGMA 法对3个群体进行聚类分析,结果显示遂溪野生群体和乐民养殖群体聚为一支,覃斗养殖群体为一支(见图1,仅列出邻接图)。
种群的遗传分化系数(Fst)表明种群遗传分化的水平,Fst在0~0.05表明种群遗传分化很弱,Fst在0.05~0.15表明种群遗传分化处于中等水平,Fst在0.15~0.25表明种群遗传分化较大,Fst大于0.25表明种群遗传分化很大。本研究中方格星虫3个群体的Fst在0.008~0.012,均不显著。AMOVA分析表明,99%的遗传变异源于群体内,群体间的遗传变异仅占1%(见表4)。
3 讨论
遗传杂合度(H)是指微卫星位点为杂合子的比例,可反映各群体在多个位点上的遗传变异,是描述群体遗传变异的一个重要指标。群体H高,表明该群体的遗传变异多,群体遗传多样性高[15]。郭昱嵩等在2012年報道了北部湾三个光裸星虫野生群体的遗传多样性[9],我们用与该研究相同的11个多态性位点对其平均杂合度进行矫正,得到湛江乌石群体的平均Ho、平均He分别为0.458、0.500,北海山口群体的平均Ho、平均He分别为0.471、0.484,越南锦普群体的平均Ho、平均He分别为0.484、0.532。矫正后的数据,本研究遂溪野生群体的平均Ho和平均He略高于郭昱嵩等报道的湛江乌石野生群体,2个养殖群体的平均Ho和平均He除了乐民群体的平均Ho低于乌石群体外,其他均略高于乌石群体,但差异很小,这表明光裸星虫遂溪群体的遗传变异程度目前仍处于较高水平。
多态信息含量(PIC)反映了微卫星标记所包含的遗传信息含量。在一个群体中该杂合子的比例越大,PIC值越大,该杂合子提供的遗传信息越多。根据Botstein等[16]提出的标准,本研究的11个多态性位点中有4个高度多态性位点(PIC>0.5)、6个中度多态性位点(0.25 遗传距离(DA)的大小可以用来确定群体之间的亲缘关系,群体间的DA与亲缘关系呈正比,与遗传相似度(I)呈反比。DA越大,表明群体间的亲缘关系越远,I越小;反之亦然[17]。本研究采用11个微卫星位点对3个光裸星虫群体进行遗传多样性分析,根据Nei's指数法对光裸星虫3个群体的遗传结构进行分析,结果显示遂溪野生群体和覃斗养殖群体之间的I最大(0.980)、DA最小(0.020),邻接法和UPGMA法聚类分析为一支。遗传分化指数(Fst)是指群体内的变异位点在群体位点中所占的比值,比值越大,群体遗传分化程度越高,本研究中光裸星虫3个群体间的Fst值在0.008~0.012,均小于0.05,3个群体均分化较小[18],这可能与养殖群体为遂溪野生群体繁育的后代有关。AMOVA分析表明,群体间的变异仅1%,提示群体内的遗传变异是引起种群总体变异的主要因素。这与光裸星虫海球幼体时期浮游生活相吻合,因为这增加了群体间的基因交流,导致群体间的变异程度远不如群体内部。总的来说,光裸星虫养殖群体和野生群体之间分化很小,养殖群体暂未出现明显的退化,这与周于娜等[12]报道的结果相似。
整体而言,本研究筛选的11个微卫星位点具有较高的多态性,适用于3个光裸星虫群体的遗传多样性研究。研究结果表明遂溪野生群体与2个养殖群体的各项遗传多样性参数之间总体上差异不大,群体间分化水平较低,光裸星虫养殖群体暂未出现明显的退化现象,这与现阶段光裸星虫大群体亲本育苗、沙虫苗养殖群体沙滩放养等养殖模式吻合。但是,近年来海洋环境污染特别是海滩垃圾污染导致的滩涂破坏有加重趋势[19-21],加上人为无序引种、苗种异地交易等,可能会进一步导致光裸星虫资源的遗传多样性水平下降,因此保护光裸星虫种质资源已经刻不容缓。我们认为只有加强对海洋垃圾源头的控制,定期检测光裸星虫资源的遗传多样性水平,同时推广科学合理的养殖模式,才能保护好光裸星虫资源,达到星虫资源的可持续利用。
参考文献:
[1] 罗少杰,杨创业,王庆恒,等.光裸星虫4个野生群体的营养成分分析与品质评价[J].广东海洋大学学报,2016,36(1):25-30.
[2] 王红勇.海南沿海光裸方格星虫生态习性及其资源保护[J].河北渔业,2014(10):16.
[3] 蒋艳,蔡德建,邹杰,等.方格星虫苗种池塘中间培育试验研究[J].广西科学,2010,17(2):175-177.
[4] 邹杰,彭慧婧,蒋艳,等.方格星虫亲体培育试验[J].渔业现代化,2010,37(3):30-33.
[5] 文雪,蒋艳,邹杰,等.方格星虫滩涂人工养殖试验[J].科学养鱼,2011(1):38-39.
[6] 陈振国,班庭辉,陈振华,等.方格星虫土池育苗技术规程[J].海洋与渔业,2015(8):64-65.
[7] 王庆恒,邓岳文,张家炜,等.一种光裸星虫室内苗种培育的方法:CN106035246A[P].2019.
[8] 王庆恒,杜晓东,李康.光裸星虫遗传多样性的RAPD分析[J].海洋水产研究,2006(3):57-61.
[9] 郭昱嵩,王庆恒,黎幸连,等.北部湾光裸星虫3个地理群体遗传多样性[J].中国水产科学,2012,19(1):62-69.
[10] Du X, Chen Z, Deng Y, et al. Comparative Analysis of Genetic Diversity and Population Structure of Sipunculus nudus as Revealed by Mitochondrial COI Sequences[J]. Biochemical Genetics, 2009,
47(11-12):884-891.
[11] 彭银辉,周于娜,刘旭佳,等.基于线粒体控制区序列的光裸方格星虫群体遗传多样性分析 [J].水产学报,2017,41(10):1542-1551.
[12] 周于娜,彭银辉,刘旭佳,等.光裸方格星虫野生与养殖群体线粒体控制区序列的遗传差异分析[J].水生生物学报,2017,41(2):384-390.
[13] Sambrock J, Russel DW. Molecular Cloning: a Laboratory Manual (3rd Edition)[M]. NewYork:Cold Spring Harbor Laboratory Press, 2002: 461-471.
[14] Wang Qingheng, Guo Yusong, Wang Zhongduo, et al. Isolation and Characterization of Microsatellite DNA Loci From the Peanut Worm, Sipunculus nudus[J]. Genetics and Molecular Research: Gmr, 2012, 11(2): 1662-1665.
[15] 劉楚吾,黎锦明,刘丽,等.中国龙虾微卫星标记的筛选及遗传多样性分析[J].遗传,2010,32(7):737-743.
[16] Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am Jhum Genet, 1980, 32(3): 314-331.
[17] 张爱兵,王正军,谭声江,等.分子生态学重要概念——遗传距离及其测度的理论研究概况[J].生态学报,2002,22(6):149-155.
[18] Balloux F, Lugon-moulin N. The Estimation of Population Differentiation with Microsatellite Markers[J]. Molecular Ecology, 2002, 11(2): 155-165.
[19] 黎树式,林俊良,黄鹄,等.广西海滩侵蚀原因与修复[J].北部湾大学学报,2019,34(12):30-37.
[20] 莫珍妮,曹庆先,陈圆,等.广西沿海典型海滩海洋垃圾调查研究初探[J].化学工程与装备,2018(7):299-301.
[21] 赵肖,綦世斌,廖岩,等.我国海滩垃圾污染现状及控制对策[J].环境科学研究,2016,29(10):1560-1566.
(责任编辑:丁志祥)
