迪庆藏猪FUT1 基因遗传变异分析
2020-12-21胡瑞举杨明华刘韶娜赵彦光赵素梅
方 晨 ,胡瑞举,杨明华,张 斌,刘韶娜,郭 飞,黄 英,赵彦光*,赵素梅*
(1.云南农业大学云南省动物营养与饲料重点实验室,云南昆明 650201;2.云南省畜牧兽医科学院,云南昆明 650224)
断奶仔猪腹泻是由多种因素引起的急性、致死性传染病。肠毒素大肠杆菌(Enterotoxigenic Escherichia coli,ETEC)F18 通过黏附于仔猪小肠黏膜上皮细胞的受体蛋白释放肠毒素,引起仔猪断奶前后腹泻和水肿[1]。α(1,2)岩藻糖转移酶基因1(α-1,2-Fucosyltransferase gene,FUT1)是调节大肠杆菌F18 黏附受体表达的一个候选基因,影响仔猪的腹泻和水肿,位于猪6 号染色体的6q11 区段[2]。相关研究表明,FUT1基因与猪的产仔性能、发育性状和腹泻相关[3-9]。
迪庆藏猪是云南省优良的地方品种,位于高原严寒地区,具有耐寒、抗逆性强、肉质优良等特点[10-11]。本实验以迪庆藏猪仔猪为研究对象,采用测序方法检测FUT1基因,并研究分析FUT1基因编码蛋白,为建立地方猪种抗病育种的候选基因及相关遗传标记和分子育种提供一定的理论依据。
1 材料与方法
1.1 实验动物 实验于云南省畜牧兽医科学院养猪与动物营养研究所猪场进行,选取113 头35 日龄体重8 kg左右的迪庆藏猪断奶仔猪。
1.2 样品采集 实验猪出生后一天内采集耳组织,并放入装有酒精的2 mL 离心管中,-80℃冰箱保存。
1.3 基因组DNA 提取 参照试剂盒(北京天根生化科技有限公司)说明书提取DNA。采用1% 琼脂糖凝胶电泳检测DNA 质量,核酸浓度检测仪测定基因组DNA浓度,并稀释到50 ng/μL,-20℃保存备用。
1.4 引物设计及合成 根据猪FUT1基因全序列(GenBank登录号:NC_010448.4),用Primer3 在线软件(http://primer3.ut.ee/)设计引物,引物由Invitrogen公司合成(表1)。
1.5 PCR 扩增及测序 PCR 扩增反应体系20 μL:Template(10×)2 μL,上、下引物(10 µmol/L)各1 μL,2×Taq PCR Master Mix(10 mmol/L)12.5 μL(擎科生物有限公司),ddH2O 3.5 μL。PCR 产物由通用生物公司测序,FUT1基因PCR 反应条件:94℃预变性5 min,94℃变性40 s,60℃退火40 s,72℃延伸1 min,40 个循环,72℃后延伸7 min 。
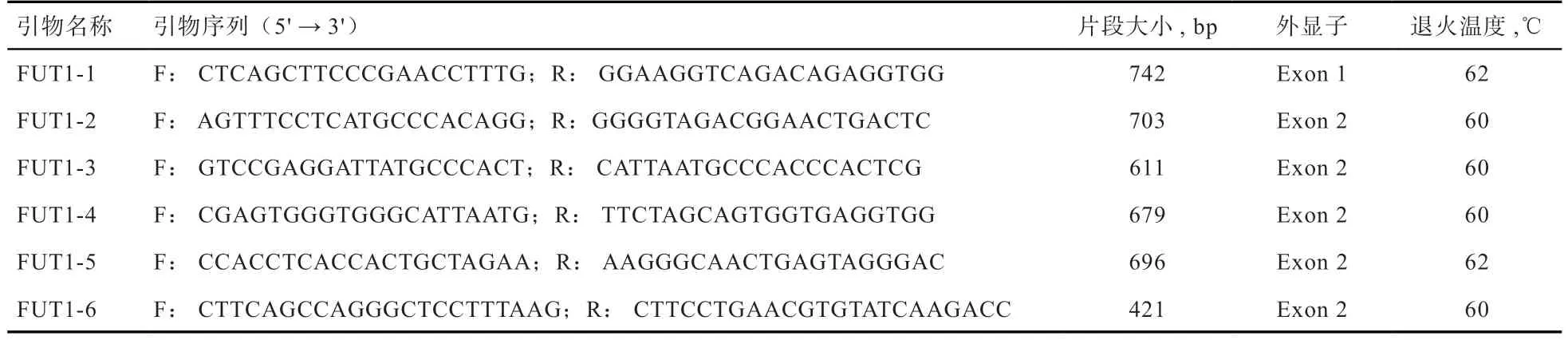
表1 FUT1 基因分段扩增引物
1.6 生物信息学分析 将PCR 扩增的FUT1基因6 段序列进行拼接。应用DNAMAN8.0 软件翻译FUT1基因序列,分析氨基酸突变。使用在线软件ProParam 工具(https://web.expasy.org/protparam/)进行蛋白质理化性质分析;使用ProtScale 程序(https://web.expasy.org/protscale/)分析蛋白疏水性;使用TMHMM2.0(http://www.cbs.dtu.dk/services/TMHMM/)进行蛋白跨膜分析;使用NetPhot(http://www.cbs.dtu.dk/services/NetPhos/)分析蛋白磷酸化位点;使用NetNGlyc(http://www.cbs.dtu.dk/services/NetNGlyc/)分析蛋白糖基化位点;使用SignalP 3.0 Server(http://www.cbs.dtu.dk/services/SignalP-3.0/)分析蛋白信号肽;使用PSIPED(http://bioinf.cs.ucl.ac.uk/psipred/)预测蛋白二级结构;使用SMART(http://smart.embl-heidelberg.de/)分析蛋白结构域;使用Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/)预测蛋白三级结构。
1.7 统计分析 使用DNAstar 软件比对序列,DNAMAN软件进行同源性分析,MEGA8.0 软件构建系统发育树。
2 结果与分析
2.1 基因组DNA 的PCR 扩增产物检测 PCR 产物用1.5%琼脂糖凝胶电泳检测。FUT1基因扩增产物条带单一、清晰明亮,与预期片段大小相符,符合DNA 测序的要求(图1)。
2.2 氨基酸变异分析 将6 段测序序列拼接,得到FUT1基因全长3 045 bp。通过比对迪庆藏猪FUT1 氨基酸变异位点与猪FUT1基因全序列(GenBank 登录号:NC_010448.4),发现FUT1基因有7 个突变位点,其中C299T、C862G、C2418A、A306G 这4 个位点为错义突变(图2)。
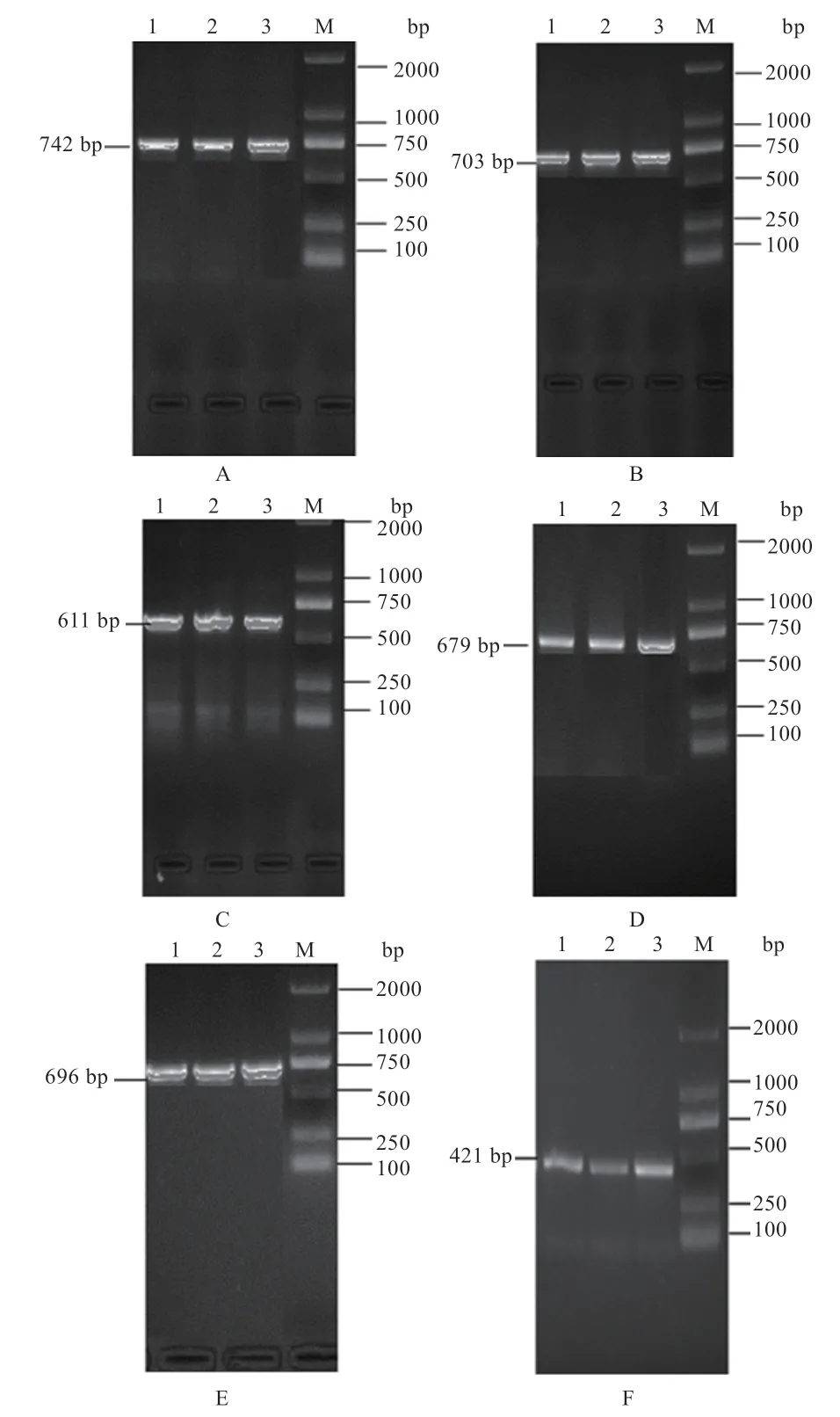
图1 FUT1 基因PCR 扩增产物结果
2.3 生物信息学分析
2.3.1FUT1基因编码蛋白理化性质FUT1基因编码蛋白分子式为C4916H7548N1390O1353S46,共包含15 253 个原子,分子质量约为109 ku,理论等电点pI 为9.21。在组成FUT1蛋白的20种氨基酸中,Leu所占的比例最高(11.4%),Tyr 所占比例最低(1.7%)。根据Guruprasad 方法,FUT1 蛋白的不稳定指数为56.71,脂肪指数为75.23,说明FUT1 蛋白不稳定。
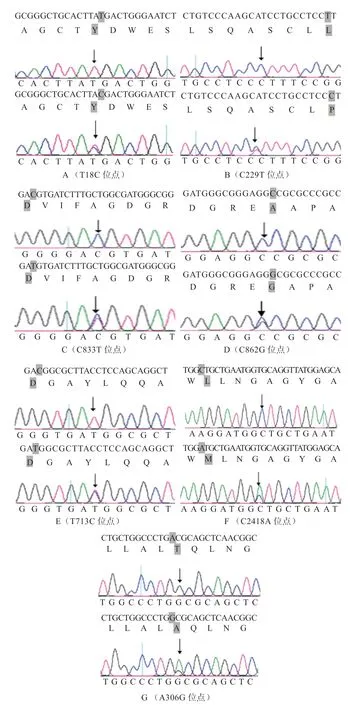
图2 氨基酸变异位点
2.3.2FUT1基因编码蛋白疏水性分析 FUT1 蛋白的1~30 位、700~780 位极具亲水性(图3)。总体看来,FUT1基因编码蛋白是亲水性蛋白。
2.3.3FUT1基因编码蛋白跨膜分析FUT1基因编码残基的所有氨基酸都位于细胞膜表面,与蛋白的疏水性区域结果分析基本一致,表明该蛋白没有跨膜结构(图4)。

图3 FUT1 基因编码蛋白亲水/疏水曲线
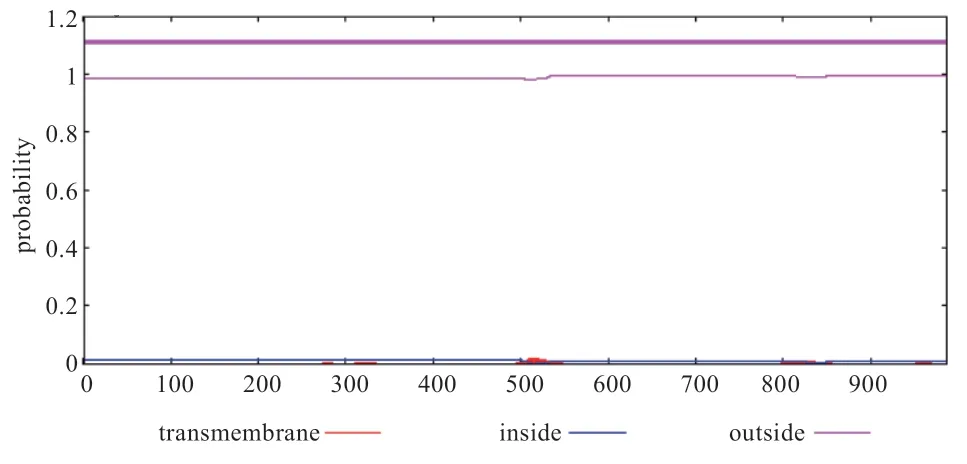
图4 FUT1 基因编码蛋白跨膜区预测结果
2.3.4FUT1基因编码蛋白磷酸化位点分析 FUT1 编码蛋白中发现58 个Ser、28 个Thr 和9 个Tyr 可能是磷酸化位点,真正的磷酸化位点还需要进一步通过试验来确认(图5)。
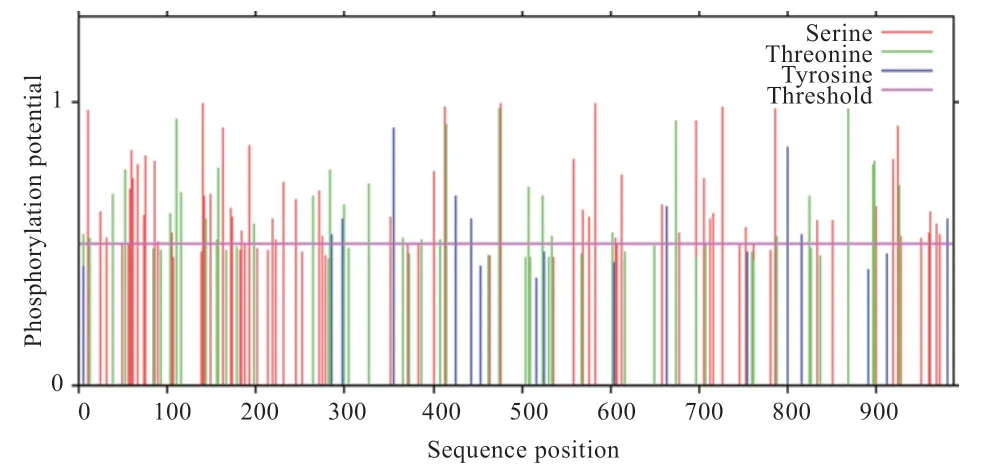
图5 FUT1 基因编码蛋白中磷酸化位点
2.3.5FUT1基因编码蛋白糖基化位点分析 FUT1 蛋白进行糖基化位点预测结果发现3 个典型的N 糖基化位点,分别位于136、195、694 位(图6)。
2.3.6FUT1基因编码蛋白的信号肽分析FUT1基因编码蛋白不具有信号肽结构(图7 和图8)。
2.3.7FUT1基因编码蛋白二级结构预测FUT1基因编码蛋白二级结构主要由α螺旋为主(图9)。
2.3.8FUT1基因编码蛋白超二级结构—结构域分析FUT1基因编码蛋白第264~554 位是一个高度保守的结构功能域——Glyco_transf_11,该结构功能域具有结构分子活性。

图6 FUT1 基因编码蛋白中糖基化位点
图7 FUT1 基因编码蛋白基于神经网络算法预测结果
图8 FUT1 基因编码蛋白基于HMM 算法预测结果
2.3.9FUT1基因编码蛋白三级结构分析FUT1基因编码蛋白的同源蛋白为c2hlhA,该蛋白属于岩藻糖基转移酶(Nodulation Fucosy Ltrans Ferase)家族成员。目标蛋白中有236 个残基以100%的置信度建模。c2hlhA与提交的目的序列匹配性较好,预测准确度为100%,该折叠子的X 射线晶体衍射结构折叠识别出FUT1基因编码蛋白三级结构(图10)。
2.4 不同物种FUT1基因比较及其系统进化树构建 从NCBI 数据库中下载各物种FUT1基因序列。利用DNAMAN 8.0 软件将迪庆藏猪分别与参考物种比对,结果显示,迪庆藏猪与滇南小耳猪的同源性最高(99.8%),迪庆藏猪与人、小鼠、牛、黑猩猩、猴子、马、狗、兔和中国树鼩同源性分别为69.8%、59.4%、80.2%、69.5%、69.5%、79.3%、78.7%、67.2% 和70.2%(表2)。利用MAGE 8.0 软件构建基因系统进化树,发现迪庆藏猪与滇南小耳猪最先聚为一支,小鼠独自为一支(图11)。2 种分析结果均表明同一物种间亲缘关系近,不同物种间亲缘关系较远,不同物种间迪庆藏猪与牛的同源性最高,小鼠最低。
3 讨 论
遗传性抗病力可以发挥机体免疫抗病系统及其相互作用等多方面遗传潜力。抗病育种指利用现有的品种资源,通过常规或分子育种的方法,培育抗病力强的新品种(系),选择培育具有体貌特征好、抗病力强、遗传性能好、适应性强、抗逆性好的个体,经若干世代的选育,最终培育出抗病力强的新品种(系)[12]。腹泻是最常见、严重的仔猪疾病之一,也是引起断奶仔猪死亡的重要原因,不但导致仔猪成活率降低,饲料报酬降低,生长发育停滞甚至形成僵猪,而且会降低仔猪免疫力,继发感染其他疾病,影响经济效益,威胁养猪业的健康发展。吴圣龙等[13-14]证实FUT1基因的M307 位点多态性与断奶仔猪腹泻和水肿病存在直接的相关性,且抗病性AA 基因型不仅对断奶仔猪腹泻具有抗性,并具有较高的一般抗病性。周利华等[15]对10 个国内外猪种FUT1基因新cSNPs 鉴别,并进行仔猪抗大肠杆菌F18侵染研究,结果发现M229C/T 和M714T/C 这2 个位点为错义突变,其中M229 位点可能是决定中国地方猪水肿与腹泻病的变异位点。本研究也发现迪庆藏猪FUT1基因存在7 个突变位点,其中有4 个错义突变,包括已发现的C299T、A306G,以及新的C862G、C2418A,这或许与种质资源或遗传进化相关。迪庆藏猪FUT1基因错义突变影响相关编码蛋白的结构与功能,可能导致迪庆藏猪产生耐严寒、抗逆性强等相关特性。
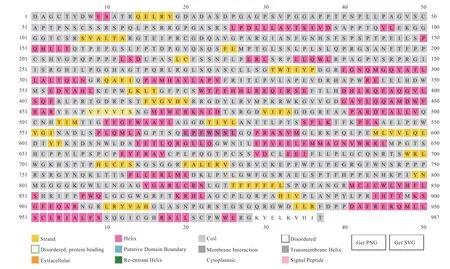
图9 FUT1 基因编码蛋白二级结构预测

图10 FUT1 基因编码蛋白三级结构分析
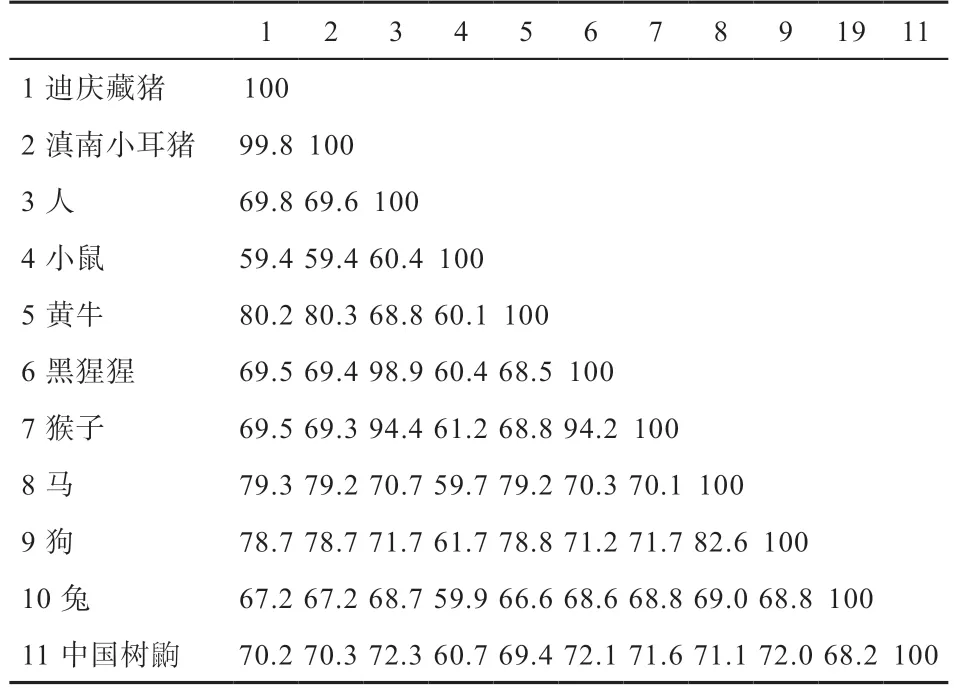
表2 不同物种FUT1 基因同源性比较
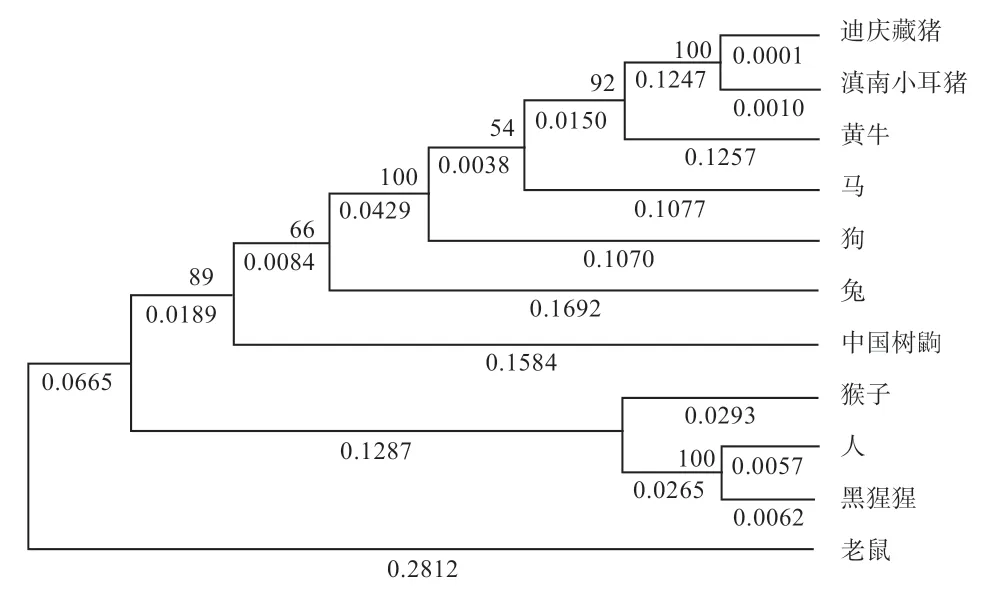
图11 不同物种FUT1 基因进化树
目前关于猪FUT1基因的研究主要集中在仔猪腹泻和生长性能等方面[16-20],生物信息学方面的研究尚未见报道。本研究结果发现,迪庆藏猪FUT1基因全长3 045 bp,共编码987 个氨基酸,FUT1基因编码蛋白是一个亲水不稳定蛋白,没有跨膜结构和信号肽结构。FUT1基因编码蛋白二级结构主要由α螺旋为主,存在一个高度保守的结构功能域Glyco_transf_11,并具有结构分子活性。氨基酸在蛋白质内部,由于其疏水的相互作用,在保持蛋白质三级结构的形成和稳定中起着重要作用。三级结构分析目标蛋白中有236 个残基以高置信度建模,c2hlhA 与目的序列匹配性较好,预测准确度高。从物种遗传进化方面看,各物种FUT1基因序列各不相同。同一物种间迪庆藏猪FUT1基因与滇南小耳猪的同源性高达99.8%,但也存在差异,不同物种间FUT1基因突变差异较大,亲缘关系远,这可能与物种进化或自然环境有关。
4 结 论
迪庆藏猪FUT1基因共有7 个突变位点,其中4 个为错义突变。在FUT1基因编码蛋白结构方面,FUT1蛋白是一个亲水性不稳定蛋白,不存在跨膜结构和信号肽结构,有95 个潜在的磷酸化位点和3 个N 糖基化位点,1 个高度保守的结构功能域Glyco_transf_11,同源蛋白c2hlhA 匹配性较好。在物种遗传进化方面,迪庆藏猪与滇南小耳猪亲缘关系近,与其他物种亲缘关系远。
