122份栽培桃品种(系)黄白肉性状的分子标记辅助鉴定
2020-08-15鲁振华沈志军牛良潘磊崔国朝曾文芳王志强
鲁振华,沈志军,牛良,潘磊,崔国朝,曾文芳,王志强
122份栽培桃品种(系)黄白肉性状的分子标记辅助鉴定
鲁振华1,沈志军2,牛良1,潘磊1,崔国朝1,曾文芳1,王志强1
(1中国农业科学院郑州果树研究所/国家桃葡萄改良中心/农业部果树育种技术重点实验室,郑州 450009;2江苏省农业科学院果树研究所,南京 210014)
【】类胡萝卜素裂解双脱氧酶基因(Carotenoid Cleavage Dioxygenases 4,)控制桃果肉颜色(白/黄),存在3种等位基因。本研究利用Indel、SSR荧光标记毛细管电泳及SNP鉴定等基因分型技术分析我国主要桃黄白肉品种(系)中等位基因的差异,为主要黄/白肉品种(系)的基因型鉴定、亲本选配和选择相应的分子标记对不同来源子代的果肉颜色进行鉴定奠定基础。利用已经报道的桃不同果肉颜色中等位基因3种突变类型,合成不同引物进行PCR扩增,LTR反转录转座子插入突变经1%的琼脂糖凝胶电泳检测,CT单元重复的PCR产物在ABI3730XL测序仪上进行SSR荧光标记毛细管电泳检测,SNP标记经Sanger测序后利用ContigExpress软件分析等位基因的碱基替换(A→T)。综合以上结果,统计每份材料中等位基因的突变类型与果肉颜色的一致性。通过对不同来源的122份桃品种(系)材料进行基因型分析,发现发生LTR反转录转座子插入突变材料的基因型共有31份,占总材料的25.4%,其中纯合插入突变材料的片段扩增长度为729 bp,共有8份,占总突变的25.8%;发生微卫星重复序列突变材料存在2 bp的插入,扩增片段长度为179 bp,该类型共有68份,占总材料的55.7%,其中纯合插入材料25份,占总突变的36.8%;发生A→T碱基替换突变的材料较少,仅有1份,占总材料的0.82%,实际应用中可以不考虑该种类型。CT和LTR插入的两种突变类型的黄肉品种(系)有7份,占总材料的5.7%。研究结果表明,LTR反转录转座子插入突变和微卫星序列重复突变是黄肉桃中等位基因的主要突变类型。其中发生一种纯合突变或两种杂合突变桃品种(系)为黄肉类型,分子标记鉴定结果与调查的122份桃品种(系)黄白肉表型性状完全一致,准确率为100%。采用分子标记明确了122个桃品种(系)黄/白肉性状的基因型,为不同亲本组合子代表型鉴定的标记类型选择提供了技术支撑,为建立桃种质材料黄/白性状的分子辅助育种体系和黄肉桃的选育奠定了基础。
桃;;分子辅助选种;果肉颜色
0 引言
【研究意义】桃[(L.)Batsch]作为我国第三大落叶果树,是近年来发展最快的树种之一。果肉颜色是果实重要的经济性状之一,由于桃果肉颜色(白/黄)表型特征明显,是桃常见的分类标准。黄肉类型桃富含类胡萝卜素,其风味浓郁、营养丰富[1],而类胡萝卜素又是维生素A合成的前体,和人类健康保健密切相关。欧美国家桃品种选育多以黄肉类型为主,近几年,中国、日本和韩国等逐渐转向黄肉桃类型的品种选育,并有迅速发展的趋势[2]。作为多年生果树,桃树童期较长,果实性状鉴定需要2—3年[3],不利于早期选择,一定程度上延缓了育种进程。同时,由于桃黄肉性状受隐性单基因控制,亲本选配是提高黄肉桃育种效率的前提。【前人研究进展】早在1920年,Connors等[4]确定了桃黄肉和白肉是受一对等位基因(Y/y)控制,且白肉相对黄肉为显性遗传。之后研究者对该基因进行研究并分析了不同果肉颜色的代谢成分差异[5-6]。Warburton等[7]、ARÚS等[8]、CANTÍN等[9]和俞明亮等[10]分别获得与Y基因连锁的标记。BRANDI等[5]研究发现黄肉桃品种中的类胡萝卜素含量远高于白肉品种。ADAMIN等[11]通过基因定位发现黄肉桃中调控类胡萝卜素裂解双加氧酶的同源基因位于桃基因组scaffold 1上,且存在3种等位变异形式:LTR反转录转座子插入,微卫星重复序列差异和A→T碱基替换。的突变降低了黄肉桃中类胡萝卜素的降解速率,进而在桃黄肉形成中起到至关重要的作用。FALCHI等[12]进一步确定了不同等位基因控制桃果肉颜色的遗传机制。【本研究切入点】传统的育种方法中,育种工作者常利用一些生物学性状的相关性对杂交后代进行早期鉴定,但鉴定工作繁杂,周期长,随着分子生物学与分子遗传学的发展,在苗期对果肉颜色鉴定已成为可能。【拟解决的关键问题】基于等位基因不同变异形式,采用InDel、微卫星SSR基因分型和Sanger测序分析对122份栽培品种(系)进行分子鉴定,确定我国主要栽培品种黄白肉性状的等位基因类型,为后续选择相应的标记对子代桃黄/白肉类型的分子鉴定和品种选育奠定基础。
1 材料与方法
1.1 材料
试验材料1—79号来源于国家果树种质南京桃资源圃(National Fruit Germplasm Repository of Nanjing,NFGRN),80—122号来源于中国农业科学院郑州果树研究所(Zhengzhou Fruit Research Institute CAAS,ZFRI),具体见表1。全部试验于2017—2018年在中国农业科学院郑州果树研究所的农业农村部果树育种技术重点实验室进行。

表1 CCD4等位基因变异与桃黄白肉表型的关系
1.2 基因组DNA提取
每份桃品种(系)材料取幼嫩叶片约30 mg,液氮研磨,然后用高通量提取叶片基因组DNA,具体提取方法参考张南南等[13]。提取基因组DNA后利用1%琼脂糖凝胶电泳和NanoDrop 1000 spectrophotometer(Themo Scientific)紫外分光光度计检测DNA浓度和纯度,然后将DNA浓度稀释到工作液浓度(约20—50 ng·μL-1),用于后续的基因分型。
1.3 引物设计及PCR扩增
引物序列根据重测序数据设计和参考FUKAMATSU等[14],具体序列信息请见表2,引物和Rox荧光标记引物在上海生工生物工程技术服务有限公司合成。PCR反应体系为2×Taq Master Mix(Mg2+)10 μL,浓度为10 μmol·L-1的正、反向引物各0.2 μL,20—50 ng·μL-1的DNA模板1 μL,使用Eppendorf Mastercycler PCR扩增仪进行DNA扩增。PCR反应程序为95℃ 3 min;95℃ 15 s,55℃ 15 s,72℃ 40 s,共35个循环;72℃ 5 min。
1.4 CCD4序列、微卫星序列和反转录转座子插入分析
根据控制桃黄肉基因的等位基因形式,目的片段和LTR反转录转座子插入突变的目的片段在PCR扩增后经1%琼脂糖凝胶电泳检测;CCD44-SSR扩增产物在ABI 3730XL测序仪(擎科生物技术有限公司)进行毛细管电泳分析,然后采用GeneMapper 4.0软件分析SSR标记在122份桃品种(系)中的基因型信息;CCD4-SNP扩增产物进行Sanger测序,利用ContigExpress软件分析测序结果,检测序列是否发生了碱基A→T替换。

表2 桃中CCD4等位基因序列测序引物
2 结果
2.1 CCD4等位基因的LTR插入分析
研究发现[11],桃成熟时由于黄桃肉中的发生突变而降低了果肉类胡萝卜素的降解速率,从而使桃果肉颜色由白色变为黄色,其中一种突变形式是黄肉桃中的内含子有一个6 254 bp的LTR反转录转座子插入。本研究利用引物CCD4-F/R和LTR-F(CCD4)/CCD4-R对122份桃栽培品种(系)中的等位基因进行扩增(图1),分别扩增片段大小为594 bp(图2-A)和729 bp(图2-B),其中729 bp的片段为LTR插入片段。
统计发现在122份桃品种(系)材料中有8份材料中的存在LTR插入纯合突变,31份材料存在LTR插入杂合突变,存在LTR插入的品种(系)占比相对较高,为25%。由于桃黄/白肉颜色中,白肉性状为显性,因此在LTR杂合突变的材料中存在其他突变类型,从而调控桃果肉黄色性状。
2.2 CCD4等位基因的SSR微卫星分析
黄肉桃品种中存在一种的微卫星重复序列由(TC)7突变为(TC)8,即二者存在2 bp的差异,从而使CCD4蛋白翻译时提前终止而丧失功能。利用SSR分子标记结合荧光毛细管电泳技术,准确分析了122份桃栽培品种(系)中的第一个外显子区域SSR重复序列,发现在黄肉桃品种中等位基因序列中的微卫星重复序列比白肉桃多2 bp的碱基,如‘霞脆’为纯合白肉桃,扩增片段大小为177 bp;‘瑞光22号’为纯合黄肉桃,扩增片段为179 bp;‘春美’为杂合白肉桃,扩增片段为177/179 bp(图3)。本研究所采用的122份材料中有25份材料存在SSR纯合突变,68份材料存在SSR杂合突变,占总材料的55.7%,可能与该类型突变材料在实际育种中作为亲本的比例较高有关。

图1 检测3种CCD4等位基因的分子标记

M:DL2000 DNA marker。A:122份桃品种(系)引物CCD4-F/R扩增的琼脂糖检测;B:122份桃品种(系)引物CCD4 LTR扩增的琼脂糖检测
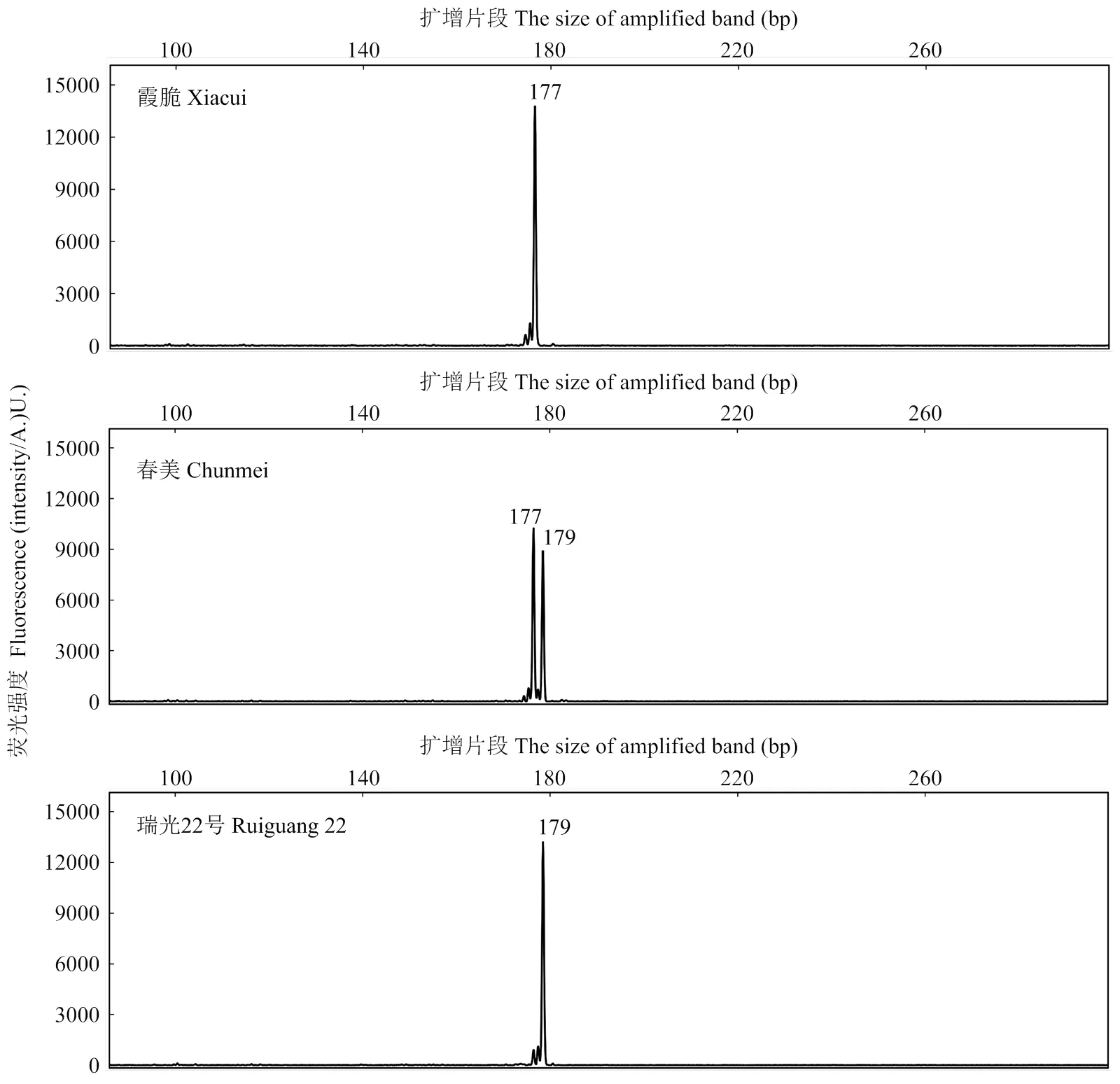
图3 CCD4 3种不同的SSR基因型
2.3 CCD4等位基因序列SNP位点检测
结合Sanger测序对122份桃栽培品种(系)中的等位基因序列进行分析,发现在122份材料中仅有‘弗尔蒂尼莫蒂尼’一份材料等位基因发生了A→T替换突变(图4),说明在黄肉桃中等位基因发生核苷酸替换的突变率极低或者该品种极少用于品种选育的亲本。
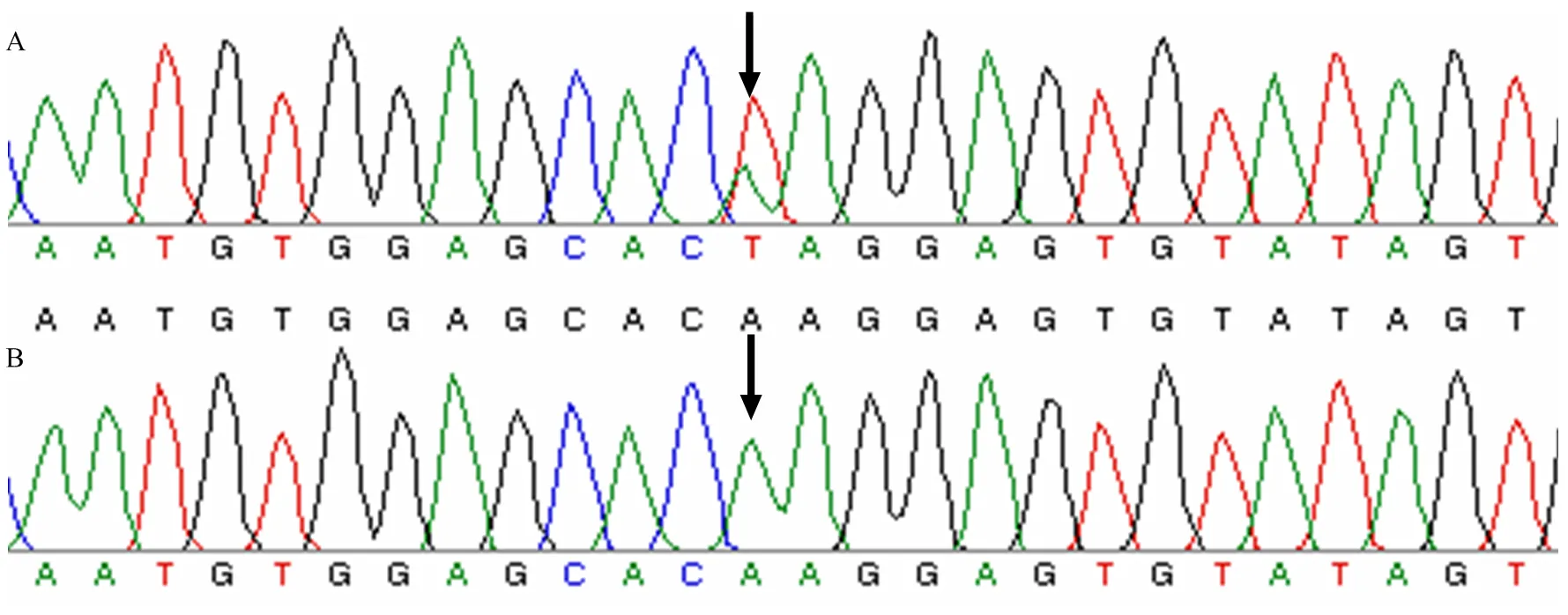
图4 基于Sanger测序的SNP基因分型(A:杂合SNP位点;B:纯合SNP位点)
2.4 122份桃品种(系)的等位基因类型
利用等位基因片段扩增、微卫星基因分型分析、Sanger测序分析结果对122份桃品种(系)材料中的果肉黄白颜色进行区分。在122份桃种质材料中可以扩增出目的基因及LTR插入的目的片段记为1,无法扩增的记为0,统计结果和SSR基因分型及SNP位点测序结果如表1所示。发现SSR基因型为179/179时,对应桃品种的果肉颜色为黄色;LTR扩增片段为0/1时,对应桃品种的果肉颜色为黄色;SSR基因型为177/179,和LTR扩增片段为0/1时,对应材料的果肉颜色为黄色;SSR基因型为177/179,SNP位点基因型为A/T时,对应桃品种的果肉颜色为黄色,其余组合对应材料的果肉颜色为白色(表1)。通过分析3种不同类型等位基因发现,纯合CT插入导致果肉颜色为黄色的品种(系)为25个;纯合LTR插入导致果肉颜色为黄色的品种(系)为8个;LTR插入和CT插入变异导致果肉颜色为黄色的品种(系)为7个;CT插入和SNP替代导致果肉颜色为黄色的品种(系)为1个(表3),分析结果与122份桃品种(系)果肉黄白颜色性状的田间调查结果一致。说明可以综合利用等位基因扩增、SSR基因分型和Sanger测序等分子标记,准确又快速地对桃的杂交后代幼苗进行黄/白肉性状的区分。
3 讨论
桃栽培品种为二倍体,其基因组小、童期短,相对其他多年生木本植物遗传改良周期较短[15]。桃全基因组的测序和高质量的组装[16],加速了对目标性状基因的定位,为桃的分子辅助选种和果树遗传改良奠定了重要的基础[17]。基因定位是实现目标性状分子鉴定的前提,而确定控制目标性状的基因则可实现表型的直接分子鉴定。采用分子标记进行辅助选种可以通过两种途径;(1)通过对控制性状的基因序列开发分子标记可以直接完成表型鉴定;(2)根据亲本基因型,在目标性状位点两侧开发分子标记,进而实现表型的鉴定。其中第一种可实现100%的表型预测,第二种准确性主要取决于定位的区间大小。目前,桃上已经克隆多个控制质量性状的候选基因,包括分枝角度[18-19]、矮化[20-21]、黏离核[22]、果形[23]、桃果实毛/油[24]、肉质[25]以及桃果肉颜色(红、黄和白肉)等基因[11-12,26],实现了对表型性状的直接分子鉴定。

表3 不同黄肉桃品种(系)CCD的等位基因类型
在植物长期的进化和人工选择过程中产生不同类型的等位变异,包括LTR插入、微卫星重复序列和单碱基变异等。其中逆转录转座子(retrotransposons)LTR和non-LTR是真核植物基因组中常见的变异类型之一,特别是长末端重复序列(long terminal repeat,LTR)的插入导致基因的突变在作物中最为常见,可改变植物基因表达和转录完整性[27]。如葡萄的果皮颜色[28]、苹果果肉颜色[29]和柑橘颜色[30]等均是由于LTR的插入导致。微卫星在植物基因组分布较为广泛,微卫星重复数改变多见于内含子区,有些也发生在基因编码区。在对已报到的3种等位变异形式进行基因型鉴定发现,白肉包括3个外显子、1个内含子、7个短的CT重复。在黄肉类型中,由于8个CT重复导致翻译提前终止,果肉表现黄色。自然界植物中也存在单碱基的突变导致表型的改变[31],如LEE等[32]发现编码区一个单个碱基A突变为G,进而导致水稻胚大小的改变。在桃和番茄中也发现了单个碱基的突变导致编码蛋白提前终止,从而使植株产生突变表型[33]。
本研究对122份品种(系)的基因型分析发现,LTR插入和微卫星重复数变化所占比例较高,仅有1个品种是A→T替换突变导致果肉颜色由白色变为黄色,该结果与FUKAMATSU等[14]研究结果一致,即对39份日本桃品种进行分析,并未检测到等位基因A→T替换突变类型。综合分析认为,可能CT重复数差异突变的单株最早作为亲本用于品种的选育,主导了现有品种的谱系,才导致该类型占比较多,也可能多个单株在重组过程中,出现相同的变异类型。微卫星重复数量差异和LTR插入是引起桃果肉颜色表型自然突变的主要途径。
4 结论
利用等位基因扩增、微卫星SSR基因分型分析和Sanger测序分析对122份桃种质资源进行果肉黄/白等位基因突变鉴定,明确了苗期可以快速区分桃子代果肉颜色的分子鉴定方法,加快了桃品种选育及后代果肉颜色鉴定进程,为桃品种的选育及改良提供了重要的参考依据。
[1] LAYNE D R, BASSI D.. 2008: 17.
[2] ROBERTSON A J, HORVAT R J, LYON B G, MEREDITH F I, SENTER S D, OKIE W R. Comparison of quality characteristic of selected yellow and white-fleshed peach cultivar., 1990, 55: 1308-1311.
[3] ARANZANA M J, ILLA E, HOWAD W, ARÚS P. A first insight into peach [(L.) Batsch] SNP variability., 2012, 8: 1359-1369.
[4] CONNORS C H. Some notes on the inheritance of unit characters in the peach., 1920, 16: 24-36.
[5] BRANDI F, BAR E, MOURGUES F, HORVATH G, TURCSI E, GIULIANO G, LIVERANI A, TARTARINI S, LEWINSOHN E, ROSATI C. Study of ‘Redhaven’ peach and its white-fleshed mutant suggests a key role of4 carotenoid dioxygenase in carotenoid and norisoprenoid volatile metabolism., 2011, 11: 24.
[6] MA J J, LI J, ZHAO J B, ZHOU H, REN F, WANG L, GU C, LIAO L, HAN Y P. Inactivation of a gene encoding(4) leads to carotenoid-based yellow coloration of fruit flesh and leaf midvein in peach., 2014, 32: 246-257.
[7] WARBURTON M L, BECERRA-VELÀSQUEZ V L, GOFFREDA J C, BLISS F A. Utility of RAPD markers in identifying genetic linkages to genes of economic interest in peach., 1996, 93: 920-925.
[8] ARÚS P, VERDE I, SOSINSKI B, ZHEBENTYAYEVA T, ABBOTT A G. The peach genome., 2012, 8: 531-547.
[9] Cantín C M, Gogorcena Y, Moreno M A. Phenotypic diversity and relationships of fruit quality traits in peach and nectarine [(L.) Batsch] breeding progenies., 2010, 171: 211.
[10] 俞明亮, 马瑞娟, 沈志军, 章镇. 桃果肉颜色、果皮茸毛和花粉育性性状的分子标记. 园艺学报, 2006, 33(3): 511-517.
YU M L, MA R J, SHEN Z J, ZHANG Z. Molecular markers linked to specific characteristics of(L.) Batsch., 2006, 33(3): 511-517. (in Chinese)
[11] Adami M, Franceschi P D, Brandi F, Liverani A, Giovannini D, Rosati C, Dondini L, Tartarini S. Identifying a carotenoid cleavage dioxygenase (ccd4) gene controlling yellow/white fruit flesh color of peach., 2013, 31:1166-1175.
[12] Falchi R, Vendramin E, Zanon L, Scalabrin S, Cipriani
G, Verde I, Vizzotto G, Morgante M. Three distinct mutational mechanisms acting on a single gene underpin the origin of yellow flesh in peach., 2013, 76: 175-187.
[13] 张南南, 牛良, 崔国朝, 潘磊, 曾文芳, 王志强, 鲁振华. 一种高通量提取桃DNA方法的建立与应用,中国农业科学, 2018, 51(13): 2614-2621.
ZHANG N N, NIU L, CUI G C, PAN L, ZENG W F, WANG Z Q, LU Z H. Establishment and application of a high-throughout protocol for peach () DNA extraction., 2018, 51(13): 2614-2621. (in Chinese)
[14] FUKAMATSU Y, TAMURA T, HIHARA S, ODA K. Mutations in the4 carotenoid cleavage dioxygenase gene of yellow-flesh peaches.,,, 2013, 77(12): 2514-2516.
[15] ARANZANA M J, ABBASSI E, HOWAD W, ARÚS P. Genetic variation, population structure and linkage disequilibrium in peach commercial varieties., 2010, 11: 69.
[16] VERDE I, ABBOTT A G, SCALABRIN S, JUNG S, SHU S Q, MARRONI F, ZHEBENTYAYEVA T, DETTORI M T, GRIMWOOD J, CATTONARO F, ZUCCOLO A, ROSSINI L, JENKINS J, VENDRAMIN E, MEISEL L A, DECROOCQ V, SOSINSKI B, PROCHNIK S, MITROS T, POLICRITI A, et al. The high-quality draft genome of peach () identifies unique patterns of genetic diversity, domestication and genome evolution., 2013, 45: 487-494.
[17] FRESNEDO-RAMÍREZ J, FRETT T J, SANDEFUR P J, SALGADO- ROJAS A, CLARK J R, GASIC K, PEACE C P, ANDERSON N, HARTMANN T P, BYRNE D H, BINK M C A M, VAN DE WEG W E, CRISOSTO C H, GRADZIEL T M. QTL mapping and breeding value estimation through pedigree-based analysis of fruit size and weight in four diverse peach breeding programs., 2016, 12: 25.
[18] DARDICK C, CALLAHAN A, HORN R, RUIZ K B, ZHEBENTYAYEVA T, HOLLENDER C, WHITAKER M, ABBOTT A, SCORZA R.1 promotes the horizontal growth of branches in peach trees and is a member of a functionally conserved gene family found in diverse plants species., 2013, 75: 618-630.
[19] HOLLENDER C A, PASCAL T, TABB A, HADIARTO T, SRINIVASAN C, WANG W P, LIU Z C, SCORZA R, DARDICK C. Loss of a highly conserved sterile alpha motif domain gene () results in pendulous branch growth in peach trees., 2018, 115(20): E4690-E4699.
[20] HOLLENDER C A, HADIARTO T, SRINIVASAN C, SCORZA R, DARDICK C. A brachytic dwarfism trait () in peach trees is caused by a nonsense mutation within the gibberellic acid receptor1., 2016, 210: 227-239.
[21] 鲁振华, 牛良, 张南南, 姚家龙, 崔国朝, 曾文芳, 潘磊, 王志强. 基于SNP标记桃矮化基因的精细定位. 中国农业科学, 2017, 50(18): 3572-3580.
LU Z H, NIU L, ZHANG N N, YAO J L, CUI G C, ZENG W F, PAN L, WANG Z Q. Fine mapping of dwarfing gene for peach based on SNP markers., 2017, 50(18): 3572-3580. (in Chinese)
[22] GU C, WANG L, WANG W, ZHOU H, MA B Q, ZHENG H Y, FANG T, OGUTU C, VIMOLMANGKANG S, HAN Y P. Copy number variation of a gene cluster encoding endopolygalacturonase mediates flesh texture and stone adhesion in peach., 2016, 67(6): 1993-2005.
[23] LÓPEZ-GIRONA E, ZHANG Y, EDUARDO I, MORA J R H, ALEXIOU K G, ARÚS P, ARANZANA M J. A deletion affecting an LRR-RLK gene co-segregates with the fruit flat shape trait in peach., 2017, 7: 6714.
[24] VENDRAMIN E, PEA G, DONDINI L, PACHECO I, DETTORI M T, GAZZA L, SCALABRIN S, STROZZI F, TARTARINI S, BASSI D, VERDE I, ROSSINI L. A unique mutation in agene cosegregates with the nectarine phenotype in peach., 2014, 9(3): e90574.
[25] PAN L, ZENG W F, NIU L, LU Z H, WANG X B, LIU H, CUI G C, ZHU Y Q, CHU J F, LI W P, FANG W C, CAI Z G, LI G H, WANG Z Q.11, a strong candidate gene for the stony hard phenotype in peach (L. Batsch), participates in IAA biosynthesis during fruit ripening., 2015, 66(22): 7031-7044.
[26] SHEN Z J, CONFOLENT C, LAMBERT P, POËSSEL J L, QUILOT-TURION B, YU M L, MA R J, PASCAL T. Characterization and genetic mapping of a new blood-flesh trait controlled by the single dominant locus DBF in peach., 2013, 9: 1435-1446.
[27] LISCH D. How important are transposons for plant evolution?, 2013, 14: 49-61.
[28] KOBAYASHI S, GOTO-YAMAMOTO N, HIROCHIKA H. Retrotransposon-induced mutations in grape skin color., 2014, 304(5673): 982.
[29] ZHANG L Y, HU J, HAN X L, LI J J, GAO Y, RICHARDS C M, ZHANG C X, TIAN Y, LIU G M, GUL H, WANG D J, TIAN Y ,YANG C X, MENG M H,YUAN G P, KANG G D,WU Y L,WANG K, ZHANG H T, WANG D P, CONG P H. A high-quality apple genome assembly reveals the association of a retrotransposon and red fruit colour., 2019, 10:1494.
[30] BUTELLI E, LICCIARDELLO C, ZHANG Y, LIU J, MACKAY S, BAILEY P, REFORGIATO-RECUPERO G, MARTIN C. Retrotransposons control fruit-specific, cold-dependent accumulation of anthocyanins in blood oranges., 2012, 24: 1242-1255.
[31] CHEN T, ZHANG Y D, ZHAO L, ZHU Z, LIN J, ZHANG S B, WANG C L. Fine mapping and candidate gene analysis of a green- revertible albino gene() in rice., 2009, 36(2): 117-123.
[32] LEE G, PIAO R H, LEE Y J, KIM B, SEO J H, LEE D Y, JANG S, JIN Z, LEE C S, CHIN J H, KOH H. Identification and characterization of, a new gene controlling embryo size in rice (L.)., 2019, 12: 22.
[33] FERREIRA D S, KEVEI Z, KUROWSKI T, FONSECA M E N, MOHAREB F, BOITEUX L S, THOMPSON A J. Bifurcate flower truss: a novel locus controlling inflorescence branching in tomato contains a defectivekinase gene., 2018, 69 (10): 2581-2593.
Molecular Marker-Assisted Identification of Yellow/White Flesh Trait for 122 Peach Cultivars (Lines)
LU ZhenHua1, SHEN ZhiJun2, NIU Liang1, PAN Lei1, CUI GuoChao1, ZENG WenFang1, WANG ZhiQiang1
(1Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences/National Peach and Grape Improvement Center/Key Laboratory of Fruit Breeding Technology of Ministry of Agriculture, Zhengzhou 450009;2Institute of Pomology, Jiangsu Academy of Agricultural Sciences, Nanjing 210014)
【】It shows that peach flesh color (yellow/white) is controlled by the gene(carotenoid cleavage dioxygenase 4). Based on three types ofallele variations, molecular markers of indel, SSR-CE, and Sanger sequence for SNP were used to analyze the genotypes of 122 peach cultivars (lines), with the aim to determine the correlation between flesh color and genetic variation. This result could provide information for parental matching and the selection of corresponding molecular markers for phenotyping in their offspring. 【】Three types of variations were detected via PCR. LTRtransposable element insertion was detected by 1% agarose gel electrophoresis, and SSR repeat numbers were detected using CE-SSR in ABI3730XL. Nucleotide substitution was detected using the Sanger sequence and analyzed with the ContigExpress software. In total, the genotypes of 122 cultivars (lines) were analyzed, and the correlation between phenotype and genetic variation was determined.【】After genotyping of the 122 cultivars, it was found that 31 accessions were LTRtransposable element insertions with 729-bp amplified fragments, accounting for 25.4% of the total accessions, of which eight accessions (25.8%) were homozygous. Sixty-eight cultivars (lines) were SSR repeat number variations, accounting for 55.7% of the total accessions and including 25 (36.8%) homozygous types with 2-bp insertions. Of the 122 cultivars (lines), only one cultivar (Fertilia Morettini) was caused by nucleotide substitution and SSR repeat number variation, accounting for 0.82%, which was not widely used in the breeding program. LTRtransposable element insertion and SSR repeat number variation were the key types affecting flesh color. Of the 122 cultivars (lines), seven yellow-flesh cultivars (lines) were caused by SSR repeat number variation and LTRtransposable element insertion, accounting for 5.7%. One homozygous or two heterozygous sequence variations were both responsible for yellow flesh. The results showed that genotypes were identical with phenotypes of the 122 accessions, with 100% accuracy. 【】The genotypes of 122 peach cultivars (lines) (white and yellow flesh color) were identified using molecular markers, which could be applied for parental selection, offspring identification in breeding programs, and flesh color selection (white or yellow) using molecular marker-assisted selection.
peach;; MAS; flesh color

10.3864/j.issn.0578-1752.2020.14.016
2019-12-25;
2020-05-18
国家自然科学基金(31870669)、中国农业科学院创新工程(CAAS-ASTIP-2019-ZFRI)、河南省重点研发项目(182102110134)
鲁振华,E-mail:luzhenhua@caas.cn。通信作者王志强,E-mail:wangzhiqiang@caas.cn
(责任编辑 赵伶俐)
