基于Miseq测序技术分析黄颡鱼不同养殖模式下池塘微生物群落结构多样性
2020-08-07王一亭王厚红吴法龙
王一亭 李 波 王厚红 吴法龙 张 磊
(1.中国科学院水生生物研究所中国科学院水生生物多样性与保护重点实验室,武汉 430072;2.中国科学院水生生物研究所淮安研究中心,淮安 223002;3.武汉市农业科学院,武汉 430207;4.淮安市洪泽区三河镇农业技术服务站,淮安 223124)
微生物群落是养殖生态系统的重要组成部分[1],其在一定程度上决定养殖生态系统的稳定性[2],并能够较好地指示养殖环境健康状况[3],因此,通过养殖环境微生物群落结构的研究来比较不同养殖模式对养殖环境及鱼类健康的影响受到越来越多学者的关注[4,5]。
黄颡鱼Pelteobagrus fulvidraco(Richardson)是典型的底栖杂食性鱼类,是我国重要的水产养殖品种。近年来,我国黄颡鱼养殖产业发展迅速,据《2018年中国渔业统计年鉴》,2017年全国黄颡鱼总产量超过48×107kg,产量位居淡水名优鱼类第三。目前,黄颡鱼养殖模式以黄颡鱼单养、黄颡鱼混养草鱼及黄颡鱼混养草鱼、鳙、鲢三种养殖模式最为常见。草鱼是典型的草食性鱼类,有助于清除池塘杂草;鲢、鳙是典型滤食性鱼类,在控制水体生态环境及水质健康中起重要作用[6,7]。通过比较黄颡鱼不同养殖模式下池塘水体及底泥中微生物的群落结构及差异,有助于掌握不同养殖模式环境微生物群落结构的变化规律,从而有效调整或改进现有养殖模式。高通量测序(HTS)技术由于其快速、准确、数据量大的优点,被广泛应用到动物个体微生物及环境微生物基因组学、功能基因组学的研究中[8,9]。对全面分析样本的微生物群落多样性等意义重大[10]。本研究基于构建16S rDNA基因测序的方法,分析黄颡鱼不同养殖模式下池塘养殖水体及底泥中微生物群落结构及差异,旨在探讨三种养殖模式下环境微生物组成及变化规律,为黄颡鱼生态绿色健康养殖模式开发提供理论依据。
1 材料与方法
1.1 实验池塘及实验鱼
实验池塘位于江苏省淮安市淮阴区水产良种场,选择9个标准化养殖池塘,每个池塘面积为6666.7 m2,水深2.2 m,进排水方便。池塘水引自同一进水渠(水源为运河水),池底淤泥约30 cm,底质一致性良好。在实验期间,水温为自然水温。实验所用鱼苗由中国科学院水生生物研究所淮安研究中心自行繁殖的同批次鱼苗,实验饲料由潜江加益加水产饲料有限公司生产,饲料蛋白含量为40%。
1.2 实验设计
实验分为黄颡鱼单养组A、黄颡鱼草鱼混养组B、黄颡鱼草鱼鲢鳙混养组C三种养殖模式,每种养殖模式分别由3个独立的养殖池塘进行实验(即每种养殖模式3个重复)。每种鱼类的放养平均质量及放养密度为: 黄颡鱼(8.20±0.10) g、15 ind./m2,草鱼(10.03±0.04) g、0.05 ind./m2,鲢(9.85±0.11) g、0.04 ind./m2,鳙(11.56±0.32) g、0.03 ind./m2。实验时间为2017年8月至11月,共84d。实验期间每天定时投喂2次(9:00和16:00),每次投喂量约为黄颡鱼鱼体总重的4%,视黄颡鱼吃食情况适量增减投喂量,并记录。
1.3 样品采集和处理
实验开始、结束时分别对池塘养殖水体及底泥取样1次。水样采用5 L采水器分别在池塘的中央和4个角水面下50 cm处采集,并将5个点的水样混合后取2 L带回实验室待测。在水样沉淀后,取上清液通过0.22 μm微孔滤膜真空过滤,将过滤后的滤膜保存在5 mL离心管中,液氮速冻3—5min,然后转移至-80℃冰箱中保存待测。泥样采集用彼得森采泥器分别在池塘中央和4个角采集水底表层泥,混合后装入无菌收集瓶。泥样带回实验室后分装至 5 mL离心管中,液氮速冻3—4h,然后转移至-80 ℃冰箱中保存待测。样品命名见表1。
1.4 样品DNA提取
水样中微生物DNA提取,取出样品滤膜充分剪碎(使样品表面菌群更好的与裂解液接触),置于1.5 mL灭菌离心管中,加入裂解液,使其释放DNA,随后使用E.Z.N.A.TMWater DNA Kit(Omega,美国)试剂盒提取;泥样中微生物DNA提取使用E.Z.N.ATMMag-Bind Soil DNA Kit(Omega,美国)试剂盒,具体方法见试剂盒说明书。
1.5 PCR扩增及测序分析
本研究选择细菌16S rDNA V3-V4可变区作为目的扩增片段,进行后续高通量测序,采用16S rDNA V3-V4区通用引物341F/805R。引物序列为:341F: 5′-CCCTACACGACGCTCTTCCGATCTG-3′,805R: 5′-GACTGGAGTTCCTTGGCACCCGAGA ATTCCA-3′,同时在3′端添加Barcode区分序列样品。
扩增V3-V4可变区采用30 μL PCR体系。PCR体系为: 2×Taqmaster Mix 15 μL,primer F(10 μmol/L)1 μL,Primer R (10 μmol/L) 1 μL,DNA模板 10—20 ng,添加去离子水至30 μL。
PCR条件为: 95℃变性3min;94 ℃变性20s,55℃退火20s,72℃延伸30s,扩增25个循环;最后于72℃延伸5min。扩增后PCR产物经纯化后,交生工生物工程有限公司(上海)进行Miseq(Illumina)高通量测序。

表 1 水样及泥样具体命名表Tab.1 List of names of water samples and mud samples
1.6 数据分析
Illumina Miseq测序得到的原始图像数据文件经碱基识别(Base Calling)分析转化为原始测序序列(Raw Reads),根据barcode区分每个样本,并对序列质量进行质控和过滤;根据overlap关系进行拼接,拼接后的序列再次进行质控和过滤,最后得到优化序列,进行下游分析。优化序列进行OTU 聚类分析和物种分类学分析。
OTU(操作分类单元,Operational Taxonomic Units)是在系统发生学或群体遗传学研究中,为了便于进行分析,人为给某一个分类单元(品系,属,种、分组等)设置的同一标志。根据不同的相似度水平,对所有序列进行OTU划分,本研究在97%的相似水平下的OTU进行生物信息统计分析。
2 结果
2.1 序列丰度
对测序所得的序列进行拼接、质控、去除低质量序列和嵌合体,12个样本共获得655100条合格16S rDNA序列,每个样本的有效序列数目41256—77508条(表2)。细菌的有效序列长度主要分布在400—440 bp(图1)。通过97%相似度的OTU进行稀释性曲线分析,结果发现各个样本曲线均趋向平坦,说明测序数据量合理,更多的数据量只会产生少量新的OTU,因此测序所得序列能够完全展示样品微生物群落多样性(图2)。

表 2 样品序列数统计及菌群多样性分析Tab.2 Numbers of reads and cliversity indes of different samples
2.2 细菌群落的α-多样性分析
利用测序划分的OTU对不同养殖模式下水体和底泥的α-多样性分析结果如表2所示: 所有样品的覆盖度均在97%以上,在三种养殖模式下底泥样本在实验前后OTU数量变化不大,水体样本OTU数量在养殖后期明显增加。
Chao1和ACE指数反映群落分布丰度,指数越大,丰度越高,Shannon和Simpson指数指示群落分布多样性,Shannon指数越大、Simpson指数越小,说明群落多样性越高。在三种养殖模式下底泥样本的Chao1、ACE和Shannon均高于水体样本,说明底泥的群落丰度及多样性明显高于水体。养殖初期与末期样本群落丰度比较,底泥样本的Chao1和A C E 指数前后变化不明显,而水体末期样本(WAE、WBE和WCE)的Chao1和ACE指数明显高于初期样本,其中WBE最高,WCE最低。养殖初期与末期样本群落多样性比较,底泥样本的Shannon和Simpson指数前后变化不明显,而水体末期样本(WAE、WBE和WCE)的Shannon指数明显高于初期样本,其中WCE最高,WAE最低;水体末期样本的Simpson指数明显低于初期样本,其中WCE最低,WAE较高。因此,在三种养殖模式下水体中末期的群落丰度和多样性均高于养殖初期,并且黄颡鱼草鱼混养组水体群落丰度最高,黄颡鱼草鱼鲢鳙混养组水体群落多样性较高。
2.3 水样与底泥样品菌群之间的关系
基于OTU丰度的样本聚类图可知: 底泥和水体样本分别聚为一簇,底泥样本间的空间距离较短,相似性更高。水体和底泥初期样本与末期样本各自具有较高的相似性。三种养殖模式对比,A、B模式与C模式下的样本表现出更大的空间距离(图3)。
为进一步探究每个样本中细菌多样性的相互关系,通过维恩分析观察水体和底泥样本OTU数目组成相似性及重叠情况。
对水体初期和末期各样本中只有一条序列的OTU进行统计分析,发现其数量已经达到总有效OTU数量的60%以上,表明各样本细菌种类多但大多数细菌丰度较小。养殖初期各水样共有OTU数为151条(图4a),占三者OTU总数的8.36%,养殖末期各水样共有OTU数为476条(图4b),占三者OTU总数的14.88%,表明随着养殖活动的进行,水体样本间细菌群落相似度有所增加。
从底泥样本的菌群相似性来看,养殖初期各泥样共有OTU数为1568条(图4c),占三者OTU总数的31.17%,养殖末期各泥样共有OTU数为1104条(图4d),占三者OTU总数的21.64%,养殖前后底泥样本之间共有OTU的比例均较高,说明在不同养殖时期、不同养殖模式下底泥群落相似性均比较高,但养殖后期三者共有OTU数占比下降,说明不同的养殖鱼类使底泥样本微生物群落结构逐渐分化。
PcoA分析各样品间的差异和距离,从图5a可以看出,6个水体样本可以分为2个相似群落,其中初期样本和末期样本分别聚集在一起,而6个泥样样本全部聚集在一起,这与聚类分析的结果一致。对水样进行PcoA分析发现(图5b),沿PC1坐标方向(贡献率为37%),初期样本与末期样本分别趋于收敛,沿PC2坐标方向(贡献率为20%),初期样本得以分开,说明黄颡鱼的养殖使水体微生物群落构成相似度逐渐升高。对泥样进行分析发现(图5c),养殖初期各样本在PC1(贡献率为32%)及PC2(贡献率为24%)方向均高度收敛,距离很近,说明初期样本相似度较高,而末期各样本在2个方向上都趋于分化,三种养殖模式下底泥的微生物群落构成相似度降低,这与聚类分析及维恩分析的结果一致。

图1 样品中有效序列长度分布Fig.1 Distribution of bacterial sequence lengths in the samples
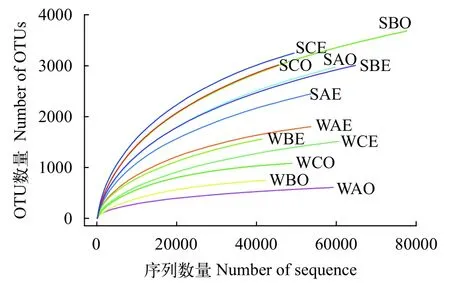
图2 各样品高通量测序结果的稀释性曲线Fig.2 The rarefaction curve basing on high-through sequencing results

图3 基于高通量测序结果的聚类分析结果Fig.3 Clustering analysis results basing on high-through sequencing results of different samples
2.4 各样本微生物菌群构成
高通量测序结果表明水样和泥样的核心微生物群明显不同,养殖前后样本之间也存在较大差异。所有样本的序列分属43个门,其中共有门有8个,分别为变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)、拟杆菌门(Bacteroidetes)、疣微杆菌门(Verrucomicrobia)、浮霉菌门(Planctomycetes)、放线菌门(Actinobacteria)。除此之外,还检测到广古菌门(Euryarchaeota)、梭杆菌门(Fusobacteria)、芽单胞菌门(Gemmatimonadetes)、硝化螺旋菌门(Nitrospirae)、衣原体门(Chlamydiae)、螺旋体门(Spirochaetes)、绿菌门(Chlorobi)、互养菌门(Synergistetes)等不常见的类群,在样本中检测量极低,这表明基于Illumina Miseq平台的高通量测序可作为全面了解黄颡鱼养殖环境微生物结构多样性的有效研究手段。
对水体样本在门水平的物种相对丰度(图6a)分析发现,各样本中丰度占比最大的是变形菌门,但其在养殖末期占比明显下降,3种养殖模式的水体样本分别从初期的75%、91.39%和80.17%,下降到养殖末期的39.5%、42.64%和30.14%。同样明显下降的还有厚壁菌门。丰度上升的有放线菌门、浮霉菌门、疣微杆菌门、绿弯菌门、酸杆菌门。其中,A模式水样拟杆菌门丰度明显上升(1.95%到10.98%),而B模式变化不大(2.11%到2.39%),C模式则略有下降(1.63%到0.76%)。从属水平(图6b)分析水样的组成及丰度发现,养殖末期水体较养殖前期水体的细菌属更丰富。部分水体样本中包含了一些致病菌或条件致病菌,如弧菌属(Vibrio)、假单胞菌属(Pseudomonas)、气单胞菌属(Aeromonas)等。其中,A模式下水体样本中黄颡鱼条件致病菌气单胞菌属(Aeromonas)在养殖末期丰度占优势(5.81%),而其B、C模式下检出量极少甚至未检出。
对底泥样本在门水平的物种组成丰度进行分析(图6a)发现,底泥样本相似度较高,丰度占比最大的门同样为变形菌门,但其在养殖后期丰度均略有上升。其他丰度较高的还有厚壁菌门、绿弯菌门、浮霉菌门等。其中A、B模式酸杆菌门丰度下降,而C模式则有所上升。从属水平分析(图6b),三种养殖模式泥样的假单胞杆菌属在养殖末期丰度显著增加。A、C模式不动杆菌属(Acinetobacter)丰度下降,而B模式丰度升高。A、B模式的Litorilinea属丰度有所下降,而C模式下丰度升高。

图4 维恩图Fig.4 Bacterial Venn diagram at distance 0.03
3 讨论
本研究提取了黄颡鱼不同养殖模式下养殖水体和底泥的微生物宏基因组DNA,采用Illumina Miseq高通量测序平台分析了微生物多样性。得到有效序列41256—77508条,可归纳为608—3686个分类操作单元。基于测序数据,评估了黄颡鱼不同养殖模式下养殖水体和底泥的微生物多样性及群落结构差异。
3.1 养殖水体及底泥微生物群落结构及多样性分析
维恩图分析及主坐标分析表明,随着养殖活动的进行,不同养殖模式下的水体样本共有OTU比例上升,水体样本在主坐标分析图中趋于收敛,表明水体样本间相似性逐渐增加。底泥样本虽然初期样本相似度较高,但后期样本共有OTU比例下降,样本在主坐标分析图中距离增大,表明样本细菌群落分布趋于分化,这可能与黄颡鱼的底栖特性相关。由于池塘底泥与水接触层中微生物含量丰富[11],不同养殖模式造成的微生物群落分布差异性可能通过黄颡鱼对底层水体及底泥的搅动而在底泥样本中被放大,显示出分化现象[12]。
样本聚类分析结果表明,黄颡鱼单养及黄颡鱼、草鱼混养模式下的水体和底泥的菌群组成及群落结构相似性较高,黄颡鱼、草鱼、鲢、鳙混养模式下样本单独形成一类,即样本聚类按照有无鲢鳙聚在一起,表明鲢鳙对混养水体菌群组成和群落结构有更明显的影响。这与唐永涛等[6]关于混养鲢鳙对池塘养殖水体微生物群落结构影响的结果一致。
对样本的α-多样性分析结果表明,不同养殖模式下的黄颡鱼养殖均可显著促进养殖水体微生物群落丰度和多样性增加,但水体样本相似度也有所增加。不同养殖模式末期样本之间相比较,黄颡鱼草鱼混养模式下水体的群落丰度最高,引入鲢、鳙混养后水体微生物群落丰度明显下降,这与Xia等[13]研究发现草鱼混养鲢鱼可以有效减轻草鱼养殖对环境的影响结论一致。草鱼是一种食草性鱼类,主要食用自然环境中的水生植物,但有研究表明草鱼更喜欢养殖池塘中的颗粒饲料[14]。草鱼与黄颡鱼混养,不仅会与黄颡鱼争夺饲料,其排泄的饲料残渣、鱼粪等废物经过发酵分解,为细菌提供适宜环境,使水体微生物大量生长,进而培育出浮游生物,败坏水质[13]。而适量的滤食性鱼类鲢鳙引入养殖模式后,鲢鳙可以将水体中富余的微生物和浮游生物作为饵料,有效减轻草鱼养殖对环境的影响[15]。同时,黄颡鱼、草鱼、鲢、鳙混养模式下的水体微生物群落多样性最高,表明鲢鳙的引入还可以增加水体微生物群落多样性。这与田相利等[16]发现的草鱼、鲢、鲤(Cyprinus carpio,Linnaeus)混养使养殖环境中微生物的结构组成与代谢功能更加多样化的结论一致。

图5 PcoA分析Fig.5 PcoA analysis
3.2 养殖水体及底泥微生物门水平和属水平差异
在本研究中,门类水平上的测序结果表明,在三种养殖模式下水体及底泥样本中,变形菌门的物种丰度明显高于其他微生物丰度,尤其是在养殖初期占绝对地位,这与Hugenholtz等[17]、陈香碧等[18]认为的变形菌门是自然环境中最常见菌群结论一致。同时变形菌门在很多养殖环境中也占据优势地位[19],赵晓伟等[20]对红鳍东方鲀(Takifugu rubripes,Temminck &Schlegel)养殖水体和底泥的微生物群落分析结果表明,变形菌门为优势菌。但随着养殖时间的延长,各种细菌的分布逐渐均匀,变形菌门细菌优势逐渐减弱。
放线菌门在物质代谢上具有独特的优点,在微生物修复水体环境中也有应用[21],尤其以溶藻作用在防治赤潮方面具有重要作用[22]。在本研究中,放线菌门在养殖初期丰度不高,但到养殖末期,却成为水体样本中占比第二的优势门,尤其在黄颡鱼草鱼混养模式下,放线菌丰度占比超过三分之一。这可能是由于鱼类养殖过程中,养殖水体中的残饵、鱼粪等逐渐富集,造成浮游植物生物量变大,为放线菌的生长繁殖提供营养。
拟杆菌门是溶解性有机物的主要消费者。有研究表明,养殖环境中残留饵料及养殖动物粪便在水体中未被及时分解并沉积在水体及底泥中时,会造成养殖池塘底部拟杆菌门细菌含量增加[23]。本研究黄颡鱼单养及混养草鱼时,养殖末期水体及底泥拟杆菌门细菌含量均明显增加,这可能是因为混养草鱼后饵料残渣和鱼粪沉积在水体尤其是底泥中,导致拟杆菌门细菌大量繁殖。而黄颡鱼、草鱼、鲢、鳙混养模式下的环境样本中拟杆菌门细菌含量大幅下降,指示滤食性鱼类鲢鳙的引入有效消耗水体中的草鱼粪便及残余饵料[13],抑制拟杆菌门细菌生长。
酸杆菌门是土壤中重要的一类微生物,是苯酚的主要降解菌[24]。有学者推测酸杆菌门的部分菌种对纤维素有一定的分解能力,而纤维素是植物细胞壁的主要组成成分[22]。在本研究中,底泥样本中酸杆菌门占比较高,这可能是由于底泥中沉积的饵料残渣尤其是草鱼粪便中含有大量纤维素。
在本研究中,厚壁菌门也是样本中丰度较高的优势门,但近年来针对该门的研究主要集中在其作为主要肠道菌群与肥胖的关系上[25]。对于本研究中养殖水体厚壁菌门丰度下降的原因还有待进一步研究。
水产养殖动物细菌性疾病的爆发往往与肠道细菌生态平衡异常相关[26]。养殖鱼类肠道菌群组成与养殖池塘底泥及水体微生物群落结构组成密切相关[27],尤其是淡水鱼肠道内优势菌与养殖水体优势菌组成一般具有相似性[28]。有研究表明,鱼类肠炎的病发与肠道内气单胞菌大量繁殖相关[29]。周金敏等[26]对患细菌性出血病的黄颡鱼与健康黄颡鱼肠道中的气单胞菌进行比较,发现患病黄颡鱼肠道内的气单胞菌数量大幅增加。在本研究中黄颡鱼单养模式下水体样本中气单胞菌在养殖末期丰度占优势,极有可能是单养模式下的黄颡鱼肠道内气单胞菌数量上升所致,该模式下黄颡鱼患出血性肠炎的概率大大增加。因此,有针对性的增加一些套养鱼类可以降低黄颡鱼单养潜在的养殖风险。

图6 样品在门和属水平物种相对丰度Fig.6 Relative abundance of species in phylum and genus level
4 结论
本研究结果显示,不同养殖模式下的黄颡鱼养殖环境微生物均体现了高多样性和高丰度特征,这可能对池塘微生态系统稳定和致病菌的抑制起到积极调控作用。然而,在黄颡鱼单养模式下水体条件致病菌数量有所上升,存在潜在致病风险;在黄颡鱼草鱼混养模式下,草鱼排泄物造成水体中具有分解有机质功能的部分微生物丰度显著增大,同时整体群落丰度也较高,促进浮游生物的生长(相关数据未发表),可能导致水体富营养化升高。只有在适量的滤食性鱼类鲢、鳙引入养殖模式后,通过鲢、鳙的混养有效减轻黄颡鱼养殖对水体及底泥菌群组成和群落结构的负面影响,同时增加水体微生物多样性,帮助恢复水质健康。因此,从微生物群落结构和多样性的角度考虑,黄颡鱼、草鱼、鲢、鳙混养是一种值得推荐的养殖模式。
