日本落叶松LoERF017基因的克隆及生物信息学分析
2020-07-13马苗苗李成浩杨静莉
马苗苗 李成浩 刘 晓 张 馨 杨静莉
(东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040)
日本落叶松(Larixkaempferi)为松科(Pinaceae)落叶松属(Larix)的落叶乔木,为我国东北林区的主要森林树种,分布于大、小兴安岭海拔300~1 200 m地带。喜光性强,对水分要求较高,在各种不同环境下均能生长,而以生于土层深厚、肥润、排水良好的北向缓坡及丘陵地带生长旺盛[1]。日本落叶松作为北方林区的主要造林树种,具有生长快、适应性强、经济价值高的特点,因而常被用于东北地区的荒山造林及纸浆用材林营建[2~3]。然而北方春季易发生干旱,干旱胁迫作为一种最普遍的树木逆境形式,影响树木的生长发育,严重时可降低林木的产量和品质[4],进而会严重影响日本落叶松的成活率。为提高日本落叶松利用效率,我国对落叶松进行的遗传改良早期主要集中在规模化繁殖配套技术,但是该过程操作复杂周期长,工程量大[5],不适合短时间的育种改良体系。随着时间的推进以及基因工程的飞速发展,基因工程为缩短育种周期提供了重要选择,因此研究落叶松遗传改良的基因工程越来越受到重视。
植物激素中的乙烯对植物的生长发育有着多方面的影响,同时也参与植物生物胁迫和非生物胁迫的调控[6~8]。ERF转录因子即乙烯应答因子,又称为乙烯应答键结合蛋白,是乙烯响应元件结合蛋白家族的一个重要的亚族[9],其所包含的AP2区域广泛参与到植物的各种生理反应中,并且积极地响应各种逆境胁迫和发育信号,在植物的生长发育过程中起着十分重要的作用。ERF类转录因子的分类,根据氨基酸序列的不同分为三类,第一和第三类起到激活转录的作用,第二类可以抑制转录[10]。依据进化关系分类,ERF家族又可以分为B1、B2、B3、B4、B5和B6等6个组[11~12]。ERF转录因子的结构,主要由DNA结合域、核定位信号域(NLS)以及转录调控域构成其蛋白结构[13]。而ERF转录因子的功能,也是植物所特有的[9]。ERF不仅自身表达处在复杂的调控途径中,并且在植物的生物与非生物胁迫诱导不同信号之间起调控作用,并参与逆境胁迫的应答反应。相关研究表明,ERF转录因子在提高农作物抗旱性和耐盐性中起到了至关重要的作用[14~15]。例如:在水稻中过量表达TERF1后增强了转基因水稻对干旱和高盐胁迫的耐受力[16];在番茄中ERF5的过量表达可以增加番茄对干旱和盐胁迫的适应性[17];在拟南芥的研究中表明ERF4的过量表达可以提高其抗旱和抗盐的能力[18];在杨树中ERF5.1可能是参与干旱胁迫的重要转录因子[19];而在烟草中ERF3的过量表达可以提高烟草对干旱和盐胁迫的抗性[20]。随着研究的发展与深入,在大豆[21]、葡萄[22]、棉花[23]、大薯[24]等多种植物中发现了该类转录因子。
近年来ERF转录因子成为分子生物学的热点之一,而落叶松作为一种重要的林业树种还未涉及,因此本文以落叶松为研究对象,通过生物信息学技术分析预测了ERF基因的功能及结构,同时分析了该基因在落叶松干旱胁迫下的表达模式。
1 材料与方法
1.1 植物材料
利用本实验室播种培养的日本落叶松土培苗,温室生长环境为25℃,光周期为16 h/8 h。培养基质是蛭石和土(1∶1),培养120 d左右,选取株高10~15 cm的落叶松幼苗为实验材料。
1.2 实验方法
1.2.1 日本落叶松RNA的提取及cDNA的合成
取日本落叶松幼苗顶芽向下5 cm左右放入液氮中研磨,利用无锡百泰克生物技术有限公司生产的通用植物总RNA提取试剂盒,提取落叶松总RNA,使用南京诺唯赞生物公司生产的Hi Script®Ⅱ QRT SuperMix for qPCR(+gDNAwiper)进行反转录为cDNA。反转录的总cDNA置于-20℃冰箱内储存备用。
1.2.2 日本落叶松LoERF017基因的克隆
通过本实验室前期的转录组数据,得到该基因的序列,根据序列设计ERF5-2的基因引物,经聚合酶链式反应(PCR)扩增技术得到LoERF017基因。
在该基因序列两端分别设计引物,由擎科生物公司合成正向引物F:5′-ATGGAGATATTCCCCCAGCAGG-3′以及反向引物R:3′-TCAAACGTCCTGGAAGTTCC-5′。以东洋纺生物公司生产的KOD酶进行扩增PCR,克隆出LoERF017基因的全长片段。PCR总反应体系为200 μL,共10管,每管20 μL,其中cDNA模版1 μL、正反向引物各1 μL、dNTP 4 μL、10*KOD Buffer10 μL、KOD酶0.4 μL、去离子水2.6 μL。
PCR反应条件为:95℃预变性3 min,98℃变性10 s,60℃退火30 s,68℃延伸1 min,共35个循环,68℃延伸7 min。
PCR产物经过1.2%琼脂糖凝胶电泳验证,使用DL2000 DNA Marker做目的片段长度的比对,在电泳检测目的条带长度正确后,使用AXYGEN回收试剂盒对PCR扩增产物进行胶回收,具体步骤见试剂盒说明书。胶回收产物使用AXYGEN纯化试剂盒做产物纯化,纯化后的产物连接T载体,利用热激法将其转化于大肠杆菌中,摇菌2 h,菌液离心抽取上清于150 μL重悬涂板,次日挑取平板内生长的大肠杆菌单个菌体12个37℃摇菌,菌液浑浊后PCR,产物用1.2%琼脂糖凝胶电泳进行初步检测,挑选条带长度正确的菌液送擎科生物公司测该基因序列。
1.2.3 植物表达载体pCAMBIA1300-LoERF017-GFP的构建
1.2.3.1 设计载体的双酶切引物
通过分析LoERF017基因全长序列及pCAMBIA1300-GFP载体图谱,设计带有酶切位点的基因引物。在载体图谱的多个插入位点中找出载体可用的全部酶切位点,以筛选可使用的限制性内切酶,然后将该基因全长序列输入到BioEdit软件中,通过软件分析得到不能切断该基因的限制性内切酶,以此选取出既可以切断载体又不会切断基因的限制性内切酶作为最终所用酶。本实验选取BamHⅠ和SalⅠ两个限制性内切酶,并根据所使用的内切酶确定酶切位点序列,由此设计正向酶切引物F:5′-CGCTAGGATGGAGATATTCCCCCAGCAGG-3′反向酶切引物R:3′-CGTCGAGTCAAACGTCCTGGAAGTTCC-5′。
1.2.3.2 目的基因PCR扩增及胶回收
酶切引物合成后,以测序比对序列成功的菌液提取的质粒为模板进行目的基因的扩增,PCR产物在120 V电压下,以电泳检测目的条带长度是否正确,若条带正确则利用购自南京诺唯赞生物公司的2×Easy Taq Master Mix以胶回收体系进行PCR扩增,然后使用AXYGEN胶回收试剂盒对其进行胶回收,并按AXYGEN产物纯化试剂盒说明书的操作步骤进行纯化。
1.2.3.3 pCAMBIA1300-GFP质粒的提取
在20 mL含浓度为50 mg·L-1的卡那霉素(Kana)的LB液体培养基加入20 μL转入pCAMBIA1300-GFP质粒的大肠杆菌菌液,置于37℃摇床上160 r·min-1震荡培养15 h,使用AXYGEN质粒小提试剂盒提取pCAMBIA1300-GFP质粒,具体操作方法见试剂盒说明书。
1.2.3.4 目的基因与载体的双酶切反应及纯化
将胶回收产物和pCAMBIA1300-GFP质粒同时进行BamHⅠ/SalⅠ双酶切,酶切体系如下(100 μL),反应条件为37℃,时间为2~3 h。最后使用AXYGEN纯化试剂盒对酶切产物分别进行纯化。双酶切总反应体系均为100 μL,其中pCAMBIA1300-GFP质粒50 μL、0×T Buffer 10 μL、BamHⅠ 5 μL、SalⅠ 5 μL、去离子水30 μL。胶回收产物50 μL、0×T Buffer 10 μL、BamHⅠ 5 μL、SalⅠ 5 μL、去离子水30 μL。
1.2.3.5 双酶切产物的连接
将酶切后的基因与载体使用全式金T4连接酶进行连接,连接体系如下。基因:载体3∶1;T4酶1 μL;5×Buffer 2 μL;去离子水2 μL;总体积10 μL。反应条件:20℃,2 h。
1.2.3.6 大肠杆菌的转化及PCR检测
将连接所得产物通过热激法转化到大肠杆菌中,摇菌2 h后离心,抽离剩下100 μL菌液打到无菌的含50 mg·L-1Kana的LB固体培养基平板中,用酒精灯烧过的无菌涂棒涂至均匀。在37℃摇床中倒置过夜培养后,次日挑取单克隆菌落摇菌,然后进行菌液PCR及电泳检测,并将阳性的菌液抽取500 μL送去测序,剩余菌液和50%甘油按1∶1比例混匀加入无菌的1.5 mL离心管中,充分混匀放入液氮快速冷凝,-80℃冷冻保存。测序比对正确后的菌液使用高纯度质粒小提试剂盒提取质粒pCAMBIA1300-LoERF017-GFP。
1.2.4 日本落叶松LoERF017基因生物信息学及进化树分析
利用NCBI(https://www.ncbi.nlm.nih.gov/orffinder/)在线软件分析该基因的开放阅读框;通过http://www.bio-soft.net/sms/index.html查询该基因的同源核苷酸及氨基酸序列;利用Ex PASy服务器中的Protparam(https://web.expasy.org/protparam/)工具分析蛋白的理化性质;根据ExPASy(https://web.expasy.org/protscale/)服务器中的ProtScale程序分析该基因蛋白的疏水性;通过SOPMA程序(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测蛋白的二级结构。利用Swiss-Model程序对该基因进行三级结构同源建模;通过TMHMM Server v.2.0(https://www.cbs.dtu.dk/)对蛋白进行跨膜区分析;使用Signal P 3.0 server(http://www.cbs.dtu.dk/services/SignalP-3.0/)对蛋白进行信号肽预测,并预测该蛋白质N端信号肽的有无及其酶切位点;使用COILS Server(https://embnet.vital-it.ch/software/COILS_form.html)对该蛋白卷曲螺旋预测分析;https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi在线预测蛋白结构域;通过http://multalin.toulouse.inra.fr/multalin/multalin.html进行氨基酸多序列比对;通过http://multalin.toulouse.inra.fr/multalin/cgi-bin/multalin.pl网址进行氨基酸结构比对;利用MEGA5.05软件采用邻接法构建系统进化树。
1.2.5 亚细胞定位
构建pCAMBIA1300-LoERF017-GFP基因融合绿色荧光蛋白(GFP)瞬时表达载体,利用AXYGEN质粒试剂盒提取质粒。将pCAMBIA1300-LoERF017-GFP质粒和pCAMBIA1300-GFP质粒分别与金粉包裹,用基因枪(PDS-1000/He Particle Delivery System)轰击放置在1/2MS培养基的2 cm×2 cm的洋葱内表皮细胞,暗培养24 h,之后用激光共聚焦显微镜(OlympusFV1000MPE)观察亚细胞定位情况。
1.2.6 日本落叶松LoERF017基因的表达模式分析
采用实验室播种培养的日本落叶松幼苗,生长环境为25℃组培室,光周期为16 h/8 h,3~5 d浇一次水,生长120 d左右停止浇水,以停止浇水的第3天为0 d取材生长状态良好且一致的土培苗,2棵为1组取3组,分别提取根茎叶的RNA,反转录成cDNA,进行qRT-PCR以分析该基因在日本落叶松不同组织中的特异性表达。
以0 d的落叶松为对照组,干旱胁迫1、3、5、7 d的材料为实验组,分别提取各个胁迫时间实验材料根的RNA,反转录成cDNA,进行qRT-PCR以分析干旱胁迫下该基因在日本落叶松根中的表达情况。干旱胁迫第7天取材后,对落叶松幼苗浇水,在恢复正常间隔3 d浇水期间,对落叶松根进行间隔1 d的连续4次取材,即复水的第1、3、5、7天,分别提取根的RNA,反转录成cDNA,进行qRT-PCR以研究复水后,该基因在根中的表达情况。
使用落叶松基因作为内参基因,设计内参引物由库美公司合成正向引物tubulin-F:ATTATGAAGAGGTCGGAGCAG-3′以及反向引物tubulin-R:CCCCACCAGTACTACCTATCAA-3′,长度为199 bp。同时合成基因正向引物qPCR-F:5′-GAGATATTCCCCCAGCAGGAAC-3′,反向引物qPCR-R:3′-TCGGATTCCCTTATATTGGCGG-5′,长度为84 bp。
以RNA反转录的cDNA为模版,使用南京诺唯赞生物生产的Cham QTMUniversal SYBR® qPCR Master Mix进行qRT-PCR,通过荧光定量PCR仪(Applied Bio systems,Foster City,CA,USA)进行PCR,用2-ΔΔCT计算法计算其相对表达量。
PCR总反应体系为20 μL,其中cDNA模版1 μL、正反向引物各1 μL、2×Easy Taq PCR Super Mix酶10 μL、去离子水7 μL。反应条件:94℃ 30 s;94℃ 5 s、60℃ 30 s,40个循环;60℃ 6 s;70个循环。
2 结果与分析
2.1 日本落叶松LoERF017基因的克隆
以日本落叶松为材料提取顶芽向下5 cm左右的总RNA(如图1所示)。利用落叶松总RNA反转录成cDNA,以Marker2000为片段长度对照,扩增ERF基因的CDS序列,扩增产物长度为624 bp(如图2所示)。
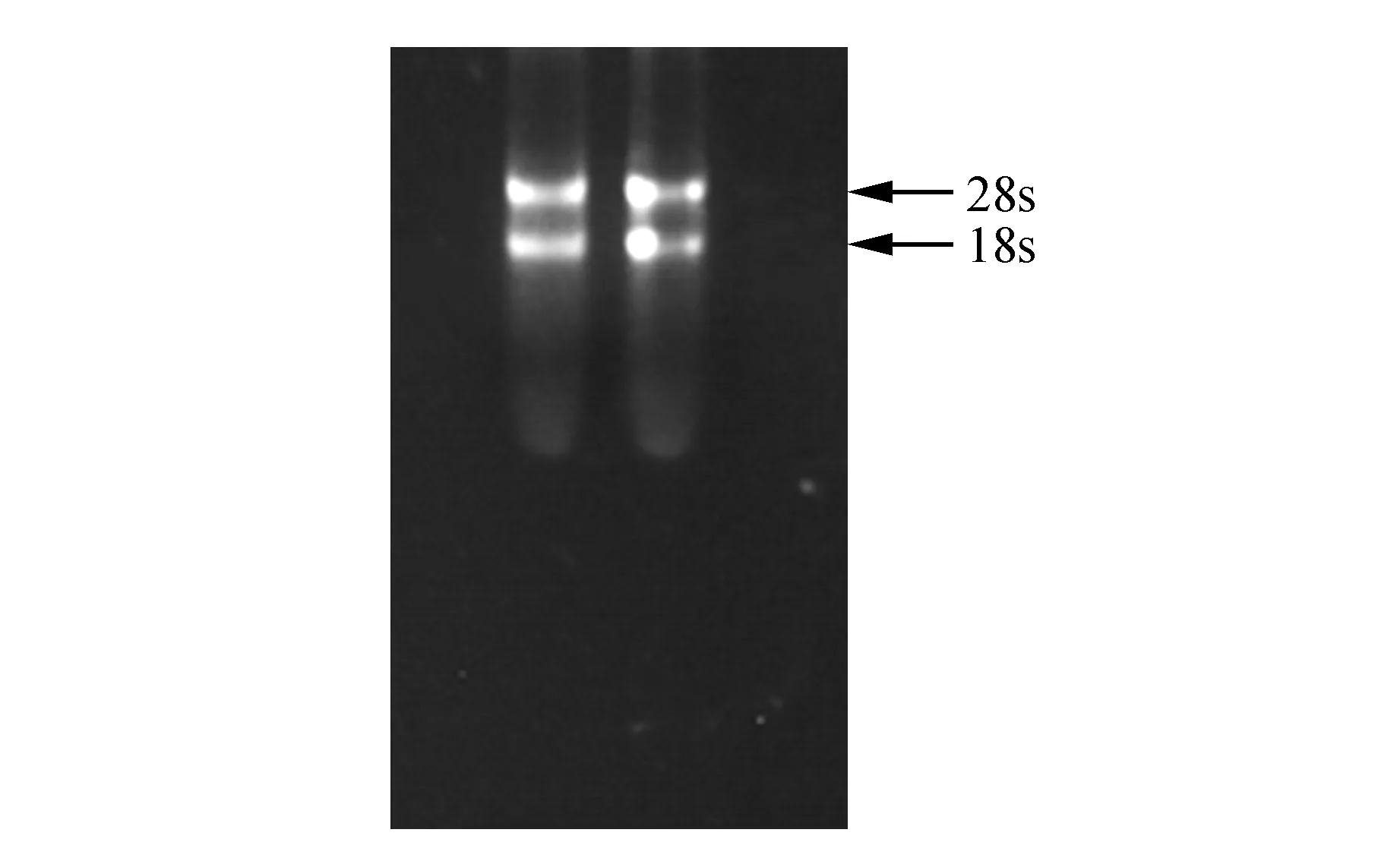
图1 落叶松RNA的提取 M. DNA Marker DL2000;1~5.RNA产物Fig.1 The RNA production of L.kaempferi M.DNA Marker DL2000;1-5.RNA production

图2 落叶松LoERF017基因的克隆 M.DNA Marker DL2000;1~5.LoERF017克隆产物Fig.2 PCR production of LoERF017 M.DNA Marker DL2000;1-5.PCR production of LoERF017
2.2 日本落叶松LoERF017基因编码的氨基酸序列及其理化特性
2.2.1 日本落叶松LoERF017基因编码的氨基酸序列
通过在线查询LoERF017基因共编码206氨基酸(如图3所示)。利用ExPASy服务器中的Protparam工具分析蛋白的理化性质,分析结果氨基酸的数量为206,分子质量为23 458.370理论等电子点为5.43,总共包括3 278个原子,分子式为C1042H1624N284O323S5,在组成蛋白的20种氨基酸中,丝氨酸(Ser)所占比例最高,占10.6%,半胱氨酸(Cys)所占比例最低,占0.5%,不包括含硒半胱氨酸(Sec)和吡咯赖氨酸(Pyl)。蛋白的不稳定指数为61.41,脂肪指数为76.86,总平均亲水性为-0.632。
2.2.2 日本落叶松LoERF017基因的理化特性
2.2.2.1 日本落叶松LoERF017基因编码的蛋白疏水性
根据ExPASy服务器中的ProtScale程序分析落叶松ERF蛋白的疏水性,计算基于K-D法的蛋白质疏水性。分析结果发现,第196位的氨基酸为亮氨酸(Iie),疏水性最大,分值为4.500;第26位的氨基酸为精氨酸(Arg),亲水性最大,分值为-4.500。根据图4中数据,分值为负值的氨基酸数量大于分值为正值的氨基酸数量,表示亲水性大于疏水性,因而推测该蛋白为亲水性蛋白。
2.2.2.2 落叶松LoERF017基因编码的蛋白的二级结构及功能结构域预测
通过在线预测蛋白的二级结构,如图5所示该蛋白的二级结构主要是由α-螺旋(30.43%),无规卷曲(54.11%),延伸链(12.56%),β-折叠(2.9%)组成。
2.2.2.3 蛋白三级结构预测
通过把氨基酸序列输入到Swiss-Model程序中,对LoERF017基因所编码蛋白的三级结构进行预测(如图6所示)。
2.2.2.4 对蛋白跨膜区进行分析
通过把氨基酸序列输入到TMHMM Server v.2.0中,对LoERF017该基因所编码的蛋白进行跨膜区分析,该蛋白长度为206;预测TMHs的数量为0;TMHs中AAs的Exp号是0;Exp编号,前60个AAs为0;其在N端的可能性为0.03768,如图7所示,在细胞膜表面,该基因并没有典型的跨膜螺旋区,细胞膜内也没有氨基酸,同时通过对该蛋白的疏水性区域分析结果,可以得出该蛋白与细胞信号传导无关。
2.2.2.5 蛋白信号肽及蛋白卷曲螺旋预测分析
通过SignalP3.0 server对该基因所编码的蛋白进行蛋白信号肽预测,结果预测蛋白质Non-secretory蛋白质信号肽概率:0.000;信号锚定概率:0.000;最大劈理位点概率:pos.-1和0之间的0.000,结果如图8所示。利用COILS Server对该蛋白卷曲螺旋结构预测分析,结果如图9所示,该蛋白不具有肝蛋白卷曲螺旋结构。
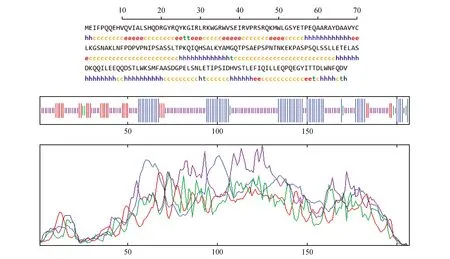
图5 LoERF017蛋白的二级结构预测Fig.5 Predicted secondary structure prediction of LoERF017 protein

图6 LoERF017蛋白的三级结构预测Fig.6 Predicted tertiary structure prediction of LoERF017 protein

图7 LoERF017蛋白的跨膜区分析Fig.7 Transmembrane region prediction of LoERF017 protein

图8 LoERF017蛋白的信号肽预测Fig.8 Signal peptide prediction of the LoERF017 protein

图9 LoERF017蛋白的卷曲螺旋预测Fig.9 Coiled Coils prediction of LoERF017 protein
2.2.2.6 蛋白结构域预测
通过https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi在线分析蛋白的功能结构域,如图10所示,通过分析得出该基因所编码的蛋白,包括AP2和KRBA1两个结构域。LA2是1994年由Jofuku等从拟南芥鉴定出来的,在LA蛋白中发现两个重复的DNA结构域,称这类DNA结构域为AP2结构域[3]。AP2结构域由第27到90的64个氨基酸构成AP2结构域,AP2结构域广泛存在于植物蛋白中。该结构域可以利用乙烯、脱落酸、茉莉酸和水杨酸等信号来调控下游基因的表达,从而提高植物的抗逆性。

图10 LoERF017蛋白的结构域预测Fig.10 Conserverd domain prediction of LoERF017 coded protein
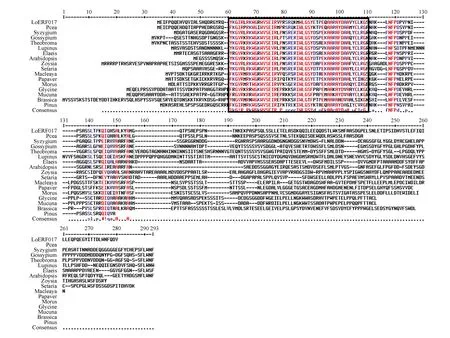
图11 日本落叶松LoERF017氨基酸与其他植物氨基酸序列比对框选为保守区域Fig.11 L.kaempferi alignment analysis of LoERF017 conservative structure domain in different plants Boxes selected as conservative areas

图12 不同植物LoERF017基因的系统进化树(邻接法)及保守区域Fig.12 Molecular phylogenetic tree analysis of LoERF017 in plant species

图13 LoERF017基因的亚细胞定位分析Fig.13 Subcellular localization analysis of the LoERF017 gene
2.2.2.7 氨基酸序列比对及系统进化树的构建
通过在线比对氨基酸序列,将日本落叶松LoERF017基因所翻译的氨基酸序列与相同基因的其他物种的氨基酸序列进行同源性比对,结果所示:日本落叶松(Larixkaempferi)LoERF017基因与火炬松(Pinustaeda)、北美云杉(Piceastichensis)等物种的同源蛋白序列有较高保守性(如图11所示)。利用MEGA5.05软件对该同源蛋白序列进行系统进化树的构建,如图12所示,日本落叶松(Larixkaempferi)与火炬松(Pinustaeda)、北美云杉(Piceastichensis)亲缘关系最近,同时与蒲桃(Syzygiumoleosum)、陆地棉(Gossypiumhirsutum)、油棕(Elaeisguineensis)、可可(Theobromacacao)、鼠耳芥(Arabidopsisthaliana)、博落回(Macleayacordata)、结缕草(Zoysiajaponica)、罂粟(Papaversomniferum)、狭叶羽扇豆(Lupinusangustifolius)、川桑(Morusnotabilis)、狗尾草(Setariaviridis)有较远的遗传距离,所以预测他们有较远的亲缘关系。同时与大豆(Glycinemax)、欧洲油菜(Brassicanapus)、刺毛黧豆(Mucunapruriens)遗传距离最远,因此预测他们亲缘关系更远。
2.3 日本落叶松LoERF017基因的亚细胞定位实验结果
利用基因枪将pCAMBIA1300-LoERF017-GFP质粒和pCAMBIA1300-GFP质粒分别与金粉包裹,轰击放置在1/2MS培养基的2 cm×2 cm的洋葱内表皮细胞,暗培养24 h后,通过激光共聚焦显微镜观察,结果如图13所示。对照组pCAMBIA1300-GFP空载体的绿色荧光充斥整个洋葱细胞,为组成型表达。pCAMBIA1300-LoERF017-GFP的融合表达载体的绿色荧光只在细胞核中,表明该基因定位在细胞核中,并且具有转录因子的一般特征。
2.4 落叶松LoERF017基因的表达模式分析
分别提取0 d的落叶松根、茎、叶RNA,以检测LoERF017基因在日本落叶松中的组织特异性表达,如图14所示,该基因在茎中的表达量是根的0.29倍,在叶中的表达是根的0.55倍,因此可以预测,该基因在日本落叶松的根中表达量最高,在茎中最低,在叶中的表达量居中。
以0 d根的表达量为对照组,分别以干旱胁迫后1、3、5、7 d根的表达量为实验组,分析干旱胁迫下该基因在根中的表达情况,同时在恢复对落叶松的正常浇水间隔之后,分析该基因在落叶松根中的表达情况,结果如图15所示,干旱胁迫下根中的表达量呈上升趋势,干旱胁迫复水后,该基因在根中的表达量有下降趋势,表明复水后该基因的调控作用趋缓,由此可以推测该基因主要在日本落叶松的根中表达并行使功能,并且该基因可能与抗旱性相关。

图14 LoERF017基因在日本落叶松不同组织中的特异性表达情况Fig.14 Specific expression of LoERF017 gene in different tissues of L.kaempferi

图15 LoERF017基因在干旱处理及复水后在根中的表达情况Fig.15 Quantitative real-time PCR analysis of the expression pattern of LoERF017 gene in roots after drought treatment and rehydration
3 讨论
日本落叶松树体高大、树干通直、材质坚韧,是一种重要的造林用材树种,在造纸业和园林绿化中也被广泛应用,具有重要的生态和经济价值。因此提高日本落叶松的抗逆性对我国林业具有多方面的意义。而且利用基因工程的育种手段无疑加快了整个育种进程,缩减了育种工程量,为育种工程提供了极大的便利。
本文从基因工程角度出发,在日本落叶松中克隆获得的LoERF017基因,共含624 bp的CDS序列,共编码206个氨基酸。预测该基因所编码的蛋白只包括AP2一个结构域,不含有指导蛋白的信号肽和跨膜运输结构,且为亲水蛋白。该基因所编码蛋白的二级结构由α-螺旋(30.43%),无规卷曲(54.11%),延伸链(12.56%),β-折叠(2.9%)组成。通过亚细胞定位,得到LoERF017基因定位在细胞核中。QRT-PCR结果表明,在日本落叶松的组织特异性表达中,LoERF017基因在根中的表达量最高,在茎中的表达量最低,在叶中的表达量居中。并且干旱胁迫下,该基因在根中的表达量有明显的上升趋势,对其复水后该基因在根中的表达量呈现下降趋势。这一结果与研究干旱胁迫条件下长春花Str和Pme基因表达变化中Str基因在干旱及复水后的表达结果相一致[25]。由此预测LoERF017与日本落叶松的抗旱调控相关。
通过对实验结果的分析,可以判断出本文所克隆的LoERF017基因属于ERF转录因子中的激活转录类,该基因可以在日本落叶松受到逆境胁迫时提高自身表达量,进而提高落叶松的抗逆性。这个研究结果与在其他一些物种中的ERF转录因子与干旱诱导反应密切相关所一致。并且相关研究表明,在拟南芥中转入LoERF017基因所得到的转基因株系在干旱胁迫下,转基因株系的根长、鲜重及长势都明显优于野生型株系,证明了LoERF017基因可以提高拟南芥的抗旱性[26]。除拟南芥中对LoERF017在植物中抗旱性的探讨,还并未在其他物种中对该基因的抗旱性做深入研究,由此该结论可直接证明本文对LoERF017在日本落叶松中的研究结果。
通过以上研究,可以初步证明落叶松LoERF017基因与日本落叶松的抗旱能力相关,但是关于该转录因子对植物抗逆性的调控机制和信号传导等具体的功能还有待进一步的研究证明。该基因的发现,为日本落叶松物种的基因组测序工作的完成提供了更多的数据参考,也为日本落叶松的抗旱研究提供了更多的理论基础。
