兼容型maizeSNP384 标记筛选与玉米杂交种DNA 指纹图谱构建
2020-07-02田红丽杨扬王易红梅许理文张云龙葛建镕王凤格赵久然
田红丽杨 扬王 璐 王 蕊 易红梅 许理文 张云龙 葛建镕 王凤格 赵久然
北京市农林科学院玉米研究中心 / 玉米DNA 指纹及分子育种北京市重点实验室, 北京100097
玉米(Zea maysL.)是我国和全球第一大作物,是杂交良种应用最早最普及的作物, 在中国农业经济结构中具有重要作用。随着种业市场的发展, 截至2018年底, 我国已有玉米审定品种12,000 个以上,申请植物新品种权7000 件以上, 每年仍有5000 多品种组合在参试(http://www.zys.moa.gov.cn/gsgg/pzsdgs/, http://www.nybkjfzzx.cn/p_pzbh/pzbh.aspx)。与日俱增的品种数量在繁荣市场的同时, 也存在品种间同质化严重、套牌侵权泛滥、种质资源难以正常流动的问题, 这些问题严重制约着现代种业的发展。同时随着分子标记辅助育种技术的应用增加了派生品种(Establishing Essential Derivation, EDV)、近似品种的数量, 给品种确权鉴定带来新的挑战, 迫切需要增加标记密度进而准确评估品种之间的遗传背景。
SNP 标记技术被国际植物新品种保护联盟(International Union for the Protection of New Varieties of Plants, UPOV)推荐为农作物品种鉴定和指纹数据库的方法之一。该技术具有共显性, 检测内容为DNA 序列信息差异, 以二等位基因变异为主, 易实现数据整合共享, 位点在基因组上分布密度更高更均匀, SNP 位点在基因内区域分布较多, 增加了与功能基因相关的概率, 易实现高通量检测, 单个数据点成本低[1-2]。高通量分型平台的快速发展极大地推动了SNP 标记技术在农作物品种鉴定领域中的应用, 如位点高通量的芯片平台, 样本高通量的原位扫描平台KASP (Kompetitive Allele Specific PCR)、Taqman 技术, 以及位点和样本均高通量的靶向测序技术等。基于上述平台玉米等主要农作物开展了相关探索工作, Mammadov 等报道了玉米SNP 位点在上述平台的兼容性情况[3]; 大豆方面, 筛选了14 个SNP 标记区分599 份大豆种质资源材料[4]; 甘蓝方面, 筛选了50 个核心SNP 标记并构建了59 个品种的指纹图谱[5]; 辣椒方面, 筛选了40 个SNP 位点用于品种鉴定[6]; 中国陆地棉方面, 筛选了23 对核心SNP 位点用于品种鉴定[7]。
就芯片技术而言, 全球主要应用美国Illumina和Thermo Affymetrix 公司推出的GGGT、infinium、axiom 3 种平台; 前两者属于光纤微珠芯片, 后者为激光微刻芯片; 3 种技术在数据分析时均对采集的荧光信号进行标准化, 获得数据点的坐标值, 然后所有数据点被划分成3 簇分别代表2 种纯合、1 种杂合基因型。基于上述主流芯片平台, 报道了多款农作物SNP 芯片产品[8-11]。玉米为二倍体杂交作物, 具有重组率高、遗传多样性丰富以及较好的基因组学研究基础, 其SNP 位点开发、评估具有较好的研究基础[12-14]。同时多款SNP 芯片产品也被推出应用,如在全基因组关联分析、QTL 定位、分子育种中应用的maizeSNP50、maize55K 芯片; 在玉米种质资源遗传评估、分子育种中应用包含768、1536 个位点的芯片; 适于玉米品种DNA 指纹分析和分子育种的通用型SNP 芯片maizeSNP3072 等[15-19]。尽管有多种不同通量玉米SNP 芯片可供使用, 但是商业化的芯片评估材料以美国和欧洲为主, 定制芯片基本上是以分析应用于自交系、群体材料、种质资源为主;而玉米品种真实性鉴定和指纹数据库构建的样品类型以杂交种为主, 筛选位点需考虑杂交种的特性,因此并不能直接采用已报道的位点组合。
本研究基于已有研究基础, 利用代表性玉米自交系和杂交种材料, 筛选确定玉米分子鉴定位点组合maizeSNP384, 此384 个核心SNP 位点具有兼容多平台、高稳定性、高重复性、高品种区分能力; 基于核心位点建立了 335 个国家审定玉米品种SNP-DNA 指纹数据, 并进行了相关分析; 以期为玉米分子鉴定、指纹数据构建以及分子育种提供支撑。
1 材料与方法
1.1 供试材料
筛选30 套包含杂交种及其双亲的玉米样品作为SNP 位点试验效果评估材料(表1), 这30 份杂交种具有较大推广种植面积, 或者为区试组的对照品种。选取1984—2013年国家审定玉米品种335 个样品评估SNP 位点的多态性、品种区分能力, 同时建立标准指纹图谱; 样品具体信息参考《国家审定玉米品种SSR 指纹图谱》[20]。
1.2 总DNA 提取
选用混株方案制备供试样品, 每个样品混合30个以上单株, 采用改良CTAB 法提取总DNA[21]。用紫外分光光度计(Nanodrop 2000)测定DNA 浓度和质量, 工作液浓度统一调整为50 ng µL-1。
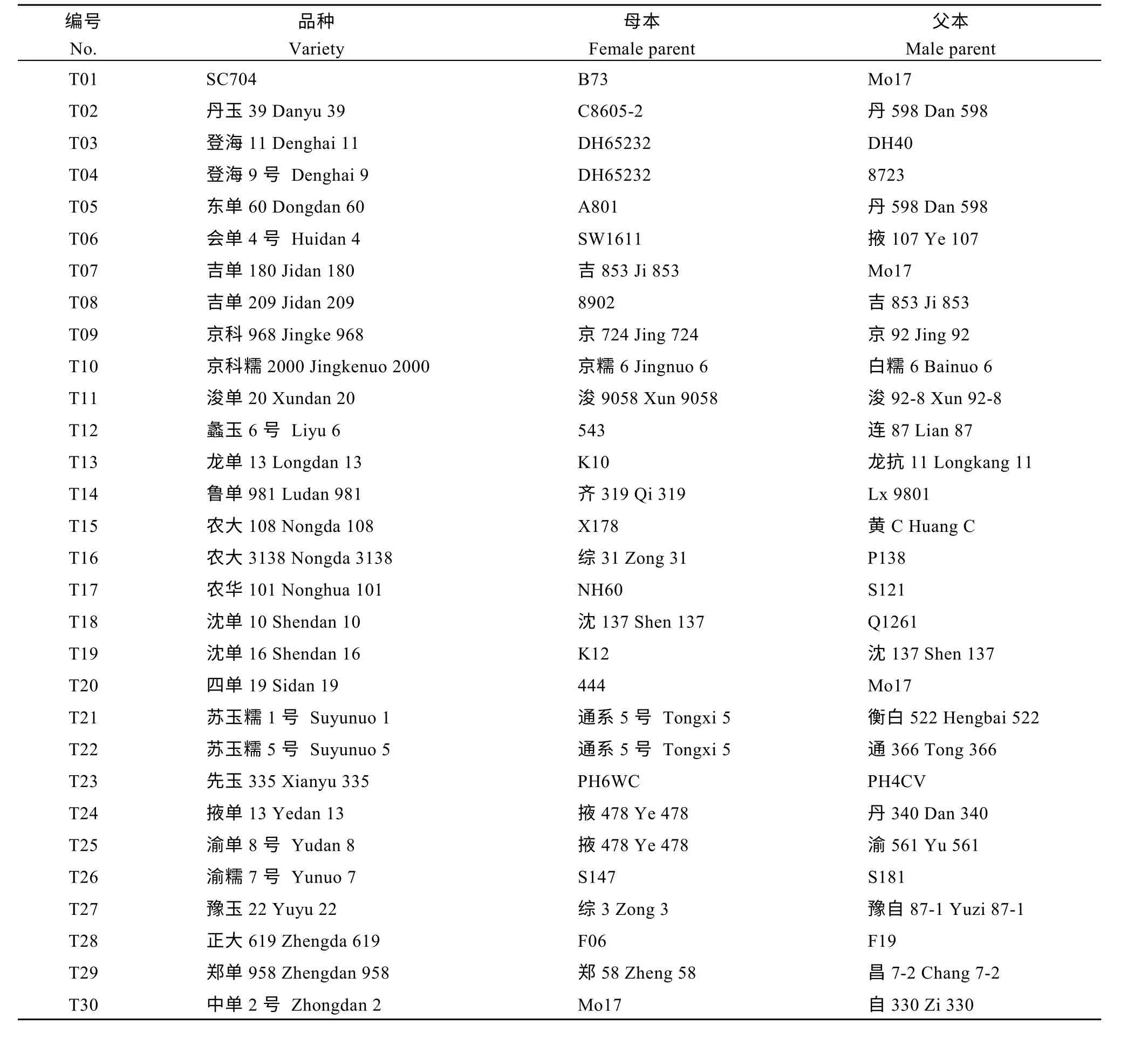
表1 用于评估SNP 位点的30 套杂交种及其双亲的样品信息Table 1 Sample information of 30 sets hybrids and their parents for SNPs evaluation
1.3 玉米分子鉴定核心SNP 位点组合的确定, 玉米杂交种SNP-DNA 指纹图谱构建
将已公布的玉米基因组3072 个SNP 位点[19], 在Infinium、GGGT、Axiom 不同平台(美国Illumina 和Thermo Affymetrix 公司)上进行平行试验, 评估获得兼容多平台、高质量的位点。基于Illumina GGGT芯片平台, 利用335 个玉米杂交种、3072 个位点, 采用iScan 芯片扫描仪进行荧光信号采集, 获得原始数据, 利用Genome Studio Genotyping Software v2011 软件分析, 获得基因型数据。同时利用335 个杂交种进一步评估试验效果, 结合多态性、品种区分能力和均匀分布的原则, 确定候选核心位点; 利用最优遗传算法[22]最终确定1 套适于玉米品种真实性鉴定和标准DNA 指纹数据构建的核心SNP 位点组合。同时抽取核心位点组合数据, 形成335 个玉米杂交种SNP-DNA 指纹图谱。
1.4 数据分析
利用python3 语言, 采用biopython 图形库绘制SNP 核心位点在染色体上的物理位置示意图(https://biopython.org/)。利用 SNP 比对统计工具(V1.0)(北京市农林科学院玉米研究中心, 软著登记号: 2018SR026743)分析核心位点的系列参数, 包括MAF (Minor Allele Frequency)、PIC (Polymorphism Information Content)和DP (Discrimination Power);DP 是指单个位点的品种识别能力, DP=1−∑(pi)2,pi是指每种基因型的频率[23]。
利用python3 语言, 采用matplotlib 图形库绘制玉米杂交种的 SNP-DNA 指纹图谱 (https://matplotlib.org/)。利用SNP 比对统计工具(V1.0)(软著登记号为2018SR026743)分析335 个杂交种的杂合率,品种两两成对比较差异位点分布, 两两比较的遗传相似系数值的分布图。遗传相似参数分别采用(1 −Nei1973)遗传距离(GD), 相同等位基因比值(GS),(1 − 差异位点比值)×100% (GL) 3 种方案。GS =(1−b/a)×100%, 其中a为比较总等位基因数目, 由于SNP 位点为二等位基因变异, 故a 为比较总位点数的2 倍,b为差异等位基因数目。GL 为1 − 差异位点数目/比较总位点数目。
利用SNP/INDEL 位点筛选工具(V1.0) (北京市农林科学院玉米研究中心, 软著登记号为2018SR 003573), 采用“基于遗传算法的植物品种真实性鉴定位点筛选方法”[22], 从本研究确定的384 个SNP核心位点中逐步抽取位点组合, 包含的位点数目从1 个增加到20 个, 分析其品种识别率的变化情况。每组位点抽取时主要依据位点和位点组合的品种区分能力, 在384 个位点中随机抽取。
2 结果与分析
2.1 适于玉米品种真实性鉴定和标准DNA 指纹数据构建的SNP 位点筛选
基于maizeSNP3072 位点组合, 根据试验效果、品种区分效率、均匀分布原则筛选确定1 套适于玉米品种真实鉴定和标准DNA 指纹数据构建的核心位点组合maizeSNP384[19]。具体筛选过程分为3 步:(1)基于30 套杂交种及其双亲样品, 利用Infinium、GGGT、Axiom 3 种不同芯片平台进行位点筛选。评估指标主要为, 在3 种芯片平台上均能成功设计探针序列, 评估分值≥0.70, 3 个平台之间数据完全一致; 数据获得率高于95%, MAF 值≥0.05; 获得候选SNP 位点为1212 个。(2)根据335 个杂交种的数据,进一步做试验质量和多态性评估。要求在不同芯片平台上3 种基因型簇AA、BB 和AB 之间具有明显的界限, 并且各自内聚(图1); 多态性指标为MAF≥0.2、PIC≥0.25、DP≥0.4; 获得候选SNP 位点为800个。(3)利用最优遗传算法, 结合在染色体上相对均匀分布的原则, 最终筛选确定了兼容多平台、高稳定性、高重复性、高品种区分能力的384 个核心SNP位点(附表1)。
2.2 maizeSNP384 位点组合的基本特征
将384 个SNP 位点的侧翼序列各35 bp 比对到B73 参考基因组(AGP-V3 版本)获得每个位点所在的染色体和物理位置信息。图2-A 展示了位点在玉米10 对染色体上的分布情况, 384 个位点基本均匀分布在长臂和短臂上, 在端粒和着丝粒区域分布极少,第2 染色体位点数目相对少。384 个核心位点全部分布在基因内区域, 其中52%分布在exon 区, 20%分布在3'UTR 区, 16%分布在promoter 区, 7%分布在5′UTR 区, 5%分布在intron 区(图2-B)。
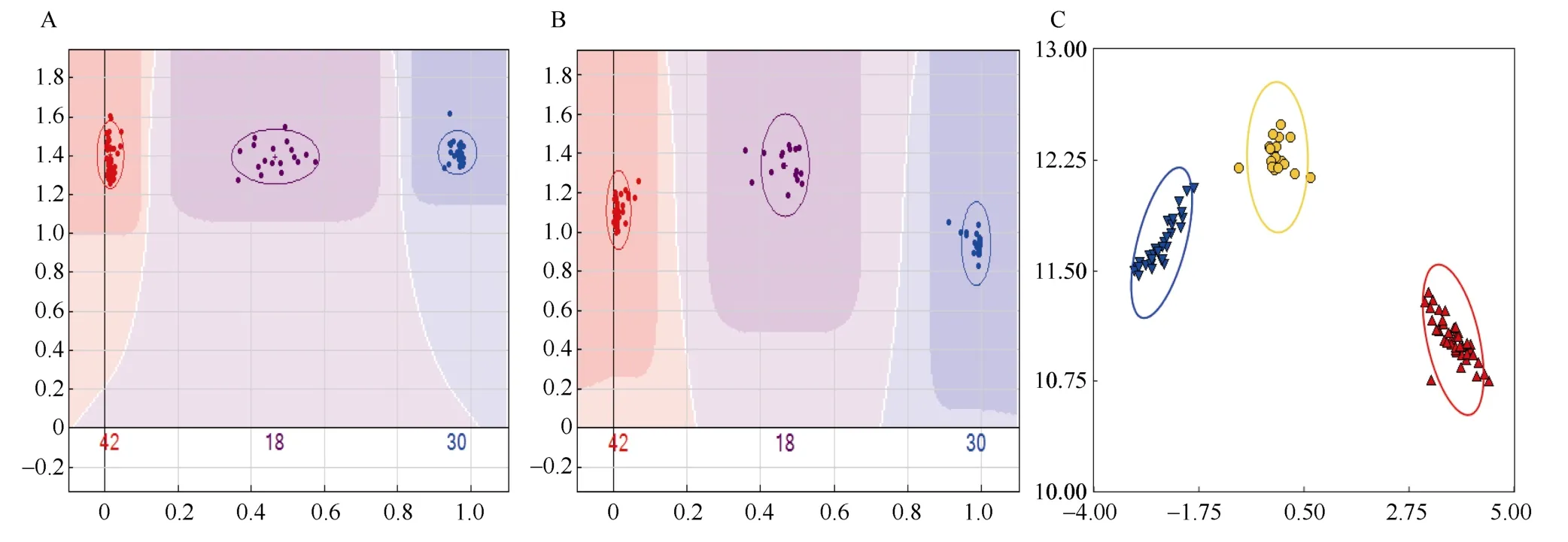
图1 SNP 位点(MG004)在3 种芯片平台上的分型图Fig. 1 Genotyping patterns of MG004 SNP locus on three different chip platforms

图2 maizeSNP384 位点组合的分布Fig. 2 Distribution of maizeSNP384 combinations
基于335 个玉米杂交种的数据评估SNP 位点的各遗传参数, 384 个位点显示了较好的多态性、品种区分能力(图3)。384 个核心SNP 位点的MAF、PIC、DP 值变异范围分别为 0.17~0.50、0.25~0.40、0.44~0.66, 平均值分别为0.39、0.36、0.60。384 个位点中88%的位点MAF 值高于0.30, 98%的位点PIC 值高于0.30, 98%的位点DP 值高于0.50 (图3,附表1)。
2.3 335 个国家审定玉米品种指纹图谱构建及分析
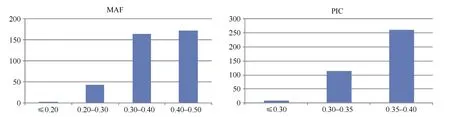
图3 384 个SNP 位点的MAF 和PIC 多态性指数变化分布图Fig. 3 Distribution of MAF and PIC values of 384 SNP loci
图4 为利用384 个SNP 标记组合构建了335 个玉米杂交种的基因型指纹图谱, 该图显示每个品种的基因型组合均不同, 即384 个SNP 位点能够有效区分所测试的杂交种。基于384 个SNP 位点的基因数据, 335 个国家审定玉米品种基因型数据获得率为97.33%; 样品杂合基因型(父母本为不同的纯合基因型情况下, 杂交种为杂合基因型)频率变异范围为0.17~0.63, 平均值为0.47, 97%的样品杂合基因型频率值介于0.35~0.60 之间(图5-A)。335 个玉米品种两两比较, 总共比较的对数有55,945 对; 结果显示差异位点数目范围为1~283 个, 差异位点数目多集中在100~270 个, 差异位点数目在20 个以内的有12对品种(图5-B)。基于384 个SNP 位点基因型数据,对335 个玉米杂交种进行遗传相似系数两两比较,GD (1 − Nei 遗传距离)、GS (相同等位基因比值)、GL (1 − 差异位点的比值) 3 种参数的值依次降低。GD 的变异范围为0.60~0.99, 平均值为0.77, 相似性数值多集中在0.65~0.90, GD≥0.98、0.95、0.90 者分别占比0.10%、0.38%、1.40%。GS 的变异范围为0.50~0.99, 平均值为 0.65, 相似性数值多集中在0.55~0.80, GS≥0.98、0.95、0.90 者分别占比0.03%、0.11%、0.35%。GL 的变异范围为0.22~0.99, 平均值为0.40, 相似性数值多集中在0.30~0.60, GL≥0.98、0.95、0.90 者分别占比0.02%、0.03%、0.09%(图5-C)。

图4 335 个玉米杂交种SNP-DNA 指纹总体展示图Fig. 4 The overall display of SNP-DNA fingerprints of 335 maize hybrids
2.4 针对于335 个国家审定玉米杂交种, 随着位点组合数目的变化, 品种区分效率变化情况
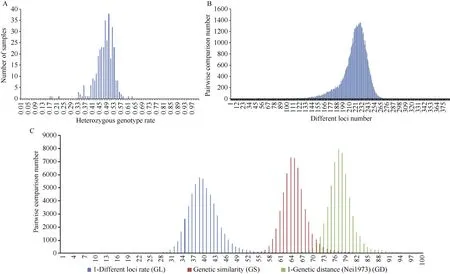
图5 335 个玉米品种的杂合基因型频率、遗传差异分析Fig. 5 Analysis of heterozygous genotype rate and genetic difference of 335 maize hybrid lines
基于384 个SNP 位点、335 个玉米杂交种的基因型数据, 采用“基于遗传算法的植物品种真实性鉴定位点筛选方法”, 从384 个位点中抽取位点组合,位点数目为从1 个增加到20 个, 分析每套位点组合的品种识别率变化曲线[22]; 横坐标为最优位点组合包含的位点数目, 纵坐标为品种识别率(图6)。在本研究中被比较的2 个品种如果有1 个位点的基因型不同则视为两者能够被识别。图6 显示, 2 个位点组合品种识别率为0.04, 3 个位点的识别率为0.10, 8 个位点的识别率超过0.90; 12 个位点或更多位点时品种识别率为0.99, 其数值趋于稳定; 最终抽取了20个位点, 能够识别所有品种; 20 个位点相对均匀分布在10 条染色体上, 每条染色体上均抽取到了位点,根据染色体的长度不同包含的位点数目亦不同。
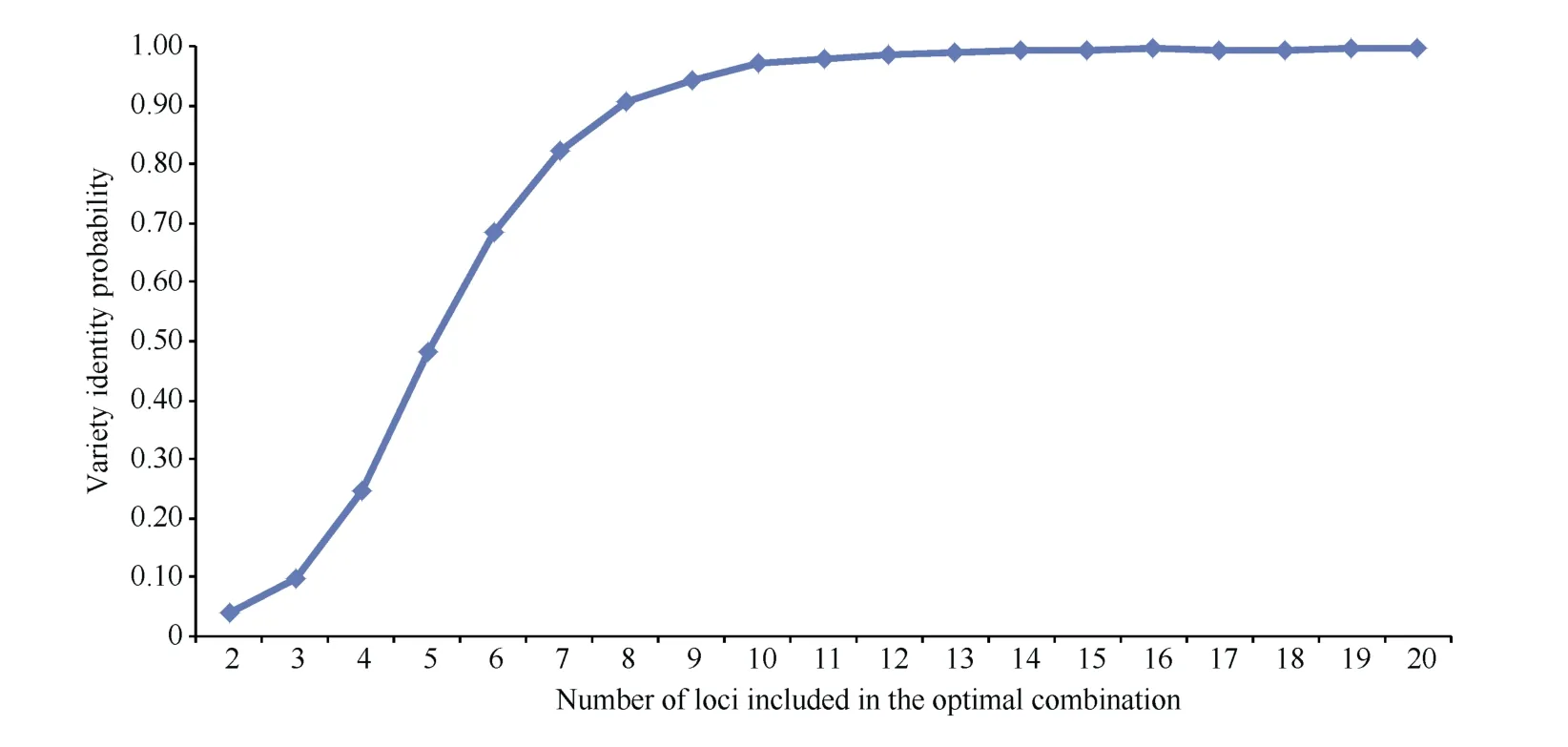
图6 基于最优位点组合的品种识别率变化曲线图Fig. 6 Variety identity probability curve based on the optimal loci combination
3 讨论
3.1 适于玉米品种分子鉴定的 SNP 标记特征,SNP-DNA 指纹数据表现形式
筛选一套适宜的核心标记位点, 确定指纹数据表现形式对于农作物品种标准DNA 指纹构建至关重要。农作物品种鉴定内容和目的不同, 筛选标记位点的标准亦不同。适于玉米品种分子鉴定的SNP标记组合需在品种间多态性高; 试验评估效果好,表现为数据信号值高, 基因型数据能够自动化统计;具有较好的重复性和稳定性; 兼容多平台; 在基因组上物理位置已知; 相对均匀分布, 引物位点间没有紧密连锁。
SNP 标记虽然以二等位基因变异为主, 其基因型数据只出现A/T/C/G 四种碱基, 但是存在相同位点在不同平台其等位基因的定义不同, DNA 双链中定义哪条链的不同, 2 个等位基因存在定义成A 或B的不同。因此如果不加以规范标准化, 不同平台之间, 相同平台不同的探针设计均会存在数据整合的问题。综上所述, 玉米品种SNP-DNA 指纹数据格式建议使用A/T/C/G 碱基, 不推荐采用其他编码转换的方式。为了不同平台、不同实验室、不同批次之间的数据整合, 必须提供至少2 个参照样品的指纹信息, 要求参照样品在每个位点上均为纯合基因型且无数据缺失, 参照样品之间具有较大的遗传差异。在玉米中, 建议选取不同杂优群的DH 系作为参照样品。
3.2 基于SNP 标记方法, 玉米品种鉴定最适位点数目分析
农作物品种鉴定最适位点数目一直是本领域关注的重点和难点, 它与鉴定目的、标记类型、检测材料等相关。一般情况下, 减少位点数目, 能够相对简便、快捷、稳定地反映品种之间的差异。但是如果位点数目太少则遗传距离评估结果不稳定, 尤其是对于亲缘关系较近的材料。这也反映出品种真实性鉴定和派生品种、近似品种鉴定对位点数目的要求不同。
真实性鉴定侧重考察品种间是否存在明显差异,鉴定的过程是“找差异”, 只要差异位点数目高于阈值即可判定结果。品种派生关系鉴定是基于已经认定具有特异性的、申请品种权保护的品种, 分析待测样品是独立的品种还是与已知品种存在派生关系;派生关系鉴定侧重考察品种间的遗传背景的相似程度, 鉴定的过程是“找相似”, 真实反映两个品种之间的相似程度。因此品种真实性鉴定和依赖性派生鉴定的最适位点数目级别不同, 前者相对少, 一般在几十至几百级别, 后者相对多, 一般在几百至几千级别[24-25]。
目前用于农作物品种鉴定标记以SSR 和SNP 为主。两种标记由于单个位点多态性的差异, 在品种鉴定时选用的标记数目亦不同。SSR 和SNP 两种标记位点组合多态性的倍数关系没有定论, 这与筛选位点的原则, 位点组合效果, 评估材料遗传背景等均有关系。ISF (International Seed Federation, 国际种子联盟)颁布的基于SNP 标记进行玉米派生品种鉴定的指南中选用了3072 个SNP 位点[26-27]。基于美国玉米自交系样品的分析, 305 个SNP 位点组合与150 个SSR 位点组合结果一致性最高, 此150 个SSR位点是ASTA (American Seed Trade Association, 美国种子贸易协会)发布的用于美国玉米品种鉴定的位点组合[28-29]。国内玉米品种鉴定SSR 标记法中公布了40 对SSR 引物, 这40 对SSR 引物已成熟应用于玉米品种鉴定[21]。本研究报道的384 个SNP 位点,从中抽取20 个位点, 能够区分335 个玉米审定品种;如果位点数目为200 个预计能够区分玉米已知品种的95%及以上, 与40 对SSR 引物区分效果相当; 但是如果应用于玉米派生、近似样品的遗传相似度分析, 需要进一步增加位点到数千个。
3.3 基于SNP 标记方法, 玉米品种鉴定分析参数和阈值的探讨
农作物品种鉴定分析参数、阈值与鉴定内容、位点数目、以及作物种类等相关。对于品种真实性鉴定, 分析参数一般为差异位点数目; 确定判定阈值的思路为首先基于真实性鉴定的核心位点组合建立已知品种的DNA 指纹数据库; 根据品种间差异位点分布, 品种内变异情况, 结合实际情况确定与预期相符的差异位点阈值。对于派生品种、近似品种鉴定, 分析参数一般为遗传相似度参数; 确定判定阈值的思路为首先基于全基因组均匀选取一套位点组合, 构建代表性样品的DNA 指纹数据库; 然后根据样品间的遗传相似度分布情况, 结合表型及系谱信息, 确定合适的遗传相似度阈值。
ISF 颁布的基于SSR 标记玉米派生品种鉴定指南中, 阈值设定为 82%~90%, 即遗传相似度低于或等于82%的判定为非派生品种, 即不同品种; 介于两者之间的为质疑区间, 需提供其他辅助材料进行判定; 高于90%的判定为派生品种[30]。ISF 颁布的基于SNP 标记玉米派生品种鉴定指南中, 阈值设定为91%和95%, 因为SNP 单位点区分效率低于SSR, 所以基于SNP 标记的阈值做了相应的提升[27]。国内玉米品种真实性鉴定SSR 标记法公布了40 对SSR 引物, 判定阈值为2 个位点, 差异位点百分比占据5%[21]。基于本研究报道的384 个位点对335个玉米品种进行遗传相似系数的两两比较, GD≥0.98、0.95、0.90 者分别占比0.10%、0.38%、1.40%。根据我国已知玉米品种实际情况和现行的SSR 行业标准, 基于SNP 标记遗传相似度阈值的设定建议高于ISF 颁布的标准, 具体阈值需要进一步深入研究。
3.4 兼容多平台的位点组合maizeSNP384 的应用前景
核心引物组合法概念早在2003年已经提出, 是指在染色体上均匀选取多态性高、稳定性高、重复性好、整体品种区分能力高的一组引物/位点组合;该方案的提出解决了农作物品种鉴定和数据整合共享的难题[24,31]。本研究报道的384 个SNP 位点为基于最优遗传算法, 结合相对均匀分布的原则, 筛选的一套兼容多平台、高稳定性、高重复性、高品种区分能力的位点组合。从中抽取了20 个位点作为一组最优位点组合, 能够区分所评估的335 个杂交种;如果抽取200 个位点预计能够区分玉米已知品种的90%及以上。故maizeSNP384 位点组合在未来基于SNP 标记的玉米品种分子鉴定中能够发挥重要作用,可作为筛选玉米品种真实性、特异性、纯度鉴定以及类群划分的候选位点组合, 为分子标记辅助育种中背景分析提供研究基础。
SNP 标记可以应用的高通量基因分型技术除了芯片平台之外, 还有样本高通量的原位扫描平台(英国LGC 公司KASP 技术, 美国Life 公司Taqman 技术), 以及位点和样本均高通量的靶向测序技术等。由于Illumina GGGT 芯片探针设计原理与KASP、Taqman 技术相似度极高, 有报道称GGGT 芯片与KASP、Taqman 技术的位点转换率高达 98%和95%[3]。本文中的384 个SNP 位点在illumina GGGT、infinium、AFFY axiom 芯片技术中均表现出了较好评估效果。如从384 个SNP 位点中选取几十或者上百个最优位点组合, 利用样本高通量的 KASP 或TaqMan 技术, 可以实现高效率、高灵活性、简便快捷、低成本的检测应用需求。
综上所述, 不管是位点高密度的芯片平台, 还是样本高通量的KASP、Taqman 技术平台, 亦或是位点样本高通量的测序平台, 本研究报道的384 个位点均能达到较高成功率, 在未来基于SNP 标记的玉米品种分子鉴定、分子育种中发挥重要作用。
4 结论
评估确定了玉米分子鉴定位点组合 maize SNP384, 此384 个核心SNP 位点具有兼容多平台、高稳定性、高重复性、高品种区分能力; 基于核心位点建立了335 个国家审定玉米品种SNP-DNA 指纹数据, 为玉米品种分子鉴定、指纹数据构建以及分子育种提供了关键数据支撑。附表 请见网络版: 1) 本刊网站http://zwxb. chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方 数 据 http://c.wanfangdata.com.cn/Periodicalzuowxb.aspx。
