广西涠洲岛底栖甲藻前沟藻属(Amphidinium)种类的形态学和系统发育学研究
2020-05-25江丽春李群吕颂辉
江丽春, 李群, 吕颂辉
海洋生物学
广西涠洲岛底栖甲藻前沟藻属()种类的形态学和系统发育学研究
江丽春, 李群, 吕颂辉
暨南大学生命科学技术学院, 水体富营养化与赤潮防治广东普通高校重点实验室, 广东 广州 510632
本文对采集自我国广西涠洲岛海域潮下带大型海藻及死珊瑚上的附着底栖甲藻样品进行了分析。利用毛细管单细胞分离方法成功分离并培养了多株前沟藻属种类, 运用光学显微镜、扫描电镜和分子生物学等技术, 对所获得的前沟藻株系进行形态学和系统发育分析, 鉴定出前沟藻属3个种, 分别为具盖前沟藻()、玛氏前沟藻()和强壮前沟藻(), 其中具盖前沟藻和玛氏前沟藻为我国新纪录种。分析比较了不同种类间形态和系统发育的差异, 发现3种前沟藻的形态特征大体与相应模式种报道的一致。本研究丰富了我国前沟藻种类多样性和系统发育进化信息, 同时将两个新记录种具盖前沟藻和玛氏前沟藻的已知分布范围拓展到了我国南部沿海水域。
涠洲岛; 前沟藻; 形态学; 系统发育
前沟藻属()隶属于甲藻纲(Dinophyceae), 裸甲藻目(Gymnodiniales) (Fensome et al, 1993)。该属形态特征为无甲板, 细胞小型到大型, 细胞形状有球形、陀螺形或双锥形; 细胞背腹或左右略扁, 腹面观中上锥部呈月牙形或三角形偏向左侧, 下锥部椭圆形或锥形; 横沟在前部, 具或不具位移, 纵沟开始于纵向鞭毛孔往细胞底部走向, 横纵沟间有腹侧脊。
前沟藻分布广泛, 大多分布于热带和亚热带海域, 为兼性底栖和浮游的甲藻类群(Murray et al, 2002; Jørgensen et al, 2004a)。前沟藻附生在大型海藻、海草和珊瑚等底栖生境, 是海洋有害底栖甲藻类群之一, 常伴随有毒的冈比亚藻()(Rhodes et al, 2010)。某些前沟藻种类会产生有害的环脂类细胞毒素或溶血性毒素(Yasumoto et al, 1987; Maranda et al, 1996), 如强壮前沟藻产生的环脂类细胞毒素会加剧西加鱼中毒, 导致鱼类死亡, 该种藻细胞裂解物还会抑制海胆发育(Baig et al, 2006)。部分前沟藻属种类也会产生一些有益的物质, 如具盖前沟藻会产生两性霉素(amphidinols)、两性霉素内酯(amphidinolides)和卡比内酯(caribenolide)等物质(Kobayashi et al, 1991; Satake et al, 1991; Bauer et al, 1995a), 两性霉素内酯显示出潜在的抗肿瘤活性, 且已用于抗白血病药物的研发(Bauer et al, 1994, 1995b)。
前沟藻属分类的早期研究是通过对野外采集的样品进行固定, 再用光学显微镜观察, 由于很多形态结构特征模糊不清(Daugbjerg et al, 2000), 研究者经常通过横沟截距与纵沟偏距、上下锥部的相对大小等形态特征进行种类区分(Claparède, 1858; Kofoid et al, 1921)。随着研究的深入, 人们认识到有些种类形态特征重叠, 不易区分, 因此基于形态学特征并结合分子系统发育分析对前沟藻属进行分类是近年常用的新方法。在最初的系统分类中, 前沟藻属种类被分在裸甲藻属(), 随着分子系统发育学分析方法的发展与应用, 系统发育研究不支持裸甲藻科(Gymnodiniaceae)的单一性(Daugbjerg et al, 2000), 也不支持前沟藻属与裸甲藻科其他属的近缘关系(Murray et al, 2004)。目前前沟藻属种类繁多, 形态多样性和物种多样性丰富, 生活方式多样, 包括异养、光合自养和混合营养等物种(Daugbjerg et al, 2000)。该属许多种类形态特征相似, 同种变形也较多, 导致很多种类在形态上分类模糊, 如玛氏前沟藻(Biecheler, 1952)和强壮前沟藻(Hulburt, 1957)的细胞大小及形状基本一致, 且都拥有位于细胞中间偏左前部位的蛋白核, 使得两者有时被误认为是同一个种。大小、代谢运动和囊泡形成等特征(Kofoid et al, 1921; Murray et al, 2004), 但代谢运动和囊泡形成在后来报道的()。由于该属许多种形态特征相似, 导致分类困难, 因此有必要利用不同的细胞形态特征进行分类(Jørgensen et al, 2004a, b)。
前沟藻分布广, 生存能力强, 在世界各地被频繁报道, 而在我国目前仅于海南岛和胶州湾水域发现了强壮前沟藻(韩笑天等, 2004)。我国研究者对前沟藻的相关研究主要集中在该属代表性种类强壮前沟藻的生理生化及代谢产物方面的研究(张珍妮等, 2016; Kong et al, 2016), 而对于该属的分类学和系统学研究还未见报道。本研究采集了我国广西北部湾涠洲岛的底栖藻类样品, 分离筛选疑似前沟藻藻株, 通过显微和超微结构的形态学观察, 同时基于rDNA分子序列分析, 对前沟藻种类进行分类鉴定, 并通过构建分子进化树, 阐明种类之间的系统发育关系。
1 材料与方法
1.1 样品采集与藻株分离培养
于2017年4月在广西涠洲岛(21°N, 109°E)选择三个采样点(图1), 检测并记录这三个采样点的光照强度、水温和海水盐度等信息。于潮间带或潜水至潮下带采集大型藻、海草和死珊瑚等底栖甲藻附着基质, 放入1L的塑料瓶中, 摇晃塑料瓶使藻细胞与附着基质脱离, 用直径2mm的筛网(10目, 大连晋丰筛网有限公司, 中国大连)粗过滤, 取过滤后的样品于倒置显微镜下用毛细管挑取单个藻细胞于96孔培养板(Corning, New York USA)中培养, 培养条件: 温度25±1℃, 盐度30‰, L1培养基, 光照周期为12L : 12D, 光强为150μmol·m2·s–1。单细胞藻株建立后转移到同等条件的三角瓶中培养。
1.2 实验方法
1.2.1 藻类形态学观察
1.2.1.1 光学显微镜观察
取对数生长期藻细胞于正置显微镜(Olympus BX 61, Tokyo, Japan)下观察, 使用QImaging Retiga 4000R数码相机(QImaging, Surrey, British Columbia, Canada)进行藻细胞形态采集, 使用Image-Pro Plus 6.0图像采集测量新保存样品的细胞大小, 统计细胞的长度、宽度等形态学数据。

图1 广西涠洲岛底栖甲藻采样站位图
W1为滴水丹屏、W2为鳄鱼湾、W3为五彩滩; 地图来自Surfer软件底图
Fig. 1 The sampling stations of benthic dinoflagellate in Weizhou Island, Guangxi
1.2.1.2 荧光显微镜观察
取对数生长期活体细胞于荧光显微镜(Olympus BX 61, Tokyo, Japan)下利用细胞的自发荧光观察细胞色素体形态、数量和分布, 同时取对数生长期活体细胞于离心管中, 加入戊二醛固定(终浓度为1%)后, 通过4', 6-二脒基-2-苯基吲哚(DAPI)染色, 避光2h之后在荧光显微镜下观察细胞核的形状、大小及位置。
1.2.1.3 扫描电子显微镜观察
取对数生长期活体细胞于离心管, 用锇酸(终浓度为2%)固定一小时, 再用注射器将固定的藻液过滤至3μm的PC滤膜(Whatman, Little Chalfont, UK)上, 并依次用梯度为100%、80%、60%、40%、20%的海水和纯水缓慢冲洗以除去盐与固定剂, 再用10%、20%、30%、40%、50%、60%、70%、80%、90%和100%的丙酮脱水, 每个梯度洗涤和脱水用液体积为4ml, 时间为4min。将脱水后的细胞进行CO2临界点干燥(Leica Microsystems, Mannheim, Germany), 取出干燥后带细胞的PC滤膜将其贴在带导电胶的铜台上, 经离子溅射仪(SCD 005, Bal-Tec, or Leica EM SCD 500, Leica, Wetzlar, Germany)喷金, 最后在扫描电子显微镜下(Carl Zeiss Inc., Oberkochen, Germany)观察细胞形态结构并拍照。
1.2.2 DNA提取, PCR扩增和测序
离心收集对数生长期细胞藻液, 使用DNA 提取试剂盒(Takara MiniBEST Universal Genomic DNA Extraction Kit), 按照使用说明对前沟藻核糖体DNA进行提取。于0.2mLPCR管中, 添加如下反应体系: 0.2μL DNA、25μL TaqPCR MasterMix (Zoman Biotechnology, Beijing, China)、正向引物与反向引物各1μL、超纯水22.81μL。反应过程按照试剂盒说明进行D1-D3(800~1000bp)序列扩增。最后将扩增获得的序列产物送至生工生物工程股份有限公司进行测序。
1.2.3 系统发育分析
将测序所得DNA正反向序列用ContigCExpress软件拼接查看并进行编辑。将编辑好的序列放入基因库(GenBank)进行比对并选择下载构建系统发育树所需的序列。使用BioEdit v7.0.5 (Hall, 1999)软件将目标藻株提取的序列与从GenBank中下载的序列进行对比及整理切除首位多余的碱基, 利用软件构建Bayses树及利用生物信息学网站(https://www. phylo.org/portal2/login!input.action%3bjsessionid=54E5409E35A57687C1CF8AB75AEFBBFF)构建ML树。Bayses树构建时将处理好的序列进行格式转换及保存, 将转换好的NEX格式文件用PAUP v4b10读取并运行Mrmodel, 再运行Mrmodel命令选择最佳模型, 结合最优模型运行MrBayes v3.1.2 (Ronquist et al, 2003)进行系统发育分析构建发育树, 最后用MEGA v5.0 (Tamura et al, 2011)选择树形及TreeView (Page, 1996)查看并添加置信值。ML树构建时, 将BioEdit整理好的序列保存为phy格式, 在CIPRES网站创建新文件夹并上传phy文件, 然后进行任务创建, 选择“RAXML-HPC2 ON XSEDE”运行工具, 参数设置时先在“Maximum Hours to Run”处选择2h, 填写输出数据的名字, 在数据类型中选择“nucleotide”, 将“estimate proportion of invariable sites”选为“yes”, 打开“advancade parameters”并选择“GTRGAMMA”, 然后选中“conduct Multiparametric Bootstrapping”,选择“print brance lengths”, 在“Bootstrap iterations”中输入1000, 最后保存参数。程序运行时会自动挑选出似然率最大的拓扑结构作为最优树, 最后下载选好的树形放入TreeView查看并添加置信值。
2 结果
本研究共分离鉴定出广西涠洲岛海域前沟藻属3个种, 分别为具盖前沟藻()玛氏前沟藻()和强壮前沟藻(), 其中具盖前沟藻和玛氏前沟藻为我国新记录种。
2.1 3种前沟藻的形态特征
2.1.1 具盖前沟藻()形态特征
细胞呈长卵圆形, 中后部最宽, 细胞长33~48μm (40±8.0μm,=30), 宽22~37μm (29±6.2μm,=30)。长宽比为1.23~1.61 (图2)。上锥部较小, 腹面观中上锥部类似直角三角形。横沟起源处到细胞上锥部顶端距离占0.1个细胞的长度, 上锥部右顶端角度接近90°, 左边形成30°的角(图2a、e)。下锥部左侧底部往外稍凸, 右侧沿着底部呈弧形, 总体呈卵圆形(图2)。横鞭毛从横沟左侧鞭毛孔伸出, 沿着横沟往背侧缠绕到横沟右侧(图2a)。纵沟位于细胞下锥部偏底端1/3处(图2a)。两个鞭毛孔处有一条腹脊(图2a)。叶绿体呈红色长条形, 从中间向四周辐射排列(图2g、h), 细胞中未见蛋白核。细胞核位于细胞下锥后部, 呈圆形或卵圆形, 直径为10~16μm (图2g)。橙黄色的眼点位于核上方(图2e), 直径为5~7μm。横鞭毛与纵鞭毛孔处各有一个泡(图2e), 直径2μm左右。

图2 具盖前沟藻扫描电镜图(a—d)与光镜图(e—l)
a. 腹面观, 箭头指向横鞭毛孔; b. 背面观; c. 底面观; d. 二分裂细胞; e. 腹面观, 黑色箭头指向囊泡, 白色箭头指向眼点; f. 黑色箭头指向眼点; g. 细胞核; h. 叶绿体形状分布; i—k. 不同形状的具盖前沟藻; l. 二分裂细胞。各图比例尺为10μm
Fig. 2 Scanning electron micrographs (a-d) and light micrographs (e-l) of. a) Ventral view; b) dorsal view; c) antapical view; d) binary division cell; e) ventral view, with black arrow pointing to the pusule and white arrow pointing to the eyespot; f) black arrow pointing to the eyespot; g) nucleus; h. distribution and shape of chloroplast; (i-k)with different shapes; l) binary division cell. Scale bars in a–l: 10 μm
2.1.2 玛氏前沟藻()形态特征
细胞呈卵圆形, 背腹扁平。细胞长10~16μm (13±3.0μm,=30), 宽7~12μm (9±2.5μm,=30), 长宽比为1.28~1.88 (图3)。腹面观中上锥部呈月牙形, 顶部弯向左侧(图3e、f)。横沟起源处到细胞上锥部顶端距离占0.2个细胞的长度。下锥部呈卵圆形(图3e、f)。细胞表面由多边形体鳞组成(图3d)。横沟从左侧开始往背面环绕到右侧往下延伸(图3b、c、e、f、g)。横鞭毛产生于横沟左侧鞭毛孔(图3c)。纵沟起源于细胞腹面中部, 也是纵鞭毛伸出的部位, 纵沟往细胞底部延伸(图3c, e)。两个鞭毛孔之间有一条腹脊(图3c)。细胞核直径5~6μm, 位于下锥部底部(图3i、j)。蛋白核直径2~4μm, 位于细胞左前部(图3f、g)。不同生长阶段叶绿体形状不一样, 生长指数期呈带小孔的网状, 分散于整个细胞, 随着细胞培育时间增长逐渐成密度低的稀疏网状结构(图3k—m)。细胞分裂是在运动的细胞中以二分裂的形式进行的(图3h)。

图3 玛氏前沟藻扫描电镜图(a—d)和光镜图(e—m)
a. 背面观; b. 侧面观; c. 腹面观, 箭头指向横鞭毛孔; d. 细胞表面; e、f. 腹面观, 箭头指向蛋白核; g. 背面观, 箭头指向蛋白核; h. 二分裂细胞; i、j. 细胞核; k—m. 叶绿体形态分布; a—c、e—m比例尺为5μm, d为2μm
Fig. 3 Scanning electron micrographs (a-d) and light micrographs (e-m) of. a) Dorsal view; b) side view; c) ventral view, with arrow pointing to the pore of transverse flagellum; d) surface of cell; (e-f) ventral view, with arrow pointing to the pyrenoid; g) dorsal view, with arrow pointing to the pyrenoid; h) binary division cell; (i-j) nucleus; (k-m) distribution and shape of chloroplast. Scale bars in a-c and e-m: 5 μm; d: 2 μm
2.1.3 强壮前沟藻()形态特征
细胞呈卵圆形, 背侧扁平, 腹侧卵圆形且底部左偏(图4a、b、e、f)。细胞长12~19μm (15±3.0μm,=30), 宽9~14μm (11±2.8μm,=30), 长宽比1.18~1.67。腹面观察中上锥部呈月牙形, 顶部向左侧弯曲(图4a、e)。横沟起源处到细胞上锥部顶端距离占0.2个细胞的长度(图4a)。腹面观察下锥部左侧呈直线形, 右侧底部呈弧线形(图4a、e)。纵沟起始于细胞腹面中间位置。腹侧脊粗短连接两个鞭毛孔。细胞核圆形或卵圆形, 位于细胞下锥部后部(图4g)。环状淀粉鞘蛋白核位于细胞中间或稍偏前部位(图4e、f), 直径约3μm。叶绿体呈网状结构, 遍布整个细胞, 密度较高(图4h)。3个前沟藻种的形态特征比较见表1。

表1 3个前沟藻种形态特征比较
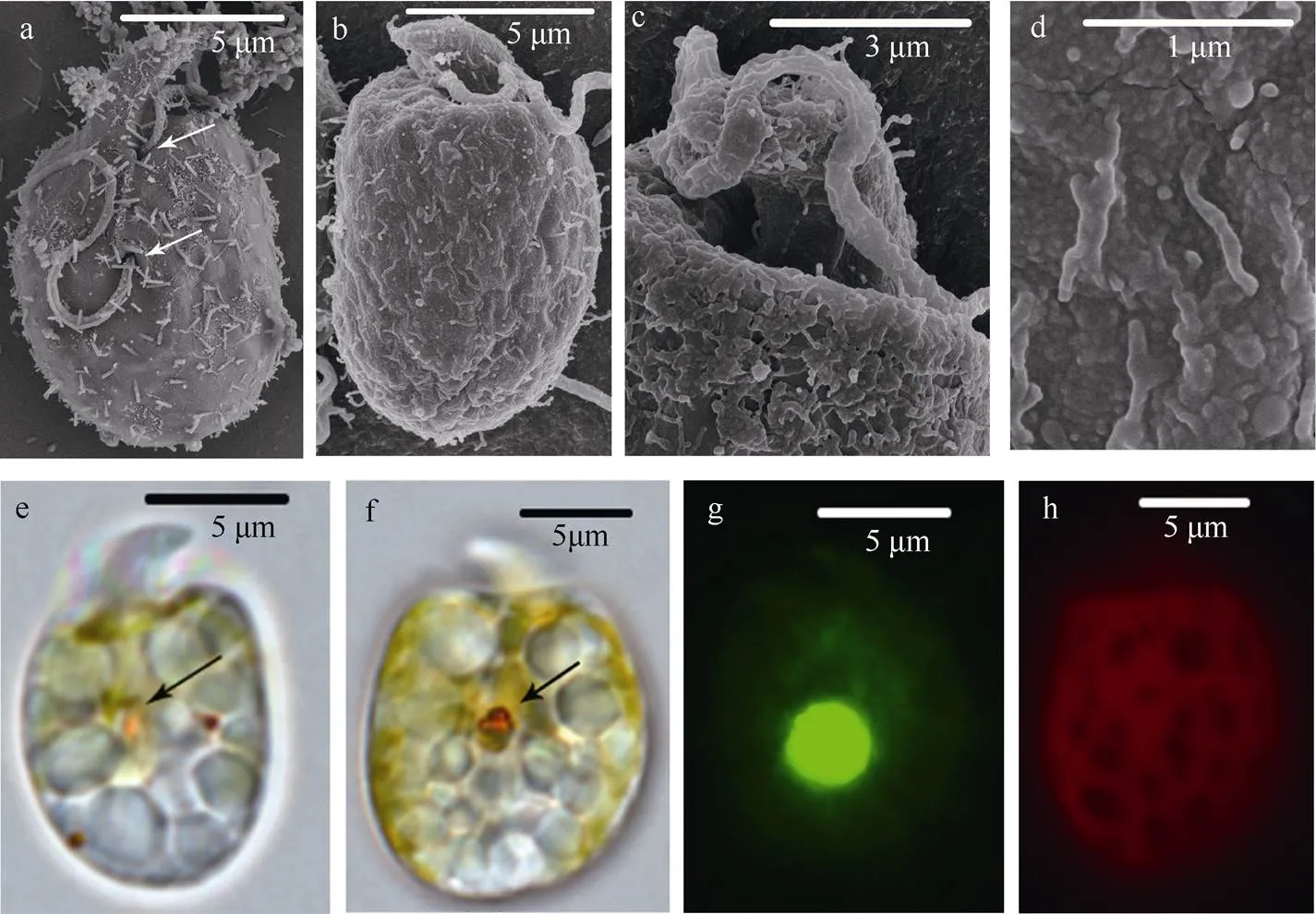
图4 强壮前沟藻扫面电镜图(a—d)和光镜图(e—h)
a. 腹面观, 箭头指向鞭毛孔; b. 背面观; c. 上锥部背面观; d. 细胞表面; e. 腹面观, 箭头指向蛋白核; f. 背面观, 箭头指向蛋白核; g. 细胞核; h. 叶绿体形态分布
Fig. 4 Scanning electron micrographs (a-d) and light micrographs (e-h) of. a) Ventral view, with arrow pointing to the pore of flagellum; b) dorsal view; c) the dorsal view of epicone; d) surface of cell; e) ventral view, with arrow pointing to the pyrenoid; f) dorsal view, with arrow pointing to the pyrenoid; g) nucleus; h) the distribution and shape of chloroplast
2.2 大亚基核糖体序列分析和系统发育
对采集的前沟藻藻株进行LSU rDNA D1-D3序列测序分析, 与基因库前沟藻属中的46个已发表序列进行比对, 选取血红哈卡藻()作为外类参考, 利用Mega和MrBayes建立系统发育树(图5)。本研究获得的具盖前沟藻WZD225株、玛氏前沟藻3WZD7株和强壮前沟藻3WZD8株系在NCBI上对应的序列号分别为MN622810、MN622808和MN622809。在ML和Bayes系统发育树中个别节点难以确定, 但主干拓扑结构基本一致。本研究中3个株系的前沟藻序列与其他46个发表的藻株序列形成4个大的进化分支, 分支节点处的数字分别代表Bayes/ML树中此节点的置信值。进化枝A与B、C、D分支间的置信值为1.00/-, 且在进化枝A的8个藻株序列间具有强的置信值(>1.00/85); 进化枝B是具盖前沟藻种, 由已报道的四个株AY455674、AY460590、AY460591、AY460592和本研究的WZD225株系组成, 置信值高达100/1.00, MN622810与序列AY460591、AY460592中度置信值为0.80/54, 且均相差19个碱基对, 相似度为98%, 与AY460590的置信值仅为0.78/-; 进化枝C由不同的前沟藻种组成, 并被分为两个亚类, 第一个亚类由、和三个种组成, 且此三个种具有高的置信值(1.00/75), 第二个亚类进一步分成两个谱系, 3WZD7在第二个谱系上属于玛氏前沟藻, 其与JQ394808相差2个碱基对, 相似度为99%, 置信值为1.00/100, 但其与另外两株美国玛氏前沟藻株在不同的姐妹进化枝上; 进化枝D具有高强度的置信值(1.00/99), 分成三个亚类, 其中一个亚类包含强壮前沟藻株系AY460586和株系KY697957, 本研究中3WZD8在第二个亚类中形成独立的姐妹分支, 但其与另外5个强壮前沟藻株系AY460578、AY460583、AY460580、AY460584和AY460579支持度为1.00/100, 相似度均达到99%。
3 讨论
前沟藻种之间的形态学特征相似, 易引起分类学混淆(Jørgensen et al, 2004a, b; Murray et al, 2004)。研究表明, 在实验室观察有限数量的细胞时, 不同种难以通过一些重要的形态学特征(如细胞形状、大小、叶绿体结构和泡等)区分开(Fukuyo, 1981)。具盖前沟藻最初因形态特征不确定及与相似物种难以区分而被命名为具盖前沟藻复合体(Murray et al, 2004)。具盖前沟藻涠洲岛株WZD225的形态特征与具盖前沟藻模式种基本相似, 两个鞭毛插入位置各有一个泡, 无环状淀粉鞘蛋白核, 略微的差异是WZD225株横沟到上锥部顶端距离与细胞长度的比率为0.1, 与Murray等 (2004)报道的模式种为0.3有所不同。而本属其他种如、、和玛氏前沟藻都具有环状淀粉鞘蛋白核, 另外具盖前沟藻与的不同除了细胞形状外, 具盖前沟藻细胞分裂不在囊泡中进行; 具盖前沟藻与的细胞形状也不同,细胞右侧凸起下部尖端, 呈驼背形状(Murray et al, 2004)。
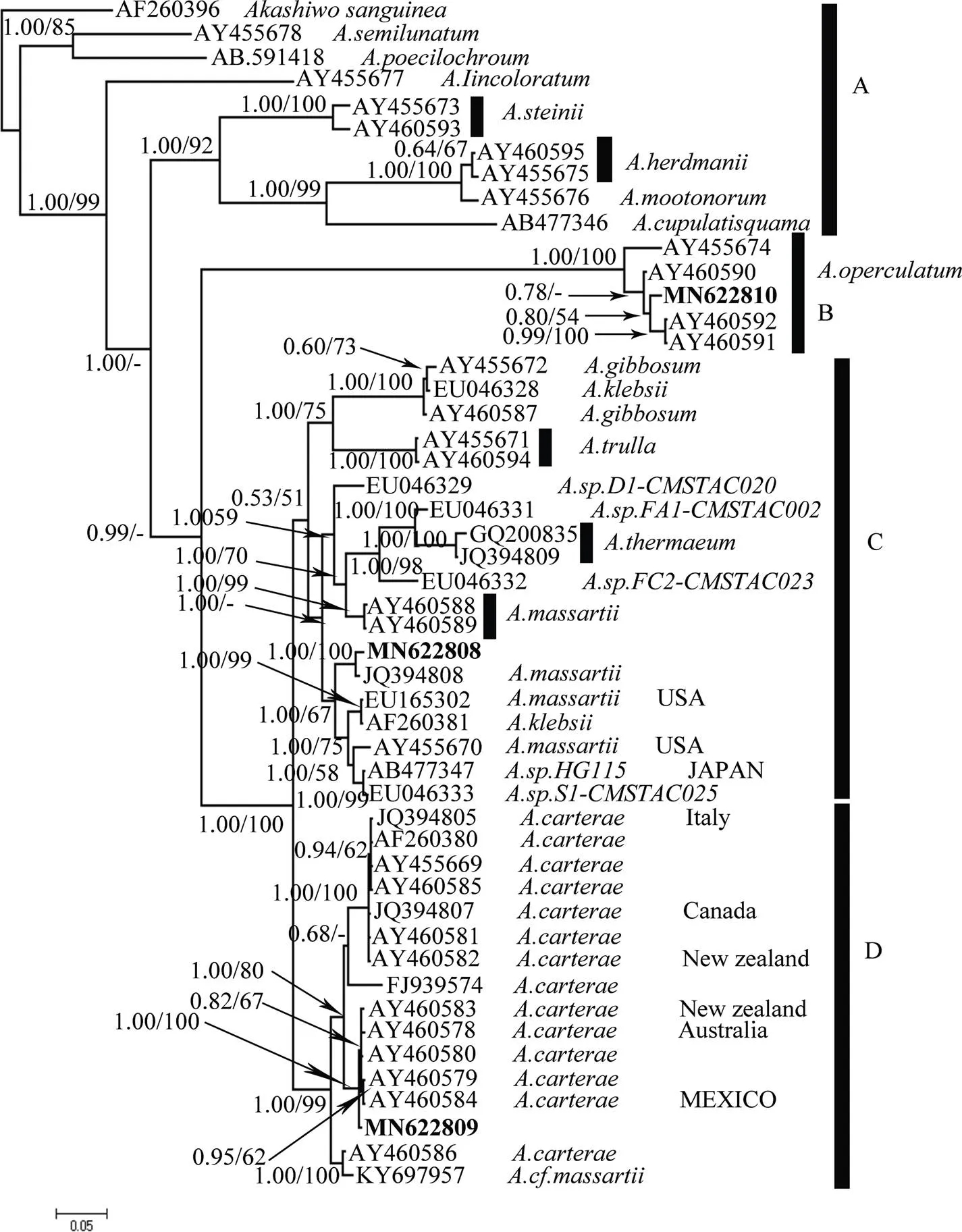
图5 前沟藻属LSU rDNA(D1-D3)的贝叶斯系统发育树
节点数字代表贝叶斯分析法的后验概率(左)和最大似然法分析的置信值(右), 50%以下的置信值已省略
Fig. 5 Bayesian inference (BI) tree of the LSU rDNA (D1-D3 regions) ofspecies. The number on the branch is the Posterior probabilities for BI analysis (left) and bootstrap values for maximum likelihood analysis (right). Support values below 50% have been omitted
玛氏前沟藻涠洲岛株3WZD7与模式种形态相似(Murray et al, 2004, 2012)。玛氏前沟3WZD7株叶绿体不同生长阶段形状会稍有不同, Murray等 (2012)和Lee等(2013)均证实了这一点, 指数生长期叶绿体多数呈带小孔的网状, 分散于整个细胞, 随着细胞培养时间增长其逐渐成密度低的稀疏网状结构。玛氏前沟3WZD7株系细胞表面及鞭毛区均有体鳞存在, 而有研究报道sp. HG114、HG115、玛氏前沟CS-259、、spp.和表面也有体鳞, 但其鞭毛处体鳞尚未报道(Morrill et al, 1981; Watanabe et al, 1990; Sekida et al, 2003; Tamura et al,2009; Murray et al, 2012)。在韩国济州岛报道的玛氏前沟细胞表面也存在体鳞(Lee et al, 2013), 所以藻株细胞表面及鞭毛体鳞的发现也可能是一个重要的分类特征。
强壮前沟藻首次发现时被误认为是(Carter, 1937), 后来对其细胞大小和叶绿体形状进行研究后, 将其分为新的种强壮前沟藻 (Hulburt, 1957)。Dodge等 (1968)对强壮前沟藻进行了详细的超微结构研究, 发现其与和的大小范围略有重叠, 并与玛氏前沟藻基本一致。强壮前沟藻与区别在于形状和分裂方式, 强壮前沟藻不通过分裂囊泡进行分裂。强壮前沟藻与区别是强壮前沟藻上锥部相对较长(Murray et al, 2004)。本研究分离的强壮前沟藻株与玛氏前沟藻株细胞大小和形状上基本一致, 区分这两个种的形态特征依据是叶绿体的形状, 在玛氏前沟藻中不同生长阶段叶绿体形状会稍有不同, 而在强壮前沟藻中叶绿体分布于细胞内呈稀疏网状, 这种结构在强壮前沟藻的所有基因型和生长阶段均一致。另外在玛氏前沟藻细胞表面和鞭毛上存在体鳞, 且这一特征在Murray等 (2012)和Lee等(2013)的文献中均有描述, 而强壮前沟藻暂未发现文献报道其体表与鞭毛区有体鳞存在。
前沟藻的分类虽结合了形态学、分子系统发育和进化等信息来开展(Vila et al, 2001; Garcés et al, 2006), 但仍存在一些问题, 像个别种如在不同的生命阶段细胞形态变化较大, 可从心形到驼峰形不等(Murray et al, 2004), 这一形态特征变化导致该种鉴定的困难, 而本研究的三个种, 具盖前沟藻、强壮前沟藻和玛氏前沟藻, 在不同生命阶段细胞表面形态特征基本稳定(Murray et al, 2004, 2012), 故在种类鉴定时均选择生长状态良好的细胞作为研究对象。另外由于前沟藻属为无甲板类的裸甲藻, 在其囊泡中缺乏厚纤维素板(Truby, 1997), 故在扫描电子显微镜拍摄的前处理如样品固定、脱水等过程中细胞极易产生形变, 从而导致某些细胞特征难以观察或丢失(Reñé et al, 2015)。为解决形态鉴定时样品处理对前沟藻形态观察带来的困难, 本研究对Botes等人的方法进行了修改, 相比Botes等人将固定的样品用聚-L-赖氨酸粘附到玻璃片上, 本研究是将锇酸固定的样品直接过滤到PC滤膜上, 并发现在PC滤膜上的细胞表面更干净, 粘附的杂质更少(Botes et al, 2002)。另外在样品脱水处理过程中, 本研究对比了梯度丙酮与梯度乙醇的脱水效果, 发现乙醇脱水后的样品皱缩较严重, 而丙酮脱水后的样品皱缩不明显, 特别是在玛氏前沟藻的鉴定时, 丙酮脱水后的样品能更清楚的显示细胞体鳞结构。
具盖前沟藻因形态特征变异性导致其在命名上一直存有争议, 分子遗传数据的使用有助于解决这种形态特征上难以进行种类鉴定的问题(Jørgensen et al, 2004a)。本研究中具盖前沟藻在系统发育树上相比其他前沟藻种, 能形成明显的具有高置信值的系统进化枝, 与Murray等 (2004)关于该种在系统进化树位置的研究结果一致。另外本研究获得的具盖前沟藻种WZD225株具有独特的rDNA序列, 其在系统进化树上形成独立的分支。没有已知的前沟藻物种或藻株具有与WZD225株完全相同的rDNA序列, 其与亲缘关系近的藻株序列AY460591、AY460592均相差19个碱基对, 相似度为98%, 与AY460590相差12个碱基对。序列间的差异也是导致WZD225株系与模式种存在形态差异的原因。强壮前沟藻和玛氏前沟藻形态特征相似, 除了依靠细胞表面超微结构区分外(Murray et al, 2004), 还有一个重要的手段是系统发育分析, 强壮前沟藻和玛氏前沟藻在LSU rDNA D1-D3系统发育树上均表现出高遗传多样性, 且两个种明显在不同的进化枝上。强壮前沟藻种内形成4个进化亚枝与Lee等 (2013)和Murray等 (2004, 2012)关于该种系统进化研究结果一致。强壮前沟藻进化枝上的序列包含本研究获得的3WZD8藻株序列和来自澳大利亚、墨西哥、新西兰、加拿大和意大利已发表的序列(Murray et al, 2004), 形态比较并未发现该进化枝上强壮前沟藻种内形态特征的明显差异, 而系统进化树上4个进化亚枝的形成表明该种可能存在基因型差异。Karafas等(2017)关于玛氏前沟藻系统发育研究结果表明, 玛氏前沟藻物种遗传多样性高于强壮前沟藻, 而本研究鉴定出的玛氏前沟藻藻株序列与已发表的相关序列间遗传进化的差异与Karafas的研究结果相一致。
前沟藻属为世界性广布种(Murray et al, 2004, 2012)。在本研究之前, 世界多个国家的不同海域已报道了具盖前沟藻、玛氏前沟藻和强壮前沟藻的存在(Murray et al, 2004, 2012; Lee et al, 2013)。本研究对我国亚热带广西涠洲岛海域强壮前沟藻和两个新记录种具盖前沟藻、玛氏前沟藻的报道扩增了该属的全球分布范围, 填补了我国前沟藻属分布的空缺, 为后续进一步丰富我国前沟藻属种类多样性和分子生物学信息奠定了基础。
韩笑天, 颜天, 邹景忠, 等, 2004. 强壮前沟藻(Hulburt)形态特征及其生长特性研究[J]. 海洋与湖沼, 35(3): 279–283. HAN XIAOTIAN, YAN TIAN, ZOU JINGZHONG, et al, 2004. Morphological features and growth characteristics of the dinoflagellateHulburt[J]. Oceanologia et Limnologia Sinica, 35(3): 279–283 (in Chinese with English abstract).
张珍妮, 姜玥璐, 李曼璐, 等, 2016. 强壮前沟藻(Hulbert)在多重逆境条件下的生长和生理响应[J]. 海洋与湖沼, 47(5): 982–989. ZHANG ZHENNI, JIANG YUELU, LI MANLU, et al, 2016. The growth and physiological changes ofHulbert under environmental stresses[J]. Oceanologia et Limnologia Sinica, 47(5): 982–989 (in Chinese with English abstract).
BAIG H S, SAIFULLAH S M, DAR A, 2006. Occurrence and toxicity ofHulburt in the North Arabian Sea[J]. Harmful Algae, 5(2): 133–140.
BAUER I, MARANDA L, SHIMIZU Y, et al, 1994. The structures of amphidinolide B isomers: strongly cytotoxic macrolides produced by a free-swimming dinoflagellate,sp[J]. Journal of the American Chemical Society, 116(6): 2657–2658.
BAUER I, MARANDA L, YOUNG K A, et al, 1995a. Isolation and structure of Caribenolide I, a highly potent antitumor macrolide from a cultured free-swimming Caribbean dinoflagellate,sp. S1-36-5[J]. The Journal of Organic Chemistry, 60(4): 1084–1086.
BAUER I, MARANDA L, YOUNG K A, et al, 1995b. The isolation and structures of unusual 1, 4-polyketides from the dinoflagellate,sp.[J]. Tetrahedron Letters, 36(7): 991–994.
BIECHELER B, 1952. Recherches sur les Peridiniens[J]. Bulletin Biologique de la France et de la Belgique, 36 (Suppl.): 1–149.
BOTES L, PRICE B, WALDRON M, et al, 2002. A simple and rapid scanning electron microscope preparative technique for delicate “Gymnodinioid” dinoflagellates[J]. Microscopy Research and Technique, 59(2): 128–130.
CARTER N, 1937. New or interesting algae from brackish water[J]. Archives für Protistenkunde, 90: 1–68.
CLAPARÈDE R É, LACHMANN J, 1859. Études sur les infusoires et les rhizopodes[J]. Mémoires de l'Institut National Genevois, 6: 261–482.
DAUGBJERG N, HANSEN G, LARSEN J, et al, 2000. Phylogeny of some of the major genera of dinoflagellates based on ultrastructure and partial LSU rDNA sequence data, including the erection of three new genera of unarmoured dinoflagellates[J]. Phycologia, 39(4): 302–317.
DODGE J D, CRAWFORD R M, 1968. Fine structure of the dinoflagellateHulbert[J]. Protistologica, 4: 231–242.
DOLAPSAKIS N P, ECONOMOU-AMILLI A, 2009. A new marine species of(Dinophyceae) from Thermaikos Gulf, Greece[J]. Acta Protozoologica, 48(2): 153–170.
FENSOME R A, TAYLOR F J R, NORRIS G, et al, 1993. A classification of living and fossil dinoflagellates[J]. Micropaleontology, Special Publication, 7: 1–351.
FUKUYO Y, 1981. Taxonomical study on benthic dinoflagellates collected in coral reefs[J]. Nippon Suisan Gakkaishi, 47(8): 967–978.
GARCÉS E, FERNANDEZ M, PENNA A, et al, 2006. Characterization of NW Mediterraneanspp. (Dinophyceae) strains using morphological, molecular, chemical, and physiological methodologies[J]. Journal of Phycology, 42(5): 1096–1112.
HALL T A, 1999. BioEdit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 41(41): 95–98.
HULBURT E M, 1957. The taxonomy of unarmored Dinophyceae of shallow embayments on Cape Cod, Massachusetts[J]. The Biological Bulletin, 112(2): 196–219.
JØRGENSEN M F, MURRAY S, DAUGBJERG N, 2004a.revisited. I. redefinition of(Dinophyceae) based on cladistic and molecular phylogenetic analyses[J]. Journal of Phycology, 40(2): 351–365.
JØRGENSEN M F, MURRAY S, DAUGBJERG N, 2004b. A new genus of athecate interstitial dinoflagellates,gen. nov., previously encompassed withinlato: inferred from light and electron microscopy and phylogenetic analyses of partial large subunit ribosomal DNA sequences[J]. Phycological Research, 52(3): 284–299.
KARAFAS S, TENG S T, LEAW C P, et al, 2017. An evaluation of the genus(Dinophyceae) combining evidence from morphology, phylogenetics, and toxin production, with the introduction of six novel species[J]. Harmful Algae, 68: 128–151.
KOBAYASHI J, SHIGEMORI H, ISHIBASHI M, et al, 1991. Amphidinolides G and H: new potent cytotoxic macrolides from the cultured symbiotic dinoflagellatesp[J]. The Journal of OrganicChemistry, 56(17): 5221–5224.
KOFOID C A, SWEZY O, 1921. The free-living unarmored dinoflagellata[M]. California: University of California Press.
KONG XIANYU, HAN XIURONG, GAO MIN, et al, 2016. Antialgal and antilarval activities of bioactive compounds extracted from the marine dinoflagellate[J]. Journal of Ocean University of China, 15(6): 1014–1020.
LEE K H, JEONG H J, PARK K, et al, 2013. Morphology and molecular characterization of the epiphytic dinoflagellate, isolated from the temperate waters off Jeju Island, Korea[J]. Algae, 28(3): 213–231.
MARANDA L, SHIMIZU Y, 1996.var. nov.(Dinophyceae), a free-swimming marine species producing cytotoxic metabolites[J]. Journal of Phycology, 32(5): 873–879.
MORRILL L C, LOEBLICH III A R, 1981. A survey for body scales in dinoflagellates and a revision ofand(Pyrrhophyta)[J]. Journal of Plankton Research, 3(1): 53–65.
MURRAY S A, GARBY T, HOPPENRATH M, et al, 2012. Genetic diversity, morphological uniformity and polyketide production in dinoflagellates (, Dinoflagellata)[J]. PLoS One, 7(6): e38253.
MURRAY S, JØRGENSEN M F, DAUGBJERG N, et al, 2004.revisited. II. Resolving species boundaries in thespecies complex (Dinophyceae), including the descriptions ofsp. nov. and. comb. nov.[J]. Journal of Phycology, 40(2): 366–382.
MURRAY S, PATTERSON D J, 2002. The benthic dinoflagellate genusin south-eastern Australian waters, including three new species[J]. European Journal of Phycology, 37(2): 279–298.
PAGE R D M. 1996. TreeView: an application to display phylogenetic trees on personal computers[J]. Computer Applications in the Biosciences, 12(4): 357–358.
REÑÉ A, CAMP J, GARCÉS E, 2015. Diversity and phylogeny of(Dinophyceae) from the NW Mediterranean Sea revealed by a morphological and molecular approach[J]. Protist, 166(2): 234–263.
RHODES L L, SMITH K F, MUNDAY R, et al, 2010. Toxic dinoflagellates (Dinophyceae) from Rarotonga, Cook Islands[J]. Toxicon, 56(5): 751–758.
RONQUIST F, HUELSENBECK J P, 2003. MrBayes 3: Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 19(12): 1572–1574.
SATAKE M, MURATA M, YASUMOTO T, et al, 1991. Amphidinol, a polyhydroxy-polyene antifungal agent with an unprecedented structure, from a marine dinoflagellate,[J]. Journal of the American Chemical Society, 113(26): 9859–9861.
SEKIDA S, OKUDA K, KATSUMATA K, et al, 2003. A novel type of body scale found in two strains ofspecies (Dinopbyceae)[J]. Phycologia, 42(6): 661–666.
TAMURA K, PETERSON D, PETERSON N, et al, 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 28(10): 2731–2739.
TAMURA M, TAKANO Y, HORIGUCHI T, 2009. Discovery of a novel type of body scale in the marine dinoflagellate,sp. nov. (Dinophyceae)[J]. Phycological Research, 57(4): 304–312.
TRUBY E W, 1997. Preparation of single-celled marine dinoflagellates for electron microscopy[J]. Microscopy Research and Technique, 36(4): 337–340.
VILA M, CAMP J, GARCÉS E, et al, 2001. High resolution spatio-temporal detection of potentially harmful dinoflagellates in confined waters of the NW Mediterranean[J]. Journal of Plankton Research, 23(5): 497–514.
WATANABE M M, SUDA S, INOUYA I, et al, 1990.gen. et sp. nov. (Gymnodinaiales, Dinophyta), a green dinoflagellate with a chlorophyll-containing and-containing endosymbiont[J]. Journal of Phycology, 26(4): 741–751.
YASUMOTO T, SEINO N, MURAKAMI Y, et al, 1987. Toxins produced by benthic dinoflagellates[J]. The Biological Bulletin, 172(1): 128–131.
Morphology and Phylogenetics study on the species of(Gymnodinials, Dinophyceae) from Weizhou Island, Guangxi
JIANG Lichun, LI Qun, LÜ Songhui
College of Life Science and Technology, Jinan University, Key Laboratory of Aquatic Eutrophication and Control of Harmful Algae Blooms of Guangdong Higher Education Institutes, Guangzhou 510632, China
In this study, strains offrom seaweeds and dead corals around Weizhou Island, Guangxi, China were isolated by single cell capillary pipette and cultured. The morphological features and phylogenetic analysis were determined based on light microscopy, scanning electron microscopy and molecular biology techiniques. Threespecies were identified, namely,,and, among whichandwere new records in China. The differences in morphology and phylogeny between different species were analyzed and compared. It was found that the morphological characteristics of the threespecies were significantly consistent with model species. This study adds information to the diversity and phylogenetic evolution ofspecies in China, and extented the distribution of two newspecies to the coastal waters of southern China.
Weizhou Island;; morphology; phylogeny
Q949.24;
A
1009-5470(2020)03-0106-10
10.11978/2019077
http://www.jto.ac.cn
2019-08-24;
2019-11-26。
林强编辑
国家自然科学基金(41576162、41876173)
江丽春(1992—), 女, 江西省九江市人, 硕士研究生, 从事海洋底栖甲藻分类学
吕颂辉。E-mail: lusonghui1963@163.com
2019-08-24;
2019-11-26.
Editor: LIN Qiang
National Nature Science Foundation of China (41576162, 41876173)
LÜ Songhui. E-mail: lusonghui1963@163.com
