HIV-1整合酶抑制剂的3D-QSAR研究
2019-12-06仝建波
仝建波, 曹 旭
(陕西科技大学 化学与化工学院, 西安 710021)
1 引 言
获得性免疫缺陷综合症(Acquired immune deficiency syndrome, AIDS), 即艾滋病病毒, 是造成人类免疫缺陷病毒(Human immunodeficiency virus HIV)的一种致命疾病, HIV(分为HIV-1和HIV-2型)属逆转录病毒的一种通过破坏人体的T淋巴细胞(CD4)、感染单核细胞和巨噬细胞, 然后经过吸附、融合过程进人宿主细胞, 进而阻断细胞免疫和体液免疫过程, 可导致机体免疫功能部分或全部丧失, 继而发生机会性感染肿瘤等[1]. HIV病毒密码是由逆转录酶、蛋白酶和整合酶3种关键酶来表达[2], 近几年来抗HIV/AIDS药物的研制主要是针对抑制上述关键酶进行的, 研制这些酶的抑制剂成为治疗艾滋病的一种有效手段[3].
香豆素衍生物可以作为一种抗艾滋病药物, 对于病毒逆转录酶表现出较高的抑制活性, 如果通过结构优化使得活性提高, 则香豆素衍生物将会成为一种高效的抗艾滋病药物(Qantitative structure activity relationship, QSAR[4])是基于配体的计算机辅助药物设计的
主要方法之一, 具有代表性的方法有CoMFA[5]与CoMSIA[6]. 本实验采用第二代CoMFA方法—Topomer CoMFA对26种二芳基苯胺衍生物进行了3D-QSAR的研究, 运用这些研究结果, 进行分子对接, 在理论上验证所构建的模型具有较高的可信性, 为实验工作者合成该系列新药物提供理论参考.
2 实验部分
2.1 易位体比较分子场
Topomer CoMFA是CoMFA与Topomer的联合技术, 能够在短时间内准确、快速构建3D-QSAR模型[7]. 采用Topomer技术将整个配体分子切割成两个或两个以上的小片段, 所切割形成的小片段会自动生成三维构象的碎片, 碎片根据一定的经验规则进行调整, 生成Topomer模型. 计算每个分子片段周围的立体场和静电场参数, 作为自变量, 以IC的负对数(pIC50[8])为建模响应值, 其中IC50值为HIV-1病毒感染细胞半数抑制浓度, 用偏最小二乘回归析方法[9], 寻找这些三维结分构的特征信息与化合物活性的联系, 从而实现模型拟合.
2.2 数据来源与分子结构构建
从文献[10]中搜集了具有确定活性的26个香豆素衍生物(见表1)作为研究对象, 其化学结构见图1, 活性标度为pIC50(-logIC50). 从文献中列举的一系列化合物中随机选择20个化合物作为训练集(Training set)用于建立模型, 剩余6个分子作为测试集(Test set)用来验证模型的外部预测能力. 利用SYBYL2.0-X软件包中的Sketch Molecule模块绘制出化合物的结构, 以分子动力学程序Minimize对所有化合物进行能量优化, 得到其最低能量构象. 优化过程中采用Tripos力场[11]、Powell能量梯度和加载Gasteiger-Huckel电荷[12], 迭代系数设置为1000次, 能量收敛限定设为0.005 kcal/mol, 其余参数采用SYBYL2.0-X的默认值.
2.3 分子对接
采用SYBYL-X2.0中的Surflex-Dock[13]进行分子对接, 对接所使用的蛋白酶晶体结构来源于RSCB数据库, 晶体结构的PDBID为:3L68, 对接模式为Surflex-Dock, 在对接前需对蛋白进行处理, 从复合物中提取配体, 删除不需要的配体及所有水分子, 给蛋白加氢加电荷, 设定距离配体分子5 A范围内的所有氨基酸残基为活性口袋进行叠合, 然后将活性最高的19号化合物与蛋白晶体进行对接. 通常用Total-Score、Crash和Polar打分来评价对接结果[14]. Total-Score为总的打分函数, 表示受体与配体的亲和能力, 打分越高越好. Crash绝对值越接近零, 表示配体与受体对接时的不适当程度越小. Polar为极性函数得分, 当结合位点位于分子表面时, 打分越高越好;当结合位点位于分子内部时, 打分越低越好[15].
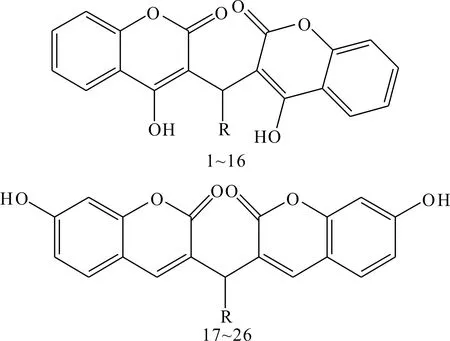
图1 化合物的基本骨架Fig. 1 Basic skeleton of the compound
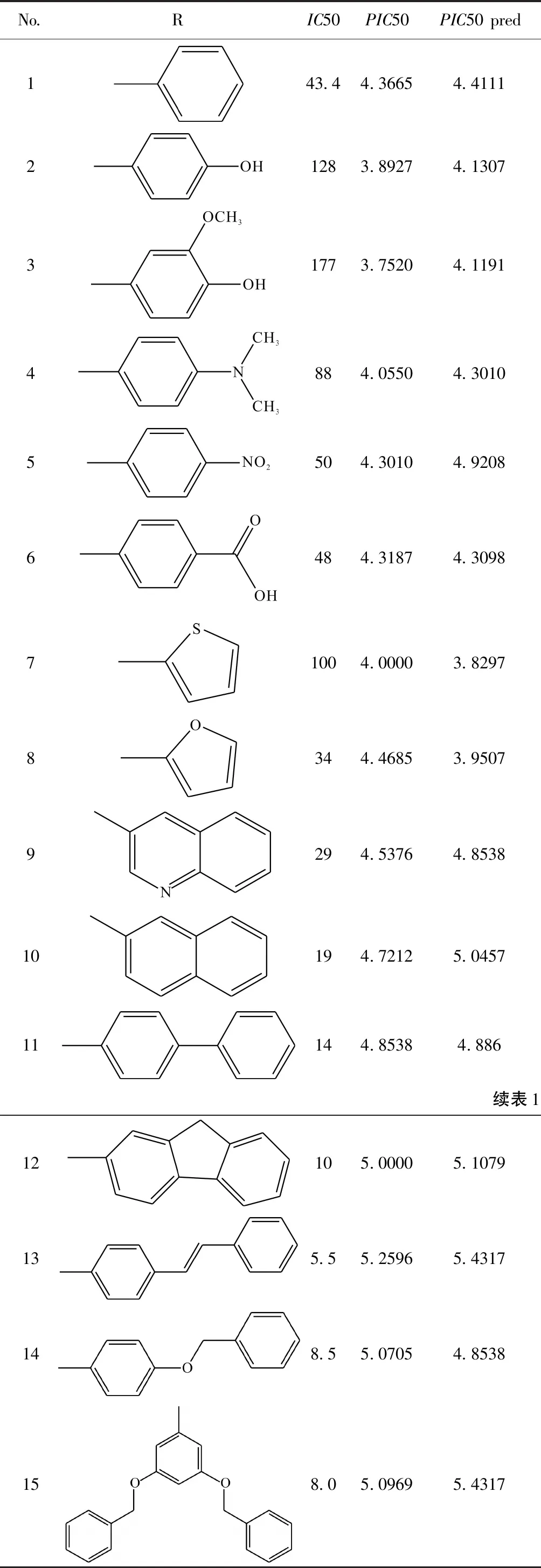
No.RIC50PIC50PIC50pred143.44.36654.411121283.89274.130731773.75204.11914884.05504.30105504.30104.92086484.31874.309871004.00003.82978344.46853.95079294.53764.853810194.72125.045711144.85384.886续表112105.00005.1079135.55.25965.4317148.55.07054.8538158.05.09695.4317
3 结果与讨论
3.1 Topomer CoMFA模型的统计结果及预测能力
通过Topomer CoMFA建立的3D-QSAR模型中, 模型的交叉验证相关系数(q2)和非交叉验证相关系数(r2)分别为0.666和0.973. 图2为训练集和测试集化合物的实验值和预测值的关系图, 从图中可以看出所有样本均匀地分布在45°直线附近, 证明所建模型具有好的可靠性及预测能力.
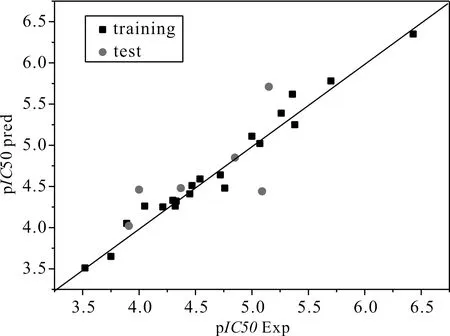
图2 香豆素衍生物活性实验值与预测值的关系图Fig. 2 Relationship between experimental values and predicted values of coumarin derivatives
3.2 3D-QSAR分析
图3为以19号分子为模板的Topomer CoMFA模型立体场和静电场的三维等势图, 其中图3A和C分别为Ra, Rb的立体场三维等势图, 图3B和D分别为Ra, Rb的静电场三维等势图, 图3A和C中绿色区域表示引入大体积的基团有利于分子抑制活性, 而黄色区域代表引进小体积的基团有利于化合物抑制活性增加;图3B和D中蓝色区域表示引入带正电荷的基团有利于分子抑制活性, 而红色区域代表引进带负电荷的基团有利于化合物抑制活性增加. 由图3A立体场等势图中可观察到在R1集团的切割点处有绿色区域, 图3C立体场等势图中可观察到在R2取代位点上也有绿色区域, 表明在这2个区域中, 将大分子基团引于此可以增加分子的活性. 如将10, 11, 12号分子的R基团分别以相应的基团取代了对应分子R基团上取代基, 其活性有明显提高, 分别由10号分子的4.7212活性值到11号分子的活性值4.8538, 到12号分子的活性值5.0000. 17, 18, 19号分子的R基团分别以相应的基团取代了对应分子R基团上取代基, 其活性有明显提高, 活性值分别由4.3344提高到了4.7644, 6.4317. 又如将24, 25, 26号分子的R基团分别以相应的基团取代了对应分子R基团上取代基, 其活性有明显提高, 活性值也相应的由3.9172提高到了4.4433,3.5228. 图3D静电场等势图可知, 在临近切割处环绕着一块蓝色等势域, 表明若此区域有正电性基团作用, 可增加化合物活性. 如8号分子的R取代基取代了7号分子的R取代基, 生物活性由4.000提高至4.4685.
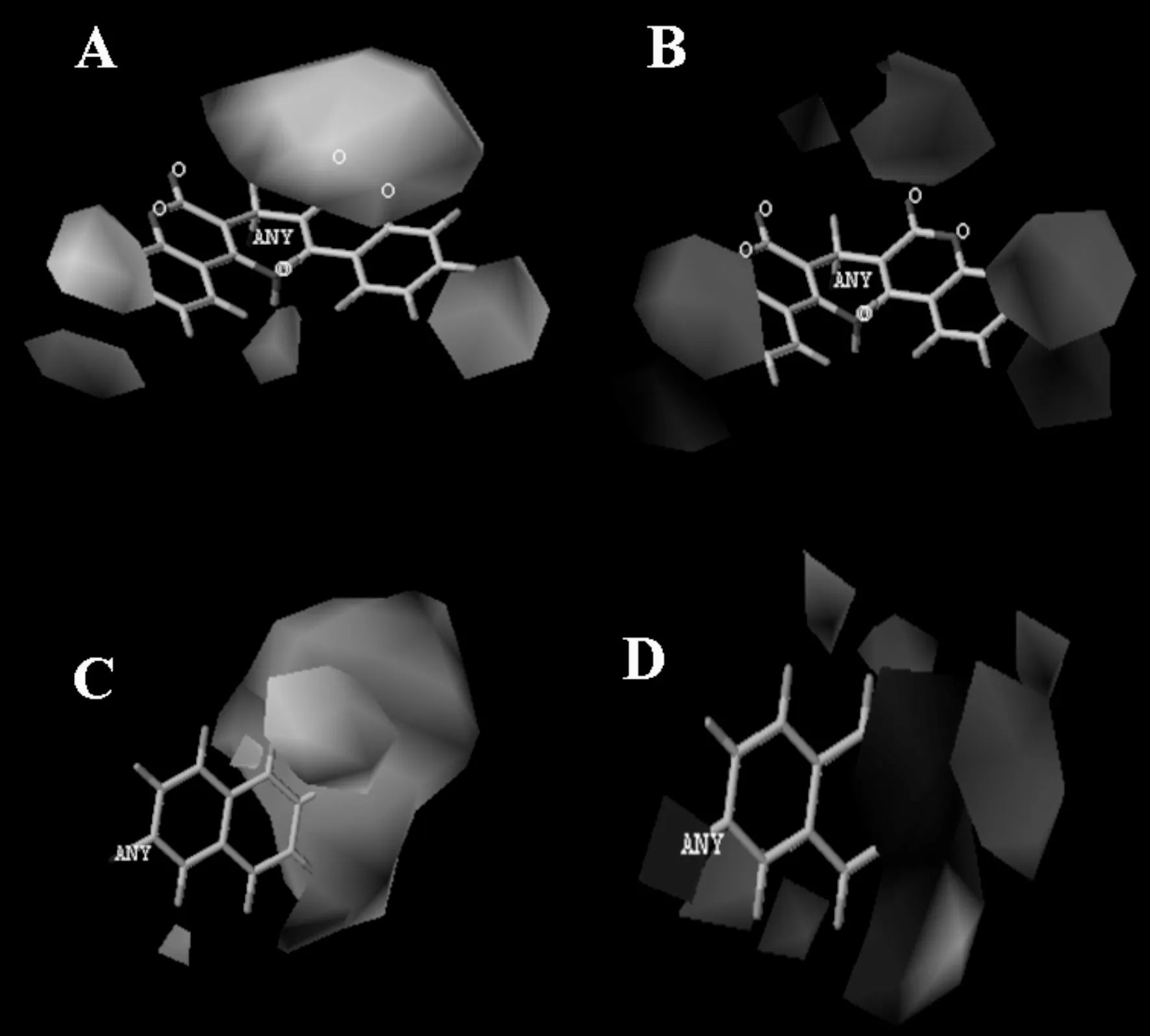
图3 Topomer CoMFA立体静电场三维等势图: (A)Ra立体等势图, (B)Ra静电等势图, (C)Rb立体等势图, (D)Rb静电等势图.Fig. 3 Contuor map of Topomer CoMFA steric and electrostatic fields: (A) steric contour map of Ra, (B) electrostatic contour map of Ra, (C) steric contour map of Rb, (D) electrostatic contour map of Rb.
3.3 分子对接
图4为分子对接的结果, 棒状表示配体, 线状表示氨基酸残基, 黄色虚线表示氢键给体[16]. 从图4B可见, 19号分子配体与晶体结构中的主要氨基酸残基共形成了3个氢键, 与A/HSI178、A/HIS181和A/HIS182形成氢键相互作用, 总打分函数(Total-Score)、配体对接进受体时的不适当程度(Crash)和极性打分函数(Polar)分别为4.8731、-2.6273和4.4605. 从图C可见, 13号分子配体与晶体结构中的主要氨基酸残基共形成了3个氢键, 与A/LYS106、A/SER25和A/ARG231形成氢键相互作用, 总打分函数(Total-Score)、配体对接进受体时的不适当程度(Crash)和极性打分函数(Polar)分别为13.3250、-1.6761和11.103. 从图4D可见, 15号分子配体与晶体结构中的主要氨基酸残基共形成了2个氢键, 与A/ARG326、A/SEP25形成氢键相互作用, 总打分函数(Total-Score)、配体对接进受体时的不适当程度(Crash)和极性打分函数(Polar)分别为7.0543、-2.4593和0.7880. 其中13号分子与3L68蛋白对接结果较好, 从而验证了所建模型具有较好的可行性.
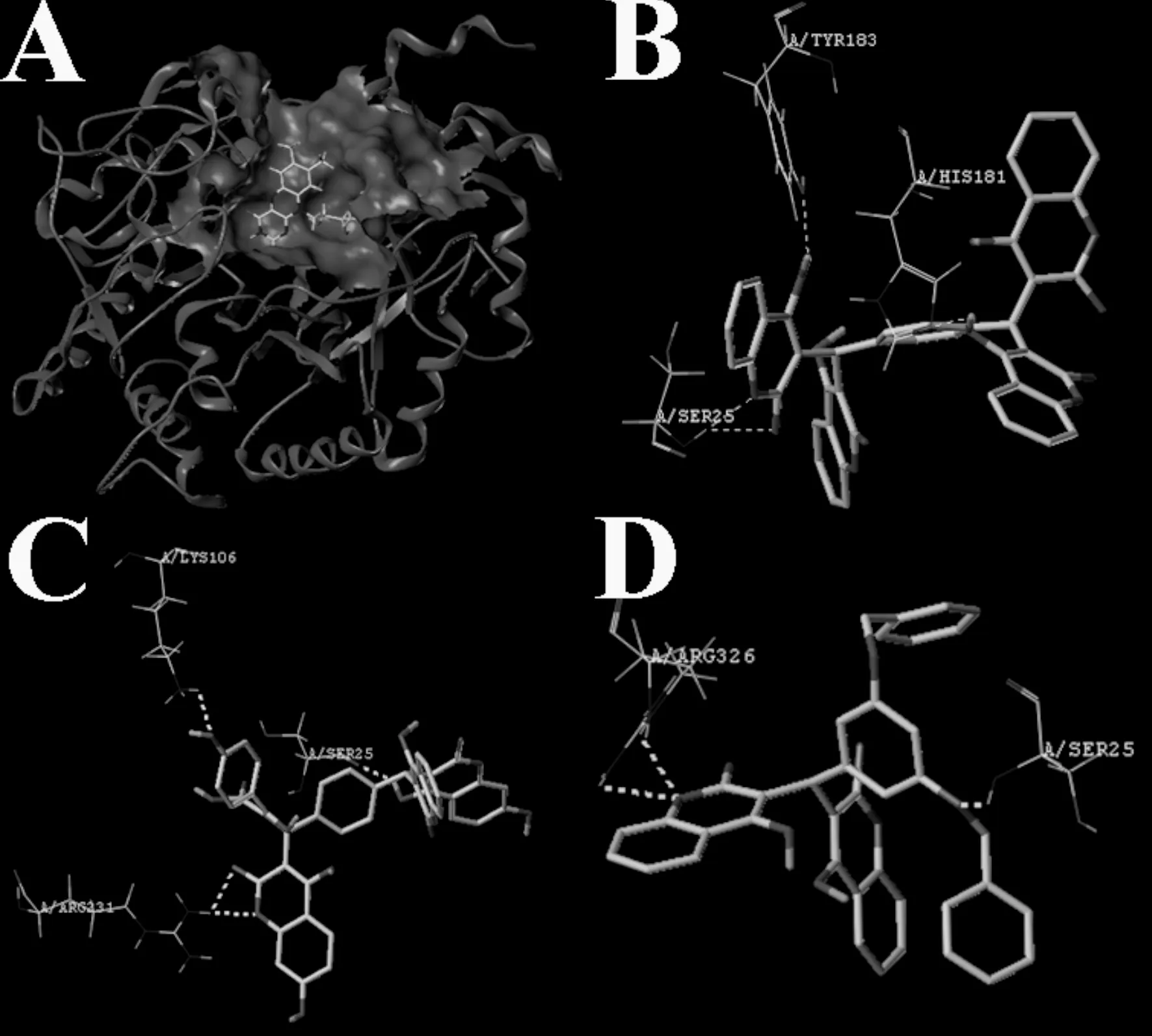
图4 化合物与3L68蛋白酶的对接作用模式: (A)19号分子对接作用模式图, (B)19号分子对接图, (C)13号分子对接图, (D)15号分子对接图.Fig. 4 Dock of compound and 3L68: (A) molecular 19 docking mode diagram,(B) molecular 19 docking map,(C) molecular 13 docking map,(D) molecular 13 docking map
4 结 论
本文利用分子对接和Topomer CoMFA方法对26个香豆素衍生物进行了研究, 建立了3D-QSAR模型, 模型等势面图提供了立体场和静电场的可视化图像, 直观地揭示了这一系列化合物中不同的取代基结构对其生物活性的影响, 并理论上通过分子对接进一步验证了所构建模型的可信性, 从而为实验工作者合成新药提供了理论参考.
