基因芯片筛选肝细胞性肝癌的差异表达基因和通路
2019-09-11钱叶本
江 勇,钱叶本,陈 朋,朱 良
肝细胞性肝癌 (hepatocellular carcinoma, HCC) 是原发性肝癌的主要类型,肝癌发病率居全球肿瘤的第5位,然而仅我国肝癌年发病例数就达到全世界的55%,且新发病例数仍在逐年增加[1]。复发和转移是困扰肝癌患者治疗和预后的主要难点。因此,深入研究肝癌发生发展过程中的相关机制,积极探讨其差异表达的基因和通路,有助于进一步认识肝癌的发病机制,提升肝癌的治疗水平和改善患者的预后。
基因芯片具有高通量、高灵敏度和自动快速等优点,可平行对比肿瘤组织及其对应的正常组织的基因表达水平,极大地提高了差异表达基因的筛选效率[2],有利于深入认识肿瘤的发病机制,提升肝癌患者早期诊断、治疗和预后的水平。该研究通过分析肝癌组织及其对应的癌旁组织的基因芯片,筛选得到差异表达基因;进一步通过生物信息学的方法,对筛选得到的差异基因进行聚类和功能富集分析,同时构建蛋白互作网络,得到核心基因。最后通过TCGA数据库中的肝癌基因表达数据RNA- sequencing初步探究核心基因的表达水平,为肝癌的诊断和治疗提供潜在的生物标记。
1 材料与方法
1.1 病例资料根据医学伦理要求和组织样本取材标准,2对肝癌组织及其对应的癌旁正常肝脏组织来自于安徽医科大学第一附属医院肝胆胰外科,组织标本取材于肝癌根治性切除术后组织。经过病理诊断后,确诊为肝细胞性肝癌。患者术前未进行过放疗、化疗或者介入治疗等操作。组织样本放入液氮中保存。该研究已经得到安徽医科大学第一附属医院伦理委员会的批准(批文号:2018- 13- 22)。
1.2 基因芯片本研究使用的基因芯片为Agilent Human Gene Expression(4×44 k),包括41 000条探针,由伯豪生物技术公司提供,实际操作流程参照Agilent表达谱芯片标准流程:总RNA反转录得到双链cDNA,再进一步用Cyanine- 3- CTP (Cy3)标记得到cRNA。将标记好的cRNA和芯片杂交,洗脱结束后,利用Agilent Scanner G2505C (Agilent Technologies) 扫描得到原始图像。
1.3 方法
1.3.1组织样本RNA提取和纯化 肝癌及其癌旁组织的总RNA通过QIAGEN RNeasy®Mini Kit(货号:#74106)试剂盒提取和纯化,同时纯化得到的RNA通过NanoDrop ND- 2000 (Thermo Scientific)进行定量检测,RNA完整性经Agilent Bioanalyzer 2100 (Agilent Technologies) 检测。
1.3.2芯片杂交与扫描 总RNA通过反转录得到双链cDNA,再进一步用Cyanine- 3- CTP (Cy3) 荧光染料标记双链cDNA,得到cRNA。经QIAGEN RNeasy® Mini Kit试剂盒纯化后,将标记好的cRNA置于60 ℃温水浴30 min,随后冰水浴1 min,进行片段化处理。随后将芯片与cRNA在65 ℃、10 r/min的条件下,于杂交炉中滚动杂交17 h。杂交结束后,进行芯片洗脱。最后利用Agilent Scanner G2505C(Agilent Technologies)扫描得到原始图像,通过Feature Extraction软件(version10.7.1.1, Agilent Technologies)处理原始图像,得到原始数据。
1.3.3芯片数据预处理和差异基因分析 利用Genespring软件(version 12.5; Agilent Technologies)对芯片原始数据进行标准化处理。标准化后的数据进行过滤,在用于比较的每组样本中,至少有1组完全标记为已被检测出的探针,进行后续分析。利用基因表达倍数变化值(Fold change)进行差异基因筛选,筛选标准:Fold change的绝对值≥3.0。
1.3.4GO功能注释和KEGG富集分析 使用DAVID(version 6.7, http://david.abcc.ncifcrf.gov/)在线分析平台[3],对筛选得到的差异基因在基因本体(gene ontology, GO)中注释,并进行KEGG (kyoto encyclopedia of genes and genomes, KEGG)功能富集分析,以判定差异基因主要影响的生物学功能和通路。P<0.05表示差异有统计学意义。
1.3.5蛋白互作网络的构建和筛选核心蛋白 通过String蛋白互作数据库(the Search Tool for the Retrieval of Interacting Genes, STRING, http://string- db.org/)分析差异表达基因的蛋白互作关系[4],并以此来构建蛋白互作网络,设置阈值条件为综合相关性评分>0.4。将String蛋白互作数据库分析得到的结果导入Cytoscape软件,通过网络分析插件CytoHubba计算节点的边(Degree,即互作连线的数量),筛选得到网络中心节点 (Hub Node)。中心节点对应的基因即为核心基因,同时也称作核心蛋白。
1.3.6核心基因的表达验证 肝癌原始的基因表达数据RNA- sequencing下载于TCGA数据库,通过Perl处理下载的原始数据,生成基因矩阵。再将Ensembl Gene ID转换为基因名称 (Gene symbol),得到最终可分析的基因表达数据,包括374例肝癌组织和50例正常肝脏组织,初步探究核心基因在肝癌组织中的表达情况。

2 结果
2.1 差异表达基因差异表达基因以Fold change≥3.0为筛选标准,在肝癌及癌旁组织中共筛选出4 324 个差异基因,其中上调表达的有2 552个基因,下调表达的有1 772个基因。挑选差异程度最大的前100个基因,热图用于展示这些基因在肝癌组织和正常组织的表达情况,见图1,红色代表高表达;绿色代表低表达。本研究的基因芯片结果已上传到gene expression omnibus (GEO) 数据库中(编号:GSE117361)。
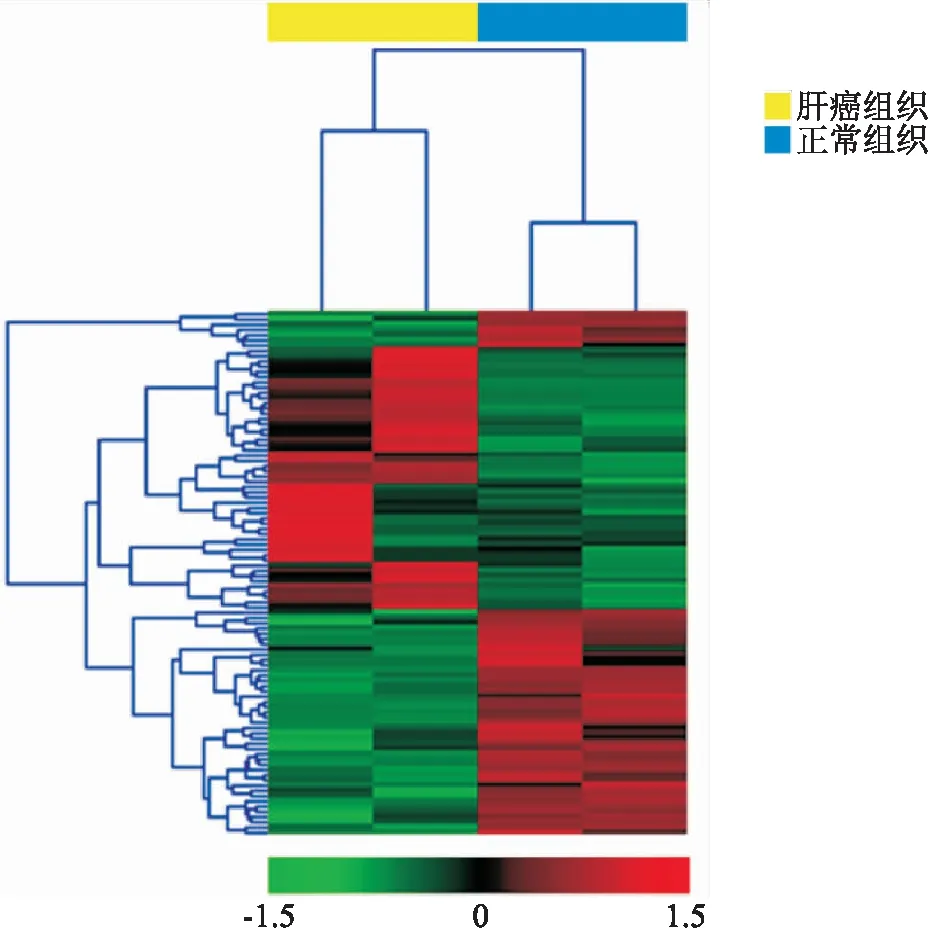
图1 前100个差异表达基因的热图
2.2 差异表达基因的GO功能注释GO功能注释的结果表明,差异表达基因在分子功能、生物过程及细胞组成的分布是存在差异的。根据显著富集GO结果,发现差异表达基因主要富集在血小板衍生生长因子结合、受体抑制剂和拮抗剂活性等相关的分子功能,同时也富集于调节细胞对血管内皮生长因子反应等生物过程和胶原蛋白复合物等细胞组成,见图2。
2.3 差异表达基因的KEGG富集分析差异基因的KEGG信号通路富集分析结果表明,在显著富集KEGG的结果中,包括TGF- β信号通路、Rap1信号通路、PI3K- Akt信号通路、细胞黏附、ECM- 受体相互作用以及趋化因子信号通路等,与肝癌发生进展密切相关,这进一步说明在肝癌的发病过程中多条信号通路失控,可能在疾病进展中起到十分重要的作用,见图3。

图2 GO功能注释
2.4 差异基因相互作用网络分析通过String蛋白互作数据库分析处理差异基因,构建蛋白互作网络,见图4。根据节点数目排序得到最相关的6个核心蛋白,对应的基因分别为DCN、TNF、COL1A1、COL3A1、FN1和COL1A2。
2.5 核心基因的表达验证通过TCGA数据库中374例肝癌组织和50例正常肝脏组织的基因表达数据,初步探究6个核心基因DCN、TNF、COL1A1、COL3A1、FN1和COL1A2的表达情况,见图5。结果表明,COL1A1(P<0.000 1)和COL1A2(P<0.000 1)在肝癌组织中的表达较正常组织显著上调,DCN在肝癌组织中的表达较正常组织显著下调(P<0.000 1);而TNF(P=0.052 2),FN1(P=0.103 0)和COL3A1(P=0.767 5)的表达无明显差异。
3 讨论
中国是肝癌发病率最高的国家,与乙肝病毒感染密切相关,其具体的发病机制不明。肝癌患者的确诊往往都在晚期,失去了根治性手术治疗的机会,且肝癌术后易复发,极大地影响了患者的预后。基因芯片技术可探索肿瘤发病过程中多种基因的表达变化及相互之间的调控关系,这有利于理解肝癌的发病机制,进一步提高诊治水平,改善预后。
本研究收集了新鲜的肝癌及癌旁组织,深入分析其基因芯片的结果,发现了4 324个差异表达基因,其中上调表达的有2 552个基因,下调表达的有1 772个基因。这一结果说明了肝癌发病过程中的基因转录变化,其发病机制可能与特定基因和表观遗传的变化密切相关。通过GO功能注释和KEGG富集分析差异表达基因潜在的生物学作用,结果发现差异表达基因主要富集于血小板衍生生长因子结合等分子功能,血管内皮生长因子反应等生物过程和胶原蛋白复合物等细胞组成;KEGG富集信号通路显示,差异表达基因主要参与了TGF- β、Rap1、PI3K- Akt和细胞黏附等信号通路,这些信号通路的变化与肝癌发生发展密切相关。TGF- β信号通路参与了肝癌发生发展的每个阶段,其在肝纤维化的进展中发挥重要作用;同时TGF- β在肝癌中表达上调,与肝癌预后直接相关[5]。Rap1是Ras家族单体G蛋白的成员之一,在肿瘤发生发展中扮演重要的角色,已经得到大家的共识[6]。 研究[7]表明Rap1可调节肿瘤细胞黏附、迁移和侵袭能力;在不同肿瘤类型中,Rap1信号通路的激活呈现出截然不同的表现,尤其是影响肿瘤转移的作用,依然有待于进一步研究。Rap1的激活对于EGFR介导胰腺癌的转移过程中是必不可少的,同时肿瘤的生长不受影响[8]。PI3K- Akt主要通过酪氨酸激酶受体传递生长因子的信号被激活[9],可调控肿瘤细胞的增殖和凋亡,与肝癌的发病过程密切相关[10]。由此说明,差异表达基因参与了肝癌发病过程中的多种细胞生物学过程,有待进一步研究其具体的生物学机制。

图3 KEGG 通路富集分析
通过蛋白互作网络分析,本研究得到了可能与肝癌发生密切相关的6个核心蛋白,对应的基因分别为DCN、TNF、COL1A1、COL3A1、FN1和COL1A2。进一步分析TCGA数据库中肝癌的基因表达数据,结果表明DCN、COL1A1和COL1A2在肝癌组织中的表达存在显著差异(P<0.000 1),而TNF、FN1和COL3A1的表达无明显差异。核心蛋白聚糖 (decorin, DCN) 是存在于细胞外基质的一类富含亮氨酸的小分子蛋白聚糖,研究表明DCN可抑制多种肿瘤细胞的增殖,可与多种细胞表面受体结合,介导肿瘤抑制的作用,包括对自噬和炎症的刺激作用以及对血管生成和肿瘤发生的抑制作用。肝硬化是肝癌重要的癌前病变,研究发现DCN在肝硬化组织的表达显著高于肝癌及癌旁组织,而DCN在肝癌组织中的表达显著低于癌旁组织;同时,DCN高表达的肝癌患者中位生存期更长,提示DCN可作为肝癌的预后标志物[11]。DCN可调控胶原纤维的生成,特异性地阻断TGF- β信号通路,发挥抗纤维化的生物活性[12]。COL1A1和COL1A2基因分别编码I型胶原蛋白的pro- α1链和pro- α2链,该胶原蛋白的三螺旋结构包括两条α1链和一条α2链,是细胞外基质的主要蛋白。COL1A1在侵袭性肝癌中表达上调,此外,COL1A1和COL1A2基因促进了I型胶原蛋白的合成,有助于HCC的侵袭和转移[13]。COL1A1的启动子发生甲基化,导致其在肝癌组织中表达下调,该表观遗传改变可能作为HCC的预后标志物[14]。微小RNA(MicroRNA)let- 7g 可抑制肝癌的转移,研究发现其抑制作用部分是通过靶向作用于COL1A2实现的[15]。

图4 蛋白相互作用网络图

图5 TCGA数据库初步验证核心基因的表达
A:DCN;B:TNF;C:COL1A1;D:FN1;E:COL3A1;F:COL1A2;与正常组织比较:****P<0.000 1
综上所述,肝癌发病过程中涉及多基因、多通路的改变,本研究通过基因芯片筛选肝癌的差异表达基因和通路,有助于进一步理解肝癌的发病机制。同时,通过TCGA数据库初步验证了核心基因的表达,为肝癌提供了新的研究方向,有待于进一步研究其具体的功能和作用。
