小鼠耳蜗性聋的关键通路和差异表达基因分析△
2019-07-23杨媛媛黄冠江陶源
杨媛媛 黄冠江 陶源
由于耳蜗部位的血液供应相对较脆弱,一旦耳蜗供血障碍易引起耳蜗的病变,从而导致耳蜗性聋[1~4],严重的耳蜗性聋患者的最佳治疗是人工耳蜗植入术,但至今耳蜗性聋的相关机制仍不十分明确[5,6]。最新研究表明,早期给予外源性脑源性神经因子可有效保护条件敲除缝隙链接蛋白26的小鼠耳蜗Corti器及螺旋神经节细胞的形态,但对于听功能则无保护作用[7]。Yoshimura等[8]通过基因芯片技术和实时荧光定量PCR,发现POU4F3、Slc17a8、TMC1及Crym的突变可导致耳蜗性聋。为进一步研究小鼠耳蜗性聋的关键通路、差异表达基因(differentially expressed genes,DEGs)及这些基因产物之间的相互作用,本研究采用数据库挖掘的方法进行基因本体分析(gene ontology analysis,GO)、京都基因和基因组百科全书分析(kyoto encyclopedia of genes and genomes, KEGG)、蛋白质互作网络分析(protein protein interaction network,PPI network)及基因集富集分析(gene set enrichment analysis, GSEA),旨在拓展耳蜗性聋机制的研究思路,并为耳蜗性聋的治疗提供参考。
1 材料与方法
1.1材料 从美国国立生物信息中心(National Center for Biotechnology Information, NCBI)基因表达数据库(gene expression omnibus, GEO)下载小鼠耳蜗性聋基因芯片表达谱数据(GSE84735),该芯片数据由东京大学的耳鼻咽喉科学者Kashio A和Someya S上传,采用Affymetrix Mouse Genome 430 2.0 Array平台,芯片数据包含10个样本,其中,耳蜗性聋小鼠样本5例(实验组),对照组小鼠样本5例,均为2月龄。实验组小鼠均采用0.15%的GeO2喂养4个月,对照组小鼠采用正常饮食喂养。均经听性脑干反应检测证实,GeO2喂养后的实验组小鼠出现了严重的听力损失,而对照组小鼠无听力损失,在小鼠6月龄的时候,运用微阵列技术分析CBA小鼠耳蜗基因的表达。
1.2分析方法
1.2.1数据处理及差异基因分析 运用R软件(版本3.5.1)进行基因芯片分析。将原始CEL(CIM Fast Event Language)数据导入R软件,对耳蜗性聋组(实验组)和正常听力组(对照组)进行差异比较,应用affyPLM和affy程序包对原始芯片数据进行质量控制,用RMA算法对数据进行标准化处理;从而应用limma程序包对基因进行筛选,选出差异表达基因(differentially expressed genes,DEGs)。筛选条件:Fold Change的绝对值大于2,且P<0.05,然后,通过R软件运行gplots程序包,做可视化的火山图。最后,使用Morpheus (https://software.broadinstitute.org/morpheus/)对排名前50的上调和下调的差异基因做可视化的热图。
1.2.2GO分析和KEGG分析 GO是一种常用的注释基因和识别生物学特性的有效方法[9, 10]。KEGG是一个对基因功能进行系统分析的数据库[11, 12],能对差异基因进行GO富集分析和KEGG通路分析[13]。为了分析差异基因,在戴维数据库(Database for Annotation, Visualization and Integrated Discovery, David) v6.8中,对差异基因进行GO富集分析和KEGG通路分析,且定义P<0.05时具有统计学意义。
1.2.3差异基因互作网络分析 交互基因的搜索工具(search tool for the retrieval of interacting genes,STRING)数据库是用于评估蛋白质相互作用信息的在线工具,String(版本10.5)涵盖了来自2 031种生物体的960万种蛋白质信息。为了评估DEGs之间的交互关系,本研究应用Cytoscape软件构建蛋白质相互作用网络(PPI network),使用cytoHubba插件筛选出前10位的关键基因,同时使用分子复合检测(molecular complex detection,MCODE)插件蛋白质相互作用对网络的模块进行分析;然后,对MCODE排名第一的模块进行KEGG通路分析,且定义P<0.05时具有统计学意义。
1.2.4GSEA分析 采用GSEA软件,对数据集GSE84735进行基因集富集分析, Spearman相关系数为基因和样本标签之间的基因的权重[14, 15],按默认的加权富集统计的方法进行富集分析,设置随机组合次数为1 000次;当通路的错误发生率(false discovery rate,FDR)<25%且P<0.01时,认为通路具有显著统计学差异。
2 结果
2.1差异基因的表达分析结果 通过实验组与对照组小鼠样本对比,总共19 191个基因被纳入研究。基于Fold change的绝对值大于2并P<0.05的标准,筛选出191个DEGs,其中,79个基因为耳蜗性聋的上调DEGs,112个基因为耳蜗性聋的下调DEGs(图1)。对排名前50的上调和下调的DEGs做可视化的热图(图2)。
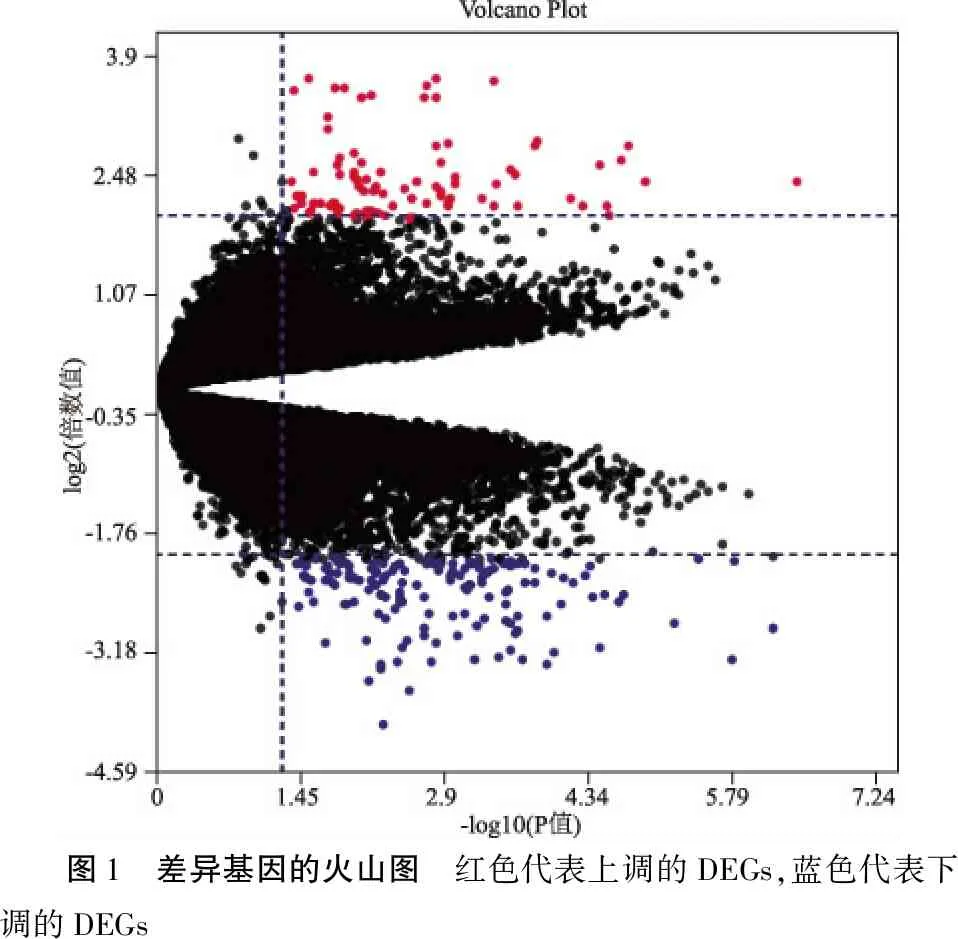
图1 差异基因的火山图 红色代表上调的DEGs,蓝色代表下调的DEGs
2.2GO分析和KEGG分析结果 上传所有的DEGs至在线分析工具-David数据库,以识别GO分析和KEGG分析。GO分析结果显示,在生物过程中,上调的DEGs显著富集的生物过程包括:蛋白质寡聚化的调节、先天免疫应答、细胞趋化性、细胞对白细胞介素-1的反应、细胞对脂多糖的反应等(表1);下调的DEGs显著富集的生物过程包括:有丝分裂核分裂、细胞周期、细胞分裂、染色体分离及红细胞分化等(表1)。在分子功能(molecular function,MF)方面,上调的DEGs显著富集的分子功能包括:趋化因子受体结合、锌离子结合、趋化因子活性、金属肽酶活性及细胞因子活性;下调的DEGs显著富集的分子功能包括:微管结合、组蛋白结合、转录抑制活性、RNA聚合酶II核心启动子近端序列特异性结合、转录调控区序列特异性DNA结合及RNA聚合酶II转录因子结合等(表1)。此外,在细胞成分(cell component,CC)方面,上调的DEGs显著富集的细胞成分包括:细胞外空间和细胞外区;下调的DEGs显著富集的细胞成分包括:着丝粒区染色体、着丝粒、纺锤体极、染色体及稠密染色体着丝粒等(表1)。

表1 小鼠耳蜗性聋差异表达基因的GO分析
KEGG分析结果显示,上调的DEGs在肿瘤坏死因子信号通路中富集,而下调的DEGs在细胞周期、孕酮介导的卵母细胞成熟、卵母细胞的减数分裂及p53信号通路中富集(表2)。
2.3PPI 网络分析结果 基于String数据库中的信息,使用Cytoscape(版本3.6.1)进行PPI网络的绘制(图3),然后,使用插件cytoHubba对关键基因进行筛选,排名前10的关键节点(hub)包括Kif11、Cdc20、Cdk1、Bub1b、Ccnb2、Cdca8、Cdca5、Sgol1、Hmmr及Ncapg,其中,Kif11节点的度(degree)最高,为31。此外,使用插件MCODE对总共91个节点和516个边界进行了分析,选择排名第1的子网络,分析网络中涉及的基因的重要通路(图4,表3),KEGG分析表明,DEGs主要与细胞周期、孕酮介导的卵母细胞成熟、卵母细胞的减数分裂、p53信号通路及病毒性癌变有关。

表2 小鼠耳蜗性聋差异表达基因的KEGG分析
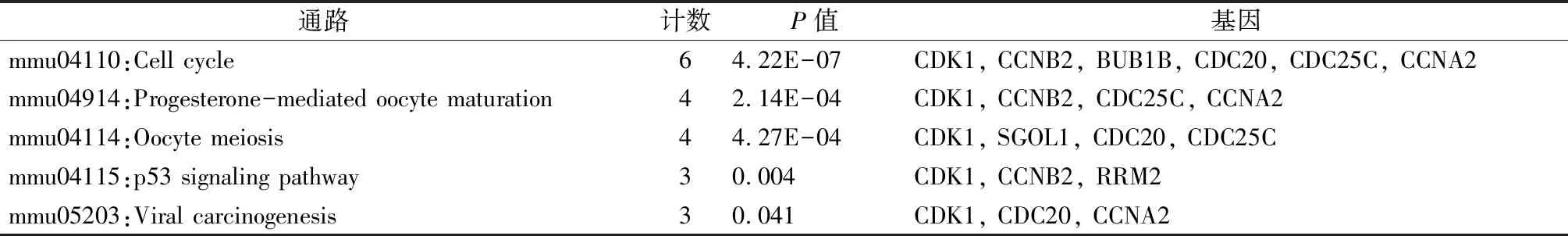
表3 关键子网络中涉及的基因的重要通路的KEGG分析

图3 蛋白质相互作用网络
2.4GSEA分析结果 GSEA分析结果显示,共有17 541个基因集在小鼠耳蜗性聋表型中表达上调,其中560个FDR<25%,621个P<0.01;共有1 836个基因集在小鼠耳蜗性聋表型中表达下调,其中703个FDR<25%,683个P<0.01;如图5所示,前三位上调的通路包括树突状细胞成熟A(图5a)、Cronquist Nras信号转导(图5b)及反应金属离子SLC转运蛋白(图5c);而前三位下调的通路包括半乳糖代谢(图5d)、氧化磷酸化(图5e)及嘌呤代谢(图5f)。
3 讨论
Qiao等[16]研究了巨细胞病毒对小鼠耳蜗的破坏机制,耳蜗的组织病理学检查显示前庭膜中淋巴细胞浸润水平增加,通过免疫组织化学染色检测到鼓膜中较高水平的TNF-α和IL-6;Zou等[17]研究了振动引起的听力损失相关动物模型的细胞和分子机制,证实耳蜗性聋与TNF-α及其受体的上调有关;Riva等[18]同样验证了这一观点;Tadros等[19]研究了随年龄增长和听力损失程度加重的小鼠耳蜗中凋亡相关基因的表达,通过基因芯片和实时荧光定量PCR技术,证实耳蜗性聋与P53相关。本研究从GEO数据集中表达谱数据(GSE84735)检索到5个小鼠耳蜗性聋的样本和5个正常听力的样本,研究中共确定191个差异表达基因。为了更好地研究DEGs的相互作用,本研究进行了GO分析和KEGG分析;在GO分析中,上调的DEGs主要参与细胞外空间、细胞外区及CXCR趋化因子受体结合等过程,下调的DEGs主要参与有丝分裂核分裂、细胞周期及细胞分裂等过程;在KEGG分析中,上调的DEGs主要参与肿瘤坏死因子信号通路,下调的DEGs主要参与细胞周期、孕酮介导的卵母细胞成熟、卵母细胞的减数分裂及p53信号通路等。

图4 关键子网络

图5 GSEA分析结果 a、b、c为前三位上调通路,d、e、f为前三位下调通路
本研究还构建了DEGs的PPI网络,并列出了前10个关键基因:Kif11、Cdc20、Cdk1、Bub1b、Ccnb2、Cdca8、Cdca5、Sgol1、Hmmr及Ncapg。Kif11被确定为具有最高连接度的关键基因,Kif11是一种蛋白质编码基因,编码驱动蛋白样蛋白家族的运动蛋白,其基因产物的功能包括染色体定位、中心体分离和建立双极纺锤体。Kovacovicova等[20]的一项研究显示,Kif11能保持双极纺锤体结构,促进卵母细胞的成熟;Johnson等[21]的研究表明KIf11参与有丝分裂纺锤体的形成过程,进而促进次级神经元和胶质细胞的发育。Cdc20一般在细胞周期中调节多种蛋白相互作用,在染色体分离和有丝分裂结束中具有重要功能[22];其他关键基因Cdk1、Bub1b、Ccnb2、Cdca8、Cdca5、Sgol1、Hmmr及Ncapg的研究,目前仍缺乏相关文献。Buniello等[23]发现wbp2表达缺失导致小鼠渐进性高频耳蜗性聋;Menardo等[24]发现SAMP8菌株能促使小鼠耳聋和耳蜗退化提前出现,还原人类耳蜗性聋的过程,促使SAMP8小鼠提前出现耳聋的相关分子机制涉及氧化应激、抗氧化酶水平的改变和复合物I、II和IV的活性降低,同时这些也导致了慢性炎症和细胞凋亡的发生。所以,耳蜗性聋的关键基因仍需要进一步研究。
本研究差异表达基因工作网络图的关键子网络分析显示耳蜗性聋的差异基因与细胞周期、孕酮介导的卵母细胞成熟、卵母细胞的减数分裂、p53信号通路及病毒性癌变有关,但相关文献目前仍缺乏;所以,耳蜗性聋的关键基因仍需要进一步研究。GSEA分析的结果显示,共有17 541个基因集在小鼠耳蜗性聋表型中表达上调,有1 836个基因集在小鼠耳蜗性聋表型中表达下调,同时前三位上调的通路包括树突细胞成熟、Croonquist Nras信号转导及反应金属离子硫蛋白转运,而前三位下调的通路包括半乳糖代谢、氧化磷酸化及嘌呤代谢。Someya等[25]研究表明耳蜗性聋与氧化磷酸化相关。而其他通路仍需要更多的基础研究来验证。然而,本研究也有一些不足:首先,样本量较小,仅包括5个耳蜗性聋小鼠样本和5个对照组样本;第二,分析结果中筛选出的基因及通路超过半数未在基础研究中得到验证,故暂无足够文献供分析,需要更多基础研究来进一步阐明基因集富集分析的结果;第三,本研究的结果全部来源于GEO数据库下载的表达谱数据,仍需进一步基础实验来验证。
总之,本研究获得的小鼠耳蜗性聋差异表达基因的生物信息学分析结果,不仅为耳蜗性聋研究方向的扩展提供了新思路,对于其发病机制及分子靶向治疗的研究也具有较好的提示作用。
