鲊肉粉发酵过程中细菌动态变化研究
2018-07-09汪雪瑞王承明
张 瑶,白 娟,汪雪瑞,王承明*
(华中农业大学 食品科学技术学院 环境食品学教育部重点实验室,湖北 武汉 430070)
鲊肉粉是一种广受人们喜爱的传统发酵食品,最早可溯源至先秦时期,在我国已经有两千多年的食用历史。传统鲊肉粉是以新鲜猪肉、米粉和红辣椒为主要原料经自然发酵而成。熟制后的成品具有适宜的口感和诱人的色泽,同时具有较长的货架期,能极大地延长产品的可食用期限。到目前为止,对其他发酵食品如豆豉[1-2]、豆瓣[3-5]、酸鱼[6-8]、发酵香肠[9]等中微生物的研究有大量文献报道,但对于鲊肉粉中细菌多样性的研究未见有文献报道。
对于传统发酵食品中细菌多样性的研究方法包括传统的纯培养法和现代分子生物学的方法[10]。传统培养法因培养条件的限制,目前能进行培养的微生物不到1%,且培养过程易导致原始菌群构成发生改变,使结果有较大偏差[11-12]。现代分子生物学技术包括聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturingdradient gel electrophoresis,PCR-DGGE)技术[13]、高通量测序技术等[14]。其中,DGGE技术因其无法检测丰度较低的微生物而具有一定的局限性[15]。随着高通量测序技术的发展,宏基因组学技术越来越多地被应用于传统发酵食品中微生物的研究[16-20]。通过对发酵制品中微生物的脱氧核糖核酸(deoxyribonucleicacid,DNA)、核糖核酸(ribonucleic acid,RNA)进行分析,宏基因组学技术不仅能检测出低丰度的微生物,而且速度快,结果准确[21-22]。因此,本实验在前期对鲊肉粉的加工工艺进行研究的基础上,采用宏基因组学技术对鲊肉粉发酵过程中细菌的组成及相对丰度进行研究,探索其发酵过程中细菌群落的动态变化,为后期深入解析鲊肉粉的发酵机理及对发酵过程的监控奠定重要的理论基础,同时可以为鲊肉粉的发酵过程设计相应的高效发酵剂,有利于鲊肉粉的工业化生产。
1 材料与方法
1.1 材料与试剂
籼米:湖北省监利县兴旺米业有限公司;新鲜猪肉、红辣椒、生姜、大蒜:市售;食盐:湖北蓝天盐化有限公司;脱氧核糖核苷酸三磷酸(deoxy-ribonucleosidetriphosphate,dNTPs)Mix、5×TransStartTM、FastPfuBuffer和Fast Pfu Fly DNAPolymerase:北京全式金生物技术有限公司;QIAGEN DNeasy mericon Food Kit:德国QIAGEN公司。
1.2 仪器与设备
BS 224S分析天平:赛多利斯科学仪器(北京)有限公司;Miseq高通量测序平台:美国Illumina公司;5810R台式高速冷冻离心机:德国Eppendorf公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司;DYY-12电泳仪:北京六一仪器厂;Vetiri梯度基因扩增仪:美国AB公司;UVPCDS 8000凝胶成像分析系统:美国Bio-Rad公司。
1.3 方法
1.3.1 鲊肉粉的加工工艺流程及操作要点

1.3.2 样品的制备
按照鲊肉粉的生产工艺流程[23]制备鲊肉粉样品。按GB/T 9695.19—2008《肉与肉制品取样方法》进行取样,取样后经组织破碎机破碎混匀后装入无菌采样管中,置于-80℃的超低温冰箱中保存,直至所有样品采集完成。试验中共采集了11组发酵时期的样品,鲊肉粉样品编号分别为:0 d、2 d、4 d、6 d、8 d、10 d、12 d、14 d、16 d、18 d和20 d。
1.3.3 宏基因组DNA的提取
按照QIAGEN DNeasy mericon Food Kit试剂盒的说明书步骤对鲊肉粉样品的宏基因组DNA进行提取,并通过微量紫外分光光度计检测DNA的纯度和浓度。
1.3.4 细菌16S rRNA扩增
PCR扩增体系:5×PCR缓冲液4 μL,2.5 mmol/L dNTPs Mix 2 μL,5 μmol/L 正向引物0.8 μL,5 μmol/L 反向引物0.8 μL,5 U/μL DNA聚合酶0.4 μL,DNA模板10 ng,体系用ddH2O补充至20 μL。细菌16S rRNA的正向引物为338F(5′-ACTCCTACGGGAGGCAGCA-3′),反向引物为806R(5′-GGACTACHVGGGTWTCTAAT-3′)。
PCR扩增条件:95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸45 s,30个循环;72℃再延伸10 min。
1.3.5 样品平衡及Miseq高通量测序
行为异常组在矛盾性和独立性维度的得分明显高于正常组(P<0.05),而在亲密度、情感表达、知识性、娱乐性、道德宗教观、组织性及控制性维度的得分则明显低于正常组(P<0.05)。见表1。
经1%琼脂糖凝胶电泳检验合格的PCR扩增产物稀释至100 nmol/L,混匀后送至上海美吉生物科技有限公司,使用Miseq平台进行高通量测序。
1.3.6 数据分析
根据序列间的重叠关系对序列进行质控,然后使用微生物生态学定量分析(quantitative insights into microbial ecology,QIIME)平台进行物种和多样性的分析,基于UniFrac距离进行主坐标分析,根据97%的相似性使用UCLUST软件进行操作分类单元(operational taxonomic units,OTU)划分,采用Origin软件作图。
2 结果与分析
2.1 宏基因组DNA的提取及PCR扩增
鲜肉粉中微生物宏基因组DNA及PCR扩增产物电泳检验结果分别见图1。由图1A可知,电泳条带存在弥散拖尾现象,这是因为宏基因组DNA中包含有大量的微生物基因组片段[24]。由图1B可知,PCR产物目的条带大小正确,浓度合适,可进行后续实验。

图1 鲊肉粉中微生物宏基因组DNA(A)及PCR扩增产物(B)电泳图Fig.1 Electrophoresis of metagenomic DNA(A)and PCR amplification products(B)from fermented meat rice
2.2 鲊肉粉中细菌多样性分析
2.2.1 稀释曲线及香农指数
稀释曲线是用于评价测序深度是否足够覆盖所有的类群,同时间接反映出样品中物种的丰富程度[25]。当曲线趋于平坦或达到平台期时就可认为测序深度已经基本覆盖到样品中的所有物种[26-27];反之,则表示样品中物种的多样性较高,还存在许多未被检测到的物种。香农指数图是利用各样品的测序量在不同测序深度时的微生物多样性指数构建的曲线,用于反映各样品在不同测序数量时的微生物多样性。当曲线趋于平缓时,说明测序数量足够大,可以反映样品中大多数的微生物物种信息[28]。鲊肉粉中细菌群落的稀释曲线及香农指数结果见图2。

图2 鲊肉粉中细菌群落的稀释曲线(A)和香农指数(B)Fig.2 Rarefaction curve(A)and Shannon index(B)of bacterial community from fermented meat rice
由图2(A)可知,随着测序深度的增加,发现物种的数量继续增加,说明随着测序量的增加可能会有新的种型被发现。由图2(B)可知,随着测序深度的增加,香农指数曲线已基本达到平衡状态,说明在此测序水平下,样品中细菌的多样性已能充分展现。
2.2.2 α多样性分析
α多样性是指一个特定区域或生态系统内的多样性[29],常用的度量指标有Chao1指数、Shannon指数、Simpson指数,不同发酵时间鲊肉粉样品的α多样性统计结果如表1所示。
Chao1指数是用于评估样品中细菌群落的OTU数目的,Chao1指数值越大表明样品中细菌的总数越多;Shannon用于评估样品中微生物的多样性,Shannon指数值越大,表明样品中细菌群落的多样性越高;Simpson指数也是用于评估样品中微生物多样性的指数,Simpson指数越大表明细菌多样性越高。由表1可知,本研究产出的序列已经能全面捕获样品中细菌微生物的多样性,因而是满足后续分子生物学分析的[30]。2.2.3β多样性分析

表1 鲊肉粉样品的α多样性统计Table 1 Alpha diversity statistics of fermented meat rice
β多样性是用于度量时空尺度上物种组成的变化[31]。其中,主成分分析(principal component analysis,PCA)是一种研究数据相似性或差异性的可视化方法,通过主成分分析可以直观反映出个体或群体间的差异,一个点表示一个样品,两点间的距离越近表示两者的菌落构成越相近[32-33]。通过数字化的矩阵对鲊肉粉的微生物多样性进行了主成分分析,结果如图3所示。
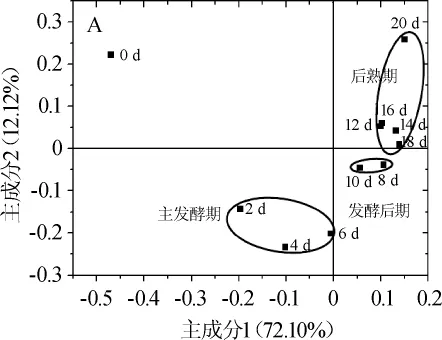
图3 鲊肉粉发酵过程中细菌群落的主成分分析Fig.3 Principal component analysis of bacterial community from fermented meat rice
由图3可知,在PC1方向上,鲊肉粉发酵0~6 d样品都聚集在负轴,其中发酵0天即未发酵的鲊肉粉样品在第二象限;2~6 d样品聚集在一起,在第三象限。8~20 d样品都聚集在正轴;在PC2方向上,2~10d样品聚集在负轴,其中,发酵8 d和发酵10 d的样品在第四象限,0 d样品和12~20 d样品也聚集在一起,12~20 d样品在第一象限。因此,可以按所在象限不同将鲊肉粉的发酵过程划分为三个阶段,依次为2~6 d的主发酵期、8~10 d的发酵后期和12~20 d的后熟期。
2.3 鲊肉粉中优势细菌科、属相对含量的比较分析
本实验运用宏基因组学技术,通过对鲊肉粉发酵过程中细菌的16S rRNA进行测定,实现了对鲊肉粉发酵过程中细菌群落组成及相对丰度的研究。结果表明,鲊肉粉发酵过程中共检出有19个门、47个纲、75个目、106个科、153个属和211个种的细菌参与鲊肉粉的发酵过程。在门水平上,厚壁菌门Firmicutes(69.02%)和变形菌门Proteobacteria(5.02%)为主要细菌门。厚壁菌门中芽孢杆菌纲Bacilli占有绝大多数(69.00%),而变形菌门中的主要细菌纲有Gammaproteobacteria(4.64%)、变形菌纲Alphaproteobacteria(0.37%)和Betaproteobacteria(0.01%)。

图4 鲊肉粉中优势细菌科相对含量的比较分析Fig.4 Comparative analysis on the relative content of the dominantbacteria family in fermented meat rice
由图4可知,在鲊肉粉的发酵过程中主要优势细菌科为葡萄球菌科Staphylococcaceae(35.81%)、明串珠菌科Leuconostocaceae(19.76%)、乳杆菌科Lactobacillaceae(7.66%)等,其中明串珠菌科细菌的相对含量在发酵开始后迅速增加,乳杆菌科细菌的相对含量在发酵后有所增加。

图5 鲊肉粉中优势细菌属相对含量的比较分析Fig.5 Comparative analysis on the relative content of the dominant bacteria genera in fermented meat rice
由图5可知,在鲊肉粉的发酵过程中主要优势细菌属为葡萄球菌属Staphylococcus(34.92%)、魏斯氏菌属Weissella(19.68%)、乳酸杆菌属Lactobacillus(7.62%)、不动杆菌属Acinetobacter(2.24%)、环丝菌属Brochothrix(2.24%)、发光杆菌属Photobacterium(1.67%)和巨球菌属Macrococcus(1.24%)。其中,葡萄球菌在鲊肉粉的发酵过程中可以分解蛋白质和脂肪,对鲊肉粉的营养和风味具有重要的作用[34];魏斯氏菌具有提高发酵食品质量缩短发酵周期等作用[35];乳酸杆菌能还原亚硝酸盐,减少亚硝胺的生成,抑制腐败微生物的生长,提高鲊肉粉的营养安全性[36]。在鲊肉粉的发酵过程中,不动杆菌属、环丝菌属和发光杆菌属的相对含量在发酵开始后迅速降低,而魏斯氏菌属的相对含量在发酵开始后迅速升高,这与关统伟等[37]的结果相似。相关性分析表明,魏斯氏菌属细菌与不动杆菌属、环丝菌属和发光杆菌属细菌间都存在显著的负相关关系,而不动杆菌属、环丝菌属和发光杆菌属细菌之间均存在显著的正相关关系,间接表明不动杆菌属、环丝菌属和发光杆菌属细菌之间存在某种互利共生的关系,而魏斯氏菌属细菌对不动杆菌属、环丝菌属和发光杆菌属细菌均有一定的抑制作用。
3 结论
本实验运用宏基因组学技术,通过对鲊肉粉中细菌16S rRNA高通量测序,对鲊肉粉发酵过程中细菌群落组成及相对丰度进行了研究。结果表明,鲊肉粉发酵过程中共有19个门、47个纲、75个目、106个科、153个属和211个种的细菌参与动态变化。该过程的优势细菌属分别为葡萄球菌属Staphylococcus(34.92%)、魏斯氏菌属Weissella(19.68%)、乳酸杆菌属Lactobacillus(7.62%)、不动杆菌属Acinetobacter(2.24%)、环丝菌属Brochothrix(2.24%)、发光杆菌属Photobacterium(1.67%)和巨球菌属Macrococcus(1.24%)。其中不动杆菌属、环丝菌属和发光杆菌属细菌的相对含量在发酵开始后迅速降低,而魏斯氏菌属细菌的相对含量在发酵开始后迅速升高,乳酸杆菌属细菌的相对含量在发酵后有所增加。相关性分析表明,不动杆菌属、环丝菌属和发光杆菌属细菌间存在互利共生关系,而魏斯氏菌属对不动杆菌属、环丝菌属和发光杆菌属细菌均有一定的抑制作用,魏斯氏菌属和乳酸杆菌属细菌有利于发酵的进行,并对腐败菌有一定的抑制作用,从而提高了产品的营养和安全性。通过本实验可以更加深入地理解鲊肉粉的发酵机制,同时可以为鲊肉粉的发酵过程设计相应的高效发酵剂,有利于鲊肉粉的工业化生产。
[1]陈廷涛.发酵乳和豆豉中微生物菌群DGGE分析及益生作用研究[D].南昌:南昌大学,2013.
[2]林榕姗.细菌型豆豉发酵机理及功能性研究[D].泰安:山东农业大学,2012.
[3]于松峰,郑 宇,杨 帅,等.传统豆瓣辣酱发酵过程细菌群落演替与发酵过程的对应关系[J].中国调味品,2017,42(4):53-58.
[4]关统伟,王鹏昊,邓奥宇,等.郫县豆瓣发酵过程可培养细菌多样性及其演化分析[J].食品与发酵工业,2017,43(4):22-27.
[5]张 琦,汪先丁,杨 虎,等.郫县豆瓣自然发酵过程中细菌群落结构的变化[J].食品与发酵科技,2010,46(6):16-18,35.
[6]于美娟,谭 欢,马美湖,等.传统固态发酵鱼中细菌群落多样性与品质特征分析[J].食品科学,2017,38(8):86-95.
[7]王华娟.酸鱼中乳酸菌的分离鉴定及其可控发酵工艺研究[D].武汉:武汉轻工大学,2016.
[8]湛剑龙.贵州侗族酸鱼、酸肉中抗氧化乳酸菌的筛选与优化[D].贵阳:贵州大学,2015.
[9]段 艳.乳酸菌的筛选及其对羊肉干发酵香肠品质特性的影响[D].呼和浩特:内蒙古农业大学,2013.
[10]吴进菊,胡佳琪,于 博,等.传统发酵食品中微生物多样性和群落结构动态变化研究进展[J].中国酿造,2016,35(9):20-23.
[11]AMANN R I,LUDWIG W,SCHLEIFER K H.Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].Microbiol Rev,1995,59(1):143-169.
[12]HANDELSMAN J.Metagenomics application of genomics to uncultured in microorganisms[J].Microb Mol Biol Rev,2004,68(4):669-685.
[13]HARUTA S,UENO S,EGAWA I,et al.Succession of bacterial and fungal communities during a traditional pot fermentation of rice vinegar assessed by PCR-mediated denaturing gradient gel electrophoresis[J].Int J Food Microbiol,2006,109(1):79-87.
[14]李 欣,王彦华,林静怡,等.高通量测序技术分析酱香型白酒酒醅的微生物多样性[J].福建师范大学学报:自然科学版,2017,33(1):51-59.
[15]夏围围,贾仲君.高通量测序和DGGE分析土壤微生物群落的技术评价[J].微生物学报,2014,54(12):1489-1499.
[16]PARK E J,KIM K H,ABELL G C J,et al.Metagenomic analysis of the viral communities in fermented foods[J].Appl Environ Microbiol,2011,77(4):1284-1291.
[17]WANG Z M,LU Z M,SHI J S,et al.Exploring flavour-producing core microbiota in multispecies solid-state fermentation of traditional Chinese vinegar[J].Sci Rep,2016,6:26818.
[18]ZHANG J,WANG X,HUO D,et al.Metagenomic approach reveals microbial diversity and predictive microbial metabolic pathways in Yucha,a traditional Li fermented food[J].Sci Rep,2016,6:32524.
[19]JI Y J,LEE S H,KIM J M,et al.Metagenomic analysis of Kimchi,a traditional Korean fermented food[J].Appl Environ Microbiol,2011,77(7):2264-2274.
[20]赵红宇,徐炜桢,杨国华,等.基于高通量测序的郫县豆瓣后发酵期细菌多样性研究[J].食品科学,2017,38(10):117-122.
[21]LI Z,RUI J,LI X,et al.Bacterial community succession and metabolite changes during doubanjiang-meju fermentation,a Chinese traditional fermented broad bean(Vicia fabaL.)paste[J].Food Chem,2017,218:534-542.
[22]WANG Q,GARRITY M G,TIEDIE M J,et al.Naive Bayesian classifier for rapid assignment of rRNA sequencesinto the new bacterial taxonomy[J].Appl Environ Microbiol,2007,73(16):5261-5267.
[23]张 瑶,白 娟,王承明.鲊肉粉的加工工艺研究[J].中国酿造,2017,36(10):175-180.
[24]郭 壮,沈 馨,董 蕴,等.襄阳大头菜腌制液膜醭细菌群落结构研究[J].中国酿造,2017,36(7):143-147.
[25]聂志强,韩 玥,郑 宇,等.宏基因组学技术分析传统食醋发酵过程微生物多样性[J].食品科学,2013,34(15):198-203.
[26]ZOU Y L,XIANG W L,RAO Y,et al.Diversity analysis of bacteria in the latter ripening of Pixian soybean paste fermentation based on 16S rDNA analysis[J].Afr J Microbiol Res,2012,6(42):7008-7012.
[27]朱 琳,高 凤,曾椿淋,等.萝卜泡菜细菌多样性的高通量测序分析[J].现代食品科技,2018,34(2):1-6.
[28]朱雯娟.发酵梅香鱼微生物种群结构分析及发酵菌株筛选研究[D].湛江:广东海洋大学,2015.
[29]LIU W,ZHENG Y,KWOK L Y,et al.High-throughput sequencing for the detection of the bacterial and fungal diversity in Mongolian naturally fermented cow's milk in Russia[J].BMC Microbiol,2015,15(1):45.
[30]苏加坤,徐 达,郭 磊,等.基于宏基因组测序的烟叶表面微生物多样性分析[J].基因组学与应用生物学,2017(4):1538-1545.
[31]陈泽斌,李 冰,王定康,等.Illumina MiSeq高通量测序分析核桃内生细菌多样性[J].江苏农业学报,2015(5):1129-1133.
[32]陈圣宾,欧阳志云,徐卫华,等.Beta多样性研究进展[J].生物多样性,2010,18(4):323-335.
[33]郭 壮.应用焦磷酸测序技术对不同人群肠道微生物群落结构的研究[D].无锡:江南大学,2013.
[34]程 燕,杨 勇,廖定容,等.腐生葡萄球菌s25在四川香肠加工过程中对脂肪和蛋白质的影响[J].食品科学,2012,33(23):223-227.
[35]李巧玉,方 芳,堵国成,等.魏斯氏菌在发酵食品中的应用[J].食品与发酵工业,2017(10):241-247.
[36]朱志远,卢士玲,孙晓斌,等.发酵剂对发酵香肠微生物和理化品质的影响[J].江苏农业学报,2009,25(4):890-893.
[37]关统伟,向慧平,王鹏昊,等.基于高通量测序的郫县豆瓣不同发酵期细菌群落结构及其动态演替[J].食品科学,2018,39(4):106-111.
