辽宁地区猪源大肠埃希菌喹诺酮类耐药基因的检测与分析
2018-01-27赵凤菊李清竹顾贵波
赵凤菊,关 淼,李清竹,顾贵波
(辽宁省动物疫病预防控制中心,辽宁 沈阳 110164)
抗生素耐药性是一个严重的公共卫生问题[1-3]。喹诺酮类药物特别是新型喹诺酮类药物是人工合成的广谱抗菌剂,在一些国家被广泛应用于家畜和宠物的临床治疗[4-5]。目前,喹诺酮类主要有3种耐药机制:药物靶位改变(质粒介导机制)、外膜通透性减低(被膜机制)和外排溢出泵(外排泵机制)[6]。其中,质粒介导的喹诺酮类耐药机制有3种:第1种是质粒介导的qnr机制,qnr基因在多种肠道细菌中存在,其编码的蛋白质属于五肽重复家族的重要成员之一,可以保护DNA解旋酶和拓扑异构酶Ⅳ,抑制喹诺酮类药物[7-8];第2种机制与aac(6′)-Ib-cr基因相关,aac(6′)-Ib-cr是aac(6′)-Ib的变异体,其编码的蛋白质能够使哌嗪基乙酰化,从而降低细菌环丙沙星或诺氟沙星的敏感性[9];第3种机制是基于喹诺酮类外排泵蛋白的qepA基因,其编码的蛋白质可以耐萘啶酸和诺氟沙星[5]。DNA解旋酶由gyrA和gyrB基因编码,拓扑异构酶Ⅳ由parC和parE基因编码,gyrA和parC之间、gyrB和parE之间的序列具有很高的同源性[10]。DNA解旋酶和拓扑异构酶Ⅳ蛋白的氨基酸突变是喹诺酮类药物的主要耐药机制之一[11]。大肠埃希菌是引起人和动物感染的主要微生物之一[12],在大肠埃希菌中gyr基因和par基因的耐药突变率很高,大多数喹诺酮耐药决定区位于gyrA67~106位、parC56~108位[10]。
为了解辽宁地区猪源大肠埃希菌中喹诺酮类耐药基因(PMQR)的分布特点及gyr基因和par基因编码的喹诺酮耐药决定区突变特征,对23株喹诺酮类抗生素耐药大肠埃希菌11个PMQR基因进行PCR检测及序列分析,为进一步研究喹诺酮类抗生素耐药机制提供理论依据,并为兽医临床喹诺酮类抗生素的合理使用、新型药物的研发以及猪大肠埃希菌病的综合防治提供科学依据。
1 材料和方法
1.1 材料
1.1.1 菌株 23株对喹诺酮类抗生素耐药大肠埃希菌从辽宁不同地区病死猪或患病猪的组织脏器、活猪粪便中分离得到,由辽宁省动物疫病预防控制中心实验室鉴定并保存。
1.1.2 培养基及试剂 营养琼脂培养基、麦康凯培养基(生产批号分别为20130608、20130708)购自北京奥博星生物技术有限公司,水解酪蛋白琼脂培养基(MH)、药敏纸片(生产批号分别为140506、140506)均购自杭州市天和微生物试剂有限公司,DNAzol提取试剂购自Invitrogen公司,PrimeSTAR® HS DNA Polymerase with GD Buffer、ExTaqDNA聚合酶、10×ExTaqBuffer、2.5 mmol/L dNTPs、DNA Marker、溴化乙锭均购自大连宝生物科技有限公司。
1.1.3 PCR扩增引物 根据GenBank上登录的gyrA、gyrB、parC、parE、aac(6′)-Ib-cr、qepA、qnrA、qnrB、qnrC、qnrD、qnrS基因序列,利用Primer Premier 5.0软件设计了11对特异性引物(表1),引物由上海生工生物工程技术服务有限公司合成。

表1 引物序列
1.2 方法
1.2.1 细菌DNA模板的制备 挑取培养18~24 h的培养物,用1 mL无菌生理盐水制备成一定浓度的菌悬液。吸取200 μL菌悬液至1.5 mL Eppendorf管中,加入800 μL DNAzol提取试剂,混匀后4 ℃、12 000 r/min离心10 min。吸取900 μL上清,置于另一个1.5 mL Eppendorf管中,加入500 μL无水乙醇,混匀后4 ℃、12 000 r/min离心5 min。弃上清,用无菌水配制的75%乙醇洗涤2次,干燥后用40 μL无菌水溶解沉淀,-20 ℃保存备用。
1.2.2 耐药基因及耐药谱检测 采用25 μL反应体系:10×PCR Buffer 2.5 μL,2.5 mmol/L dNTPs 2 μL,上、下游引物(20 μmol/L)各1 μL,ExTaq酶0.125 μL,灭菌水16.375 μL,模板 2 μL。
PCR反应条件:94 ℃ 5 min;94 ℃ 45 s,退火温度(根据引物选择合适退火温度,表1) 45 s,72 ℃ 50 s,35个循环;72 ℃ 10 min。用1%琼脂糖凝胶电泳检查PCR产物。
采用药敏纸片法测定耐药谱。
1.2.3 序列测定 将PCR产物送上海生工生物工程有限公司进行序列测定,进行gyr和par基因突变分析。并将基因序列推导为氨基酸序列,分析突变情况。
2 结果与分析
2.1 PMQR基因检测结果及菌株耐药谱分析
利用PCR检测方法,对23株喹诺酮类耐药菌株进行耐药基因检测(表2)。结果表明,20株供试菌为PMQR基因阳性,阳性率为86.96%(20/23),21.74%(5/23)的菌株携带3种PMQR基因,39.13%(9/23)的菌株携带2种PMQR基因,26.09%(6/23)的菌株携带1种PMQR基因,13.04%(3/23)的菌株未检测到PMQR基因。其中,主要PMQR基因是aac(6′)-Ib-cr基因,阳性率为73.91%(17/23);其次为qepA、qnr基因(qnrB、qnrC、qnrS基因),阳性率分别为52.17%(12/23)、39.13%(9/23);qnrA、qnrD基因未被检测到。
23株喹诺酮类耐药菌株耐药谱广,均对萘啶酸耐药。
2.2 gyr和par基因突变检测结果
23株喹诺酮类耐药菌株均对萘啶酸耐药,选取15株耐药菌株进行gyrA、gyrB、parC和parE基因的序列测定分析,将基因序列推导为氨基酸序列,分析突变情况(表3)。从表3可以看出,gyrA在第87位氨基酸的突变率为80%(12/15),其中1株该位点的天冬氨酸被谷氨酸所替代,11株该位点的天冬氨酸被天冬酰胺所替代;gyrB基因在第426位、447位氨基酸的突变率分别为80%(12/15)、100%(15/15),12株第426位的天冬氨酸被天冬酰胺所替代,15株第447位的赖氨酸被谷氨酸所替代;parC基因在第56位、80位氨基酸的突变率分别为20%(3/15)、73.33%(11/15),3株第56位的丙氨酸被苏氨酸所替代,11株第80位的丝氨酸被异亮氨酸所替代;parE基因未检出突变。在15株突变株中,K447E的突变率最高,其次为D87N、D426N、S80I和A56T。
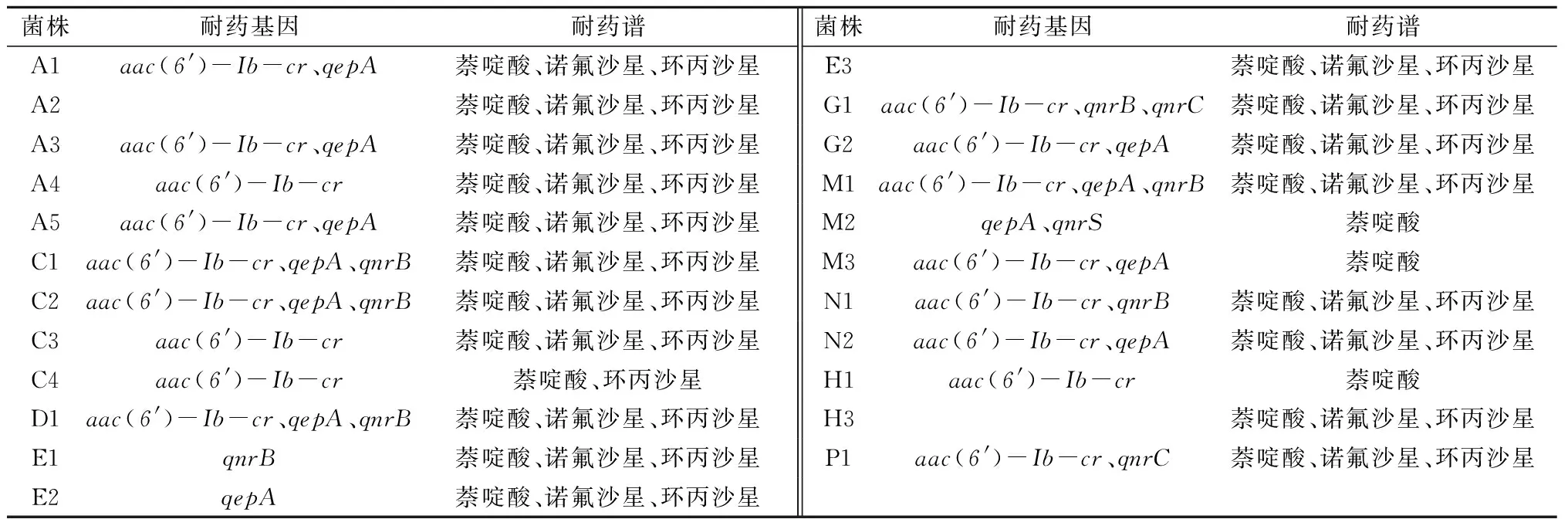
表2 大肠埃希菌中喹诺酮类耐药基因的检测结果及耐药谱

表3 喹诺酮耐药决定区突变分析
注:A.丙氨酸;D.天冬氨酸;E.谷氨酸;K.赖氨酸;I.异亮氨酸;N.天冬酰胺;S.丝氨酸;T.苏氨酸;WT.野生型。
3 结论与讨论
喹诺酮类抗生素的滥用导致细菌耐药性越来越严重,质粒介导的喹诺酮抗性基因易在不同菌属之间传播,导致常见传染病的治疗非常困难[13]。在世界各地,qnr、aac(6′)-Ib-cr、qep和oqx质粒介导的喹诺酮耐药基因在人和供食品生产动物的细菌分离株中已有报道[14]。目前,已在大肠埃希菌、克雷伯菌属、肠杆菌属的细菌中发现aac(6′)-Ib-cr基因,在大肠埃希菌中最常见[15]。qnr基因在多个国家的多种肠杆菌科细菌中均有检出,分布较为广泛,其中qnrA基因于1998年肺炎克雷伯菌的临床分离株中被确定,随后qnrB基因和qnrS基因在大肠埃希菌、沙门氏菌、肺炎克雷伯菌中被发现[16]。携带qepA基因和oqxAB基因的大肠埃希菌临床分离株分别在2002年和2008年被发现[14]。本研究中,PMQR基因的阳性率高达86.96%,其中aac-(6′)-Ib-cr基因的检出率最高,占检测菌株的73.91%,其次为qepA、qnr基因。
人们首先在金黄色葡萄球菌和假单胞菌中发现由于突变的原因产生喹诺酮类抗药性现象[10]。对喹诺酮类药物耐药的主要机制涉及gyrA基因和parC基因的喹诺酮耐药决定区的突变,最常见的突变是gyrAS83位、D87位,parCG78、S80、E84位[17]。此外,能导致大肠埃希菌对喹诺酮类药物耐药的突变是gyrBD426、K447位,parEL445位[18]。gyrA第83位氨基酸突变可能是大肠埃希菌对喹诺酮类耐药的首要条件,而gyrA第87位和parC第80位氨基酸突变是导致高水平耐药的必要条件[15]。gyrB上D426N的突变可使菌株对所有的喹诺酮类药物耐药,而K447E的突变则能增强菌株对萘啶酸的耐药性[19]。本研究中,15株耐药菌株中有12株的gyrA亚基出现氨基酸突变,15株的gyrB亚基均发生突变,11株的parC亚基发生突变,而parE亚基中未检出突变,并且突变类型与报道的一致[15,17-18]。
综上,在辽宁地区PMQR耐药基因及靶位突变成为猪源大肠埃希菌对喹诺酮类药物产生耐药性的重要原因。因此,有必要加强该地区耐药基因及耐药基因突变的监控,指导临床合理用药,降低因喹诺酮类耐药造成的经济损失。
[1] Armas-Freire P I,Trueba G,Proao-Bolaos C,etal.Unexpected distribution of the fluoroquinolone-resistance geneqnrBinEscherichiacoliisolates from different human and poultry origins in Ecuador[J].Int Microbiol,2015,18(2):85-90.
[2] 梁乔,赵凤菊,顾贵波,等.辽宁地区猪源大肠埃希菌氨基糖苷类抗生素耐药基因的检测与分析[J].河南农业科学,2017,46(6):134-137.
[3] 李进福,丁海峰,李小申,等.河南地区猪呼吸道隐性感染大肠杆菌耐药基因检测及分析[J].河南农业科学,2017,46(9):144-148.
[4] Harifi M E,Meshkat Z,Izadi N,etal.Prevalence of quinolone resistance genes among extended-spectrumβ-Lactamase-producingEscherichiacoliin Mashhad,Iran[J].Jundishapur J Microbiol,2015,8(12):e16217.
[5] Ma J,Zeng Z,Chen Z,etal.High prevalence of plasmid-mediated quinolone resistance determinantsqnr,aac(6′)-Ib-cr,andqepAamong ceftiofur-resistant Enterobacteriaceae isolates from companion and food-producing animals[J].Antimicrob Agents Chemother,2009,53(2):519-524.
[6] 孔令超,李志国,张瑞华,等.2012—2013年山东省禽源大肠杆菌中质粒介导喹诺酮类药物耐药基因的检测[J].中国动物传染病学报,2014,22(4):56-62.
[7] Yanat B,Rodríguez-Martínez J M,Touati A.Plasmid-mediated quinolone resistance in Enterobacteriaceae:A systematic review with a focus on Mediterranean countries [J].Eur J Clin Microbiol Infect Dis,2016,36(3):431-435.
[8] Tran J H,Jacoby G A.Mechanism of plasmid-mediated quinolone resistance[J].Proc Natl Acad Sci USA,2002,99(8):5638-5642.
[9] Robicsek A,Strahilevitz J,Jacoby G A,etal.Fluoroquinolone-modifying enzyme:A new adaptation of a common aminoglycoside acetyltransferase[J].Nat Med,2006,12 (1):83-88.
[10] Johnning A,Kristiansson E,Fick J,etal.Resistance mutations ingyrAandparCare common inEscherichiacommunities of both fluoroquinolone-polluted and uncontaminated aquatic environments[J].Front Microbiol,2015,6:e34953.
[11] Hopkins K L,Davies R H,Threlfall E J.Mechanisms of quinolone resistance inEscherichiacoliandSalmonella:Recent developments[J].Int J Antimicrob Agents,2005,25(5):358-373.
[12] Liu X,Liu H,Li Y,etal.High prevalence of β-lactamase and plasmid-mediated quinolone resistance genes in extended-spectrum cephalosporin-resistantEscherichiacolifrom dogs in Shaanxi,China[J].Front Microbiol,2016,7:1-9.
[13] Soundararajan N,Shanmugam P,Devanbu C,etal.A study on theaac(6′)-Ib-crgene prevalence among ciprofloxacin-resistant strains of uropathogenic Enterobacteriaceae[J].Int J Appl Basic Med Res,2016,6(4):258-261.
[14] Kawanishi M,Ozawa M,Hiki M,etal.Detection ofaac-(6′)-Ib-crin avian pathogenicEscherichiacoliisolates in Japan[J].J Vet Med Sci,2013,75(11):1539-1542.
[15] 姜晓冰.大肠杆菌质粒介导喹诺酮耐药机制研究[D].广州:华南理工大学,2013.
[16] Rezazadeh M,Baghchesaraei H,Peymani A.Plasmid-mediated quinolone-resistance(qnr) genes in clinical isolates ofEscherichiacolicollected from several hospitals of Qazvin and Zanjan provinces,Iran[J].Osong Public Health Res Perspect,2016,7(5):307-312.
[18] Jacoby G A,Corcoran M A,Hooper D C.Protective effect of qnr on agents other than quinolones that target DNA gyrase[J].Antimicrob Agents Chemother,2015,59(11):6689-6695.
[19] Ruiz J.Mechanisms of resistance to quinolones:Target alterations,decreased accumulation and DNA gyrase protection[J].J Antimicrob Chemother,2003,51(5):1109-1117.
