宏基因组学在法医学鉴定中的应用
2017-09-26萨日娜蔡令艺武会娟严江伟
萨日娜,蔡令艺,武会娟,严江伟,刘 旭,胡 荣
(1.北京市理化分析测试中心,北京100089;2.济宁市精神病防治院,山东 济宁272051;3.中国科学院北京基因组研究所,北京100101)
宏基因组学在法医学鉴定中的应用
萨日娜1,蔡令艺2,武会娟1,严江伟3,刘 旭1,胡 荣1
(1.北京市理化分析测试中心,北京100089;2.济宁市精神病防治院,山东 济宁272051;3.中国科学院北京基因组研究所,北京100101)
随着分子生物学与基因组学的发展,宏基因组学在法医学与法医学鉴定中逐渐扮演着重要的角色。近年来,宏基因组学作为研究环境微生物菌群构成与多样性、各成员之间相互关系及与环境之间相互关系的分支学科,在法医学鉴定相关领域的应用也逐渐兴起,并带来了新的契机。本文对宏基因组学研究策略及其在法医学鉴定中个体识别、案发现场生物斑迹来源鉴定及药物滥用检测等方面的应用现状进行了综述,旨在进一步阐明宏基因组学在法医学中的作用与应用价值。
法医遗传学;宏基因组学;综述;基因组,微生物;个体识别
微生物是一群体形微小、结构简单的低等生物的总称,包括细菌、真菌、病毒以及一些小型的原生生物、藻类等在内的一大类多样性生物群体。美国国立卫生研究院(National Institutes of Health,NIH)于2007年10月启动了耗资1.5亿美元的人体微生物组计划(Human Microbiome Project,HMP)[1],旨在发现人体不同部位的微生物菌落构成及分布特征,建立涵盖人体口腔、呼吸道、皮肤、肠道等部位的微生物基因组序列,进而阐明人类健康和疾病与人体微生物组的关系。
微生物群落是一个多基因组的混合物,其中可能高达99%的微生物不能单独分离培养。1998年,威斯康星大学HANDELSMAN等[2]在研究土壤微生物时首次提出了宏基因组学(metagenomics)这一概念,通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究环境样品所包含的全部微生物的遗传组成及其群落功能。而宏基因组(metagenome)是指特定环境中全部微小生物遗传物质的总和。目前,相对于其他领域而言,医学领域宏基因组学相关研究还比较少,主要集中在口腔、肠道、皮肤等人体微生物学方面,以及研究抗药基因与医院感染等方面相关的微生物[3-6]。近年来,宏基因组学在法医学鉴定相关领域的应用也逐渐兴起,并给法医学个体识别与身份匹配、案发现场鉴定与混合斑迹来源鉴定以及毒理学的研究和发展带来了新的契机。本文对微生物宏基因组学在法医鉴定中的应用与前景进行综述,旨在进一步阐明微生物宏基因组学在法医学中的作用与应用价值。
1 宏基因组学研究策略
目前宏基因组学的研究策略主要有基于全基因组测序和基于16S/18S rRNA基因测序两大类(图1)。两种宏基因组学方法各有优缺点,既可以根据研究目的选用其中的一种,也可以综合使用。
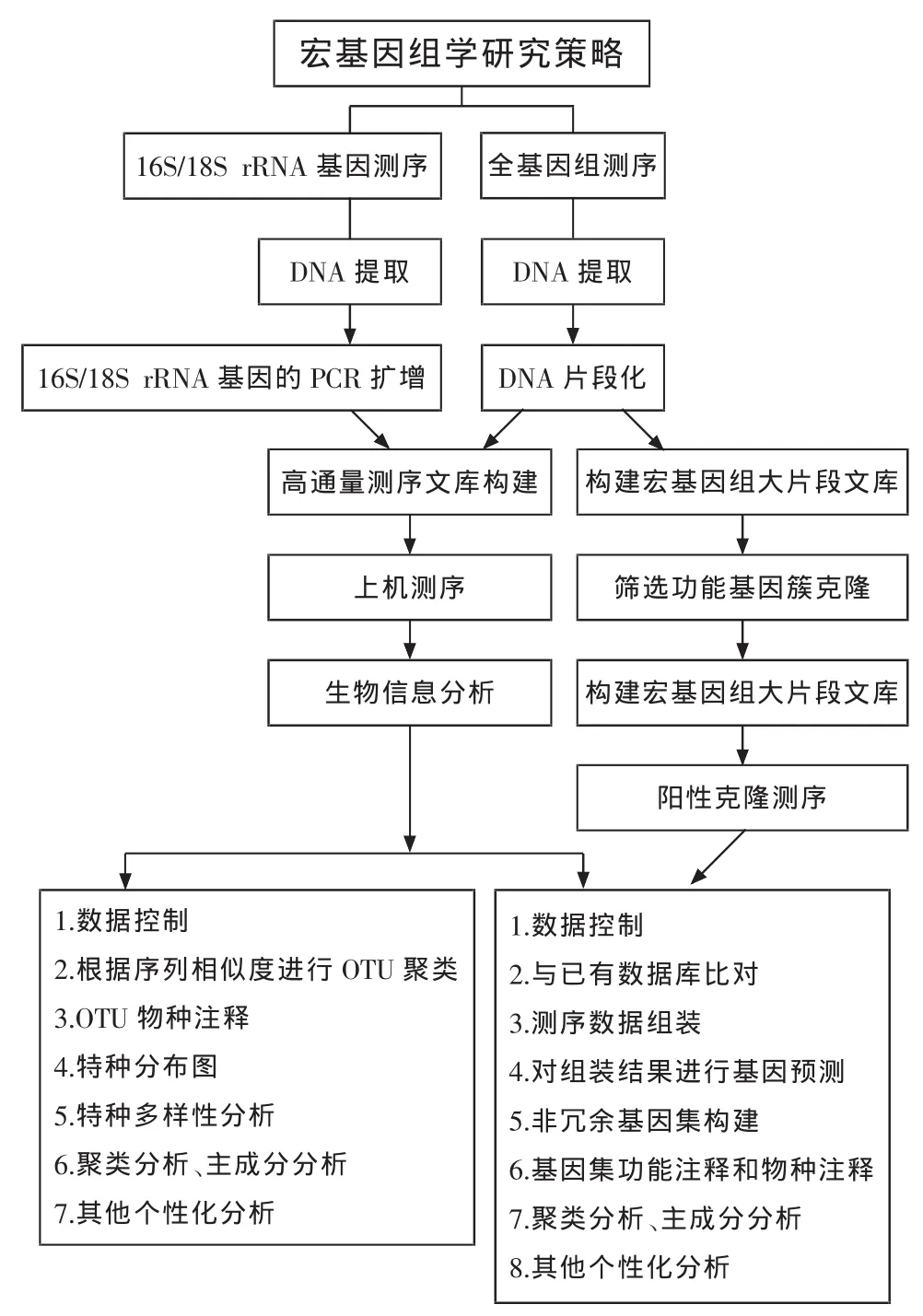
图1 宏基因组学研究策略流程图
1.1 基于全基因组测序
基于全基因组测序的宏基因组学可以得到细菌等微生物的全部基因组序列,进而研究样品中的功能基因以及其在环境中所起的作用。传统的基于全基因组测序的宏基因组学研究主要流程为:采用直接裂解提取法或膜过滤法提取土壤、水等环境中或动植物组织样品的DNA;将样品中的DNA片段化,使用克隆技术将DNA随机连接插入质粒或黏粒等合适的载体,构建宏基因组大片段文库;然后转化细菌并筛选克隆;对插入片段进行测序和功能性分析,通过外源基因赋予宿主细胞的新性状或基于某些已知DNA序列筛选,达到对微生物菌落特性与功能的基本了解(图1)。但是,传统的全基因组测序的宏基因组学分析费用高、周期长,极大地限制了宏基因组学研究的发展。
1.2 基于16S/18S rRNA基因测序
对16S/18S rRNA基因的高通量测序分析是目前最普遍与最广泛的宏基因组学研究之一。大量实验表明,最适合于揭示各类生物亲缘关系的是rRNA[7,8]。细菌等原核生物rRNA按沉降系数分为3种,分别为5S、16S和23S rRNA。16S rRNA长度适中,包含足够的遗传信息,在所有的细菌中都存在,并且16S rRNA保守性与特异性并存,保守区序列有助于设计测序引物,可变区序列用于分析种属关系,为鉴定与分类提供了便利。而真菌、一些藻类等微生物则用18S rRNA分析种属关系。因此,16S/18S rRNA是微生物群落分析、进化及分类研究最常用的靶分子,通过对样品中16S/18S rRNA基因测序可以鉴定其中微生物物种的丰度和分布情况。目前,随着新一代高通量低成本测序技术的广泛应用,让我们在获得海量的数据后,能够快速、全面地分析微生物群落结构,给宏基因组学的广泛与深度研究带来了新的契机[9]。相比于全基因组测序,基于16S/18S rRNA基因测序的宏基因组学无需构建文库,因此极大地缩减了分析周期及成本,然而其缺点是无法对不同物种的基因功能进行分析。
2 宏基因组学技术在法医学鉴定中的应用现状
随着计算生物学与分子生物学,尤其是新一代测序技术的发展,微生物在法医鉴定中的应用得到了极大的推动及革新[10]。即使对于很微量的微生物,宏基因组学技术通过分析完整的基因组DNA或RNA信息,可以辅助个体身份匹配,判断环境(土壤、水体、植物等)及斑迹中微生物的组成,以及在病原体与毒物鉴定等方面发挥作用。鉴于微生物在法医鉴定中的潜在作用,微生物法医学已经作为一个学科分支,用于探究微生物对法医科学与调查研究的影响[11]。
2.1 在个体识别中的辅助应用
不同生存环境与生活方式的个体具有不一样的微生物群落构成,COSTELLO等[12]对4个不同时间点采样的7~9名健康个体的27个身体部位进行了分析,发现不仅每个人身体不同部位拥有不同的微生物组成及多样性,而且不同个体间同一部位的微生物也复杂多变。
皮肤不同部位的微生物多样性与聚集模式差异大于口腔与消化系统[13]。人体微生物,尤其是体表(皮肤与毛发)微生物的这种个体化差异与独特性,表明其可能作为个体识别的一种辅助方式[14]。FIERER等[15]最先比较了计算机使用者手部及配套鼠标和键盘上的细菌、其他键盘上的细菌以及超过250个手部细菌群落的数据库,发现一台计算机表面与其使用者手部皮肤的细菌组合通常比任何其他样本与计算机使用者的手部皮肤细菌更加匹配;并且细菌在物体上可以持续至多两周基本不发生变化。在哈佛大学最新发表的研究[16]中,利用宏基因组学技术结合微生物学、计算机理论,研究人员通过长时间对一个人群携带微生物的分析研究,得到了个体特异的并在一段时间内相对稳定的个体宏基因组特征。对100多个个体的群体进行身体部位特异性的微生物宏基因组分析检测,发现约三分之一的个体在30~300 d之后的重新分析测试与之前的宏基因组特征相符,并且个体间的特异性也较好。因此,发现人体细菌DNA足够的特征以及把细菌“指纹”和个体有效地联系起来可能是未来法医物证鉴定分析中的新型分析策略。
俄勒冈大学的MEADOW等[17]在最新研究中提出了微生物云的概念,即人体释放到环境的所有微生物,表明每个人都携带着独一无二的混合细菌群,并散发至空气中。研究人员通过过滤设备收集试验对象释放的所有微生物并进行宏基因组学分析,通过微生物云可以辨别绝大多数试验对象,并且还发现空气中的颗粒微生物比沉积到物体表面的颗粒微生物更能体现个人特征;但是研究人员也表示,此发现建立于无菌背景,换到大的或者充满灰尘的空间,他们并不确定研究结果是否能够被重复,需要更深入的研究来验证。
针对毛发所携带的微生物宏基因组学分析及其在法医学鉴定中应用价值的探索也已经有了初步的尝试。TRIDICO等[18]对人类毛发中的头发和阴毛进行了基于新一代测序技术的宏基因组学分析,结果表明,人类阴毛有可能成为独立的证据而作为其他结果的佐证,尤其是性侵相关案件中,当其他证据不足时,有可能通过分析侵害者或受害者阴毛宏基因组而得到有用信息。
除了皮肤与毛发相关微生物,口腔微生物尤其是唾液微生物也可能具有重要的法医学应用价值。人类口腔唾液中含有约700~800种细菌,密度达到1.4× 108/mL[19]。案件中出现的咬痕可能作为法医鉴定的物证之一,但口腔或唾液DNA容易受到唾液酶的降解,这种情况下,唾液微生物组成可能会指示咬痕制造者的个体信息。然而口腔微生物组成受口腔卫生、饮食、地理环境、遗传等多方面因素的影响,需要更多研究才能验证口腔或唾液宏基因组是否能够提供准确可靠的身份匹配信息。
2.2 在案发现场生物斑迹来源鉴定中的应用
在法医学领域,土壤、水体甚至植物等与案发现场相关的可疑物证应用于案件调查已有上千年的历史[20]。土壤检验可为犯罪嫌疑人和犯罪现场是否具有关联提供极为重要的证据,并为案件的侦破提供方向和范围。以往对土壤的检验主要包括颜色、质地、物质成分以及对地质学特征加以区别。土壤含有大量多样性的微生物,随着分子生物学技术的快速发展,人们对土壤微生物的认识不断加深。早期的研究主要运用末端限制性片段长度多态性(terminal-restriction fragment length polymorphism,T-RFLP)分析[21]和扩增长度异质性聚合酶链式反应(amplicon length heterogeneity-polymerase chain reaction,ALH-PCR)[22]等常用DNA指纹技术来分析土壤等环境样品的微生物基因多态性。HORSWELL等[23]最先将细菌16S rRNA分析方法应用于法庭科学的土壤分析当中,他们对不同来源的土壤样品进行了细菌总DNA的提取后,再用细菌的16S rRNA基因特异引物对所提取的DNA进行PCR扩增,经酶切与电泳后获得各土壤样本的图谱;依据图谱所含峰的数目和位置,结合统计学公式计算样品间的相似度,从而推断样品间的关系;通过细菌的16S rRNA基因序列的比较,发现不同土壤间包含的细菌群体存在较大的差异性,且这些细菌群落在相对较长的时间内保持一定的平衡。以上方法均是在微生物种属构成不明确的情况下对所提取的土壤微生物DNA片段长度多态性的比较,不同样本间的分辨率较低。
新一代高通量测序技术的出现与发展,推动了宏基因组学应用于法医鉴定相关的土壤与环境微生物多样性分析及群落构成鉴定。KHODAKOVA等[24]对两个不同城市不同地点的土壤微生物,分别利用鸟枪法全基因组测序或基于非特异性扩增后测序的宏基因组学技术,通过种属注释、聚类分析后发现,基于非特异性扩增的宏基因组数据更能区分不同地点的土壤样本。然而,TIMS等[25]在尝试利用皮肤指纹微生物群落作为法医学依据来指示个体所在地点与环境时,没有得到理想的结果。值得注意的是,土壤及环境微生物如果在近距离和短的时间范围内变化太大,则不能代表性地指示某一现场或位置。因此,LENZ等[26]建议用土壤中的固氮菌类这一相对稳定和具有代表性的特定菌群作为法医学鉴定中指示地点相关信息的依据。
分子生物学是法医学中分析犯罪现场遗留血斑与其他生物斑迹的重要工具。在生物斑迹的物种鉴定、斑迹成分分析、DNA谱图分析中,实际可用于分析的核酸质量和含量不一定足够高。传统的基因检测方法需要100~500 pg DNA含量才能得到较好的结果,并且其中痕量DNA往往被掩盖而无法测得。BRENIG等[27]利用深度测序平台结合宏基因组学分析1个案件中不明来源的血斑,对其进行全基因组扩增后测序,通过比对已知微生物和哺乳动物数据库,发现了该血斑的宏基因组组成及来源,即使是含量很低的成分也能鉴定,表明宏基因组学可用于法医学中未知斑迹的组成与来源分析鉴定。
2.3 在药物滥用检测中的应用
除了在个体识别、案发现场鉴定中的可能作用,口腔微生物还可能在法医毒理学相关领域具有潜在的应用价值。对于毒品摄入的鉴定,常规体内检测主要是以尿液、血液或器官作为检验材料。目前,毒品检测主要是以金标法进行尿液检测,具有快速、方便、准确率高等特点。但在现场检测药物滥用与否时,唾液更加容易采集,且具有不受时间、地点的限制,无需对收集人员进行专门训练,可防止掺假替换等优点,是滥用药物现场检测较好的生物检材。然而,无论对于尿液、血液还是唾液,毒品在人体内具有一定的代谢周期,超过代谢周期的时间期限则无法检测到毒品的摄入情况。因此,开发一种时效性更长久的吸毒确认鉴定方法,对于指导公安实战、促进全面禁毒工作显得尤为重要。
甲基苯丙胺(俗称冰毒)作为新型毒品的典型代表,其滥用已在全球呈蔓延趋势。甲基苯丙胺滥用者具有的一系列健康问题中,一般都伴有口腔疾病,严重者甚至可以发展为“冰毒嘴”[28,29]。因此有理由推断,甲基苯丙胺的滥用会改变口腔微生物群落构成,通过宏基因组学技术分析检测口腔微生物可以判断被检测者是否存在滥用冰毒的情况,并且不受甲基苯丙胺在体内代谢周期的影响。对于海洛因、鸦片、大麻等其他毒品的检测,也可以比较摄入前后口腔微生物组成的变化,以及比较这几种毒品对口腔微生物的影响差异。
3 展 望
微生物是一个巨大的基因资源库。利用宏基因组学技术可揭示环境及动植物相关微生物的多样性。基于高通量测序的宏基因组学技术已经广泛应用于食品工业、医药业、环境治理等领域[30-32],在法医学鉴定方面的应用也有更广泛的前景。第一,在不能够提取到清晰可用的指纹并且没有个体DNA的情况下,宏基因组学通过分析接触物表面及环境微生物、微生物云的分布与构成,可应用到犯罪取证领域,辅助个体识别,使其有望成为继指纹之后的又一身份特征而辅助案件的侦查。第二,宏基因组学检测土壤等环境微生物以及生物斑迹的来源与组成,可以作为指示案发现场相关取证信息的依据。第三,通过宏基因组学技术检测口腔微生物的变化特征来辅助吸食毒品的检测,如果证实其可行性,则可能克服药物时效性问题而给药物滥用检测领域带来进一步的发展。最后,未来很可能开发出一个高质量、全面、系统的法医宏基因组数据库,通过促进和提升宏基因组数据的积累、挖掘和共享来快速准确地辅助法医鉴定中的相关应用。
但是宏基因组学在法医学领域的研究应用刚刚开始,还有很多理论和技术问题需要在实际应用中不断完善和提高。个体微生物检测技术与指纹和DNA技术相比,特异性和稳定性还有待进一步验证。未来在法医学实践中,除了现有的个体识别、案发现场物证来源鉴定、药物滥用检测方面的应用,宏基因组学可能在法医病理学、毒理学(如特定疾病的病理相关微生物组成分析)以及尸体微生物分析中都可能有所突破。总之,宏基因组学在法医物证学领域的发展与应用将为法医学检验与鉴定提供新的研究角度和解决方法。
[1]TURNBAUGH P J,LEY R E,HAMADY M,et al.The human microbiome project[J].Nature,2007,449(7164):804-810.
[2]HANDELSMAN J,RONDON M R,BRADY S F,et al.Molecular biological access to the chemistry of unknown soil microbes:a new frontier for natural products[J].Chem Biol,1998,5(10):R245-R249.
[3]CHEN H,JIANG W.Application of high-throughput sequencing in understanding human oral microbiome related with health and disease[J].Front Microbiol,2014,5:508.
[4]QIN J,LI R,RAES J,et al.A human gut microbial gene catalogue established by metagenomic sequencing[J].Nature,2010,464(7285):59-65.
[5]SANFORD J A,GALLO R L.Functions of the skin microbiota in health and disease[J].Semin Immunol,2013,25(5):370-377.
[6]LÓPEZ-PÉREZ M,MIRETE S.Discovery of novel antibiotic resistance genes through metagenomics[J]. Recent Adv DNA Gene Seq,2014,8(1):15-19.
[7]LUDWIG W,SCHLEIFER K H.Bacterial phylogeny based on 16S and 23S rRNA sequence analysis[J]. FEMS Microbiol Rev,1994,15(2-3):155-173.
[8]ANZAI Y,KIM H,PARK J Y,et al.Phylogenetic affiliation of the pseudomonads based on 16S rRNA sequence[J].Int J Syst Evol Microbiol,2000,50(Pt4):1563-1589.
[9]Di BELLA J M,BAO Y,GLOOR G B,et al.High throughput sequencing methods and analysis for microbiome research[J].J Microbiol Methods,2013,95(3):401-414.
[10]ALAN G,SARAH J P.Microbes as forensic indicators[J].Trop Biomed,2012,29(3):311-330.
[11]BUDOWLE B,SCHUTZER S E,BREEZE R G,et al.Microbial Forensics[M].2nd ed.San Diego:Academic Press,2011.
[12]COSTELLO E K,LAUBER C L,HAMADY M,et al. Bacterial community variation in human body habitats across space and time[J].Science,2009,326(5960):1694-1697.
[13]CHEN Y E,TSAO H.The skin microbiome:current perspectives and future challenges[J].J Am Acad Dermatol,2013,69(1):143-155.
[14]BLASER M J.Harnessing the power of the human microbiome[J].Proc Natl Acad Sci USA,2010,107(14):6125-6126.
[15]FIERER N,LAUBER C L,ZHOU N,et al.Forensic identification using skin bacterial communities[J]. Proc Natl Acad Sci USA,2010,107(14):6477-6481.
[16]FRANZOSA E A,HUANG K,MEADOW J F,et al. Identifying personal microbiomes using metagenomic codes[J].Proc Natl Acad Sci USA,2015,112(22):E2930-E2938.
[17]MEADOW J F,ALTRICHTER A E,BATEMAN A C,et al.Humans differ in their personal microbial cloud[J].PeerJ,2015,3:e1258.
[18]TRIDICO S R,MURRAY D C,ADDISON J,et al. Metagenomic analyses of bacteria on human hairs:a qualitative assessment for applications in forensic science[J].Investig Genet,2014,5(1):16.
[19]LAZAREVIC V,WHITESON K,HERNANDEZ D,et al.Study of inter-and intra-individual variations in the salivary microbiota[J].BMC Genomics,2010,11:523.
[20]RUFFELL A.Forensic pedology,forensic geology,forensic geoscience,geoforensics and soil forensics[J]. Forensic Sci Int,2010,202(1-3):9-12.
[21]MACDONALD C A,ANG R,CORDINER S J,et al. Discrimination of soils at regional and local levels using bacterial and fungal T-RFLP profiling[J].J Forensic Sci,2011,56(1):61-69.
[22]MORENO L I,MILLS D K,ENTRY J,et al.Microbial metagenome profiling using amplicon length heterogeneity-polymerase chain reaction proves more effective than elemental analysis in discriminating soil specimens[J].J Forensic Sci,2006,51(6):1315-1322.
[23]HORSWELL J,CORDINER S J,MAAS E W,et al. Forensic comparison of soils by bacterial community DNA profiling[J].J Forensic Sci,2002,47(2):350-353.
[24]KHODAKOVA A S,SMITH R J,BURGOYNE L,et al.Random whole metagenomic sequencing for forensic discrimination of soils[J].PLoS One,2014,9(8):e104996.
[25]TIMS S,VAN WAMEL W,ENDTZ H P,et al. Microbial DNA fingerprinting of human fingerprints:dynamic colonization of fingertip microflora challenges human host inferences for forensic purposes[J].Int J Legal Med,2010,124(5):477-481.
[26]LENZ E J,FORAN D R.Bacterial profiling of soil using genus-specific markers and multidimensional scaling[J].J Forensic Sci,2010,55(6):1437-1442.
[27]BRENIG B,BECK J,SCHÜTZ E.Shotgun metagenomics of biological stains using ultra-deep DNA sequencing[J].Forensic Sci Int Genet,2010,4(4):228-231.
[28]RAVENEL M C,SALINAS C F,MARLOW N M,et al.Methamphetamine abuse and oral health:a pilot study of“meth mouth”[J].Quintessence Int,2012,43(3):229-237.
[29]BROWN R E,MORISKY D E,SILVERSTEIN S J. Meth mouth severity in response to drug-use patterns and dental access in methamphetamine users[J].J Calif Dent Assoc,2013,41(6):421-428.
[30]KERGOURLAY G,TAMINIAU B,DAUBE G,et al. Metagenomic insights into the dynamics of microbial communities in food[J].Int J Food Microbiol,2015,213:31-39.
[31]MULCAHY-O’GRADY H,WORKENTINE M L.The challenge and potential of metagenomics in the clinic[J]. Front Immunol,2016,7:29.
[32]BIK H M.Deciphering diversity and ecological function from marine metagenomes[J].Biol Bull,2014,227(2):107-116.
Application of Metagenomics in Forensic Identification
SA Ri-na1,CAI Ling-yi2,WU Hui-juan1,YAN Jiang-wei3,LIU Xu1,HU Rong1
(1.Beijing Center for Physical and Chemical Analysis,Beijing 100089,China;2.Jining Hospital for Prevention and Treatment of Psychiatric Diseases,Jining 272051,China;3.Beijing Institute of Genomics, Chinese Academy of Sciences,Beijing 100101,China)
With the development of molecular biology and genomics,metagenomics is playing a more important role in forensic science and forensic identification.In recent years,as a branch discipline studying the composition profile and diversity of microbe flora as well as studying the interaction within microbe and with environment,the application of metagenomics has gradually risen and brought new opportunities for forensic identification-related area.In this review,strategy of metagenomics and its application in forensic identification including individual identification,origin determination of biological stain in crime scene and drug abuse detection are summarized.This article aims to elucidate the role and application value of metagenomics in forensic science.
forensic genetics;metagenomics;review;genome,microbial;personal identification
DF795.2
A
10.3969/j.issn.1004-5619.2017.04.014
1004-5619(2017)04-0397-05
2015-11-19)
(本文编辑:柳 燕)
萨日娜(1987—),女,蒙古族,博士,主要从事宏基因组与法医学相关研究;E-mail:benxiongsrn@126.com
武会娟,女,博士,主要从事先进分子生物学技术在司法鉴定中的应用研究;E-mail:whjmmwhj@126.com
