并基角毛藻若干分类学疑问的初步探讨
2017-08-16陈作艺
陈作艺 李 扬
(华南师范大学生命科学学院, 广州市亚热带生物多样性与环境生物监测重点实验室, 广州 510631)
并基角毛藻若干分类学疑问的初步探讨
陈作艺 李 扬
(华南师范大学生命科学学院, 广州市亚热带生物多样性与环境生物监测重点实验室, 广州 510631)
并基角毛藻是硅藻门角毛藻属的典型种类, 以“相邻角毛基部并行融合”作为标志性特征, 但该特征是否稳定, 及如何赋予其分类学价值?尚存较多争议。研究以符合现存并基角毛藻分类标准的藻株作为目标藻株,采用毛细管显微操作技术从我国沿海代表水域分离藻株, 建立了目标藻株的单克隆培养株系, 利用光学显微镜、扫描电镜和透射电镜分别对其形态学特征进行观察, 同时还扩增了核糖体大亚基编码基因的D1-D3区序列, 并构建系统进化树。综合分析形态学特征和分子系统学数据, 初步得到以下结论: (1)具有相同遗传特征的并基角毛藻株系在“相邻角毛基部并行融合”特征上具有不稳定性, 即“相邻角毛基部并行融合”不能作为并基角毛藻种类的标志性特征; (2)并基角毛藻和洛氏角毛藻作为相似种类, 均显示出较高的物种多样性, 高于目前的认知, 预示着或许存在隐形种或拟隐形种; (3)推测并基角毛藻单胞变型只是并基角毛藻原种的一个生活史阶段, 是并基角毛藻的同种异名, 不宜继续保留其独立的分类学地位。
并基角毛藻; 分类学; 角毛基部并行融合; 并基角毛藻单胞变型
角毛藻属(Chaetoceros Ehrenberg)是物种多样性最为丰富的浮游硅藻类群之一, 广泛分布于全球各类水体中[1,2]。角毛藻属的物种多样性丰富, 目前已报道528个分类单元, 但其中有一半被认为是同种异名[2—4], 仅有224个物种目前被认可[5]。
并基角毛藻(C.decipiens Cleve)是角毛藻属的典型种类, 最初报道于北大西洋海域[6]。该种具有规则的链状群体和发达的角毛, 在光学显微镜下易于识别, 同时, 它也是一个全球广布种, 从极地到热带的各类海域中都有记录[2,3,7—10]。
目前, 并基角毛藻的典型形态特征可概括为: (1) 链状群体规则, 窗孔多为椭圆形; (2) 角毛长且直, 位于同一平面上; (3) 角毛上具有4—6行交替纵向排列的孔纹和小刺; (4) 相邻角毛的基部具有长度不等的并行融合; (5) 尚未发现休眠孢子。其中第4个特征是并基角毛藻的命名依据, 也被认为是本种的标志性特征而被广泛应用[2,3,9]。
然而, 也有一些学者对此提出疑问。曾有学者发现“相邻角毛基部并行融合”存在变化情况[3,4,7—9,11],有时长, 有时短。有学者甚至认为具有“不同长度并行融合”的种类或许是过渡种类, 或应该是新的分类单位[3,8,11]。因此有学者建议寻找其他更为有效而稳定的形态特征, 作为并基角毛藻的识别特征[3,10]。截至目前, 关于“相邻角毛基部的并行融合”是否是稳定的形态特征, 如何赋予其分类学价值仍未有定论。
另外, 在以往的研究中, 尤其是基于人工培养藻株的形态学研究中, 报道了为数不少的种下分类单位, 并基角毛藻单胞变型(C.decipiens f.singularis Gran)即是其中之一。这些种下分类单位的建立仅仅是在形态特征的基础之上, 仍存在诸多争议。并基角毛藻单胞变型的建立依据是“单细胞生活或形成2—4个细胞的短链”, 而非原种“形成长的链状群体”。有学者认为该变型只是原种的某一个发育阶段, 应是同种异名[7,12,13], 但也有学者持不同观点,认为群体特征亦可作为稳定的区分依据, 支持单胞变型的独立存在[14,15]。为了验证单胞变型是否成立, 需开展基于藻株生活史的连续观察, 并开展传统形态分类学之外的分析比较工作, 例如分子分类学技术的引入。
当前, 传统形态分类学方法与现代分子生物学技术相结合的分析手段已成为藻类学研究的主流,并极大促进了重要硅藻类群的分类学和系统学发展[16—22]。分子生物学技术可以为传统的形态分类学研究提供有益的补充信息, 两者的相互结合可增强结论的客观性。本文研究正是基于建立的单克隆培养藻株, 结合生活史中形态特征的观察, 以及基于目标基因序列分析的分子数据, 针对上述分类学疑问开展的二次研究, 以期能够提供科学而客观的答案。
1 材料与方法
1.1 样品的采集
选取我国沿海的代表性海域, 利用浮游植物网(孔径20 μm)进行水平拖网, 以采集活体浮游植物样品, 尽快带回实验室进行目标藻株的分离、纯化。以前言中概括的典型形态特征作为判断并基角毛藻的关键依据, 进而建立目标藻株的培养体系。需要说明的是, 鉴于“相邻角毛基部的并行融合”的相关争议, 相邻角毛基部具有或无并行融合的藻株均作为目标藻株。另外, 针对单细胞或2—4个短链群体, 亦作为目标藻株, 以分析并基角毛藻单胞变型的分类学地位。
1.2 单克隆培养株系的建立
首先利用毛细管法在生物倒置显微镜(Mshot MI-12)下挑取目标藻细胞, 转移至滴有L培养液的48孔细胞培养板中[23], 待其存活并繁殖达到一定细胞数量(100个以上), 转移到盛有L培养液的100 mL三角瓶中培养。单克隆藻株保存于光照培养室内,培养条件是(22±2)℃、12鲶12光循环、光照强度约为50—80 μmol photons/(m2·s)。本文共建立了25个目标藻株的单克隆培养株系(表 1)。
在目标藻株中, 藻株MC1035的初始分离细胞是多细胞链状群体, 但是在该藻株后续的培养过程中, 同时出现了群体细胞和单个细胞2种生活形态,于是对MC1035进行2次分离, 将群体细胞和单个细胞分别挑出、纯化, 建立单克隆培养, 然后再分别编号, 群体细胞的株系继续编为MC1035, 单个细胞的株系则编为MC1036。
1.3 休眠孢子的诱导
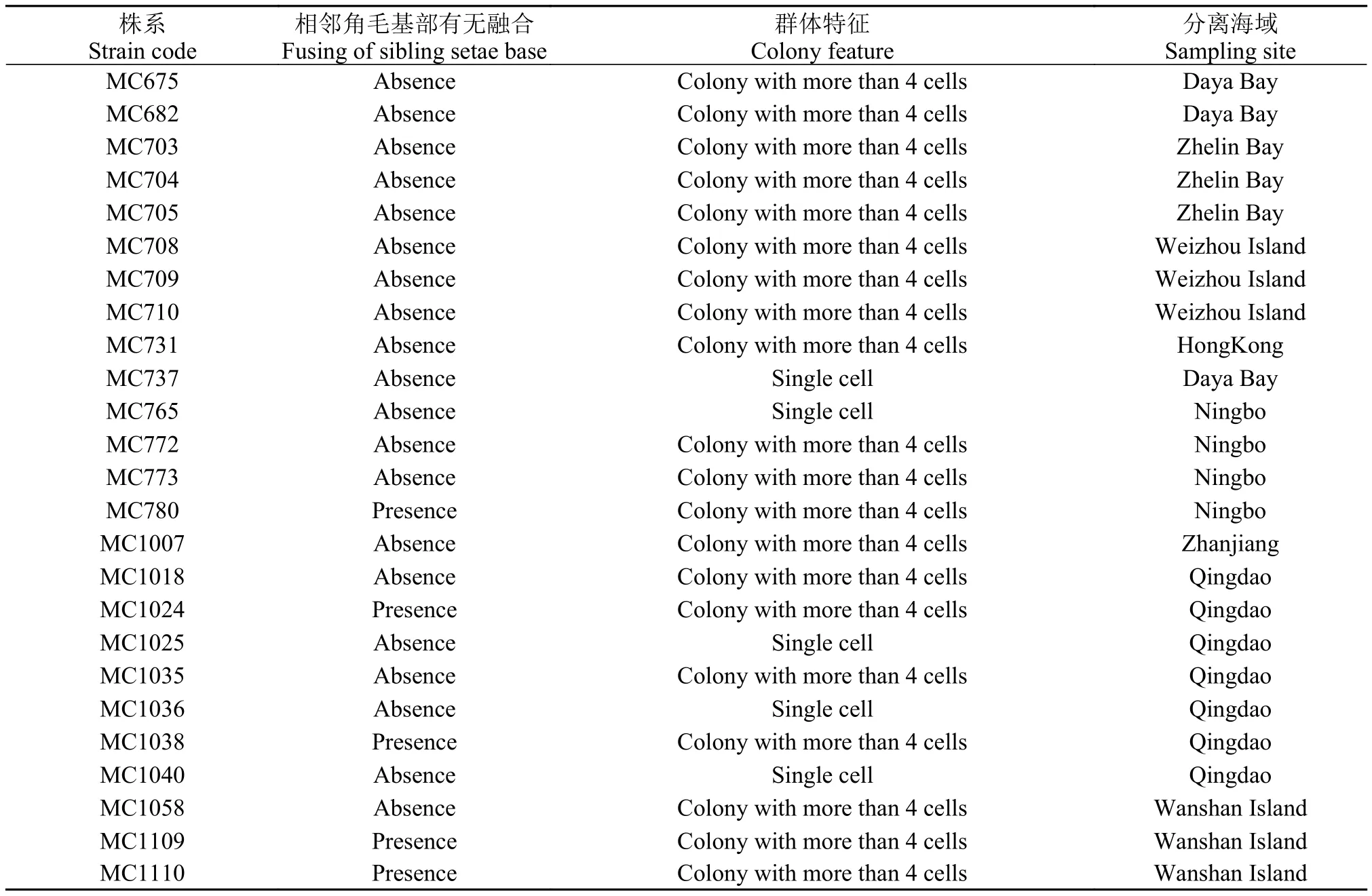
表 1 本文建立的目标藻株信息Tab.1 List and information of monoclonal strains in this study
分别吸取1 mL处于对数生长期的培养藻液, 转移到盛有L无氮培养液的100 mL三角瓶中培养[10],培养条件是(22±2)℃、12鲶12光循环、光照强度约为50—80 μmol photons/(m2·s), 而培养时间至少为两周以上, 以诱导目标藻株的休眠孢子, 每个藻株均诱导3次以上, 并定期观察有无休眠孢子产生。
1.4 形态学特征的观察
取处于对数生长期的藻液, 置于光学显微镜(Olympus BX53)下进行微分干涉(Differential interference contrast, DIC)观察, 并使用Olympus DP27数码相机进行拍照。目标藻株的群体类型、色素特征、窗孔特征、角毛形态及走势、休眠孢子在母细胞内的位置及其外形等主要基于光镜照片。
对于扫描电镜样品的处理, 先取对数生长期藻液约200 μL, 加入等体积的浓硫酸(>95%), 静置10min, 以去除有机质, 然后加入灭菌双蒸水多次水洗至中性[24]。用20 mL注射器吸取酸化后的样品,用孔径5 μm的Milipore膜过滤样品, 然后取出膜, 并待其自然晾干后, 用碳导电胶贴于铜台上, 喷金并置于扫描电镜(Zeiss Utras 55)下观察和拍照。角毛上孔纹和刺的特征、角毛基部融合的特征、窗孔超微特征、唇形突结构、休眠孢子结构等主要基于扫描电镜的观察。
对于透射电镜样品的处理, 酸化方法同上。可用移液枪吸取10 μL酸化后的样品, 滴加在喷镀碳膜的微孔铜网上, 自然晾干后置于透射电镜(Philips Tecnai 10)下观察和拍照。壳面和壳套孔纹等主要基于透射电镜的观察。
1.5 基于LSU nrDNA部分序列的分子系统学分析
用离心法收集藻细胞, 进行总DNA的提取[19]。利用正向引物D1R-F[25]和反向引物D3B-R[26]扩增核糖体大亚基编码基因(LSU nrDNA)的D1-D3区序列。PCR反应程序为: 首先94℃预变性4min, 接着进行35个循环(94℃ 35s, 55℃ 50s, 72℃ 35s), 最后72℃延伸7min。PCR产物送往深圳华大基因公司(BGI)进行纯化和测序。
利用NCBI的blast功能, 检索目标藻株序列的邻近序列信息, 同时检索NCBI现有的并基角毛藻序列, 以及洛氏角毛藻C.lorenzianus Grunow等相似种类的序列(表 2)。运用BioEdit软件进行序列的比对和矩阵[27], 有741个碱基用于最终的分析。基于MrModeltest 2.3[28]计算, 选择的最适模型和参数为GTR+I+G, ([AC=0.6762], [AG=1.7054], [AT= 0.8081], [CG]=0.1322, [CT]=3.8583, [GT]=1.0000, gamma shape=0.6573, nucleotide frequencies: A=0.2603, C=0.1869, G=0.2922, T=0.2605), 然后分别用RAxML-HPC2[29]和MrBayes 3.2[30]构建最大似然树(Maximum Likelihood, ML)和贝叶斯推理树(Bayesian inference, BI)。其中ML分析的自检值(Bootstrap test)设定为1000。选用大西洋角毛藻C.atlanticus Cleve (EF423454)为群外对照。
2 结果
2.1 基于形态特征的初步鉴定
如“材料与方法”中所述, 本文以并基角毛藻作为目标藻, 以现存的并基角毛藻形态分类特征为判断依据, 建立了25个目标藻的单克隆培养体系。也就是说, 这25个目标藻株均符合前言概括的关于并基角毛藻的前3个特征, 但在第4个特征“相邻角毛基部的并行融合”上, 则存在变化, 或长、或短、甚至无。
除了上述概括的共有特征之外, 目标藻株之间也存在一些区别特征。后续利用光学显微镜(LM)和电子显微镜(EM)对25个目标藻株进行了形态学观察, 并依据群体特征、相邻角毛基部融合特征、角毛孔纹特征等将目标藻株进行了类群划分,主要可划分为以下4个类群。
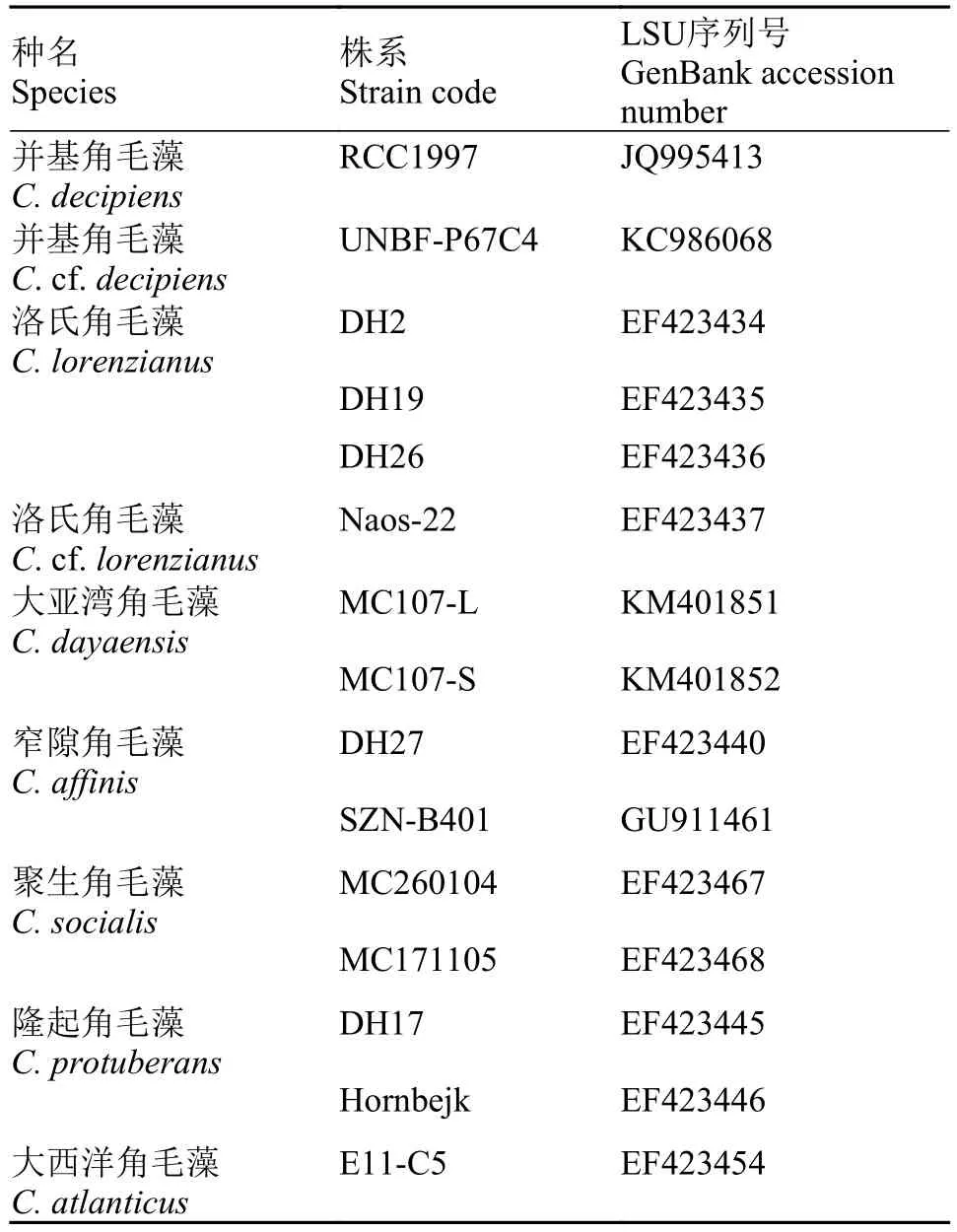
表 2 本文选用的GenBank序列信息Tab.2 Information of sequences from GenBank used in this study
类群一: 包括株系MC737、MC765、MC1025、MC1036和MC1040。LM下, 它们均符合并基角毛藻单胞变型的定义: 单细胞生活, 或由2—4个细胞组成短链, 这5个株系的初始分离细胞亦是单个细胞。营养细胞的壳面长轴34.24—52.59 μm, 贯壳轴高6.78—25.63 μm, 窗孔高6.91—14.46 μm。角毛上的孔纹椭圆形, 密度为10 μm内18—23个。基于上述形态特征, 类群一的藻株初步鉴定为并基角毛藻单胞变型C.decipiens f.singularis。
类群二: 包括MC780、MC1024、MC1038、MC1109和MC1110。LM下, 由多数细胞形成长链状群体, 相邻角毛基部具有并行融合的特征。营养细胞的壳面长轴29.64—54.18 μm, 贯壳轴高9.88—15.42 μm, 窗孔高4.43—12.74 μm。角毛上的孔纹椭圆形, 密度为10 μm内16—24个。类群二的藻株初步鉴定为并基角毛藻C.decipiens。
类群三: 包括MC705、MC708、MC731、MC773和MC1018。LM下, 由多数细胞形成长链状群体, 相邻角毛基部不具有并行融合的特征。营养细胞的壳面长轴27.04—50.11 μm, 贯壳轴高8.2—17.5 μm, 窗孔高5.86—15.14 μm。角毛上的孔纹椭圆形, 密度为10 μm内14—18个。类群三的藻株初步鉴定为形似并基角毛藻C.cf.decipiens。
类群四: 包括MC675、MC682、MC703、MC704、MC709、MC710、MC772、MC1007、MC1035和MC1058。LM下, 由多数细胞形成长链状群体, 相邻角毛基部不具有并行融合的特征。营养细胞的壳面长28.32—48.64 μm, 贯壳轴高10.78—18.33 μm, 窗孔高5.93—14.52 μm。角毛上的孔纹椭圆形, 密度为10 μm内19—25个。类群四的藻株初步鉴定为形似并基角毛藻C.cf.decipiens。
类群四与类群三的藻株在形态学上更为相似,其相邻角毛的基部均不具有并行融合, 两者之间的区别主要在于角毛孔纹密度, 类群三(10 μm内14—18个)低于类群四(10 μm内20—25个)。在以往报道中, 并基角毛藻的相似种是洛氏角毛藻C.lorenzianus Grunow, 两者之间的有效区分依据正是角毛孔纹的疏密程度[2,6,9,10,31], 因此本文亦将角毛孔纹特征作为二次分析的目标特征, 以判断角毛孔纹密度的分类学意义。
2.2 基于LSU nrDNA部分序列的分子系统学分析
整理并基角毛藻及其邻近种类的LSU nrDNA D1-D3序列, 得到40个序列信息, 其中25个目标藻株来自本文新获得的序列信息, 另外15个为NCBI下载的邻近序列和外类群序列。对矩阵后的序列信息进行了最大似然法分析和贝叶斯推理, 以大西洋角毛藻(EF423454)为外类群, 分别构建了ML和BI系统进化树。两者具有相似的拓扑结构,继而将两者合并(图 1)。
图 1显示, 无论是本文的25个目标藻株, 还是NCBI下载的并基角毛藻和洛氏角毛藻, 都很好地聚类在一个大的分支上, 并具有很高的置信值(BPP=1, ML=100), 说明基于现存形态学标准而鉴定的并基角毛藻和洛氏角毛藻具有很好的遗传一致性, 分子系统学结论也支持它们的单起源。
在并基角毛藻和洛氏角毛藻共同构成的大分支中, 最先分离出来的是巴拿马海湾(Gulf of Panama)的形似洛氏角毛藻(EF423437), 构成分支Ⅰ, 且具有较高的置信值(BPP=1, ML=100)。其次分离出来的是加拿大芬迪湾(Fundy Bay)的形似并基角毛藻(KC986068), 构成分支Ⅱ, 也具有较高的置信值(BPP=1, ML=100)。再向内, 来自意大利那不勒斯湾(Gulf of Naples)的3株洛氏角毛藻(EF423434、EF423435、EF423436)聚为一个小的分支, 构成分支Ⅲ, 置信值也较高(BPP=0.99, ML=97)。本文建立的25个目标藻株与一株分离自波福特海(Beaufort Sea)的并基角毛藻(JQ995413)聚为一支, 构成分支Ⅳ。
2.3 结合形态特征与分子系统的物种确认
分子系统树(图 1)明确显示, 本文从中国沿海建立的25个单克隆培养株系具有完全相同的LSU nrDNA D1—D3序列, 它们应该属于同一物种。
首先, 鉴于类群二(MC780、MC1024、MC1038、MC1109和MC1110)具有典型的“相邻角毛基部的并行融合”, 经与并基角毛藻模式标本手绘图对比(Cleve 1873, p.11, Pl.I, fig 5a & b), 可以确认类群二的藻株是典型的并基角毛藻。
其次, 类群一(MC737、MC765、MC1025、MC1036和MC1040)的藻株虽然多为单细胞或2—4个细胞的短链群体, 且没有“相邻角毛基部的并行融合”, 但它们与类群二藻株具有相同的LSU nrDNA D1—D3序列, 也应该是并基角毛藻, 同时也说明单细胞或2—4个细胞的短链群体只是生活史的一个阶段, 并基角毛藻单胞变型应该是并基角毛藻的同种异名。
最后, 类群三和类群四的藻株虽然都不具有“相邻角毛基部的并行融合”, 但同样基于相同的LSU nrDNA D1—D3序列信息, 它们也应该是并基角毛藻, 证实了“相邻角毛基部的并行融合”并非稳定特征。另外, 也说明本文统计的角毛孔纹密度范围(类群三, 10 μm内14—18个; 类群四, 10 μm内19—25个)均在并基角毛藻的尺度区间之内。
另外, 从NCBI下载的邻近序列也表现出一定的形态学差异和遗传多样性, 这些将在讨论部分,加以详细分析。

图 1 基于核糖体大亚基LSU nrDNA部分序列的系统树(以大西洋角毛藻为群外对照)Fig.1 Molecular phylogenetic tree inferred from LSU rDNA, with C.atlanticus as outgroup
2.4 形态学描述
并基角毛藻(图版Ⅰ)
Chaetoceros decipiens Cleve, Bihang till K Svenska Vet-Akad Handlingar 1:11, Pl.I, fig.5a & b, 1873。
细胞链直或略微弯曲, 常形成4—10个细胞的链状群体(图版Ⅰ-1—4), 也有单细胞生活, 或2—4个细胞短链的情况(图版Ⅰ-5—6)。细胞宽环面观呈矩形, 壳面长轴多大于贯壳轴高度(图版Ⅰ-1—6)。每个细胞含多个色素体, 色素体呈小盘状,散布(图版Ⅰ-1、2、4—5)。壳面观呈宽椭圆形或近圆形, 中央区域略高(图版Ⅰ-9、14), 使得壳面呈马鞍形。壳面中央有一个环纹, 环纹周围有不规则放射状排列的肋纹, 肋纹之间散布有数量众多的小孔(图版Ⅰ-15—16)。壳面边缘有圆弧形硅质肋(图版Ⅰ-9、14)。壳面长轴两端有明显隆起, 上生角毛。壳套高度约占贯壳轴的1/3(图版Ⅰ-2、4), 分布有平行状排列的硅质条纹, 条纹间散布有小孔(图版Ⅰ-17)。链端壳面近中央位置有一个唇形突,外管较短(图版Ⅰ-14—15), 链中壳面则无唇形突(图版Ⅰ-16)。相邻角毛基部与壳面长轴相接处有若干条硅质脊和硅质膜(图版Ⅰ-10)。壳套上结构与壳面相同, 有平行排列的肋纹, 以及其间分布的孔纹。壳套基部有一圈凹槽(图版Ⅰ-17)。相邻细胞之间的窗孔呈椭圆形(图版Ⅰ-1—4、6、9), 中央平, 或有略微缢缩(图版Ⅰ-6、9)。
角毛硬且直, 与链轴方向平行, 呈Brunel-I型,即角毛均分布在同一个平面上(图版Ⅰ-7)。角毛自壳面长轴两端生出后, 即与相邻角毛交叉。链中壳面的相邻角毛基部有并行融合(图版Ⅰ-1、2、7、10), 融合区域或长或短(图版Ⅰ-1、7), 有时甚至无(图版Ⅰ-3、4、6、9)。链端角毛呈U型(图版Ⅰ-3、5、6、8), 比链中角毛稍粗。角毛上分布有4—6排交替纵向排列的孔纹和小刺, 孔纹呈椭圆形(图版Ⅰ-12—13), 长0.25—0.59 μm [(0.37±0.07) μm],密度为13—25/10 μm [(20.44±3.48/10) μm], 有时光镜下可见(图版Ⅰ-11)。环带多数, 环带分布有平行排列的肋纹, 其间散布一些小孔(图版Ⅰ-18)。
营养细胞的壳面长轴27.09—54.18 μm [(43.46±10.22) μm], 贯壳轴高6.83—25.46 μm [(14.31±2.78) μm], 窗孔高4.41—15.12 μm [(9.35±2.26) μm](n=50)。
未见休眠孢子。
分布: 采自中国的青岛、宁波、柘林湾、万山岛、香港、大亚湾及湛江海域。本种还分布于北大西洋和戴维斯海峡[6,32], 北美西部沿岸海域[7], 美国罗德岛州纳拉干塞特湾[3], 加利福尼亚湾及下加利福尼亚湾海岸[9]; 南墨西哥湾[35], 丹麦沿海[4], 日本海[33], 意大利那不勒斯湾[10]。
3 讨论
3.1 并基角毛藻的分类学二次研究和信息补充
自1873年首次报道以来, “相邻角毛基部的并行融合”即被作为并基角毛藻的标志性特征, 尤其是在模式标本的手绘图中, “并行融合”这一特征显著可见[6]。在此后的研究中, 虽有学者报道“角毛基部并行融合”的长度存在变化情况[3,4,7—9,11], 但质疑多集中在具有过渡特征的种类是不是新的种类?以及“不具有并行融合”的种类是并基角毛藻还是洛氏角毛藻?因为“并行融合”曾被认可作为这两个种的区别特征[2,3,9]。但是对于具有“并行融合”即可确认是并基角毛藻的观点, 并未有学者产生怀疑,这一观点在模式种手绘图中即有体现, 在后续的研究中也被验证[2,3,9]。
因此, 本研究首先要解决的问题, 即是确认具有典型特征的并基角毛藻。本研究的类群二具有典型的“相邻角毛基部的并行融合”(图版Ⅰ-1、2、7、10), 经与并基角毛藻模式标本手绘图对比[6], 可以确认类群二的藻株是典型的并基角毛藻。
在此基础上, 通过分子系统学的分析, 发现不具有“并行融合”特征的类群三、类群四藻株与类群二藻株呈现出极为保守的遗传多样性, 同时除了“并行融合”特征之外, 它们在其他形态特征上也表现出较高的一致性。因此, 结合形态学和分子系统学数据, 我们可以认为类群三、类群四藻株也是并基角毛藻。继而可以确认“相邻角毛基部的并行融合”特征并非稳定特征, 以往研究中赋予其重要的分类学权重是不妥的, 尤其是作为并基角毛藻的标志性特征。基于此, 我们认为并基角毛藻具有一定的形态变化范围, 尤其是“相邻角毛基部的并行融合”特征, 时长时短, 甚至有时无, 这是对并基角毛藻分类学信息的最新补充。综上所述, “相邻角毛基部的并行融合”特征仍可作为并基角毛藻的特征之一, 需要补充说明的是, 这一特征并非持续特征,没有“并行融合”特征的也可能是并基角毛藻。
3.2 并基角毛藻与并基角毛藻单胞变型
并基角毛藻单胞变型由Gran[34]于1904年首次提出, 主要特征是细胞单个生活或组成2—4个细胞的短链, 这一观点被之后的部分学者认同和采用[9,14,15]。但也有学者认为本变型是原种的同种异名[12,13], Cupp[7]也不认同本变型的独立分类学地位,指出本变型可能为原种的一个发育阶段或从原种的小孢子发育而来。
本研究建立了单细胞或短链群体的单克隆培养株系(类群一), 并将其与典型并基角毛藻进行了对比分析。首先, 发现它们在形态特征上具有较高的一致性。除了群体的长短之外, 并未发现其他形态差异, 只是单细胞或短链群体不具有“相邻角毛基部的并行融合”特征, 如前所述, 这一特征并不稳定, 不应作为分类学特征。其次, 我们对类群一藻株的生活史进行了跟踪观察, 发现在培养过程中,会有长的链状群体出现, 表明不同的群体特征可能是处于不同生活史阶段。本研究中MC1035和MC1036即是从同一培养株系中二次分离出来的,但它们在形态学和遗传特征上均表现出较高的保守型, 说明它们应该是同一物种, 也说明群体特征并不能作为重要的分类学依据。最后, 具有不同群体特征的藻株(类群一、类群二、类群三、类群四)均具有完全相同的LSU nrDNA D1—D3序列信息, 遗传多样性上极为保守, 说明它们是同一物种。综上所述, 本研究认为并基角毛藻单胞变型应该是并基角毛藻的同种异名, 不应具有独立的分类学地位。
3.3 并基角毛藻与洛氏角毛藻的遗传多样性
如前所述, “相邻角毛基部的并行融合”并非稳定特征, 没有“并行融合”特征的也可能是并基角毛藻。这也带来了新的问题。因为在以往研究中,“相邻角毛基部的并行融合”被作为并基角毛藻和洛氏角毛藻的区别性特征[2,3,9]。如果不具有“并行融合”特征的藻株也可能是并基角毛藻的话, 那么它与洛氏角毛藻的有效区别特征又是什么呢?曾有学者认为壳面是否具有孔纹可以作为两者的区别特征[8,11], 也有学者认为休眠孢子可以作为区别特征[3,9]。目前都难有确定的结论, 因为我们首先需要确认洛氏角毛藻模式标本的特征, 继而获得活体藻株, 并建立形态数据和分子信息, 这些都还需要后续深入研究。
然而, 分子系统树(图 1)也提供了一些有价值的信息, 为后续研究指出了方向。从NCBI下载的邻近序列表现出较高的遗传多样性, 例如都被鉴定为并基角毛藻的加拿大芬迪湾株系(KC986068)与本文建立的25个株系, 以及波福特海株系(JQ995413)之间具有明显的遗传差异; 同样被鉴定为洛氏角毛藻的那不勒斯湾株系(EF423434、EF423435、EF423436), 与巴拿马株系(EF423437)也具有较明显的遗传差异。这说明基于现存形态分类学体系确立的并基角毛藻和洛氏角毛藻可能存在较大的不确定性, 某些重要的分类学特征或许并不具有真实的分类学价值, 前面已论述的“相邻角毛基部的并行融合”就是其中代表。另外, 这些遗传差异的存在也表明并基角毛藻和洛氏角毛藻可能存在较高的物种多样性, 只是目前还被隐藏在这两个种类里面。其实在以往的形态学研究中, 已有较多的形态多样性被报道, 例如壳面孔纹的有无、角毛孔纹的形态差异和密度区别, 还有休眠孢子的形态差异等等[4,8—11,33,35], 这些都预示着潜在的物种多样性的存在。
[1]Round F E, Crawford R M, Mann D G.The Diatoms: Biologyand Morphology of the Genera [M].Cambridge: Cambridge University Press.1990, 747
[2]Hasle G R, Syvertsen E E.Marine diatoms [A].In: Tomas C R (Eds.), Identifying Marine Phytoplankton [M].London: Academic Press.1997, 5—387
[3]Rines J E B, Hargraves P E.The Chaetoceros Ehrenberg (Bacillariophyceae) flora of Narragansett Bay, Rhode Island, USA [J].Bible Phycology, 1988, 79(3): 5—196
[4]Jensen K G, Moestrup Ø.The genus Chaetoceros (Bacillariophyceae) in inner Danish coastal waters [J].Nordic Journal of Botany, 1998, 18(1): 88
[5]Guiry M D, Guiry G M.Algaebase.World-wide electronicpublication, National University of Ireland, Galway.Availableat: http://www.algaebase.org.
[6]Cleve P T.On diatoms from the Arctic Sea [J].Bihang till K Svenska Vet-Akad-Handlingar, 1873, 1(1): 1—28
[7]Cupp E E.Marine Plankton Diatoms of the West Coast of North America [M].Scripps Institution of Oceanography.1943, 1—237
[8]Evensen D L, Hasle G R.The morphology of some Chaetoceros (Bacillariophyceae) species as seen in the electron microscopes [J].Nova Hedwigia, Beiheft, 1975, 53(4): 152—174
[9]Hernández-Becerril D U.A morphological study of Chaetoceros species (Bacillariophyta) from the plankton of the Pacific Ocean of Mexico [J].Bulletin of the Natural History Museum (Botany series), 1996, 26(1): 1—73
[10]Kooistra W H C F, Sarno D, Hernandez-Becerril D U, et al.Comparative molecular and morphological phylogenetic analyses of taxa in the Chaetocerataceae (Bacillariophyta) [J].Phycologia, 2010, 49(5): 471—500
[11]Okuno H.Electron microscopical study on fine structure of diatom frustules.XIV.Observation on the genus Chaetoceros [J].Botanical Magazine, 1956, 69(829—830): 182—200
[12]Hustedt F.Die Kieselalgen Deutschlands, Österreichs und der Schweizmit Berücksichtigung der übrigen Länder Europassowie der angrenzenden Meeresgebiete, Teil 1 [A].In: Rabenhorst’s Kryptogamen e Flora [M].Akademische Verlagsgesellschaftm.b.H, Leipzig.1930, 920
[13]VanLandingham S L.Catalogue of the Fossil and Recent Genera and Species of Diatoms and Their Synonyms.Part II.Bacteriastrum through Coscinodiscus [M].Verlag von J.Cramer, Lehre, Germany.1968, 48
[14]Chin T G, Chen J H, Huang K G.Marine Planktonic Diatoms from China Sea [M].Shanghai: Shanghai Science and Technology Press.1965, 115 [金德祥, 陈金环, 黄凯歌.中国海洋浮游硅藻类.上海: 上海科学技术出版社.1965, 115]
[15]Guo Y J, Qian S B.Flora Algarum Marinarum Sinicarum, Tomus V.Bacillariophyta, No.1 Centricae [M].Beijing: Science Press.2003, 345—346 [郭玉洁, 钱树本.中国海藻志: 第5卷(硅藻门).北京: 科学出版社.2003, 344]
[16]Zingone A, Perpoco I, Sims P, et al.Diversity in the genus Skeletonema (Bacillariophyceae).I.A reexamination of thetype material of S.costatum with thedescription of S.grevilleasp.nov.[J].Journal of Phycology, 2005, 41(1): 140—150
[17]Sarno D, Kooistra W H C F, Medlin L K, et al.Diversity in the genus Skeletonema (Bacillariophyceae).II.An assessment of the taxonomy of S.costatum likespecies, with the description of four new species [J].Journal of Phycology, 2005, 41(1): 151—176
[18]Sarno D, Kooistra W H C F, Balzano S, et al.Diversity in the genus Skeletonema (Bacillariophyceae).III.Phylogenetic position and morphological variabilityof Skeletonema costatum and Skeletonema grevillei, withthe de-scription of Skeletonema ardens sp.nov.[J].Journal of Phycology, 2007, 43(6): 156—170
[19]Lundholm N, Daugbjerg N, Moestrup Ø.Phylogeny of the Bacillariaceae with emphasis on the genus Pseudonitzschia (Bacillariophyceae) based on partial LSU rDNA [J].European Journal of Phycology, 2002, 37(1): 115—134
[20]Lundholm N, Moestrup Ø, Hasle G R, et al.A study of the Pseudo-nitzschia pseudodelicatissima/cuspidata complex (Bacillariophyceae): What is P.pseudodelicatissima [J]? Journal of Phycology, 2003, 39: 797—813
[21]Lundholm N, Moestrup Ø, Kotaki Y, et al.Inter- and intraspecific variation of the Pseudo-nitzschia delicatissima complex (Bacillariophyceae) illustrated by rRNA probes, morphological data and phylogenetic analyses [J].Journal of Phycology, 2006, 42(5): 464—481
[22]Lundholm N, Bates S S, Baugh K A, et al.Cryptic and pseudo-cryptic diversity in diatoms - with descriptions of Pseudo-nitzschia hasleana sp.nov.and P.fryxelliana sp.nov.[J].Journal of Phycology, 2012, 48(4): 436—454
[23]Guillard R R L, Hargraves P E.Stichochry sisimmobilis is a diatom, not a chrysophyte [J].Phycologia, 1993, 32(2): 234—236
[24]Cheng Z D, Gao Y H, Liu S C.Nano-diatoms in Fujian Coast [M].Beijing: Ocean Press.1993, 91 [程兆第, 高亚辉, 刘师成.福建沿岸微型硅藻.北京: 海洋出版社.1993, 91]
[25]Scholin C A, Herzog M, Sogin M, et al.Identification of group- and strain-specific genetic markers for globally distributed Alexandrium (Dinophyceae).II.Sequence analysis of a fragment of the LSU rDNA [J].Journal of Phycology, 1994, 30(6): 999—1011
[26]Nunn G B, Theisen B F, Christensen B, et al.Simplicitycorrelated size growth of the nuclear 28S ribosomal RNA D3 expansion segment in the crustacean order Isopoda [J].Journal of Molecular Evolution, 1996, 42(2): 211—223
[27]Hall T A.BioEdit: a user-friendly biological sequence alignment editor and analysis program for window 95/98/NT [J].Nucleic Acids Symposium Series, 1999, 41(1): 95—98
[28]Nylander J A A.MrModeltest v2.Program distributed by the author.Evolutionary Biology Center, Uppsala University, Uppsala, Sweden.2004, 65
[29]Miller M A, Pfeiffer W, Schwartz T.Creating the CIPRES science gateway for inference of large phylogenetic trees [C].In: Proceedings of the gateway computing environments workshop (GCE), New Orleans, LA.2010, 8
[30]Ronquist F, Teslenko M, Mark P, et al.MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space [J].Oxford Journals, 2012, 3(4): 539—542
[31]Grunow A.About Some New and insufficiently known species and genera of diatoms [J].Negotiations the Imperial Royal Zoological-Botanical Society in Vienna, 1863, 13(1): 137—162
[32]Cleve P T.Diatoms from Baffins Bay and Davis Strait [J].K Svenska Vet-Akad Handlingar, 1896, 22(1): 3—22
[33]Shevchenko O G, OrlovaT Y, Hernández-Becerril D U.The genus Chaetoceros (Bacillariophyta) from Peter the Great Bay, Sea of Japan [J].Botanic Marina, 2006, 49(3): 236—258
[34]Gran H H.The diatoms of the Arctic seas.Part I: The diatom plankton [J].Fauna Arctica Deutsch, 1904, 3(4): 511—554
[35]Hernández-Becerril D U, Granados C F.Species of the diatom genus Chaetoceros (Bacillariophyceae) in the plankton from the Southern Gulf of Mexico [J].Botanic Marina, 1998, 41(4): 505—519
PRELIMINARY STUDY ON SOME TAXONOMIC PUZZLES OF CHAETOCEROS DECIPIENS CLEVE
CHEN Zuo-Yi and LI Yang
(Guangzhou Key Laboratory of Subtropical Biodiversity and Biomonitoring, College of Life Science, South China Normal University, Guangzhou 510631, China)
As one of the largest marine planktonic diatom genera, Chaetoceros Ehrenberg has a very wide biogeographical distribution from polar to tropical waters.C.decipiens Cleve is a representative species of the genus Chaetoceros characterized by the regular chains and stiff setae.The fusing part on the sibling setae bases was the unique characters for C.decipiens in the present taxonomy system that mainly built on morphology, which has been accepted and used by some diatom taxonomists.However, some other researchers hold different opinion, in which the fusing on the sibling setae bases was a variable morphological structure that could not give much significant taxonomic value.Meanwhile, some questions remain about the taxonomic position of the intraspecific units of C.decipiens, which is difficult to answer whether these intraspecific units are independent taxonomic units or just a living stage of life cycle of C.decipiens.To answer this question, the monoclonal stains and the combination of morphology features and molecular analy-sis will be required.To do this, several monoclonal strains were established from several Chinese representative sites using glass micropipette under invert microscopy according with the present taxonomic morphology of C.decipiens.The morphology was studied by light microscopy, scanning electron microscopy and transmission electron microscopy.Meanwhile, the hypervariable region (D1-D3) of nucleic large subunit ribosomal rRNA encoding gene was amplified and sequenced as molecular evidence.The result indicated that the fusing of sibling setae base was not a steady structure, which could not be considered as significant morphological feature for the accurate identification of C.decipiens, and that some similar species without fusing of sibling setae base could be the true C.decipiens.Moreover, according to the molecular phylogenetic analysis, high diversity was hidden in the morphologically-identified C.decipiens and C.lorenzianus, which might show the presence of cryptic and pseudo-cryptic species.Inaddition, C.decipiens f.singularis Gran was confirmed as a temporary stage of whole life cycle of C.decipiens based on the continuous observation during cultures maintainance.Finally, they totally shared the same LSU sequences.Thus, C.decipiens f.singularis Gran was proposed as the synonym of C.decipiens.
Chaetoceros decipiens; Taxonomy; Fusing of sibling setae base; C.decipiens f.singularis
Q949.27
A
1000-3207(2017)04-0914-09

图版ⅠPlateⅠ
1—6并基角毛藻(LM): 1.细胞链宽环面观 (MC1109); 2.链中角毛并行融合(箭型)(MC1110); 3.宽环面观及U型端角毛(MC1035); 4.链中角毛无并行融合(箭型)(MC1058); 5.单细胞宽环面观(MC1036); 6.短链群体宽环面观(MC1036).标尺=50 μm (1, 3), 25 μm (5, 6), 10 μm (2, 4); 7—18并基角毛藻(LM.11; SEM.7—10, 12—14; TEM.15—18).7.链状群体, 示角毛基部并行融合(MC1109); 8.单细胞宽环面观(MC765); 9.链中壳面及窗孔(MC765); 10.角毛基部并行融合(箭型), 硅质翼(三角)及硅质脊(箭头)(MC1024); 11—13.角毛结构(MC773); 14—15.链端壳面, 示唇形突(箭型)(MC731); 16.链中壳面, 示中央环纹(箭型)(MC731); 17.壳套(MC731); 18.环带(MC731).标尺=10 μm (7, 8, 11), 5 μm (16, 18), 4 μm (9, 14), 2 μm (13, 15), 1 μm (10, 12, 17)
1—6 LM of Chaetoceros decipiens.1.Broad girdle views (MC1109); 2.Fusing parts of sibling setae (arrows) (MC1110); 3.Broad girdle views and U-shaped terminal setae (MC1035); 4.Sibling setae base without fusing (arrows) (MC1058); 5.Solitary living cell (MC1036); 6.Short chain in broad girdle view (MC1036).7—18 Chaetoceros decipiens.LM (Fig.11), SEM (Figs 7—10, 12—14) and TEM (Figs 15—18).7.Chain in broad girdle view, with fusing at the sibling setae base (MC1109); 8.Solitary cell (MC1036); 9.Intercalary valves and aperture (MC765); 10.Detail of fusing setae base (arrow), silica wings (triangle) and silica ridges (arrowhead) (MC1024); 11—13.Structure of setae (MC773); 14—15.Terminal valves with rimoportulae (arrowheads) (MC731); 16.Intercalary valve with central annulus (arrow) (MC731); 17.Mantle (MC731); 18.Girdle bands (MC731).1, 3 scale bars, 50 μm.5, 6 scale bars, 25 μm.2, 4 scale bars, 10 μm.7, 8, 11 scale bars, 10 μm.16, 18 scale bars, 5 μm.9, 14 scale bars, 4 μm.13, 15 scale bars, 2 μm.10, 12, 17 scale bars, 1 μm
10.7541/2017.114
2016-07-04;
2016-11-06
国家自然科学基金(31570205和31370235); 广东省科技计划项目(2015A030401084)资助 [Supported by the National Natural Science Foundation of China (31570205,31370235); Guangdong Province Science and Technology Program (2015A030401084)]
陈作艺(1988—), 男, 广东肇庆人; 硕士研究生; 主要从事藻类学研究。E-mail: 875999102@qq.com
李扬, E-mail: liyang@scnu.edu.cn
